DNA損傷修復蛋白XRCC1的研究進展
徐誠,嚴麗鋒,王心如,顧愛華
(南京醫科大學 公共衛生學院,江蘇 南京 211100)
1 XRCC1結構
1.1 基因結構
人類XRCC1基因是參與DNA單鏈斷裂修復(single- strand break repair,SSBR)過程中被克隆的第一個基因,位于19號染色體長臂1區3帶2亞帶,有31.9 kb大小,包含了17個外顯子[1];其小鼠同源基因位于7號染色體,有26 kb大小[2],外顯子的位置與人類的相似[1]。XRCC1基因在人類和小鼠有84%相同的編碼區域,并且外顯子和內含子高度保守,編碼的蛋白有86%相同的氨基酸序列。
1.2 蛋白質結構
目前,人類XRCC1蛋白發現有三個活性結構域,一個核定位信號區域和一個酪蛋白激酶(CK2)磷酸化位點[3]。具體是氨基端結構域(NTD,1- 183氨基酸區域),乳腺癌易感基因羧基端1(BRCT1, 315- 403氨基酸區域),乳腺癌易感基因羧基端2(BRCT2, 538- 633氨基酸區域),核定位信號(NLS,nuclear localization signal)區域和酪蛋白激酶(CK2)磷酸化位點(圖1)。其中,NTD對DNA聚合酶β(POLβ)具有高度的親和力[4- 6],可形成一個包繞有缺口DNA的“三明治”結構。BRCT結構域主要是在參與DNA損傷應答的蛋白質中被發現,功能類似于磷酸化依賴蛋白相互作用結構域[7]。BRCT1是DNA損傷后的修復及復制所必需的[8- 9],可與多聚ADP核糖聚合酶1(PARP1)和多聚ADP核糖聚合酶2(PARP2)相互作用[10- 11],并且能夠維持高效率SSBR和細胞存活[8]。NTD和BRCT1之間的連接區域被證實是可以和其他蛋白相互作用的結構域,即核定位信號區域,比如能與脫嘌呤/脫嘧啶核酸內切酶(APE1)、羥基鳥嘌呤糖苷酶1(OGG1)[12]和增殖細胞核抗原(PCNA)結合[13]。CK2在染色體DNA斷裂修復和維持基因的完整性方面有著直接作用,并且這個功能是通過與XRCC1磷酸化而實現[3]。
2 XRCC1的功能
2.1 XRCC1相關蛋白復合體的組成成分
研究認為XRCC1與POLβ和LIG3有較強的親和力,提示XRCC1作為支架蛋白或臨時對接平臺來參與堿基切除修復(BER)這個過程。然而,體外實驗觀察到XRCC1能構成二聚體和異四聚體,提示XRCC1復合物可能包含更多的因子[13- 16]。體內研究顯示BER過程中的酶能結合到DNA,進而充當修復中間物來防止細胞遺傳毒性和致突變毒性的發生,直到傳遞到下一個修復酶[17]。
XRCC1在復制灶(foci)能夠與細胞周期蛋白A免疫共沉淀,提示XRCC1參與DNA修復中的復制過POLβ:DNA聚合酶β;PCNA:增殖細胞核抗原;APE1:脫嘌呤/脫嘧啶核酸內切酶1;PARP1:聚腺苷酸二磷酸核糖轉移酶1;PARP2:聚腺苷酸二磷酸核糖轉移酶2;PNKP:多聚合苷酸激酶3磷酸化酶;APLF:aprataxin和PNKP樣因子;Lig3:DNA連接酶Ⅲ;APTX:aprataxin;N- terminal domain:氨基段結構域;NLS(nuclear localization signal):核定位信號;BRCT1:乳腺癌抑癌基因1羧基端;BRCT2:乳腺癌抑癌基因2羧基端;CK2:酪蛋白激酶程[13,18]。從S期細胞中分離出與XRCC1和尿嘧啶DNA糖苷酶(UNG2)相關的蛋白復合體。共定位實驗顯示在復制灶的內部和外部均有XRCC1,多聚合苷酸激酶3磷酸化酶(PNKP)和POLβ,而UNG2只與復制機制相關。因此,不同復合體的BER效率也是不同的。在BER過程中,XRCC1復合體比UNG2復合體更高效,提示S期存在獨特的BER復合體[19- 20]。
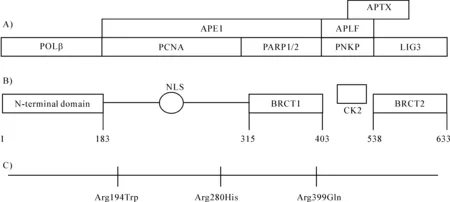
A).XRCC1蛋白和其他蛋白相互作用的區域;B).XRCC1蛋白結構域;C).3個常見的XRCC1突變體
圖1XRCC1蛋白結構簡示圖
XRCC1和LIG3較強的相互作用以及發現少量XRCC1復合體具有AP核酸內切酶活性,提示該復合體除了PNKP和POLβ,也包含LIG3和APE1[19- 22]。當損傷的程度達到一定的閾值后,該復合體能在DNA復制、LP(long patch) BER和其他修復通路中發揮作用。
PCNA在DNA復制和LP BER過程中起到中心支架蛋白的作用,另外一些BER蛋白已被證實能和PCNA免疫共沉淀[13,23- 24]。研究顯示,PCNA能具體結合到XRCC1的氨基酸166- 310區域,但在細胞S期卻未發現。XRCC1通過BRCT1結構域和PCNA在氨基酸310- 436區域存在復制灶的共定位,提示在復制灶上XRCC1可能與其他蛋白相互作用,而不是PCNA[13,25]。另外,在復制灶發現XRCC1氨基酸310- 436區域與PARP1、N- 烷基嘧啶DNA糖基化酶(MPG)、UNG2和PCNA相互作用[19,23,26- 29]。
2.2 生物體中XRCC1的功能
XRCC1基因全身敲除的小鼠表現胚胎致死,可能是SSBR能力的缺失和隨后的細胞凋亡發生所導致[30]。單倍劑量不足的敲基因小鼠在正常飼養條件下,表現出生命周期、體重、器官功能、血細胞形態、染色體狀態等正常,并且未觀察到自發腫瘤的明顯增加和不完整的染色體結構[31],提示在無毒害環境下大約50%的XRCC1蛋白足夠維持動物基因組的完整。而且之前研究顯示大約10%蛋白水平足夠維持胚胎發育和分娩后發育,并且通過非定量差的細胞毒性檢測發現,這些轉基因小鼠的成纖維細胞對甲磺甲酸酯呈現很少或基本沒有敏感性的增加[32]。進一步評價這些轉基因動物發現,與同窩出生的比較,體重表現出大約25%的下降,因而XRCC1缺失對生物體影響的具體機制仍不明確[33]。
3 XRCC1突變體
XRCC1作為BER過程中的支架蛋白維持基因組的穩定,常見的3個XRCC1多態性Arg194Trp(rs1799782)、Arg280His(rs25489)和Arg399Gln(rs25487)與疾病相關聯。研究發現Arg194Trp突變體能保護由DNA損傷誘導劑引起基因組紊亂,而另兩個突變體與降低基因組的穩定有關[34]。XRCC1多態性的變異影響BER的效率可能因為影響了蛋白與蛋白的相互作用,具體表現為影響了XRCC1的募集能力,或間接改變了BER酶活性。體外實驗顯示,EM9細胞,一種XRCC1蛋白缺陷細胞株和構建XRCC1多態性的細胞證實Arg280His和Arg399Gln分別能積累更少或解離由長波黑斑效應紫外線誘導的DNA損傷。單細胞凝膠電泳顯示這些突變體對于過氧化氫誘導的氧化損傷在修復水平有微弱的差別,支持了XRCC1的細微改變可能影響DNA的修復,進而影響基因組的穩定[25,35]。
4 XRCC1和疾病
堿基損傷、無堿基位點和單鏈斷裂可以由內源性活性物質和DNA錯配復制持續產生。XRCC1改變可能改變基因組的穩定性,進而導致疾病或癌癥。XRCC1的3個突變體與癌癥的關聯有廣泛報道。然而,流行病學薈萃分析至今也沒有得出XRCC1變異與癌癥明確發生與否的結論[36- 38]。這可能與研究中人群樣本的數目和基因SNP顯著的不同有關。比如,在亞洲人群突變純合子Arg280His觀察到與乳腺癌有關,但在高加索人群中沒有被發現[39]。突變純合子Arg194Trp在亞洲人群中能增加罹患肺癌的風險,但在高加索人群中能降低這種風險[40]。另外,Arg399Gln多態性與晚期非小細胞肺癌患者對鉑類藥物化療的敏感性相關[41],有可能成為鉑類藥物化療后生存期的預測指標[42]。
XRCC1能作為酶的輔因子來影響DNA單鏈損傷末端修復進程,XRCC1功能的改變能導致疾病。神經元條件敲除XRCC1的小鼠能影響神經的發育和導致DNA鏈斷裂的積累,初步認為XRCC1突變體能導致神經退行性疾病和膠質瘤的發生[43]。最近的流行病學研究提示XRCC1的Arg399Gln突變體和帕金森病、散發型肌萎縮側索硬化和膠質瘤有關[44- 47],而Arg194Trp突變體發現能降低膠質瘤的風險[48]。
XRCC1的突變體可能改變XRCC1形成蛋白復合體的功能,進而影響DNA損傷時復合體的募集以及相互作用蛋白酶的活性,這都可能導致癌癥的發生。
5 結 語
XRCC1作為在DNA修復過程中的支架蛋白,對DNA修復過程有著重要影響,但是具體的分子機制還沒有研究透徹。另外,流行病學研究也沒有明確得出人群中XRCC1的突變體與疾病是否有關聯。動物和細胞模型均在不同角度和不同程度揭示了XRCC1在生物體中的作用,但仍需更多研究。研究XRCC1多態性和疾病易感特征的聯系有助于從分子和細胞水平提高疾病的早期診斷率、治療有效率和預后,對于篩查易感人群、早期預防及臨床治療有重要意義。
[1] LAMERDIN J E,MONTGOMERY M A,STILWAGEN S A,et al.Genomic sequence comparison of the human and mouse XRCC1 DNA repair gene regions[J].Genomics,1995,25(2):547- 554.
[2] BROOKMAN K W,TEBBS R S,ALLEN S A,et al.Isolation and characterization of mouse Xrcc- 1,a DNA repair gene affecting ligation[J].Genomics,1994,22(1):180- 188.
[3] LOIZOU J I,EL- KHAMISY S F,ZLATANOU A,et al.The protein kinase CK2 facilitates repair of chromosomal DNA single- strand breaks[J].Cell,2004,117(1):17- 28.
[4] CALDECOTT K W,AOUFOUCHI S,JOHNSON P,et al.XRCC1 polypeptide interacts with DNA polymerase beta and possibly poly (ADP- ribose) polymerase,and DNA ligase III is a novel molecular “nick- sensor”invitro[J].Nucleic Acids Res,1996,24(22):4387- 4394.
[5] KUBOTA Y,NASH R A,KLUNGLAND A,et al.Reconstitution of DNA base excision- repair with purified human proteins:interaction between DNA polymerase beta and the XRCC1 protein[J].EMBO J,1996,15(23):6662- 6670.
[6] DIANOVA,I I,SLEETH K M,ALLINSON S L,et al.XRCC1- DNA polymerase beta interaction is required for efficient base excision repair[J].Nucleic Acids Res,2004,32(8):2550- 2555.
[7] BORK P,HOFMANN K,BUCHER P,et al.A superfamily of conserved domains in DNA damage- responsive cell cycle checkpoint proteins[J].FASEB J,1997,11(1):68- 76.
[8] TAYLOR R M,THISTLETHWAITE A,CALDECOTT K W.Central role for the XRCC1 BRCT I domain in mammalian DNA single- strand break repair[J].Mol Cell Biol,2002,22(8):2556- 2563.
[9] KUBOTA Y,HORIUCHI S.Independent roles of XRCC1’s two BRCT motifs in recovery from methylation damage[J].DNA Repair (Amst),2003,2(4):407- 415.
[10] PLESCHKE J M,KLECZKOWSKA H E,STROHM M,et al.Poly(ADP- ribose) binds to specific domains in DNA damage checkpoint proteins[J].J Biol Chem,2000,275(52):40974- 40980.
[11] SCHREIBER V,AME J C,DOLLE P,et al.Poly(ADP- ribose) polymerase- 2 (PARP- 2) is required for efficient base excision DNA repair in association with PARP- 1 and XRCC1[J].J Biol Chem,2002,277(25):23028- 23036.
[12] MARSIN S,VIDAL A E,SOSSOU M,et al.Role of XRCC1 in the coordination and stimulation of oxidative DNA damage repair initiated by the DNA glycosylase hOGG1[J].J Biol Chem,2003,278(45):44068- 44074.
[13] FAN J,OTTERLEI M,WONG H K,et al.XRCC1 co- localizes and physically interacts with PCNA[J].Nucleic Acids Res,2004,32(7):2193- 2201.
[14] BEERNINK P T,HWANG M,RAMIREZ M,et al.Specificity of protein interactions mediated by BRCT domains of the XRCC1 DNA repair protein[J].J Biol Chem,2005,280(34):30206- 30213.
[15] LEVY N,MARTZ A,BRESSON A,et al.XRCC1 is phosphorylated by DNA- dependent protein kinase in response to DNA damage[J].Nucleic Acids Res,2006,34(1):32- 41.
[16] CUNEO M J,GABEL S A,KRAHN J M,et al.The structural basis for partitioning of the XRCC1/DNA ligase III- alpha BRCT- mediated dimer complexes[J].Nucleic Acids Res,2011,39(17):7816- 7827.
[17] PRASAD R,SHOCK D D,BEARD W A,et al.Substrate channeling in mammalian base excision repair pathways:passing the baton[J].J Biol Chem,2010,285(52):40479- 40488.
[18] PARLANTI E,LOCATELLI G,MAGA G,et al.Human base excision repair complex is physically associated to DNA replication and cell cycle regulatory proteins[J].Nucleic Acids Res,2007,35(5):1569- 1577.
[19] AKBARI M,SOLVANG- GARTEN K,HANSSEN- BAUER A,et al.Direct interaction between XRCC1 and UNG2 facilitates rapid repair of uracil in DNA by XRCC1 complexes[J].DNA Repair (Amst),2010,9(7):785- 795.
[20] HANSSEN- BAUER A,SOLVANG- GARTEN K,SUNDHEIM O,et al.XRCC1 coordinates disparate responses and multiprotein repair complexes depending on the nature and context of the DNA damage[J].Environ Mol Mutagen,2011,52(8):623- 635.
[21] CALDECOTT K W,MCKEOWN C K,TUCKER J D,et al.An interaction between the mammalian DNA repair protein XRCC1 and DNA ligase Ⅲ[J].Mol Cell Biol,1994,14(1):68- 76.
[22] AKBARI M,KROKAN H E.Base excision repair efficiency and mechanism in nuclear extracts are influenced by the ratio between volume of nuclear extraction buffer and nuclei- implications for comparative studies[J].Mutat Res,2012,736(1- 2):33- 38.
[23] OTTERLEI M,WARBRICK E,NAGELHUS T A,et al.Post- replicative base excision repair in replication foci[J].EMBO J,1999,18(13):3834- 3844.
[24] EISSENBERG J C,AYYAGARI R,GOMES X V,et al.Mutations in yeast proliferating cell nuclear antigen define distinct sites for interaction with DNA polymerase delta and DNA polymerase epsilon[J].Mol Cell Biol,1997,17(11):6367- 6378.
[25] HANSSEN- BAUER A,SOLVANG- GARTEN K,GILLJAM K M,et al.The region of XRCC1 which harbours the three most common nonsynonymous polymorphic variants,is essential for the scaffolding function of XRCC1[J].DNA Repair (Amst),2012,11(4):357- 366.
[26] MASSON M,NIEDERGANG C,SCHREIBER V,et al.XRCC1 is specifically associated with poly(ADP- ribose) polymerase and negatively regulates its activity following DNA damage[J].Mol Cell Biol,1998,18(6):3563- 3571.
[27] CAMPALANS A,MARSIN S,NAKABEPPU Y,et al.XRCC1 interactions with multiple DNA glycosylases:a model for its recruitment to base excision repair[J].DNA Repair (Amst),2005,4(7):826- 835.
[28] XIA L,ZHENG L,LEE H W,et al.Human 3- methyladenine- DNA glycosylase:effect of sequence context on excision,association with PCNA,and stimulation by AP endonuclease[J].J Mol Biol,2005,346(5):1259- 1274.
[29] BRYANT H E,PETERMANN E,SCHULTZ N,et al.PARP is activated at stalled forks to mediate Mre11- dependent replication restart and recombination[J].EMBO J,2009,28(17):2601- 2615.
[30] TEBBS R S,FLANNERY M L,MENESES J J,et al.Requirement for the Xrcc1 DNA base excision repair gene during early mouse development[J].Dev Biol,1999,208(2):513- 529.
[31] MCNEILL D R,LIN P C,MILLER M G,et al.XRCC1 haploinsufficiency in mice has little effect on aging,but adversely modifies exposure- dependent susceptibility[J].Nucleic Acids Res,2011,39(18):7992- 8004.
[32] TEBBS R S,THOMPSON L H,CLEAVER J E.Rescue of Xrcc1 knockout mouse embryo lethality by transgene- complementation[J].DNA Repair (Amst),2003,2(12):1405- 1417.
[33] LADIGES W C.Mouse models of XRCC1 DNA repair polymorphisms and cancer[J].Oncogene,2006,25(11):1612- 1619.
[34] GINSBERG G,ANGLE K,GUYTON K,et al.Polymorphism in the DNA repair enzyme XRCC1:utility of current database and implications for human health risk assessment[J].Mutat Res,2011,727(1- 2):1- 15.
[35] BERQUIST B R,SINGH D K,FAN J,et al.Functional capacity of XRCC1 protein variants identified in DNA repair- deficient Chinese hamster ovary cell lines and the human population[J].Nucleic Acids Res,2010,38(15):5023- 5035.
[36] XUE H,NI P,LIN B,et al.X- ray repair cross- complementing group 1 (XRCC1) genetic polymorphisms and gastric cancer risk:A HuGE review and meta- analysis[J].Am J Epidemiol,2011,173(4):363- 375.
[37] HUANG J,ZHANG J,ZHAO Y,et al.The Arg194Trp polymorphism in the XRCC1 gene and cancer risk in Chinese Mainland population:a meta- analysis[J].Mol Biol Rep,2011,38(7):4565- 4573.
[38] GSUR A,BERNHART K,BAIERL A,et al.No association of XRCC1 polymorphisms Arg194Trp and Arg399Gln with colorectal cancer risk[J].Cancer Epidemiol,2011,35(5):e38- 41.
[39] LI H,HA T C,TAI B C.XRCC1 gene polymorphisms and breast cancer risk in different populations:a meta- analysis[J].Breast,2009,18(3):183- 191.
[40] JIANG J,LIANG X,ZHOU X,et al.DNA repair gene X- ray repair cross complementing group 1 Arg194Trp polymorphism on the risk of lung cancer:a meta- analysis on 22 studies[J].J Thorac Oncol,2010,5(11):1741- 1747.
[41] 劉歡年,劉永萍,薛宏波,等.修復基因多態性與晚期肺癌順鉑化療近期療效的研究[J].現代醫學,2013,41(9):659- 661.
[42] 成紅艷,陳寶安,孫新臣,等.ERCC1、XRCC1單核苷酸多態性與非小細胞肺癌鉑類藥物化療預后關系的研究[J].東南大學學報:醫學版,2011,30(5):717- 721.
[43] LEE Y,KATYAL S,LI Y,et al.The genesis of cerebellar interneurons and the prevention of neural DNA damage require XRCC1[J].Nat Neurosci,2009,12(8):973- 980.
[44] WEI X,CHEN D,LV T.A functional polymorphism in XRCC1 is associated with glioma risk:evidence from a meta- analysis[J].Mol Biol Rep,2013,40(1):567- 572.
[45] GENCER M,DASDEMIR S,CAKMAKOGLU B,et al.DNA repair genes in Parkinson’s disease[J].Genet Test Mol Biomarkers,2012,16(6):504- 507.
[46] COPPEDE F,MIGHELI F,LO GERFO A,et al.Association study between XRCC1 gene polymorphisms and sporadic amyotrophic lateral sclerosis[J].Amyotroph Lateral Scler,2010,11(1- 2):122- 124.
[47] YOSUNKAYA E,KUCUKYURUK B,ONARAN I,et al.Glioma risk associates with polymorphisms of DNA repair genes,XRCC1 and PARP1[J].Br J Neurosurg,2010,24(5):561- 565.
[48] RAJARAMAN P,HUTCHINSON A,WICHNER S,et al.DNA repair gene polymorphisms and risk of adult meningioma,glioma,and acoustic neuroma[J].Neuro Oncol,2010,12(1):37- 48.

