大黃魚(Larimichthys crocea)Tristetraprolin基因的克隆與功能分析*
汪慧娟 何志巧 石 戈 周素明 張曉林 申 望
大黃魚()Tristetraprolin基因的克隆與功能分析*
汪慧娟1何志巧1石 戈1周素明2張曉林1申 望1①
(1. 浙江海洋大學(xué)海洋科學(xué)與技術(shù)學(xué)院 浙江舟山 316022; 2. 寧波大學(xué)海洋學(xué)院 浙江寧波 315211)
Tristetraprolin (TTP)是廣泛存在于真核生物的RNA結(jié)合蛋白。TTP通過促進(jìn)mRNA降解或抑制翻譯在轉(zhuǎn)錄后水平抑制炎癥因子表達(dá), 是炎癥性疾病的潛在治療靶點。采用RACE技術(shù)獲取了大黃魚() TTP (命名為TTP) cDNA的全長序列。TTP全長cDNA 1 508 bp, 包括77 bp的5′-非編碼區(qū)(5′-UTR)、183 bp的3′-UTR和1 248 bp的開放閱讀框(ORF), 編碼418個氨基酸殘基。推導(dǎo)的TTP理論分子量44 897.78 Da, 預(yù)測等電點pI 8.37; 哺乳動物TTP的重要功能結(jié)構(gòu)域: N-端核輸出序列(NES)、中央串聯(lián)鋅指結(jié)構(gòu)域(TZF)、C-端NOT1-結(jié)合結(jié)構(gòu)域(NOT1-BD)也在TTP中保守存在; 系統(tǒng)發(fā)育分析顯示TTP和其他脊椎動物TTP聚為一支, 并與哺乳動物ZFP36家族其他成員ZFP36L1、ZFP36L2和ZFP36L3的進(jìn)化支分離。組織表達(dá)特異性分析顯示檢測的9個組織均表達(dá)TTP mRNA, 但不同組織間表達(dá)水平差異大, 其中肌肉組織表達(dá)水平最高。大黃魚頭腎細(xì)胞系LYC-hK細(xì)胞中過表達(dá)TTP上調(diào)LPS誘導(dǎo)早期(1 h) TNF-α mRNA表達(dá)量, 之后TNF-α mRNA表達(dá)量快速下調(diào)至接近對照組水平(1.5 h); 放線菌D抑制轉(zhuǎn)錄后過表達(dá)TTP上調(diào)TNF-α mRNA降解速率, 表明TTP可通過促進(jìn)TNF-αmRNA降解調(diào)節(jié)TNF-α表達(dá)。以上研究結(jié)果提示TTP可能是大黃魚炎癥反應(yīng)平衡關(guān)鍵調(diào)控蛋白, 在大黃魚感染性病害防治策略開發(fā)中有潛在應(yīng)用價值。
大黃魚; RNA結(jié)合蛋白; Tristetraprolin蛋白; 炎癥反應(yīng); 轉(zhuǎn)錄后調(diào)控
炎癥反應(yīng)是病原感染、組織損傷等脅迫作用誘導(dǎo)的機(jī)體適應(yīng)性反應(yīng)。受控的適度炎癥反應(yīng)清除入侵病原或修復(fù)組織損傷, 而失控的高炎癥反應(yīng)則導(dǎo)致組織器官損傷, 誘發(fā)炎癥性疾病甚至死亡(Medzhitov, 2008)。因此, 炎癥因子表達(dá)在轉(zhuǎn)錄水平和轉(zhuǎn)錄后水平都受到嚴(yán)格調(diào)控(Yoshinaga, 2019)。RNA結(jié)合蛋白(RNA binding protein, RBP)介導(dǎo)的在轉(zhuǎn)錄后水平調(diào)節(jié)炎癥因子mRNA加工、定位、降解和翻譯, 是炎癥因子表達(dá)調(diào)控的關(guān)鍵機(jī)制(Kafasla, 2014; Uchida, 2019)。
Tristetraprolin (TTP, 別名ZFP36、TIS11、GOS24、NUP475)是ZFP36家族RBP的原型蛋白, 廣泛存在于真核生物中(Makita, 2021)。哺乳動物TTP都有3個保守功能結(jié)構(gòu)域: N-端的核輸出信號(nuclear export signal, NES)、中央串聯(lián)的2個CCCH型鋅指結(jié)構(gòu)域(tandem zinc-finger domain, TZF)和C-端的NOT1-結(jié)合結(jié)構(gòu)域(NOT1-binding domain, NOT1-BD) (Akira, 2021)。TZF結(jié)構(gòu)域識別、結(jié)合靶mRNA 3′-非翻譯區(qū)(untranslating region, UTR)的AU-富集元件(AU-rich element, ARE), N端和C端結(jié)構(gòu)域募集CCR4-NOT1脫腺嘌呤復(fù)合體(CCR4-NOT1 deadenylase complex)、脫帽復(fù)合體(decapping complex, Dcp)、核酸外切體(exosomes)、5′~3′外切核糖核酸酶1 (5′~3′ exoribonuclease 1, XRN1)等促進(jìn)靶mRNA降解, 調(diào)節(jié)靶基因表達(dá)(Akira, 2021)。TTP靶RNA組研究顯示TTP的靶mRNA主要是炎癥和免疫相關(guān)基因mRNA, 包括經(jīng)典炎癥因子TNF-α、IL-1β、IL-6的mRNA (Kratochvill, 2011; Mukherjee, 2014)。與之相對應(yīng), TTP參與調(diào)控多種炎癥和免疫相關(guān)疾病的發(fā)生和發(fā)展 (Zhang, 2021)。例如, 基因敲除, TTP-KO小鼠表現(xiàn)出嚴(yán)重的炎癥型疾病綜合征, 包括惡液質(zhì)、關(guān)節(jié)炎、皮膚炎、結(jié)膜炎、白血病等, TNF-α中和抗體處理可顯著減輕癥狀(Taylor, 1996; Carballo, 2001)。反之, 敲除TTP mRNA 3′-UTR的AREs順式元件過表達(dá)TTP, 下調(diào)LPS誘導(dǎo)的巨噬細(xì)胞和小鼠炎癥因子表達(dá), 減輕風(fēng)濕性關(guān)節(jié)炎、內(nèi)毒素血癥、牛皮癬、多發(fā)性硬皮病等免疫相關(guān)疾病小鼠模型的臨床癥狀(Patial, 2016b)。因此, TTP被認(rèn)為是炎癥性疾病的潛在治療靶點(Mahmoud, 2019; Li, 2022)。感染性養(yǎng)殖病害一直是制約水產(chǎn)養(yǎng)殖業(yè)發(fā)展的瓶頸問題。例如, 在亞洲魚類養(yǎng)殖業(yè)總經(jīng)濟(jì)損失中, 僅弧菌病所造成的經(jīng)濟(jì)損失就約占50% (Xu, 2022)。高炎癥反應(yīng)也是魚類感染性病害的病理基礎(chǔ)(Dai, 2023), 但迄今還未見魚類TTP功能的研究報道, 特別是TTP對魚類炎癥因子表達(dá)的調(diào)節(jié)作用。
大黃魚()是我國養(yǎng)殖規(guī)模最大的海水魚(農(nóng)業(yè)農(nóng)村部漁業(yè)漁政管理局等, 2022)。隨著養(yǎng)殖規(guī)模和養(yǎng)殖密度的增大, 大黃魚感染性病害防治壓力也越來越大(王凡等, 2019)。而抗生素等傳統(tǒng)感染性病害防治方法由于環(huán)境安全和食品安全等問題, 已被禁用或嚴(yán)格限制使用(胡洋等, 2022), 開發(fā)抗生素替代物等大黃魚感染性病害防治新方法成為保障產(chǎn)業(yè)健康發(fā)展的迫切需求。因此, 本研究以大黃魚TTP (命名為TTP)為研究對象, 采用RACE (rapid amplification of cDNA end)技術(shù)克隆基因全長cDNA序列并進(jìn)行生物信息學(xué)分析, 研究TTP的結(jié)構(gòu)特征和系統(tǒng)發(fā)生; 采用實時熒光定量技術(shù)(Real-time Quantitative PCR, RT-qPCR)檢測基因的組織表達(dá)模式; 并通過在大黃魚頭腎細(xì)胞系LYC-hK中過表達(dá)TTP, 研究TTP對LPS誘導(dǎo)的TNF-α mRNA表達(dá)水平的調(diào)節(jié)作用及其機(jī)制。研究結(jié)果可為進(jìn)一步探討TTP對大黃魚抗感染炎癥反應(yīng)的調(diào)控作用以及評估其應(yīng)用價值提供參考。
1 材料與方法
1.1 實驗材料
健康大黃魚[平均體重(142.7±12.2) g]購買自浙江省舟山市半島水產(chǎn)養(yǎng)殖有限公司, 在實驗室循環(huán)海水養(yǎng)殖系統(tǒng)中使用過濾海水飼養(yǎng)一周適應(yīng)環(huán)境后取樣, 水溫24~25 °C。
為檢測基因的組織表達(dá)特異性, 5條健康大黃魚浸泡于0.02% MS-222 (國藥, 中國)麻醉后, 取肝、腸、胃、肌肉、心、脾、腎、鰓和腦等9個組織的樣本放置于1.5 mL EP管中, 加入RNALater?動物組織RNA穩(wěn)定保存液(碧云天, 上海), –80 °C凍存?zhèn)溆谩?/p>
1.2 全長cDNA克隆
從大黃魚NCBI基因組數(shù)據(jù)庫(登錄號: 12197)中檢索到一個基因同源物(基因登錄號: 104928501)及其預(yù)測的mRNA序列(登錄號: XM_010742797.3) (Ao, 2015)。以預(yù)測的大黃魚TTPmRNA序列為參考, 使用NCBI在線引物設(shè)計工具Primer-Blast設(shè)計大黃魚TTP mRNA 5′-RACE、3′-RACE和開放閱讀框(opening reading frame, ORF) PCR引物(表1) (Ye, 2012)。Trizol法提取總RNA, 按TaKaRa RNAiso Plus (TaKaRa, 日本)說明書操作。5′-RACE和3′-RACE使用TaKaRa SMARTer?RACE 5′/3′ Kit (TaKaRa, 日本), 操作按說明書進(jìn)行。3′-RACE引物組合: 第一輪PCR引物TTPF1/3′- RACE UPM long primer, 第二輪巢式PCR引物TTPF2/3′-RACE UPM short primer; 5′-RACE引物組合: 第一輪PCR引物TTPR2/5′-RACE UPM long primer, 第二輪巢式PCR引物TTPR1/5′-RACE UPM short primer。ORF PCR擴(kuò)增使用TaKaRa?DNA Polymerase (TaKaRa, 日本), 模板為5′/3′-RACE cDNA, 引物為ETTPF/ ETTPR, 擴(kuò)增程序: 95 °C 2 min; 95 °C 15 s, 56 °C 30 s, 72 °C 1 min, 35次循環(huán)。PCR產(chǎn)物經(jīng)電泳檢測、瓊脂糖膠回收后連接simple pMD-19T vector (TaKaRa, 日本), 轉(zhuǎn)化DH5α (TaKaRa, 日本), 挑陽性單克隆送上海生工測序, 拼接5′-RACE、3′-RACE和ORF序列獲得全長cDNA序列。
1.3 生物信息學(xué)分析
使用ExPASy在線工具包Compute pI/MW (https://web.expasy.org/compute_pi/)在線預(yù)測理論分子量和等電點; 使用NCBI在線工具包CD-Search識別保守結(jié)構(gòu)域(Marchler-Bauer, 2017); 使用SignalP 6.0預(yù)測信號肽(Teufel, 2022); 使用Euk-mPLoc 2.0預(yù)測TTP的亞細(xì)胞定位(Chou, 2010); 使用NetNES 1.1工具包預(yù)測核輸出信號(NES) (Fu, 2011); 使用BLASTP程序在NCBI非冗余(nr)數(shù)據(jù)庫中搜索同源序列(Johnson, 2008); 多序列比對使用ClustalW1.83軟件包(Larkin, 2007);使用MEGA version 7軟件包的鄰接(Neighbor-Joining, NJ)法構(gòu)建系統(tǒng)發(fā)育樹(Kumar, 2016); 本研究中多序列比對和系統(tǒng)發(fā)育樹分析使用的序列見表2。
表1 引物信息

Tab.1 Information of the primers
表2 多序列比對和系統(tǒng)樹構(gòu)建所用的ZFP36家族成員信息

Tab.2 The sequences of ZFP36 family used for multiple sequence alignment and phylogenetic tree analysis
1.4 RT-qPCR
Trizol法提取總RNA, 按TaKaRa RNAiso Plus (TaKaRa, 日本)說明書操作。取1 μg總RNA按TaKaRa PrimeScript? RT reagent Kit with gDNA Eraser試劑盒(TaKaRa, 日本)使用說明合成cDNA第一鏈。20 μL RT-qPCR反應(yīng)體系按BeyoFast? SYBR Green qPCR Mix (碧云天, 中國)試劑盒說明書配制, 即SYBR Green qPCR Mix 10 μL、上下游引物混合物2.0 μL、cDNA 2.0 μL、超純水6 μL, 引物序列見表1; 反應(yīng)程序: 95 °C 2 min; 95 °C 15 s, 60 °C 30 s, 40次循環(huán), 反應(yīng)在ABI QuantStudio 1實時檢測系統(tǒng)(Applied Biosystems, 美國)上運行; 擴(kuò)增完成后, 采用熔解曲線分析驗證擴(kuò)增特異性。以為內(nèi)參基因, 采用2–ΔΔCt法分析目的基因的相對表達(dá)水平; 在mRNA穩(wěn)定性分析中, 以放線菌素D處理0 hTNF-α mRNA表達(dá)水平為參照, 采用2–ΔCt法分析其他時間點TNF-α mRNA的相對表達(dá)水平(Livak, 2001)。
1.5 構(gòu)建pEGFP-N1-LcTTP重組載體
(1.2)中測序驗證正確ORF單克隆擴(kuò)大培養(yǎng)后提取pMD-19T-TTP重組質(zhì)粒, pMD-19T-TTP重組質(zhì)粒和pEGFP-N1表達(dá)載體質(zhì)粒(Clontech, 美國)分別用限制性內(nèi)切酶I和GI (TaKaRa, 日本)雙酶切, 酶切片段膠回收純化后使用T4 DNA連接酶(TaKaRa, 日本)連接, 連接產(chǎn)物轉(zhuǎn)化DH5α挑單克隆送上海生工測序, 測序驗證正確的pEGFP- N1-TTP單克隆擴(kuò)大培養(yǎng)后使用Endo-free Plasmid Mini Kit II (Omega, 美國)提取無內(nèi)毒質(zhì)粒。重組表達(dá)載體pEGFP-N1-TTP表達(dá)產(chǎn)物為GFP-TTP融合蛋白。
1.6 細(xì)胞培養(yǎng)和轉(zhuǎn)染
大黃魚頭腎細(xì)胞系LYC-hK按之前報道培養(yǎng)(Zhou, 2023)。簡而言之, 使用含15%胎牛血清(FBS; 四季青, 中國)、100 U/mL青霉素/鏈霉素(P/S)的DMEM/HAM’S F-12完全培養(yǎng)基(Hyclone, 美國)置于26 °C、5% CO2的二氧化碳培養(yǎng)箱中培養(yǎng)。
細(xì)胞轉(zhuǎn)染按GP-transfect-Mate轉(zhuǎn)染試劑(吉瑪, 中國)說明書操作。簡而言之: 6孔板按2×105細(xì)胞/孔接種LYC-hK細(xì)胞, 24 h后3 μg質(zhì)粒DNA與6 μL GP-transfect-Mate試劑混合于400 μL無FBS的DME/F-12培養(yǎng)基靜置20 min后轉(zhuǎn)染LYC-hK細(xì)胞, 轉(zhuǎn)染6 h換完全培養(yǎng)基繼續(xù)培養(yǎng)48 h, 熒光顯微鏡(Leica, 德國)觀察轉(zhuǎn)染效率后進(jìn)行后續(xù)實驗。
1.7 數(shù)據(jù)分析
所有數(shù)據(jù)均采用GraphPad Prism 6.0軟件(GraphPad software Inc., 美國)進(jìn)行分析, 并以均數(shù)±均數(shù)標(biāo)準(zhǔn)誤差(SEM)表示。組間統(tǒng)計學(xué)差異采用非配對雙尾學(xué)生檢驗,值小于0.05為有統(tǒng)計學(xué)意義。
2 結(jié)果
2.1 LcTTP全長cDNA序列分析
將5′-RACE、3′-RACE和ORF擴(kuò)增序列拼接得到的全長cDNA序列共1 508 bp, 其中ORF 1 248 bp, 5′-UTR 77 bp, 3′-UTR 183 bp, polyA結(jié)構(gòu)上游63 bp處有一加尾信號AATAA (圖1)。
注: 下劃線示核輸出信號(NES); 陰影區(qū)示串聯(lián)重復(fù)鋅指結(jié)構(gòu)域(TZF); 星號示CCCH型鋅指結(jié)構(gòu)域的保守氨基酸殘基位點; 粗體示起始密碼子和終止密碼子; 方框表示加尾信號
推導(dǎo)的TTP蛋白序列包含415個氨基酸殘基, 理論分子量(MW) 44 897.78 Da, 預(yù)測等電點(pI) 8.37。SignalP 6.0分析TTP未在N端檢測到信號肽, Euk-mPLoc 2.0亞細(xì)胞定位分析預(yù)測TTP定位在細(xì)胞核, NetNES 1.1分析顯示在TTP的N端有一NES序列(T10KNFMNLALDDGLL23; 圖1)。CD-Search分析顯示ZFP36家族特征結(jié)構(gòu)域TZF也保守存在于TTP中央?yún)^(qū)域。多序列比對顯示TTP氨基酸序列與黃鰭鯛(XP_036937641.1)、條紋鱸魚(XP_035535819.1)、人(NP_003398.3)和小鼠(NP_035886.1) TTP氨基酸序列的同源性分別為91.46%、91.28%、40.69%和39.58%; 且哺乳動物TTP保守的重要功能結(jié)構(gòu)域: 中央TZF結(jié)構(gòu)域和C端NOT1-BD結(jié)構(gòu)域也在TTP中保守存在(圖2)。
2.2 LcTTP的系統(tǒng)發(fā)生分析
系統(tǒng)發(fā)育分析顯示: 脊椎動物TTP聚為一支(置信度99%), 與哺乳動物ZFP36家族的其他成員ZFP36L1、ZFP36L2和ZFP36L3分開。在脊椎動物中, TTP又聚類為哺乳動物、爬行動物、兩棲動物和魚類4個進(jìn)化支,TTP與其他硬骨魚TTP聚為一支(圖3)。以上結(jié)果提示本研究克隆得到的TTP是大黃魚ZFP36家族成員TTP。
2.3 LcTTP基因的組織表達(dá)特異性
基因的組織表達(dá)特異性與其生理功能密切相關(guān)。為檢測基因的組織表達(dá)特異性, 本研究采集5條健康大黃魚的腦、胃、鰓、肝、脾、腎、頭腎、肌肉、心臟等9個組織, RT-qPCR檢測不同組織中TTP mRNA的表達(dá)水平。結(jié)果顯示9個組織均檢測到TTP mRNA表達(dá), 但不同組織表達(dá)量差異較大(圖4)。肌肉組織TTPmRNA表達(dá)量最高(53.63±27.43), 其次為胃(16.99±14.38)和鰓(10.18±4.27), 而腦(1.10±0.23)、肝(2.58±1.02)、腎(2.98±1.25)、脾(3.01±1.03)、頭腎(4.42±1.26)、心臟(4.89±1.93)等組織TTP mRNA表達(dá)量均較低。
2.4 LPS誘導(dǎo)LYC-hK細(xì)胞LcTTP和炎癥因子基因表達(dá)
LPS是誘導(dǎo)炎癥因子表達(dá)的經(jīng)典病原相關(guān)分子模式(pathogen associated molecular pattern, PAMP)分子。哺乳動物研究顯示LPS通過誘導(dǎo)巨噬細(xì)胞、淋巴細(xì)胞、嗜中性粒細(xì)胞等免疫細(xì)胞TTP表達(dá), 促進(jìn)TNF-α、IL-1β等炎癥因子mRNA降解, 下調(diào)LPS誘導(dǎo)的炎癥因子表達(dá)(Mahtani, 2001; Fairhurst, 2003; Tchen, 2004)。因此, 本研究首先調(diào)查LPS是否誘導(dǎo)LYC-hK細(xì)胞炎癥因子和基因表達(dá)。研究結(jié)果顯示(圖5): LPS (1 μg /mL)處理LYC-hK細(xì)胞3 h顯著上調(diào)TNF-α mRNA表達(dá)量(1.48倍;<0.01), 6 h時顯著下調(diào)(0.77倍;<0.05); 而IL-1β mRNA表達(dá)量在LPS處理2 h時顯著上調(diào)(1.98倍;<0.01), 6 h時顯著下調(diào)(0.69倍;<0.01), 之后逐漸上升, 24 h時又顯著上調(diào)(1.64倍;<0.05);TTP mRNA表達(dá)量在LPS處理3 h時顯著上調(diào)(1.57倍;<0.001), 其他時間點均出現(xiàn)小幅下調(diào)(1 h 0.80倍、2 h 0.83倍、6 h 0.75倍、12 h 0.78倍、24 h 0.81倍)。以上結(jié)果提示LYC-hK細(xì)胞中TTP可能參與調(diào)控LPS誘導(dǎo)的炎癥因子表達(dá)。
注: 黑色陰影示相同氨基酸位點; 灰色示相似氨基酸殘基位點; 上劃線以人TTP為參考, 示保守結(jié)構(gòu)域; NES: 核輸出序列; TZF: 串聯(lián)鋅指結(jié)構(gòu)域; CTD: C端結(jié)構(gòu)域; 星號示CCCH型鋅指結(jié)構(gòu)域保守氨基酸位點
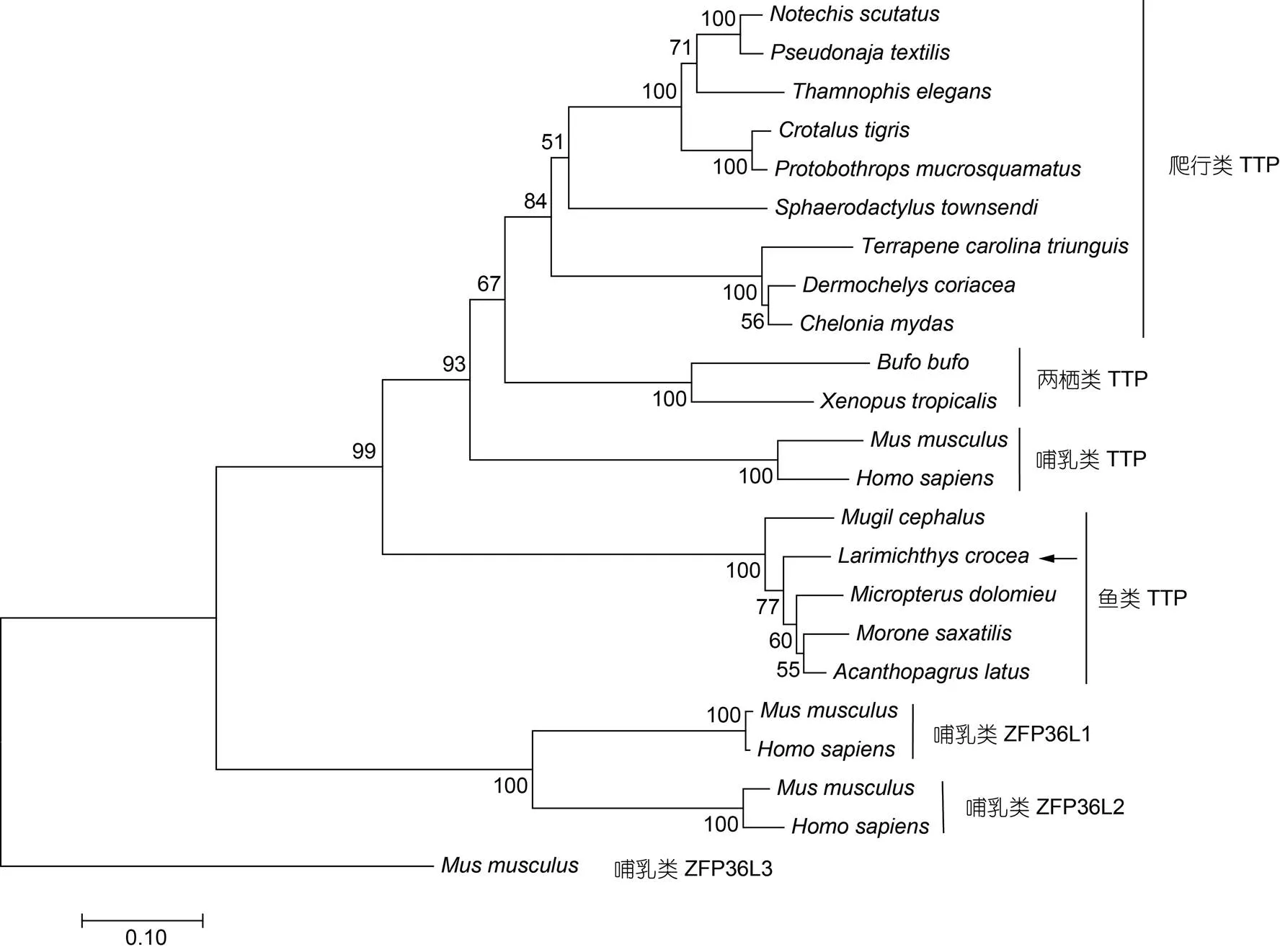
圖3 LcTTP與其他脊椎動物ZFP36家族代表性成員的系統(tǒng)發(fā)生分析
注: 使用MEGA 7.0軟件包采用NJ法構(gòu)建系統(tǒng)發(fā)育樹; 分支上數(shù)值為通過1000 次分析得到的bootstrap值; 比例尺代表分支長度; 箭頭示TTP在NJ樹中的位置; 構(gòu)建系統(tǒng)樹所用序列的GenBank登錄號見表2

圖4 LcTTP基因的組織表達(dá)譜(n=5)
2.5 LYC-hK細(xì)胞過表達(dá)LcTTP調(diào)節(jié)LPS誘導(dǎo)的TNF-α基因表達(dá)
炎癥因子TNF-α mRNA是哺乳動物中最早鑒定研究的TTP靶mRNA, TTP通過結(jié)合TNF-α mRNA 3′-UTR的ARE順式元件促進(jìn)TNF-α mRNA降解, 抑制TNF-α表達(dá)(Taylor, 1996; Carballo, 1997)。因此, 本研究在大黃魚LYC-hK細(xì)胞中過表達(dá)TTP, 調(diào)查TTP是否調(diào)節(jié)LPS誘導(dǎo)的TNF-α表達(dá)及其作用機(jī)制。研究結(jié)果顯示(圖6): 與空載體pEGFP-N1轉(zhuǎn)染對照組相比, pEGFP-N1-TTP轉(zhuǎn)染的過表達(dá)組在LPS (1 μg/mL)處理0.5 h (3.48 vs 4.80;<0.01)、1 h (3.44 vs 11.27;<0.001)、1.5 h (3.50 vs 4.52;<0.05)、3 h (2.24 vs 3.12;<0.01)、6 h (1.34 vs 2.36;<0.01)均顯著上調(diào)TNF-α mRNA表達(dá)量, 峰值在LPS處理1 h時。表明過表達(dá)TTP在LPS誘導(dǎo)早期大幅上調(diào)TNF-α mRNA表達(dá)量, LPS處理1 h后TNF-α mRNA表達(dá)量快速下調(diào)到接近對照組水平, 提示大黃魚TNF-αmRNA可能是TTP的靶mRNA。

圖5 LPS誘導(dǎo)LYC-hK細(xì)胞LcTTP和炎癥因子表達(dá)
注: a.; b.; c.

圖6 過表達(dá)LcTTP調(diào)節(jié)LYC-hK細(xì)胞LPS誘導(dǎo)的TNF-α基因表達(dá)
2.6 LcTTP促進(jìn)TNF-α mRNA降解
促進(jìn)靶mRNA降解是哺乳動物TTP調(diào)節(jié)炎癥因子表達(dá)的主要機(jī)制之一(Bataclan, 2021)。本研究在LPS (1 μg/mL)處理LYC-hK細(xì)胞1 h后, 以放線菌D (5 mg/mL)抑制轉(zhuǎn)錄, RT-qPCR分析轉(zhuǎn)錄抑制后pEGFP-N1轉(zhuǎn)染對照組和pEGFP-N1-TTP轉(zhuǎn)染過表達(dá)組TNF-αmRNA的降解速率, 調(diào)查TTP對靶mRNA穩(wěn)定性的影響。研究結(jié)果如圖7所示, 過表達(dá)TTP顯著上調(diào)TNF-α mRNA的降解速率, pEGFP-N1轉(zhuǎn)染對照組和pEGFP-N1-TTP轉(zhuǎn)染過表達(dá)組TNF-α mRNA的半衰期(1/2)分別為115 min和40 min, 表明TTP可通過促進(jìn)TNF-α mRNA降解調(diào)節(jié)TNF-α表達(dá)。

圖7 LcTTP促進(jìn)TNF-αmRNA降解
3 討論
哺乳動物研究顯示TTP可通過促進(jìn)細(xì)胞因子mRNA降解調(diào)節(jié)免疫反應(yīng)(Makita, 2021), 是炎癥和免疫相關(guān)疾病的潛在治療靶點(Patial, 2016a)。顯然, 借鑒哺乳動物研究成果開展魚類TTP的功能研究, 可為魚類感染性養(yǎng)殖病害的防治提供新線索。本研究采用RACE技術(shù)克隆了大黃魚的全長cDNA序列, 推導(dǎo)的氨基酸序列分析顯示哺乳動物TTP重要功能結(jié)構(gòu)域在TTP中也保守存在, 系統(tǒng)發(fā)生分析顯示TTP與其他脊椎動物TTP聚為一支且與哺乳動物ZFP36家族的其他成員ZFP36L1、ZFP36L2和ZFP36L3等分離; 組織表達(dá)特異性分析顯示基因在檢測的9個大黃魚組織中廣泛表達(dá), 但不同組織表達(dá)水平差異較大; 在大黃魚頭腎細(xì)胞系LYC-hK中, LPS處理誘導(dǎo)炎癥因子表達(dá)時也調(diào)節(jié)基因的表達(dá), 提示LYC-hK細(xì)胞中TTP可能參與調(diào)控LPS誘導(dǎo)的炎癥因子表達(dá); LYC-hK細(xì)胞過表達(dá)TTP證實TTP可通過促進(jìn)TNF-αmRNA降解調(diào)節(jié)LPS誘導(dǎo)的TNF-α表達(dá)。這些研究結(jié)果提示脊椎動物中TTP的結(jié)構(gòu)和功能可能非常保守, 大黃魚TTP也可通過促進(jìn)炎癥因子mRNA降解調(diào)節(jié)炎癥因子表達(dá), 可能是大黃魚炎癥反應(yīng)的關(guān)鍵調(diào)控蛋白, 在大黃魚感染性病害防治策略開發(fā)中有潛在應(yīng)用價值。
ZFP36蛋白家族是以中央TZF結(jié)構(gòu)域為結(jié)構(gòu)特征的一類RNA結(jié)合蛋白, 廣泛存在于從單細(xì)胞真核生物、無脊椎動物到哺乳動物的整個真核生物界(Blackshear, 2014)。哺乳動物ZFP36蛋白家族通常有3個成員: TTP、ZFP36L1和ZFP36L2, 小鼠ZFP36蛋白家族還有一個在胎盤和胚胎外組織特異表達(dá)的ZFP36L3 (Blackshear, 2005)。魚類和兩棲類包含哺乳動物ZFP36家族的直系同源物和一個獨特的C3H-4蛋白, 該蛋白有一個典型的TZF結(jié)構(gòu)域和2個退化的鋅指結(jié)構(gòu)域(De, 1999)。TTP是ZFP36家族首先報道的原型蛋白, 通常有3個保守結(jié)構(gòu)域: N-端的NES信號序列、中央TZF結(jié)構(gòu)域和C-端的NOT1-BD (Ciais, 2013)。多序列比對顯示哺乳動物TTP的3個功能結(jié)構(gòu)域在TTP中也保守存在, 但LocNES工具包分析顯示TTP的NOT1-BD結(jié)構(gòu)域可能還是潛在的NES信號序列。ZFP36家族成員ZFP36L1和ZFP36L2缺失N端的NES信號序列, NES定位在C端替代TTP的NOT1-BD結(jié)構(gòu)域(Ciais, 2013)。而在系統(tǒng)樹中,TTP與其他硬骨魚TTP聚在一起, 合并其他脊椎動物TTP聚為一支, 與哺乳動物的ZFP36L1、ZFP36L2、ZFP36L3等分離。這些結(jié)果表明本研究克隆的TTP屬于典型的TTP蛋白, 也提示TTP可能與哺乳動物TTP有相似的功能。
基因組織表達(dá)水平與其生理功能密切相關(guān), 調(diào)查目的基因的組織表達(dá)特異性可為其功能研究提供線索(Lu, 2004)。TTP mRNA在成年小鼠各種組織中表達(dá)量均較低, 腎和肺中TTP mRNA表達(dá)量相對較高, 脂肪、心、肝、脾相對較低, 而在骨骼肌、睪丸和腦中只有痕量表達(dá)(Lai, 1990)。在蛋白水平, 肝、卵巢和睪丸中TTP水平高, 而腦、心、腸、脾、胸腺、腎、骨骼肌TTP水平較低, 肺中幾乎檢測不到TTP蛋白(Lu, 2004)。本研究檢測的9個大黃魚組織器官中均檢測到TTP mRNA表達(dá), 這與小鼠中觀察到TTP mRNA廣泛表達(dá)現(xiàn)象類似, 提示TTP可能也如哺乳動物TTP廣泛參與機(jī)體免疫反應(yīng)的調(diào)節(jié)。但大黃魚基因的組織表達(dá)特異性與小鼠基因存在差異,TTP mRNA在肌肉中的表達(dá)量最高, 其余依次為胃、腮、心、頭腎、脾、腎、肝和腦, 提示基因的組織表達(dá)特異性可能存在種間差異, 而種間差異的生理、病理意義還需在后續(xù)功能研究進(jìn)一步探討。
炎癥是宿主清除入侵病原的必需生理反應(yīng), 但高炎癥反應(yīng)和炎癥因子過量分泌也導(dǎo)致組織損傷甚至死亡, 因此, 免疫細(xì)胞炎癥因子的表達(dá)在轉(zhuǎn)錄水平和轉(zhuǎn)錄后水平都受到嚴(yán)格調(diào)控(Akira, 2021)。TTP是在轉(zhuǎn)錄后水平通過促進(jìn)炎癥因子mRNA降解下調(diào)炎癥因子表達(dá)的抑炎蛋白(Mukherjee, 2014)。哺乳動物巨噬細(xì)胞、樹突狀細(xì)胞、嗜中性粒細(xì)胞、T細(xì)胞、B細(xì)胞等免疫細(xì)胞中均表達(dá)TTP (Makita, 2021; Zhang, 2021)。哺乳動物巨噬細(xì)胞是TNF-α等炎癥因子的主要分泌細(xì)胞, 研究顯示LPS等炎癥誘導(dǎo)物處理巨噬細(xì)胞早期TTP與炎癥因子表達(dá)水平同步上調(diào), 處理后期TTP通過促進(jìn)炎癥因子mRNA降解抑制炎癥因子表達(dá)(Ostareck, 2019), 所以巨噬細(xì)胞是研究TTP調(diào)控炎癥因子表達(dá)的首選細(xì)胞類型。本研究曾嘗試轉(zhuǎn)染大黃魚原代單核/巨噬細(xì)胞過表達(dá)TTP研究TTP對TNF-α mRNA表達(dá)的調(diào)控效應(yīng)及其機(jī)制, 但因轉(zhuǎn)染效率低(<10%)放棄。而大黃魚頭腎細(xì)胞系LYC-hK轉(zhuǎn)染效率較高(50%~80%), 且LPS處理LYC-hK細(xì)胞早期, 炎癥因子TNF-α (3 h)和IL-1β (2 h)mRNA表達(dá)量顯著上調(diào), 并在誘導(dǎo)6 h時回復(fù)到基礎(chǔ)水平之下;TTP mRNA表達(dá)量小幅下調(diào)(1 h和2 h)后顯著上調(diào)(3 h)。這與LPS誘導(dǎo)哺乳動物炎癥因子和TTP mRNA表達(dá)的模式相似(Ostareck, 2019), 提示LYC-hK細(xì)胞中TTP可能參與調(diào)控LPS誘導(dǎo)的炎癥因子表達(dá)。因此, 本研究選擇大黃魚LYC-hK細(xì)胞系研究TTP對炎癥因子表達(dá)的調(diào)節(jié)效應(yīng)及其作用機(jī)制。
小鼠巨噬細(xì)胞TTP的主要靶RNA是炎癥因子(TNF-α、IL-1α、IL-1β、IL-6等)以及其他免疫相關(guān)蛋白的mRNA (Mukherjee, 2014; Sedlyarov, 2016)。基因敲除巨噬細(xì)胞TNF-α (Carballo, 1997, 1998)、GM-CSF (Carballo, 2000) mRNA的穩(wěn)定性增強(qiáng), mRNA及其編碼蛋白的表達(dá)量均上調(diào); 反之, 過表達(dá)TTP則顯著下調(diào)巨噬細(xì)胞LPS誘導(dǎo)的TNF-α、IL-1β、Cxcl2等mRNA的表達(dá)量(Patial, 2016b)。但在本研究中, LYC-hK細(xì)胞過表達(dá)TTP顯著上調(diào)LPS誘導(dǎo)早期(1 h) TNF-α mRNA表達(dá)量, 之后迅速下降到對照組水平(2 h)。Lai等(1999)報道HEK293細(xì)胞低水平過表達(dá)TTP下調(diào)TNF-α mRNA表達(dá)量, 但高水平過表達(dá)TTP反而上調(diào)TNF-αmRNA表達(dá)量(Lai, 1999); 以重組nLuc-ARE為靶mRNA, 過表達(dá)野生型TTP和組成型激活TTP-AA也觀察到相同現(xiàn)象, 即低水平過表達(dá)TTP下調(diào)nLuc-ARE mRNA表達(dá)量, 而高水平過表達(dá)TTP反而上調(diào)nLuc-ARE mRNA表達(dá)量(Mahmoud, 2019)。這些研究提示TTP作為核糖核酸外切酶復(fù)合體(exoribonuclease complex)支架蛋白, 高水平表達(dá)后可能由于復(fù)合體其他組分不足, 組裝完全的活性復(fù)合體反而減少, 導(dǎo)致核糖核酸外切酶總活性降低, 上調(diào)靶mRNA表達(dá)量; 隨著LPS誘導(dǎo)時間延長, 核糖核酸外切酶復(fù)合體其他結(jié)構(gòu)組分表達(dá)水平上調(diào), 組裝完全活性復(fù)合體增多, 靶mRNA降解加速而急劇下降(Burack, 2000; Mahmoud, 2019)。同時在本研究中也發(fā)現(xiàn): 放線菌素D抑制轉(zhuǎn)錄后, 過表達(dá)TTP促進(jìn)TNF-α mRNA降解, 下調(diào)TNF-α mRNA的半衰期, 表明TTP也如哺乳動物TTP, 可通過促進(jìn)靶RNA降解下調(diào)靶基因表達(dá)(Carballo, 1998; Deleault, 2008)。因此,TTP可能是大黃魚炎癥反應(yīng)的關(guān)鍵調(diào)控蛋白。
4 結(jié)論
本研究采用RACE技術(shù)克隆鑒定了基因的全長cDNA序列, 序列分析顯示哺乳動物TTP重要的功能結(jié)構(gòu)域在TTP中保守; 組織表達(dá)特異性分析顯示基因在大黃魚機(jī)體中廣泛表達(dá), 但不同組織表達(dá)水平差異較大; LYC-hK細(xì)胞過表達(dá)TTP發(fā)現(xiàn)TTP通過促進(jìn)TNF-αmRNA降解調(diào)節(jié)LPS誘導(dǎo)的TNF-α表達(dá)。這些結(jié)果提示脊椎動物中TTP的結(jié)構(gòu)和功能可能非常保守, 大黃魚TTP可通過促進(jìn)炎癥因子mRNA降解調(diào)節(jié)炎癥因子表達(dá), 可能是大黃魚炎癥反應(yīng)的關(guān)鍵調(diào)控蛋白, 在大黃魚感染性病害防治策略開發(fā)中有潛在應(yīng)用價值。
王凡, 廖碧釵, 孫敏秋, 等, 2019. 福建大黃魚產(chǎn)業(yè)發(fā)展形勢分析[J]. 中國水產(chǎn)(3): 45-49.
農(nóng)業(yè)農(nóng)村部漁業(yè)漁政管理局, 全國水產(chǎn)技術(shù)推廣總站, 中國水產(chǎn)學(xué)會, 2022. 2022中國漁業(yè)統(tǒng)計年鑒[J]. 北京: 中國農(nóng)業(yè)出版社: 22.
胡洋, 張旭, 王歡, 等, 2022. 藥用植物資源在水產(chǎn)動物疾病控制中的研究進(jìn)展[J]. 中國生物工程雜志, 42(11): 43-58.
Akira S, Maeda K, 2021. Control of RNA stability in immunity [J]. Annual Review of Immunology, 39: 481-509.
AO J Q, MU Y N, XIANG L X,, 2015. Genome sequencing of the perciform fishprovides insights into molecular and genetic mechanisms of stress adaptation [J]. PLoS Genetics, 11(4): e1005118.
BATACLAN M, LEONI C, MONTICELLI S, 2021. RNA‐binding proteins and RNA methylation in myeloid cells [J]. Immunological Reviews, 304(1): 51-61.
BLACKSHEAR P J, PERERA L, 2014. Phylogenetic distribution and evolution of the linked RNA-binding and NOT1-binding domains in the tristetraprolin family of tandem CCCH zinc finger proteins [J]. Journal of Interferon & Cytokine Research, 34(4): 297-306.
BLACKSHEAR P J, PHILLIPS R S, GHOSH S,, 2005., a rodent X chromosome gene encoding a placenta-specific member of the Tristetraprolin family of CCCH tandem zinc finger proteins [J]. Biology of Reproduction, 73(2): 297-307.
BURACK W R, SHAW A S, 2000. Signal transduction: hanging on a scaffold [J]. Current Opinion in Cell Biology, 12(2): 211-216.
CARBALLO E, BLACKSHEAR P J, 2001. Roles of tumor necrosis factor-α receptor subtypes in the pathogenesis of the tristetraprolin-deficiency syndrome [J]. Blood, 98(8): 2389-2395.
CARBALLO E, GILKESON G S, BLACKSHEAR P J, 1997. Bone marrow transplantation reproduces the tristetraprolin-deficiency syndrome in recombination activating gene-2 (-/-) mice. Evidence that monocyte/macrophage progenitors may be responsible for TNFalpha overproduction [J]. The Journal of Clinical Investigation, 100(5): 986-995.
CARBALLO E, LAI W S, BLACKSHEAR P J, 1998. Feedback inhibition of macrophage tumor necrosis factor-α production by tristetraprolin [J]. Science, 281(5379): 1001-1005.
CARBALLO E, LAI W S, BLACKSHEAR P J, 2000. Evidence that tristetraprolin is a physiological regulator of granulocyte- macrophage colony-stimulating factor messenger RNA deadenylation and stability [J]. Blood, 95(6): 1891-1899.
CHOU K C, SHEN H B, 2010. A new method for predicting the subcellular localization of eukaryotic proteins with both single and multiple sites: Euk-mPLoc 2.0 [J]. PLoS One, 5(4): e9931.
CIAIS D, CHERRADI N, FEIGE J J, 2013. Multiple functions of tristetraprolin/TIS11 RNA-binding proteins in the regulation of mRNA biogenesis and degradation [J]. Cellular and Molecular Life Sciences, 70(12): 2031-2044.
DAI Y F, LV Z M, YOU M X,, 2023. PPARα alleviates inflammation via inhibiting NF-κB/Rel pathway inchallenged[J]. Fish & Shellfish Immunology, 135: 108701.
DE J, LAI W S, THORN J M,, 1999. Identification of four CCCH zinc finger proteins in, including a novel vertebrate protein with four zinc fingers and severely restricted expression [J]. Gene, 228(1/2): 133-145.
DELEAULT K M, SKINNER S J, BROOKS S A, 2008. Tristetraprolin regulates TNF TNF-α mRNA stability via a proteasome dependent mechanism involving the combined action of the ERK and p38 pathways [J]. Molecular Immunology, 45(1): 13-24.
FAIRHURST A M, CONNOLLY J E, HINTZ K A,, 2003. Regulation and localization of endogenous human tristetraprolin [J]. Arthritis Research & Therapy, 5(4): 1-12.
FU S C, IMAI K, HORTON P, 2011. Prediction of leucine-rich nuclear export signal containing proteins with NESsential [J]. Nucleic Acids Research, 39(16): e111.
JOHNSON M, ZARETSKAYA I, RAYTSELIS Y,, 2008. NCBI BLAST: a better web interface [J]. Nucleic Acids Research, 36: W5-W9.
KAFASLA P, SKLIRIS A, KONTOYIANNIS D L, 2014. Post-transcriptional coordination of immunological responses by RNA-binding proteins [J]. Nature Immunology, 15(6): 492-502.
KRATOCHVILL F, MACHACEK C, VOGL C,, 2011. Tristetraprolin‐driven regulatory circuit controls quality and timing of mRNA decay in inflammation [J]. Molecular Systems Biology, 7(1): 560.
KUMAR S, STECHER G, TAMURA K, 2016. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets [J]. Molecular Biology and Evolution, 33(7): 1870-1874.
LAI W S, CARBALLO E, STRUM J R,, 1999. Evidence that tristetraprolin binds to AU-rich elements and promotes the deadenylation and destabilization of tumor necrosis factor alpha mRNA [J]. Molecular and Cellular Biology, 19(6): 4311-4323.
LAI W S, STUMPO D J, BLACKSHEAR P J, 1990. Rapid insulin-stimulated accumulation of an mRNA encoding a proline-rich protein [J]. Journal of Biological Chemistry, 265(27): 16556-16563.
LARKIN M A, BLACKSHIELDS G, BROWN N P,, 2007. Clustal W and Clustal X version 2.0 [J]. Bioinformatics, 23(21): 2947-2948.
LI S M, LIU S Y, CHEN R A,, 2022. Activation of the MKK3-p38-MK2-ZFP36 Axis by coronavirus infection restricts the upregulation of AU-rich element-containing transcripts in proinflammatory responses [J]. Journal of Virology, 96(5): e0208621.
LIVAK K J, SCHMITTGEN T D, 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCtmethod [J]. Methods, 25(4): 402-408.
LU J Y, SCHNEIDER R J, 2004. Tissue distribution of AU-rich mRNA-binding proteins involved in regulation of mRNA decay [J]. Journal of Biological Chemistry, 279(13): 12974-12979.
MAHMOUD L, MOGHRABI W, KHABAR K S A,, 2019. Bi-phased regulation of the post-transcriptional inflammatory response by Tristetraprolin levels [J]. RNA Biology, 16(3): 309-319.
MAHTANI K R, BROOK M, DEAN J L E,, 2001. Mitogen-activated protein kinase p38 controls the expression and posttranslational modification of tristetraprolin, a regulator of tumor necrosis factor alpha mRNA stability [J]. Molecular and Cellular Biology, 21(19): 6461-6469.
MAKITA S, TAKATORI H, NAKAJIMA H, 2021. Post-transcriptional regulation of immune responses and inflammatory diseases by RNA-binding ZFP36 family proteins [J]. Frontiers in Immunology, 12: 711633.
MARCHLER-BAUER A, BO Y, HAN L Y,, 2017. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures [J]. Nucleic Acids Research, 45(D1): D200-D203.
MEDZHITOV R, 2008. Origin and physiological roles of inflammation [J]. Nature, 454(7203): 428-435.
MUKHERJEE N, JACOBS N C, HAFNER M,, 2014. Global target mRNA specification and regulation by the RNA-binding protein ZFP36 [J]. Genome Biology, 15(1): R12.
OSTARECK D H, OSTARECK-LEDERER A, 2019. RNA-binding proteins in the control of LPS-induced macrophage response [J]. Frontiers in Genetics, 10: 31.
PATIAL S, BLACKSHEAR P J, 2016a. Tristetraprolin as a therapeutic target in inflammatory disease [J]. Trends in Pharmacological Sciences, 37(10): 811-821.
PATIAL S, CURTIS II A D, LAI W S,, 2016b. Enhanced stability of tristetraprolin mRNA protects mice against immune-mediated inflammatory pathologies [J]. Proceedings of the National Academy of Sciences of the United States of America, 113(7): 1865-1870.
SEDLYAROV V, FALLMANN J, EBNER F,, 2016. Tristetraprolin binding site atlas in the macrophage transcriptome reveals a switch for inflammation resolution [J]. Molecular Systems Biology, 12(5): 868.
TAYLOR G A, CARBALLO E, LEE D M,, 1996. A pathogenetic role for TNFα in the syndrome of cachexia, arthritis, and autoimmunity resulting from tristetraprolin (TTP) deficiency [J]. Immunity, 4(5): 445-454.
TCHEN C R, BROOK M, SAKLATVALA J,, 2004. The stability of tristetraprolin mRNA is regulated by mitogen-activated protein kinase p38 and by tristetraprolin itself [J]. Journal of Biological Chemistry, 279(31): 32393-32400.
TEUFEL F, ALMAGRO ARMENTEROS J J, JOHANSEN A R,, 2022. SignalP 6.0 predicts all five types of signal peptides using protein language models [J]. Nature Biotechnology, 40(7): 1023-1025.
UCHIDA Y, CHIBA T, KURIMOTO R,, 2019. Post-transcriptional regulation of inflammation by RNA- binding proteins via-elements of mRNAs [J]. The Journal of Biochemistry, 166(5): 375-382.
XU K P, WANG Y S, YANG W X H,, 2022. Strategies for prevention and control of vibriosis in Asian fish culture [J]. Vaccines, 11(1): 98.
YE J, COULOURIS G, ZARETSKAYA I,, 2012. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction [J]. BMC Bioinformatics, 13: 134.
YOSHINAGA M, TAKEUCHI O, 2019. Post‐transcriptional control of immune responses and its potential application [J]. Clinical & Translational Immunology, 8(6): e1063.
ZHANG Y W, ZHOU J, WEI Z Y,, 2021. TTP-mediated regulation of mRNA stability in immune cells contributes to adaptive immunity, immune tolerance and clinical applications [J]. RNA Biology, 18(12): 2150-2156.
ZHOU S M, WANG Y, SHU F L,, 2023. Functional insights of a two-component system sensor kinase GacS in a fish pathogen,[J]. Aquaculture, 562: 738866.
MOLECULAR CHARACTERIZATION AND FUNCTIONAL ANALYSIS OF TRISTETRAPROLIN FROM LARGE YELLOW CROAKER
WANG Hui-Juan1, HE Zhi-Qiao1, SHI Ge1, ZHOU Su-Ming2, ZHANG Xiao-Lin1, SHEN Wang1
(1. Marine Science and Technical College, Zhejiang Ocean University, Zhoushan 316022, China; 2. School of Marine Sciences, Ningbo University, Ningbo 315211, China)
Tristetraprolin (TTP) is an RNA binding protein and widely distributed in eukaryotes. In mammals, TTPs decrease the expression of inflammatory factors by promoting mRNA decay or inhibiting translation to regulate the balance of inflammatory response, and has been considered as a potential therapeutic target for inflammatory diseases. A full-length cDNA sequence of TTP in large yellow croaker (; denoted asTTP) was obtained by rapid amplification of cDNA ends (RACE) method. The full-length cDNA ofTTP was 1 508 bp, including a 77-bp 5'-untranslated region (5'-UTR), a 183-bp 3'-UTR, and a 1 248-bp open reading frame (ORF) encoding 418 amino acid residues. The theoretical molecular weight (MW) ofTTP is 44 897.78 Da, and the predicted isoelectric point (pI) was 8.37. The important functional domains of mammalian TTP are well conserved inTTP, including the N-terminal nuclear export sequence (NES), the central CCCH-type tandem zinc finger domain (TZF), and the C-terminal NOT1-binding domain (NOT1-BD). Phylogenetic analysis showed thatTTP is clustered into one clade with other vertebrate TTPs and separated from the other mammalian ZFP36 family members, including ZFP36L1, ZFP36L2, and ZFP36L3. Tissue expression pattern analysis indicated that the mRNA transcriptsofTTP were detected in all the 9 examined tissues with the highest expression level in muscle. In LYC-hK cells (a large yellow croaker kidney cell line), the overexpression ofTTP significantly increased the expression level of TNF-αin the early phase after LPS challenge and peaked in 1 h, and then rapidly decreased near to the control level in 1.5 h. After inhibition of transcription by actinomycin D, the overexpression ofTTP significantly increased the degradation rate of TNF-α mRNA, indicating thatTTP can regulate the expression of TNF-α by promoting mRNA decay. These results imply thatTTP is the key regulator of inflammatory responses and a potential target for the control of infectious diseases in large yellow croaker.
; RNA binding protein; Tristetraprolin; inflammatory response; posttranscriptional regulation
* 浙江海洋大學(xué)科研啟動經(jīng)費, 2020。汪慧娟, 碩士研究生, E-mail: wang_hui_j@163.com
申 望, 副教授, E-mail: shenwang@zjou.edu.cn
2023-05-08,
2023-07-29
Q789; S965
10.11693/hyhz20230500103

