dsRNA和Aza-CdR轉(zhuǎn)染豬腎細胞的全基因組差異甲基化區(qū)域分布特征的比較
王 勇,王懷棟,郭永清,俞 英,王楚端,王曉鑠, *
(1. 內(nèi)蒙古農(nóng)業(yè)大學(xué)職業(yè)技術(shù)學(xué)院,包頭014109;2. 中國農(nóng)業(yè)大學(xué)動物科學(xué)技術(shù)學(xué)院,北京100193)
豬RNA病毒的模板有正鏈病毒RNA模板、負鏈病毒RNA模板和全長正負鏈反基因組RNA模板,即雙鏈RNA(double-stranded RNA,dsRNA)模板。dsRNA作為單鏈RNA5 OAS)/ RNaseL系統(tǒng)所識別,并誘導(dǎo)Ⅰ型干擾素(IFN-α / β)的產(chǎn)生。Poly I:C(Polyinosinic: polycytidylic acid)為多聚肌苷酸Poly(I)和多聚胞苷酸Poly(C)的共聚物,通常用于模擬病毒的復(fù)制中間體雙鏈RNA(dsRNA)。通過合成不同長度的Poly I:C,研究dsRNA作為病毒或者免疫刺激劑轉(zhuǎn)染宿主細胞的分子免疫機制。Liu等認為短片段的 polymer(~300 bp) 不會激活MDA-5,但它是RIG-I潛在的配體。相反的,長片段的 Poly I:C 更偏好激活MDA-5,長度≥63 kb的PolyI:C 通常用于研究RNA sensing。但關(guān)于dsRNA轉(zhuǎn)染豬細胞的全基因組甲基化機制仍沒有被研究。
全基因組甲基化是近些年發(fā)展較快的新興學(xué)科,就像分子生物學(xué)的發(fā)展由單個基因進入全基因組的研究一樣,DNA甲基化也由對少數(shù)位點的修飾分析進入了全基因組水平的“全景式”研究階段。DNA甲基化與人類發(fā)育和腫瘤等疾病密切相關(guān),特別是CpG島高甲基化導(dǎo)致抑癌基因轉(zhuǎn)錄失活。近些年,畜禽重要經(jīng)濟性狀的DNA甲基化已經(jīng)成為表觀基因組學(xué)的重要研究內(nèi)容之一。差異甲基化區(qū)域(differential methylation region,DMR)是指在不同組織類型或疾病狀態(tài)下的細胞水平上發(fā)生的差異甲基化區(qū)域。2019年,Wang等解析了PolyI:C結(jié)合Aza-CdR共同轉(zhuǎn)染豬腎細胞的甲基化圖譜,定量分析DMR和甲基化標記,揭示病毒模擬物Poly I:C對甲基化抑制劑Aza-CdR具有潛在的抑制作用,這些結(jié)果將提高人類對哺乳動物疾病的了解和診斷具有重要的實用價值。全基因組DNA甲基化圖譜通過分析試驗組與對照組的DMR,以尋找重要的表觀遺傳標記(epigenetic marker),這將有助于了解正常和疾病狀態(tài)下不同基因相互作用的網(wǎng)絡(luò)調(diào)控關(guān)系,不僅可為病毒感染、腫瘤和糖尿病等復(fù)雜疾病的深入研究提供新的理論依據(jù),還可為環(huán)境因素、營養(yǎng)和衰老等研究提供新的方法。
DNA甲基化轉(zhuǎn)移酶抑制劑5-氮脫氧胞苷[5-aza-2′-deoxycytidine(脫氧胞核嘧啶),Aza-CdR],具有很強的甲基化抑制作用,在細胞培養(yǎng)中可以介導(dǎo)細胞分化和基因表達。含Aza-CdR的DNA與甲基化酶形成的穩(wěn)定共價復(fù)合體使甲基基團不能轉(zhuǎn)移到胞嘧啶,導(dǎo)致DNA甲基化反應(yīng)受阻,從而使基因組甲基化水平降低。有研究表明,Aza-CdR會抑制DNA的復(fù)制、轉(zhuǎn)錄,還可能在哺乳動物細胞內(nèi)誘發(fā)基因表達變化、細胞程序性死亡等,也可以抑制病毒RNA成熟所需要的甲基化反應(yīng)。Aza-CdR常用于兩個領(lǐng)域:1)在DNA甲基化抑制基因表達的生物功能試驗中做驗證對照,進而深入挖掘DNA甲基化標記;2)用于治療某些特異的腫瘤。2019年,Chen等利用5-Aza-CdR抑制DNMT1表達,增加腸癌細胞凋亡的速率。2015年,Roulois等證明低劑量5-Aza-CdR可通過誘導(dǎo)病毒模擬物靶向應(yīng)對結(jié)直腸癌啟動細胞(CICs),這與部分源自內(nèi)源性逆轉(zhuǎn)錄病毒元件的dsRNA誘導(dǎo)、MDA5/MAVS RNA識別通路的激活以及IRF7的下游激活有關(guān)。2009年,Koga等利用MeDIP-chip方法鑒定5個新的人類黑色素瘤甲基化標記,并結(jié)合Aza-CdR進行驗證。基于此,本研究利用Poly I:C和Aza-CdR轉(zhuǎn)染豬腎上皮細胞系PK15,模擬dsRNA病毒的感染及抗病毒作用。通過與非轉(zhuǎn)染的PK15細胞進行對比,分析豬腎細胞全基因組差異甲基化基因并篩選抗病毒相關(guān)通路,為腎感染的病毒性疾病研究提供分子依據(jù),進而揭示 Aza-CdR 和 dsRNA 對豬腎細胞的甲基化效應(yīng)。
1 材料與方法
1.1 試驗用細胞及 MeDIP-chip 芯片設(shè)計
以豬腎上皮細胞系PK15(購自中國武漢典型培養(yǎng)物保藏中心,細胞編號:3115CNCB00260)為試驗材料,10%熱滅活胎牛血清、100單位·mL青鏈霉素37 ℃、5% CO條件下培養(yǎng)(具體培養(yǎng)方法參考文獻[4])。當細胞生長至70%~80%時,饑餓培養(yǎng)2 h, 以達到細胞平衡狀態(tài)。使用Poly I:C(P)和Aza-CdR(A)轉(zhuǎn)染PK15細胞,收集處理的細胞,進行DNA甲基化芯片檢測和分析。共3組細胞:1)Mock 細胞(C組);2)10 μg·mLPolyI:C轉(zhuǎn)染10 h的PK15細胞(P組);3)5 μmol·LAza-CdR轉(zhuǎn)染6 h的PK15細胞(A組)。細胞的時間和劑量依賴試驗結(jié)果見參考文獻[18]。每組3個重復(fù),共計9個樣本,用于MeDIP-chip芯片檢測。9張芯片來源于Nimbgen公司設(shè)計的Pig 385 K CpG Island plus Ensembl Promoter Array Methylation Chip,最終完成本試驗豬DNA甲基化差異區(qū)域的注釋。
1.2 PK15細胞DNA的抽提
使用Wizard基因組DNA純化試劑盒(Promega)提取細胞基因組DNA。利用NanoDropND-2000c分光光度計測定DNA濃度后,再使用EZ DNA甲基化金標準試劑盒(Zymo Research)對每個樣本1 μg基因組DNA進行重亞硫酸鹽轉(zhuǎn)化。用20 μL緩沖液洗脫轉(zhuǎn)化的DNA用于免疫共沉淀。
1.3 DNA免疫共沉淀和芯片雜交
基因組DNA 經(jīng)超聲波破碎后,樣本用于免疫共沉淀試驗。每個樣本的免疫共沉淀處理分為3份,第1份是Input DNA,第2份是 MeDIP DNA,第3份是 Mock IP DNA。分別對9張 MeDIP 芯片進行qPCR 質(zhì)檢。1)Input 試驗:加入0.4 μg樣本基因組DNA,不加任何抗體;2)MeDIP 試驗:加入2 μg 樣本基因組DNA 和5 μg樣本甲基化DNA抗體(抗體來源于豬 Anti-5-methylcytidine antibody,Diagenode);3)Mock 試驗:加入5 μg的小鼠IgG和2 μg的樣本DNA(小鼠IgG不能和樣本DNA 發(fā)生作用)。MeDIP DNA 和Input DNA 分別用Cy 5和 Cy 3標記,檢測合格后,與豬385 K全基因組CpG島(CGI)和啟動子芯片進行雜交。
1.4 原始數(shù)據(jù)校正
免疫共沉淀的樣本經(jīng)芯片雜交后得到原始數(shù)據(jù)(raw data),原始數(shù)據(jù)包括每個探針的熒光信號強度,這些數(shù)據(jù)以log(MeDIP / Input)(即 logratio)及value來表示。logratio即 MeDIP DNA 的熒光信號和 Input DNA的熒光信號比值的對數(shù)值。為了避免技術(shù)因素對試驗結(jié)果的影響,以便更真實地反映樣品間DNA甲基化的差異,試驗對芯片得到的原始logratio進行校正。利用中位數(shù)中心和分位數(shù)校正(median-centering and quantile normalization)方法對試驗組中每個樣品的logratio 值進行校正,經(jīng)過校正后所有芯片數(shù)據(jù)的中值都在0左右,并且樣本間在上下區(qū)間的分布也趨于相似,表明數(shù)據(jù)可用于后續(xù)分析。
1.5 芯片數(shù)據(jù)分析
由NimbleScan v2.6(Roche NimbleGen,05933315001)計算logratio值。logratio值代表每個探針在MeDIP DNA和Input DNA中的富集強度,值代表每個探針的紅綠信號差異是由非生物因素造成的概率,值由修正的KS檢驗算法計算。值越低,表示探針越有可能代表一個甲基化事件,進而找到富集的甲基化峰(enrichment peaks,EPs)。Peaks代表可能的DNA甲基化區(qū)域,由專業(yè)商用軟件SignalMapv1.9(Roche NimbleGen,05225051001)計算。
1.6 差異甲基化區(qū)域計算
多樣本之間甲基化區(qū)域的比較由差異甲基化峰(differential enrichment peaks,DEPs)表示,通過兩兩組別之間的差異值logratio 計算M值,M值(也稱作Peak DM value值)是指組間的差異甲基化峰值(DEPs),可代表兩組之間的差異甲基化區(qū)域(DMR)。計算方法:M=Average(logMeDIP E / Input E)- Average(logMeDIP C / Input C),其中,E代表試驗組,C代表對照組。根據(jù)NimbleScan Algorithm判定DEPs時有兩個條件:1)兩組中至少有一組log(MeDIP / Input) > 0.3 和M > 0;2)在一個Peak 中,兩組中至少有一半的探針變異系數(shù)(coefficient variability,CV)≤0.8。本研究分析了PolyI:C. Control(P. C)和Aza-CdR. Control(A. C)兩個比較組間的差異甲基化區(qū)域分布特征。
1.7 DNA bisulfite PCR 和 Bisifite clone sequencing
應(yīng)用Oligo 6.0軟件設(shè)計目的基因和持家基因引物,使用Zymo Premix啟動PCR熱循環(huán)。PCR熱循環(huán)條件:95 ℃ 10 min;94 ℃ 30 s,55 ℃ 45 s,72 ℃ 45 s,40~45循環(huán);72 ℃ 10 min。PCR 產(chǎn)物用于后續(xù)測序試驗。Bisifite clone sequencing前試驗主要包括PCR產(chǎn)物切膠回收,感受態(tài)細胞制備(CaCl法),載體連接反應(yīng),質(zhì)粒DNA轉(zhuǎn)化。PCR產(chǎn)物克隆至pGM-T載體,使用ABI 377自動測序儀完成測序反應(yīng)。挑取8~10個克隆用于Bisulfite克隆測序分析。
2 結(jié) 果
2.1 Poly I:C和Aza-CdR轉(zhuǎn)染豬PK15細胞的全基因組差異甲基化峰分布
試驗分析了兩個比較組P. C和A. C基因間區(qū)域(intergenic)、基因內(nèi)區(qū)域(introgenic)和啟動子區(qū)(promoter)3個區(qū)域的差異甲基化峰(DEPs)分布。正常的豬腎上皮細胞在全基因組區(qū)域(intergenic、introgenic、promoter)均有DEPs分布,而Poly I:C(圖1A)和Aza-CdR(圖1B)分別轉(zhuǎn)染PK15細胞的DEPs則主要在高密度CpG含量啟動子(high-CpG-containing promoters,HCPs)增加顯著,中等密度CpG含量啟動子(inter mediate-CpG-containing promoters,ICPs)和低密度CpG含量啟動子(low-CpG-containing promoters,LCPs)并沒有顯著增加。結(jié)果說明,兩種化學(xué)物質(zhì)處理細胞后,DNA甲基化的變化主要發(fā)生在啟動子位置,特別是與啟動子甲基化的密度相關(guān)聯(lián)。
為更進一步確認富集的差異甲基化峰(DEPs)在全基因組的分布,試驗將DEPs分為上調(diào)的甲基化峰和下調(diào)的甲基化峰。若差異探針分布在Poly I:C處理組(或Aza-CdR組)中而不分布在Control對照組中,則為上調(diào)的甲基化峰;若差異探針分布在Control對照組中而不分布在Poly I:C處理組(或Aza-CdR組)中,則為下調(diào)的甲基化峰。在兩個比較組P. C和A. C中,高密度CpG島啟動子(HCPs)下調(diào)的DEPs 較多,分別為394個下調(diào)DEPs(81%,92個上調(diào)DEPs,圖1C)和223個下調(diào)DEPs(54%,190個上調(diào),圖1D),初步推斷Poly I:C和Aza-CdR對豬PK5細胞均具有去甲基化作用,而這一現(xiàn)象在ICPs和LCPs中并沒有被發(fā)現(xiàn)。
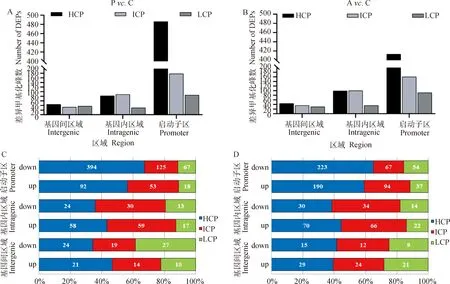
A. Poly I:C轉(zhuǎn)染豬PK15 細胞的差異甲基化峰(DEPs)數(shù);B. Aza-CdR轉(zhuǎn)染豬PK15 細胞的DEPs數(shù);C. Poly I:C轉(zhuǎn)染豬PK15 細胞的DEPs分布;D. Aza-CdR轉(zhuǎn)染豬PK15 細胞的DEPs分布A. The number of differential methylation peaks (DEPs) of Poly I: C transfected porcine PK15 cells; B. The number of DEPs of porcine PK15 cells transfected with Aza-CdR; C. DEPs distribution of Poly I: C transfected porcine PK15 cells; D. DEPs in porcine PK15 cells transfected with Aza-CdR圖1 Poly I:C和Aza-CdR轉(zhuǎn)染豬PK15 細胞的全基因組差異甲基化峰分布Fig.1 Genome differential methylation peaks in PK15 cells transfected with Poly I:C and Aza-CdR
2.2 Poly I:C和Aza-CdR轉(zhuǎn)染豬PK15細胞的染色體差異甲基化峰分布
為進一步研究染色體上不同密度的CpG島及甲基化峰在染色體上的分布情況,試驗分析了甲基化峰值Peak DM value與染色體的關(guān)系。利用R軟件繪制箱型圖,結(jié)果如圖2所示,橫軸代表不同的染色體,縱軸代表不同染色體上富集的差異甲基化峰值。結(jié)果說明,不同染色體上甲基化水平不同。當Poly I:C轉(zhuǎn)染PK15細胞后,染色體水平上甲基化峰值均<0.5(圖2A);當Aza-CdR轉(zhuǎn)染細胞后,染色體水平上甲基化峰值在0.5左右波動(圖2B)。結(jié)果證明,Poly I:C可能具有去甲基化作用,而Aza-CdR除了具有去甲基化作用,對某些區(qū)域也會上調(diào)甲基化,如1、10、11和18號染色體區(qū)域Peak DM value值(即M值)分別為0.52、0.58、0.57和0.56(圖2B)。
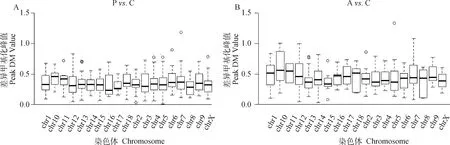
圖2 Poly I:C(A)和Aza-CdR(B)轉(zhuǎn)染豬PK15細胞的染色體差異甲基化峰分布Fig.2 Chromosome differential methylation peaks in PK15 cells transfected with Poly I:C (A) and Aza-CdR(B)
2.3 Poly I:C和Aza-CdR轉(zhuǎn)染豬PK15細胞的啟動子差異甲基化峰分布
將啟動子-1 500—+500 bp區(qū)劃分為3個區(qū)域:-1 500—-500 bp(遠端啟動子,distal promoter),-500—-200 bp(中端啟動子,middle promoter)和-200—+500 bp(近端啟動子和第一外顯子,proximal promoter and first exson)。結(jié)果揭示,在P. C和A. C兩個比較組中,-200—+500 bp區(qū)域中的甲基化陽性富集峰最多(綠色),其次為-500—-200 bp區(qū)域(紅色),最后為-1 500 —-500 bp(藍色)(圖3)。結(jié)果表明,Poly I:C和Aza-CdR具有比較相似的啟動子甲基化分布特征。

圖3 Poly I:C(A)和Aza-CdR(B)轉(zhuǎn)染豬PK15細胞的啟動子差異甲基化峰分布Fig.3 Promoter differential methylation peaks in PK15 cells transfected with Poly I:C (A) and Aza-CdR (B)
在差異甲基化區(qū)域DMR中,Peaks內(nèi)探針的平均-lg(值)反映了陽性富集的可能性大小(即Peak Score,Cut off = 2,值<0.01)。若Peak Score ≥ 2,則這個Peak極顯著代了一個甲基化事件。發(fā)現(xiàn)甲基化的富集程度在Poly I:C和Aza-CdR對Control組中的-200—+200 bp 區(qū)域富集程度較高[圖4,- lg (值) ≥ 2,<0.01]。試驗用3種不同顏色的線代表-800—+200 bp區(qū)域差異表達基因3種不同的表達水平,綠色線代表表達量最低(1 st),紅色線代表表達量最高(3rd)。通過與表達譜差異基因的結(jié)合分析,如圖4 所示,在-200—+200 bp的近端啟動子及第一外顯子區(qū)域,甲基化富集水平(代表甲基化程度)與差異基因的表達水平存在負相關(guān)關(guān)系(圖4箭頭所示)。結(jié)果表明,PK15細胞的DNA甲基化影響基因表達與甲基化修飾的位置相關(guān)。在0—+ 200 bp(P. C和A. C,圖4A和4B黑色箭頭所示)區(qū)域和-800—-600 bp (A. C,圖4B紅色箭頭所示)區(qū)域內(nèi)基因表達受DNA甲基化調(diào)控,即甲基化富集程度越高表達量越低;而在-600—0 bp區(qū)域,DNA甲基化不調(diào)控基因表達。
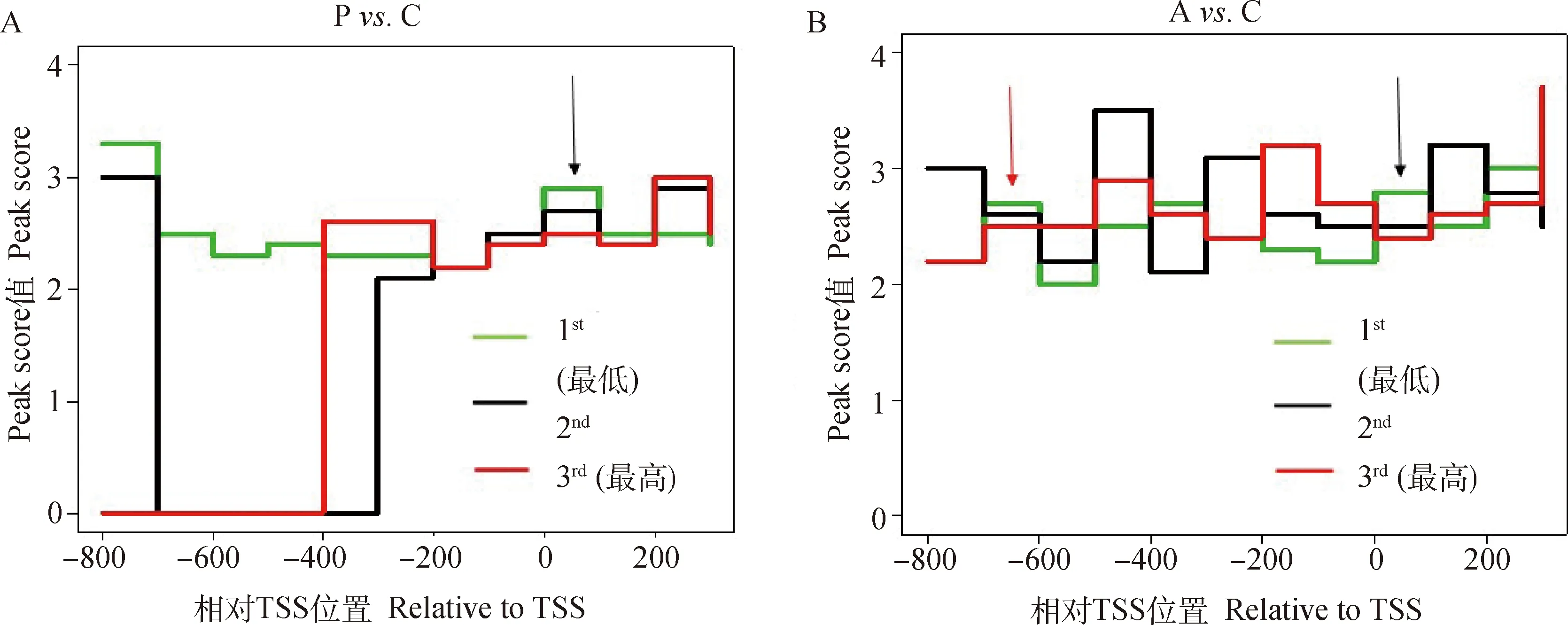
A. Poly I:C轉(zhuǎn)染豬PK15 細胞;B. Aza-CdR轉(zhuǎn)染豬PK15 細胞A. Poly I: C transfected porcine PK15 cells; B. PK15 cells transfected with Aza-CdR圖4 啟動子差異甲基化峰的分布及其與表達的關(guān)系Fig.4 Promoter methylation peaks amd its relationship with gene expression
圖5結(jié)果顯示了差異甲基化峰在染色體上的富集情況[- lg (值)≥2,<0.01]。Poly I:C處理細胞的啟動子中等密度的ICPs主要富集在9號和17號染色體上,而HCP和LCP富集程度較一致(圖5A);Aza-CdR處理細胞的啟動子CpG島密度與染色體分布無明顯的相關(guān)性(圖5B)。

圖5 Poly I:C(A)和Aza-CdR(B)轉(zhuǎn)染豬PK15細胞的染色體上啟動子差異甲基化峰分布Fig.5 Chromosome promoter differential methylation peaks in PK15 transfected withPoly I:C (A) and Aza-CdR (B)
2.4 Poly I:C和Aza-CdR轉(zhuǎn)染豬PK15細胞的全基因組差異甲基化基因GO分析
和表達譜分析一樣,本研究對差異甲基化基因進行功能分類,利用Gene Ontology(GO)注釋結(jié)合fisher’s exact test(值<0.05)檢驗方法進行分析。如圖6所示,這些在P. C比較組中上調(diào)和下調(diào)的差異甲基化基因都有各自獨特的富集GO terms,基于<0.05 的條件下,結(jié)果揭示Poly I:C 主要參與細胞生長調(diào)控、細胞成分組成及發(fā)育過程等,其中,富集程度最高的基因參與了巨噬細胞分化的調(diào)控(圖6A);Aza-CdR 主要參與調(diào)控Rho信號轉(zhuǎn)導(dǎo)、對病毒響應(yīng)的防御、調(diào)控器官生長等(圖6B)。
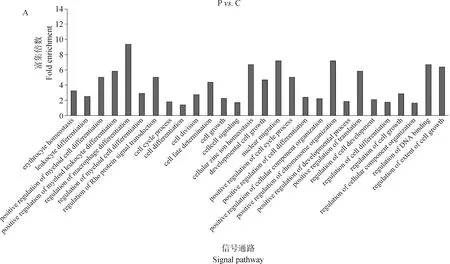
圖6 Poly I:C(A)和Aza-CdR(B)轉(zhuǎn)染豬PK15細胞的差異甲基化基因GO分析Fig.6 GO analysis of differential methylation genes in PK15 cells transfected with Poly I:C (A) and Aza-CdR (B)
2.5 篩選Poly I:C和Aza-CdR轉(zhuǎn)染豬PK15細胞的啟動子差異甲基化區(qū)域DMR
分析P. C(表1)和A. C(表2)比較組中的差異甲基化基因發(fā)現(xiàn),Poly I:C轉(zhuǎn)染的細胞中Peroxisome Proliferator-Activated Receptor Gamma(PPARG)、Follistatin(FST)等基因啟動子甲基化下調(diào),主要參與細胞分化、發(fā)育的調(diào)控。Aza-CdR除了具有調(diào)節(jié) DNA 甲基化的修飾作用外,它還具有調(diào)控其他表觀遺傳修飾、誘導(dǎo)細胞凋亡及參與機體免疫反應(yīng)的作用,例如DFP3在調(diào)控心肌細胞發(fā)育方面發(fā)揮作用、BNIP3L參與病毒的繁殖等過程,這些基因的復(fù)雜調(diào)控可通過調(diào)節(jié)DNA 的甲基化來抑制或增強基因表達。為了明確差異甲基化區(qū)域DMR,試驗選擇BCL2/腺病毒E1B相互作用蛋白3樣基因(BNIP3L,BCL2/adenovirus E1B interacting protein 3-like)進行比對,利用NimbleGen公司的Signalmap軟件分析基因啟動子區(qū)富集的差異甲基化峰,結(jié)果顯示,在14號染色體的10459946—10460615 bp區(qū)段共有669 bp Peak Length CG位點發(fā)生去甲基化(圖7)。
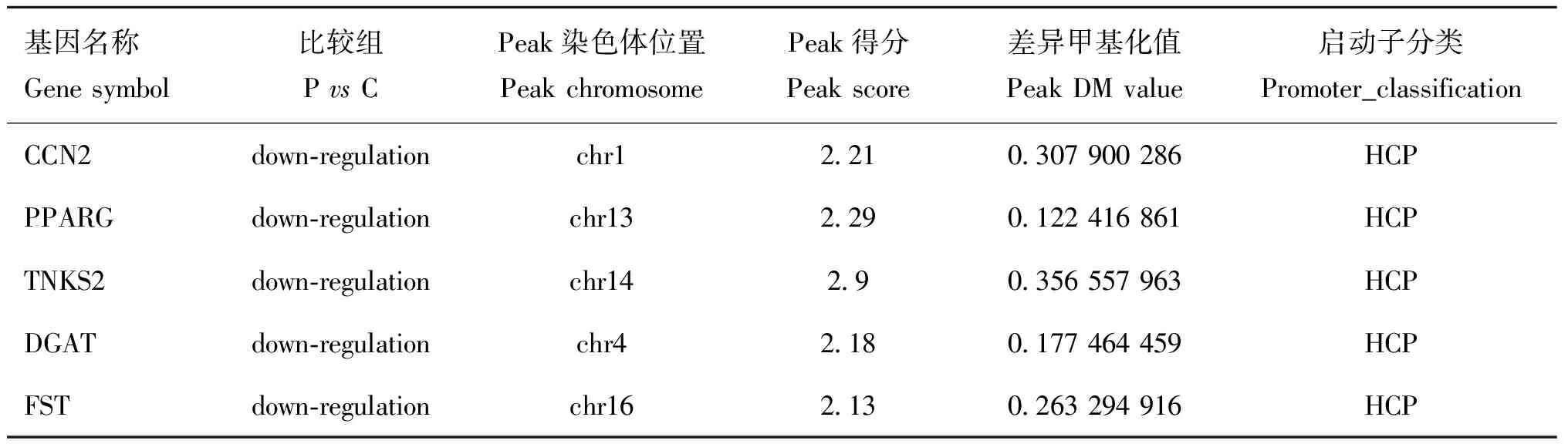
表1 Poly I:C處理豬PK5細胞后啟動子差異甲基化基因
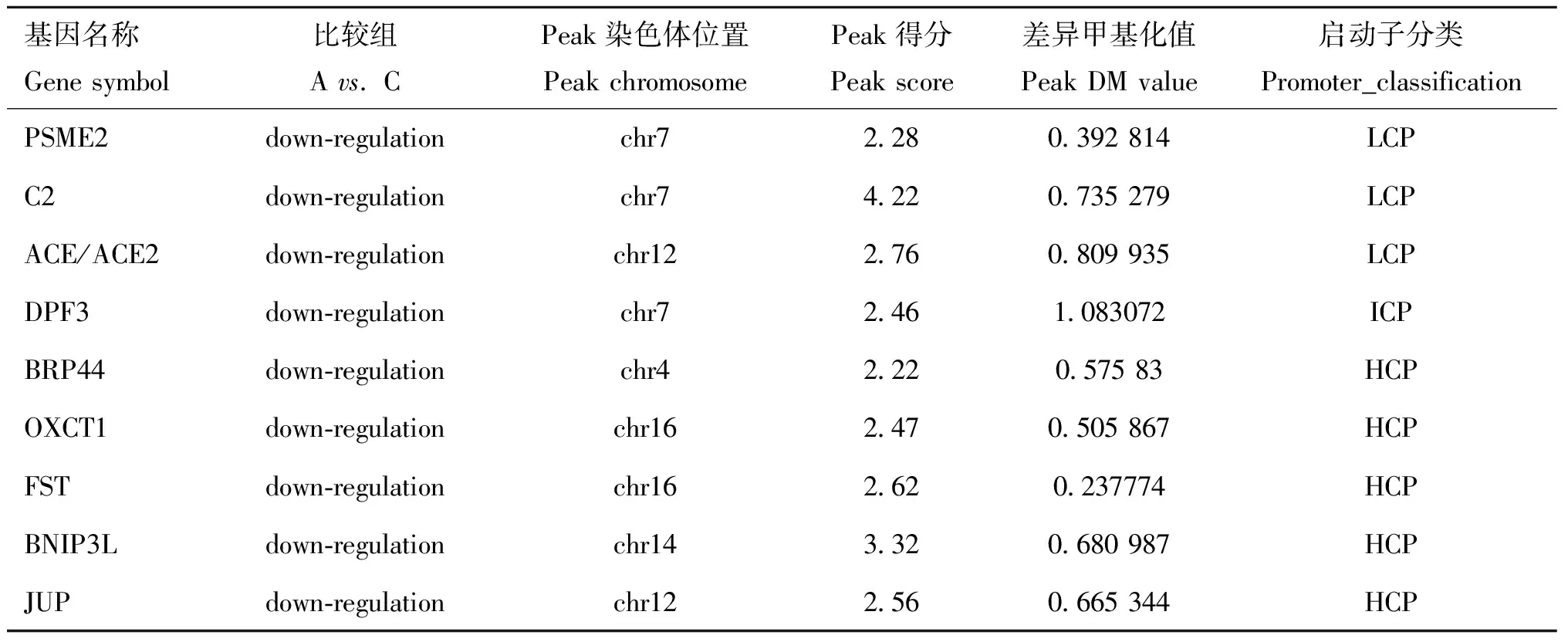
表2 Aza-CdR處理豬PK5細胞后啟動子差異甲基化基因
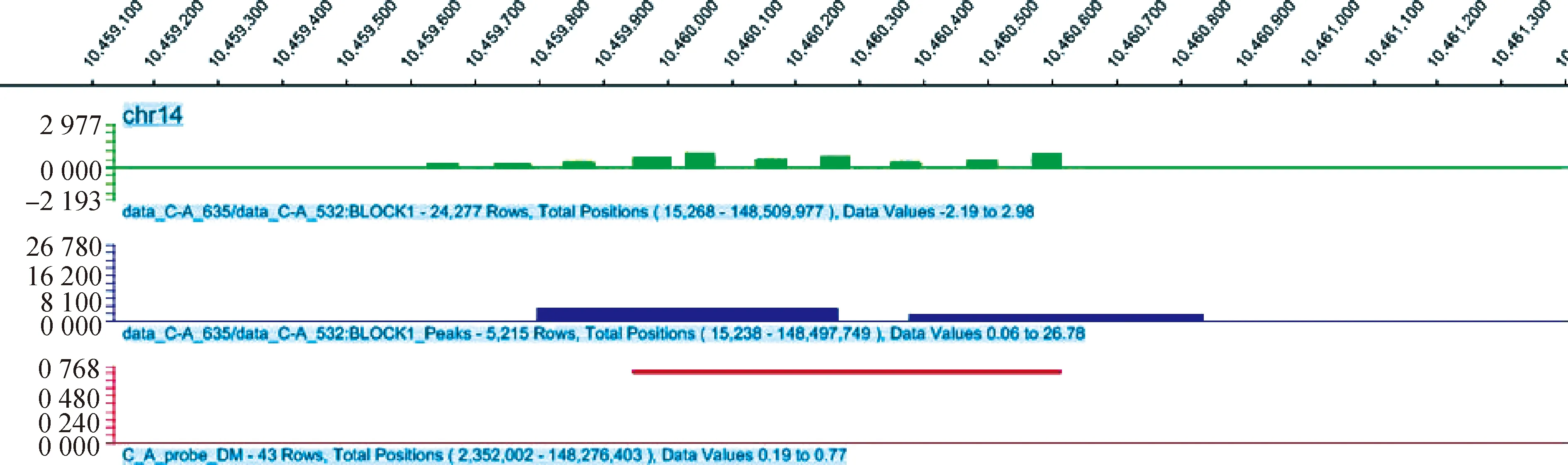
圖7 豬BNIP3L基因的啟動子差異甲基化區(qū)域DMRFig.7 Promoter differential methylation region of porcine BNIP3L gene
2.6 驗證PolyI:C和Aza-CdR轉(zhuǎn)染豬PK15 BNIP3L基因啟動子差異甲基化區(qū)域DMR
為了獲得更精確的3L啟動子甲基化差異信息,使用高敏感的bisulfite測序方法測定3L基因啟動子區(qū)甲基化差異變化,檢測上述區(qū)域20個CpG位點(Chr14: 10459946—10460615 bp)。與未轉(zhuǎn)染的細胞(DNA甲基化程度為93.8%)相比,Poly I:C處理的DNA甲基化水平顯著降低至35.0 %,Aza-CdR轉(zhuǎn)染的細胞DNA甲基化程度降低至26.3%(圖8A)。
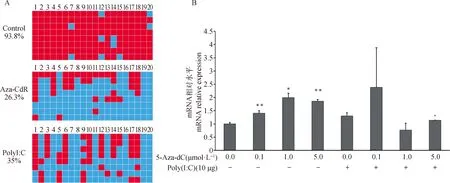
A. BNIP3L基因啟動子甲基化克隆測序分析;B. BNIP3L基因mRNA表達量分析A. Clone-sequencing of BNIP3L gene promoter; B. mRNA expression level of BNIP3L gene圖8 豬BNIP3L基因的啟動子差異甲基化區(qū)域DMR的驗證Fig.8 Validation of promoter differential methylation region of porcine BNIP3L gene
為了進一步驗證PolyI:C通過去甲基化機制啟動3L基因的轉(zhuǎn)錄表達,試驗引入甲基化抑制劑Aza-CdR分析去甲基化與轉(zhuǎn)錄表達的關(guān)系。試驗使用0.1、1.0、5.0 μmol·LAza-CdR處理細胞,同時在10.0 μg·mLPolyI:C轉(zhuǎn)染4 h后再使用Aza-CdR不同濃度處理細胞6 h,分析3L基因的轉(zhuǎn)錄表達。試驗結(jié)果顯示,0.1、1.0、5.0 μmol·LAza-CdR和10.0 μg·mLPoly I:C單獨處理細胞時均顯著表達BNIP3L基因(<0.05)(圖8B)。然而,5.0 μmol·LAza-CdR 單獨處理細胞時,與未處理的細胞相比,BNIP3L差異表達2倍以上,并達到及其差異顯著水平(<0.01)。
3 討 論
本研究首次在全基因組范圍研究豬腎細胞的 DNA 甲基化圖譜的試驗,獲得了 3組樣本(Control細胞,Poly I:C轉(zhuǎn)染細胞,Aza-CdR轉(zhuǎn)染細胞)所有特有的全基因組甲基化分布特點。豬腎細胞全基因組DNA甲基化主要分布于5′調(diào)控區(qū)域。試驗在組間比較后,特別是在P. C和A. C比較中發(fā)現(xiàn)DNA甲基化在 基因組上的分布特征與CpG島密度與距離TSS的位置有關(guān)。Poly I:C對PK15作用使得TSS附近200 bp(-200—+500 bp 處,即近啟動子區(qū)和第一外顯子區(qū))低甲基化啟動子增多(圖3),可能這種低甲基化模式更利于轉(zhuǎn)錄因子的結(jié)合進而起始轉(zhuǎn)錄。Tang等報道dsRNA轉(zhuǎn)染人胚腎細胞(HEK293T)和人肺上皮細胞(A549)后激活I(lǐng)L6啟動子,增加IL6轉(zhuǎn)錄表達。該研究同時使用DNA甲基化轉(zhuǎn)移酶抑制劑5-aza-2′-deoxycytidine處理細胞作為對照,IL6表達上調(diào)。這些結(jié)果表明dsRNA通過抑制DNMT活性從而誘導(dǎo)啟動子去甲基化激活I(lǐng)L6表達,發(fā)揮抗流感病毒的作用。Li等研究發(fā)現(xiàn)在流感病毒感染的細胞中,病毒中間產(chǎn)物dsRNA使得IL32啟動子去甲基化增加轉(zhuǎn)錄因子CREB對IL32的結(jié)合上調(diào)其表達,證明dsRNA具有抑制DNMT1活性作用。通過抑制IL32的6個亞型均在病毒感染的細胞中阻礙病毒復(fù)制,特別是IL32-γ具有較強的抗病毒活性。Wu等使用深度測序技術(shù)對Poly I:C刺激和PRRSV感染的豬肺泡巨噬細胞(PAMs)進行miRNA轉(zhuǎn)錄組分析,以比較先天免疫激活和失活狀態(tài)下的不同miRNA譜。測序后,在PAMs中觀察到267個已知成熟miRNAs和64個新miRNAs,與模擬對照細胞相比,共197個miRNAs在Poly I:C刺激的PAMs中表達顯著不同。Tran-Thi等研究了豬衛(wèi)星細胞(PSCs)在骨骼肌的構(gòu)建、發(fā)育和自我更新中起著重要的作用。Poly I:C在刺激PSCs后0、12、24和48 h可模擬病毒感染的增殖和分化。
早在1980年,Jones和Taylor就已發(fā)現(xiàn)5-氮雜胞苷(5-azacytidine)可以抑制活體細胞DNA甲基化,使DNA甲基化對自然狀態(tài)的基因組DNA影響的研究成為可能。前期研究通過引入Aza-CdR研究病毒類似物dsRNA轉(zhuǎn)染豬腎細胞后對全基因組表達的影響,結(jié)果揭示,Aza-CdR可逆轉(zhuǎn)基因表達,這與基因的甲基化狀態(tài)密切相關(guān)。Venolia等早在1982年用5-azacytidine處理過細胞DNA轉(zhuǎn)染其他細胞時,失活X染色體相關(guān)的次黃嘌呤酸核糖基轉(zhuǎn)移酶基因得到表達,而對照的沒有用5-azacytidine處理過的細胞 DNA 不能使該基因表達。另外,Aza-CdR對細胞的多樣效應(yīng)(主要包括DNA去甲基化及誘導(dǎo)細胞凋亡效應(yīng))具有細胞依賴以及時間劑量處理性特點,對細胞具有一定的毒性作用。因此,在使用時要慎重考慮其特征。
前期研究利用Agilent豬4 × 44 K全基因組表達譜芯片分析了Poly I:C和Aza-CdR單獨處理組(P. C和A. C)的差異表達基因的變化,在兩個比較組中均篩選出與免疫反應(yīng)、細胞發(fā)育分化及細胞凋亡相關(guān)的信號通路,這些通路中基因的表達差異可能與DNA甲基化的調(diào)控密切相關(guān)。與此同時,研究利用豬385 K MeDIP-chip芯片技術(shù)篩選得到DGAT、FST、BNIP3L、DPF3等與細胞發(fā)育分化、病毒復(fù)制、炎癥反應(yīng)相關(guān)的重要甲基化調(diào)控基因。這些結(jié)果揭示,Poly I:C和Aza-CdR對PK15細胞的去甲基化作用可調(diào)控免疫相關(guān)的信號通路。結(jié)合甲基化芯片與表達譜芯片研究結(jié)果發(fā)現(xiàn),Aza-CdR具有負向調(diào)控病毒繁殖的功能,并且可減弱PolyI:C對細胞的反應(yīng)。最近的研究證實,Lnc-Dpf3的新型長鏈非編碼RNA可通過抑制樹突狀細胞體內(nèi)遷移進而抑制炎癥性疾病的發(fā)生發(fā)展。BNIP3L的過表達可挽救線粒體自噬缺陷并對腦缺血損傷具有保護作用。本研究也篩選到這兩個關(guān)鍵基因,抗病毒相關(guān)基因3L 和炎癥反應(yīng)相關(guān)基因3,以期作為候選甲基化差異基因用于今后的驗證試驗,這些系統(tǒng)的分析將有利于理解病毒感染豬宿主細胞的免疫反應(yīng)分子機理,為豬和其傳染性病毒的互作提供遺傳基礎(chǔ)。
4 結(jié) 論
目前,使用Aza-CdR轉(zhuǎn)染豬腎細胞的全基因組甲基化圖譜仍未研究,本試驗首次比較研究了Aza-CdR和dsRNA對豬腎細胞轉(zhuǎn)染后的甲基化效應(yīng)。試驗初步揭示,Poly I:C與Aza-CdR的作用相似具有去甲基化作用,這與最近的一些報道相一致。Aza-CdR和dsRNA并不是對豬所有基因具有去甲基化作用,主要針對特有基因的特有啟動子,譬如在與病毒復(fù)制有關(guān)基因3L 和炎癥反應(yīng)相關(guān)基因3的啟動子區(qū)域中,這些特有啟動子的CpG島對Aza-CdR和dsRNA具有特別的敏感性。

