膝骨關節炎患者軟骨組織與血漿中miRNA表達變化及意義
荊琳,單鵬程,張洪美,王秀均
(中國中醫科學院望京醫院,北京100102)
?
膝骨關節炎患者軟骨組織與血漿中miRNA表達變化及意義
荊琳,單鵬程,張洪美,王秀均
(中國中醫科學院望京醫院,北京100102)
目的 觀察膝骨關節炎患者軟骨組織及血漿中miRNA的表達變化,并探討其臨床意義。方法 取2例因外傷導致膝關節軟骨剝脫無法回植的正常關節股骨髁非負重區軟骨組織、2例行人工關節置換術的原發膝骨關節炎股骨髁非負重區軟骨組織以及15例正常志愿者及15例骨關節炎患者的血漿標本。用RT-PCR技術及HiSeq高通量測序技術檢測軟骨組織miRNA表達,根據fold change>1且P<0.01,篩選骨關節炎致病基因;采用RT-PCR技術檢測血漿中miRNA表達。結果 軟骨組織中檢測的miRNA長度主要分布在22 nt,共篩選出5種基因:hsa-miR-378c、hsa-miR-1268a、hsa-miR-483-5p、hsa-miR-375、hsa-miR-1275,篩選出的miRNA在正常軟骨中低表達,在骨關節炎軟骨組織中高表達。篩選出的miRNA在原發膝骨關節炎患者及健康志愿者血漿中表達差異無統計學意義(P均>0.05)。結論 hsa-miR-378c、hsa-miR-1268a、hsa-miR-483-5p、hsa-miR-375和hsa-miR-1275在骨關節炎軟骨組織中高表達,在血漿中表達無規律;以上5種miRNA可能為膝骨關節炎發病的潛在致病基因。
膝骨關節炎;微小RNA;軟骨
microRNA(miRNA)是一類內源性的非編碼RNA,參與細胞生長、發育、基因轉錄和翻譯等諸多生命活動的調控過程,在細胞增殖、分化和凋亡及機體發育、疾病發生過程中發揮著重要的調控作用。據估計,人類至少有1/3的基因受miRNA調控[1]。有研究表明,miRNA在組織細胞中具有特征性的表達譜,并且身體發育過程和疾病發生會導致其表達譜發生明顯變化[2,3]。然而,國內外有關miRNA與骨關節炎關系的報道較少。2015年1月~2016年3月,本研究通過比較正常膝關節與原發膝骨關節炎關節軟骨組織中miRNA的表達差異,以探索miRNA在膝骨關節炎病理過程中的作用。
1 資料與方法
1.1 臨床資料 選行人工關節置換術的原發膝骨關節炎男性患者2例,年齡分別為45、56歲,取股骨髁非負重區軟骨組織作標本(OA1、OA2);選本院因外傷導致膝關節軟骨剝脫、無法回植的男性志愿者2例,年齡分別為42、55歲,取股骨髁非負重區軟骨組織作標本(CK1、CK2)。選擇原發膝骨關節炎患者15例(觀察組),男6例、女9例,年齡36~62(42±4.7)歲;體檢健康志愿者15例(健康對照組),男7例、女8例,年齡24~60(40±6.2)歲;均早晨空腹抽取外周靜脈血5 mL,離心,分離血漿,于-70 ℃下保存待檢;二者性別、年齡比較差異無統計學意義,具有可比性。
1.2 軟骨組織中miRNA表達檢測 將關節軟骨組織及血漿標本送于深圳華大基因科技服務有限公司進行基因測序。取軟骨組織100 mg置1.5 mL離心管中,加TRIzol 1 mL,充分勻漿,室溫靜置5 min,加入氯仿0.2 mL振蕩、離心,取上清液加入異丙醇0.5 mL離心,棄上清液,加入75%乙醇1 mL沉淀、晾干,獲取純化RNA。采用RT-PCR技術、HiSeq高通量測序技術的miRNA數字化分析,邊合成邊測序,構建樣品之間的小分子RNA差異表達譜。將原始數據進行去除接頭、去污染序列及去低質量序列處理,得到可信的目標分析序列,再對其進行RNA序列長度分布統計和樣本間公共序列統計。Ct值為待檢基因實時熒光強度明顯大于背景循環數,采用2-ΔΔCt法計算目的基因相對表達量。建立數據庫,分別使用Scatter plot圖、fold change及t檢驗比較兩標本中共同基因的差異表達,fold change=log2(OA/CK)(OA、CK為兩組基因相對表達量)。篩選條件:fold change>1且P<0.01,認為是差異基因。
1.3 血漿miRNA表達檢測 取血漿標本按照TRIzol試劑說明提取總RNA,根據篩選出的目標基因,用Pola(A)加尾法及實時RT-PCR技術進行檢測。使用One Step PrimeScript miRNA cDNA Synthesis Kit(TaKaRa)進行Poly(A)多聚尾和合成cDNA,用SYBR Premix Ex TaqTM Ⅱ(TaKaRa) 進行加樣,每個樣本均重復檢測3次,取平均值。采用2-ΔΔCt法計算目的基因相對表達量。

2 結果
2.1 膝骨關節炎及正常軟骨組織中miRNA表達比較 CK1、CK2、OA1、OA2中測得的miRNA長度主要集中于22 nt,其次為23 nt。長度為22 nt的miRNA占31.4%~51.73%,而22 nt加23 nt的miRNA占58.17%~75.88%。在fold change>1且P<0.01的條件下,共篩選出5種miRNA:hsa-miR-378c、hsa-miR-1268a、hsa-miR-483-5p、hsa-miR-375、hsa-miR-1275,以上均在OA1、OA2中低表達而在CK1、CK2中高表達。見表1、2。

表1 CK1、CK2及OA1、OA2中miRNA的相對表達量

表2 膝骨關節炎及正常軟骨組織和miRNA的fold change值
2.2 膝骨關節炎患者及健康志愿者血漿中miRNA表達比較 膝骨關節炎患者及健康志愿者血漿中hsa-miR-1268a、hsa-miR-375、hsa-miR-1275相比表達量比較差異無統計學意義(P均>0.05);兩組血漿中均無hsa-miR-483-5p、hsa-miR-378c表達。
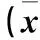
表3 膝骨關節炎患者及健康志愿者血漿miRNA相對表達量比較±s)
3 討論
miRNA能通過與靶mRNA特異性的結合而導致靶mRNA降解或抑制其翻譯,從而對基因進行轉錄后調控。組織細胞內的microRNA可通過某種方式分泌至外周血中,成為循環microRNA,后者作為一種全新的疾病診斷標記物已經得到了重視。目前,miRNA的生物學作用在許多疾病發生發展的過程中被逐漸認識,然而miRNA在骨關節炎發病機制中的作用國內外研究非常有限。所以我們針對原發膝骨關節炎患者及正常志愿者的膝關節軟骨及血漿進行研究,篩選出具有差異表達的miRNA,以期對其進一步深入研究[4~6]。
通常所說的miRNA是指長度18~24 nt的單鏈非編碼小分子RNA,其通過結合目標信使RNA(mRNA)的3′端非翻譯區降解目標基因,或抑制目標基因翻譯來調節基因表達[7~9]。miRNA長度分布的峰值能夠幫助判斷小RNA的種類,多數情況下植物樣本和動物樣本序列長度分布上有著明顯的差異,其表現是植物樣本長度分布的峰值一般出現在21 nt或24 nt,而動物樣本峰值則出現在22 nt。根據這些條件對于樣品情況和測序情況可以做初步判斷。我們檢測的4例人軟骨標本中miRNA長度主要集中在22 nt,其次為23 nt。這與理論上動物標本miRNA峰值一致。
本研究由Scatter plot圖不難發現,每組miRNA表達差異比較中,都有miRNA在骨關節炎軟骨組織中高表達,而在正常軟骨組織中低表達。而這些存在差異表達的miRNA可能在骨關節炎發病機制中發揮重要作用,有待進一步深入研究。Iliopoulos等[10]通過比較正常人與骨關節炎患者關節軟骨組織中已知的365種miRNA的表達情況,從中鑒別出的16種miRNA存在差異,通過以上16種miRNA能夠區別正常軟骨和骨關節炎軟骨。在miRNA基因庫中,目前人類已知的miRNA就有800余種。本研究中,針對所有檢測的已知miRNA,比較其在正常軟骨及骨關節炎軟骨組織中的表達,在fold change>1且P<0.01的條件下,篩選出hsa-miR-378c、hsa-miR-1268a、hsa-miR-483-5p、hsa-miR-375及hsa-miR-1275這5種miRNA,均在骨關節炎軟骨組織中高表達,而在正常軟骨組織中低表達。推斷這5種miRNA可能在骨關節炎的發病機制中發揮重要作用。
Diaz-Prado等[11]曾比較4例正常志愿者關節軟骨及6例骨關節炎患者關節軟骨中已知的729種miRNA,在fold change值>1.5及P<0.01的條件下,只有hsa-miR-483-5p在骨關節炎軟骨中表達明顯升高。而在Iliopoulos等[10]鑒別出的在正常軟骨及骨關節炎軟骨組織中表達具有顯著差異的16種miRNA中,hsa-miR-483-5p在骨關節炎軟骨中的表達,不論是通過miRNA的微陣列分析還是定量PCR分析都顯著升高。此外,Zuntini等[12]也通過實驗證明,hsa-miR-483-5p在骨關節炎軟骨中表達明顯升高。本研究中,hsa-miR-483-5p在骨關節炎軟骨中的表達明顯升高,其fold change值均>1,這說明hsa-miR-483-5p的高表達可能與骨關節炎的發病密切相關。除hsa-miR-483-5p外,hsa-miR-378c、hsa-miR-375及hsa-miR-1275的fold change值分別為1.52、2.22及4.46,這3種miRNA的表達在數據分析中比hsa-miR-483-5p更理想,所以以上幾種miRNA可能為膝骨關節炎發病的潛在基因。目前,已經有研究證明,miR-27a和miR-27b在骨關節炎患者軟骨中能夠調節基質金屬蛋白酶13的表達,從而在骨關節炎的發病過程中起調節作用[13,14]。此外還有研究報道,miR-34a可以抑制骨關節炎大鼠軟骨細胞凋亡[15]。因此,如果能夠找出調控骨關節炎發病的miRNA,可為骨關節炎的早期診斷及靶向治療提供強有力的理論依據。本研究已經鑒別出的5種miRNA在骨關節炎軟骨中高表達,以上均可能是骨關節炎發病的潛在致病基因。因此,我們將繼續針對這5種miRNA在骨關節炎發病機制中的作用進行深入研究。
本研究所篩選的目標基因在血漿中部分無表達,部分表達組間比較差異無統計學意義。推論可能是因為5種miRNA在關節軟骨組織及血漿中出現的機制不同,在血漿中出現的機制較為復雜,有待進一步研究及積累更多臨床數據。
由于正常軟骨標本獲取困難,miRNA篩選、測序復雜,且價格昂貴,本研究只在小樣本中對骨關節炎潛在的致病基因做初步篩選。但是以上miRNA在血漿中表達不穩定,無規律可循。我們計劃下一步擴大軟骨和血漿樣本量,再次進行循環驗證,并探索這些潛在致病基因在骨關節炎發病過程中的調控機制,以期通過干預發現防治骨關節炎的新方法。
[1] Lu J, Getz G, Miska EA, et al. MicroRNA expression profiles classify human cancers[J]. Nature, 2005,435(7043):834-838.
[2] Marcucci G, Radmacher MD, Maharry K, et al. MicroRNA expression in cytogenetically normal acute myeloid leukemia[J]. N Engl J Med, 2008,358(18):1919-1928.
[3] Pfeffer S, Zavolan M, Grasser FA, et al. Identification of virus-encoded microRNAs[J]. Science, 2004,304(5671):734-736.
[4] Denli AM, Tops BB, Plasterk RH, et al. Processing of primary microRNAs by the microprocessor complex[J]. Nature, 2004,432(7014):231-235.
[5] Gregory RI, Yan KP, Amuthan G,et al. The microprocessor complex mediates the genesis of microRNAs[J]. Nature, 2004,432(7014):235-240.
[6] Lee Y, Ahn C, Han J, et al. The nuclear RNase Ⅲ Drosha initiates microRNA processing[J]. Nature, 2003,425(6956):415-419.
[7] Chendrimada TP, Gregory RI, Kumaraswamy E, et al. TRBP recruits the Dicer complex to Ago2 for microRNA processing and gene silencing[J]. Nature, 2005,436(7051):740-744.
[8] Sun BK, Tsao H. Small RNAs in development and disease[J]. J Am Acad Dermatol, 2008,59(5):725-737.
[9] Farh KK, Grimson A, Jan C, et al. The widespread impact of mammalian MicroRNAs on mRNA repression and evolution[J]. Science, 2005,310(5755):1817-1821.
[10] Iliopoulos D, Malizos KN, Oikonomou P, et al. Integrative microRNA and proteomic approaches identify novel osteoarthritis genes and their collaborative metabolic and inflammatory networks[J]. PLoS One, 2008,3(11):e3740.
[11] Diaz-Prado S, Cicione C, Muinos-Lopez E, et al. Characterization of microRNA expression profiles in normal and osteoarthritic human chondrocytes[J]. BMC Musculoskeletal Disorders, 2012(13):144.
[12] Zuntini M, Salvatore M, Pedrini E, et al. MicroRNA profiling of multiple osteochondromas: identification of disease-specific and normal cartilage signatures[J]. Clin Genet, 2010,78(6):507-516.
[13] Tardif G, Hum D, Pelletier JP, et al. Regulation of the IGFBP-5 and MMP-13 genes by the microRNAs miR-140 and miR-27a in human osteoarthritic chondrocytes[J]. BMC Musculoskelet Disord, 2009(10):148.
[14] Akhtar N, Rasheed Z, Ramamurthy S, et al. MicroRNA-27b regulates the expression of matrix metalloproteinase 13 in human osteoarthritis chondrocytes[J]. Arthritis Rheum, 2010,62(5):1361-1371.
[15] Abouheif MM, Nakasa T, Shibuya H, et al. Silencing microRNA-34a inhibits chondrocyte apoptosis in a rat osteoarthritis model in vitro[J]. Rheumatology(Oxford), 2010,49(11):2054-2060.
北京市自然科學基金資助項目(7102166)。
10.3969/j.issn.1002-266X.2016.37.020
R684.3
B
1002-266X(2016)37-0061-03
2016-07-21)

