農桿菌介導法構建里氏木霉ura3基因突變體的研究
閆作梅,張 旭,李 杰
(東北農業大學生命科學學院,哈爾濱 150030)
里氏木霉作為工業菌株,常用來生產多種重要的酶類,包括纖維素酶、半纖維素酶、蛋白酶、淀粉酶等,具有巨大的商業價值[1-2]。目前,里氏木霉的基因組測序已經完成,對于其基因的功能有待研究和開發[3]。同時,里氏木霉是理想的表達真核基因的系統,具有很好的合成和分泌蛋白的能力,在理想的培養條件下發酵分泌纖維素酶可達到40~100 g·L-1發酵液[4-5],被稱為重組蛋白工廠。Harkki等報道,通過基因定位置換整合使編碼里氏木霉CBHI的基因失活,結果EGI的產量提高,不再產生CBHI;1993年,Karhunen等將編碼里氏木酶EGI的基因置于強啟動子CBHI的控制下,用此表達盒替換染色體的CBHI位點之后,EGI產量是通常強纖維素分解菌所產CBHI量的2倍或者與其一樣多;此外,還有一些關于里氏木酶的一個或幾個纖維素酶基因經基因定位置換整合而失活的報道[6-7]。
在里氏木霉轉化研究中,常以營養缺陷型互補基因作為篩選標記,這種篩選標記易于篩選獲得轉化子,被廣泛應用到絲狀真菌的轉化系統中[8]。其中乳清酸核苷-5-磷酸脫羧酶(ura3)基因缺陷株及其相應的轉化系統己被證明是一種行之有效的轉化系統。由于5-氟乳清酸(5-FOA)對原養型菌株有毒性作用,而ura3缺陷菌株則對其產生抗性,所以提供了一種可以正反雙向篩選的篩選系統。
本研究嘗試利用農桿菌介導法通過同源重組獲得里氏木霉ura3基因缺失突變體,從而為里氏木霉轉化系統研究提供材料。
1 材料與方法
1.1 材料
1.1.1 菌株和培養條件
大腸桿菌培養用LB培養基,37℃培養;里氏木霉菌種活化用土豆培養基,培養里氏木霉菌絲體用PDA培養基或MM培養基,28℃培養。
1.1.2 菌株和質粒
菌株:大腸桿菌DH5α、農桿菌GV3101、里氏木霉QM9414。
質粒:pEMT-5由本實驗室保存。1.1.3 分子試劑
膠回收試劑盒Gel Extract Kit(購自百泰克公司)。限制性核酸內切酶、T4DNA連接酶、rTaq酶、LA-Taq酶 、 dNTP、 PyrobestTMDNA Polymerase、pMD18-T vector等(購自大連寶(TaKaRa)生物工程有限公司)。
1.1.4 PCR引物設計
ura3引物序列如下:
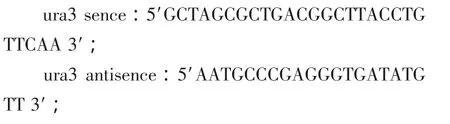
pyrA引物序列如下:

ura3和pyrA基因農桿菌PCR檢測引物序列:


1.2 方法
1.2.1 ura3和pyrA基因的克隆
以里氏木霉QM9414基因組DNA為模板,PCR擴增ura3基因片段,預期擴增片段大小為1 469 bp。PCR反應條件:94℃預變性10 min,94℃變性1 min,55℃退火30 s,72℃延伸1 min,72℃延伸10 min,4℃保存,30個循環。
以黑曲霉UV48基因組DNA為模板,PCR擴增pyrA基因片段,大小為3 370 bp。PCR反應條件:94℃預變性10 min,94℃變性1 min,55℃退火30 s,72℃延伸4 min,72℃延伸10 min,4℃保存,30個循環。
1.2.2 ura3缺失基因載體和pyrA基因載體構建
質粒提取、酶切、轉化參見文獻[9],連接參見文獻[10],DNA片段回收參見膠回收試劑盒說明。
1.2.3 重組質粒轉化根癌農桿菌
取0.5~1 μL重組質粒DNA,加入100 μL根癌農桿菌感受態細胞中,輕輕混勻;冰浴5 min,液氮冷凍8 min,迅速至37℃溫浴融化;加入800 μL YEB液體培養基,28℃,輕輕搖動4~5 h;
5 000 r·min-1離心5 min,倒掉大部分上清,將剩余約50 μL菌液用移液器轉至含有Km 100 mg·L-1、Gent 50 mg·L-1、Rif 50 mg·L-1的 YEB 固體培養基上涂勻。28℃暗培養48~96 h。
1.2.4 根癌農桿菌和里氏木霉共培養
用5 mL滅菌蒸餾水從培養5~7 d的PDA平板上洗下里氏木霉菌的分生孢子,用MM液體培養基稀釋至相應的孢子濃度。接種含雙元載體系統的pEMT-△ura3根瘤農桿菌單菌落于3 mL液體YEB培養基(含 50 mg·L-1Rif、50 mg·L-1Gent、100 mg·L-1Kan)中,200 r·min-1,28 ℃培養 2~3 d,離心收集菌體,用一定體積的IM液體培養基重懸菌體。將菌液濃度調到660 nm處的吸收值為0.25,繼續培養6 h,OD660達到0.6~0.8。取處理好的里氏木霉孢子懸液和根瘤農桿菌菌液各100 μL,混勻,涂布MM 培養基(含 200 μmol·L-1乙酰丁香酮),24 ℃共培養48 h。
1.2.5 轉化子的篩選
將在24℃孵育48 h的培養物移至含有尿嘧啶核苷(1.87 mg·mL-1)、五氟乳氰酸(5 mg·mL-1)和阿莫西林(100 mg·mL-1)的誘導培養基上28℃培養8~10 d至抗性菌落出現。
1.2.6 轉化子的鑒定
提取轉化子的基因組DNA,PCR鑒定轉化子。如果ura3基因被敲除,擴增出片段應為445 bp;如果黑曲霉的pyrA插入到里氏木霉的基因組中,擴增出650 bp的片段。
2 結果與分析
2.1 5-FOA篩選濃度的確定
將里氏木霉孢子接種于含不同濃度5-FOA的PDA固體培養基上,28℃培養7 d后,里氏木霉生長情況見圖1。5-FOA濃度在5 mg·mL-1時即可有效抑制里氏木霉的生長,因此確定5-FOA篩選濃度為 5 mg·mL-1。
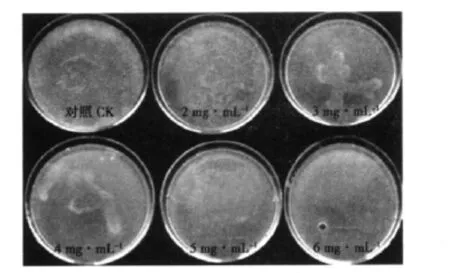
圖1 5-FOA對里氏木霉生長的抑制作用Fig.1 Inhibition effect of 5-FOA on the germination and growth of Trichoderma reesei strains
2.2 ura3和pyrA基因的克隆
ura3基因PCR擴增得到一條1 469 bp的目的條帶(見圖2);pyrA基因PCR擴增得到一條3 300 bp的目的條帶(見圖3)。將上述擴增條帶克隆至T載體,篩選陽性克隆送交測序。測序結果表明,已獲得完整的ura3基因和pyrA基因片段(序列略)。
2.3 ura3缺陷型載體的構建
ura3缺陷型載體pEMT-△ura3的構建過程見圖4。HindⅢ、XbaⅠ雙酶切質粒pMD-ura3,回收約2 600和1 470 bp的片段。再將1 470 bp的小片段用SmaⅠ酶切,回收約650和700 bp片段。將上述3個片段連接,獲得質粒pMD-△ura3。經酶切和測序證明,該載體上的ura3基因缺失110 bp,結果正確。再用Eco RⅠ、NheⅠ雙酶切質粒pEMT-5,回收約8 000 bp的載體片段,與經同樣酶切質粒pMD-△ura3回收的1 350 bp的目的片段連接,獲得載體pEMT-△ura3。通過凍融法將pEMT-△ura3轉入農桿菌GV3101,轉化菌落的PCR鑒定結果見圖5。
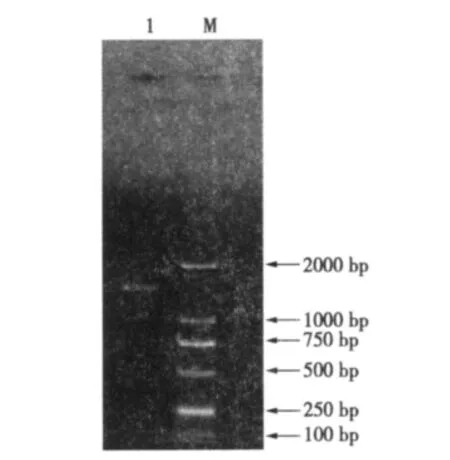
圖2 ura3基因PCR擴增結果Fig.2 Results of PCR amplification of ura3
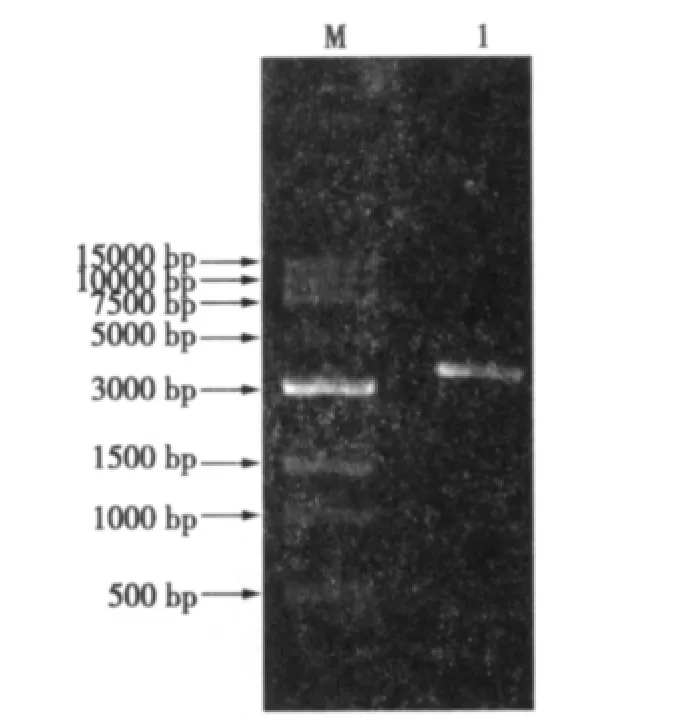
圖3 pyrA基因PCR擴增結果Fig.3 Results of PCR amplification of pyrA
2.4 黑曲霉pyrA基因載體的構建
黑曲霉pyrA基因載體pEMT-pyrA的構建過程見圖6,將pEMT-ku70載體用XbaⅠ、Sna BⅠ雙酶切,回收11 524 bp的載體片段,然后用XbaⅠ,NcoⅠ雙酶切的黑曲霉pyrA基因片段與載體連接,構建重組質粒pEMT-pyrA。
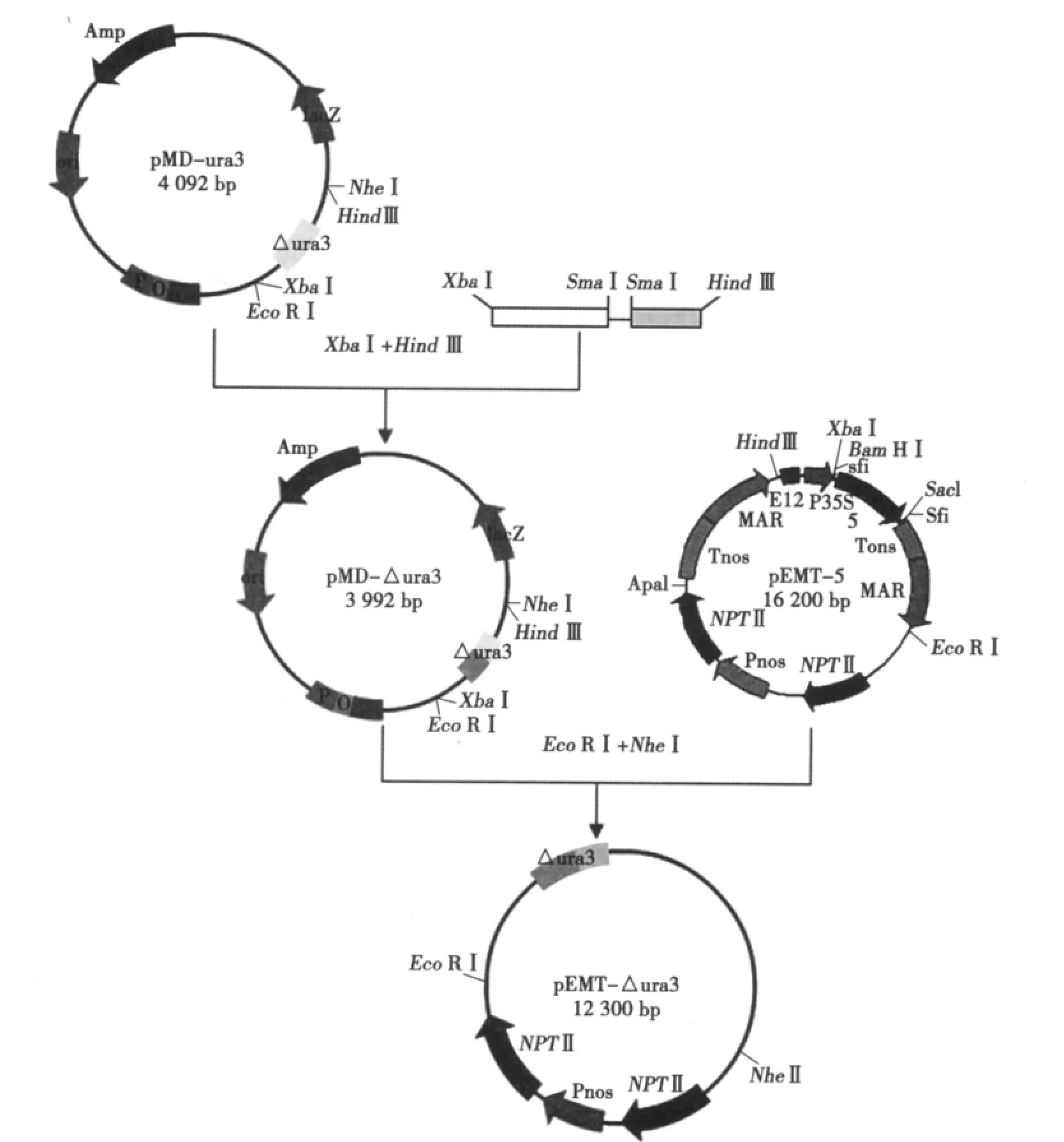
圖4 pEMT-△ura3載體的構建Fig.4 Construction of vector pEMT-△ura3
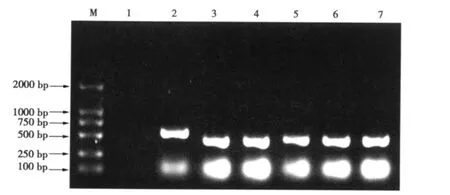
圖5 pEMT-△ura3轉入農桿菌的鑒定結果Fig.5 Characterization of pEMT-△ura3 transfer in Agrobacterium
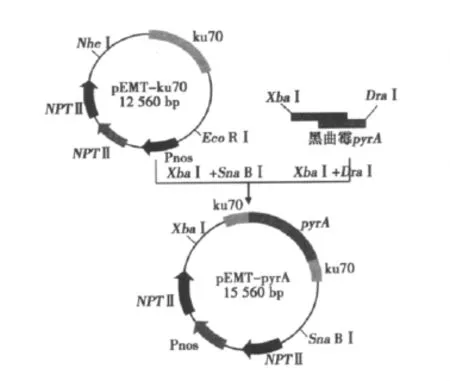
圖6 pEMT-pyrA載體的構建Fig.6 Construction of vector pEMT-pyrA
通過凍融法將pEMT-△ura3轉入農桿菌GV3101,轉化菌落的PCR鑒定結果見圖7。
2.5 ura3基因缺失突變株的分子鑒定
通過農桿菌介導法將pEMT-△ura3轉化里氏木霉QM9414,將篩選平板上獲得的菌落進行PCR驗證(見圖 8)。
結果顯示隨機挑選的20個轉化子中只有1個可以擴增得到ura3的缺失基因(445 bp,泳道13),即發生的是同源重組,將其命名為QM9414-△ura3突變株。還有5株(泳道3、7、9、10、12)同時擴增出445和555 bp特異條帶,即發生的是非同源重組,其同源重組率為16.6%(1/6)。
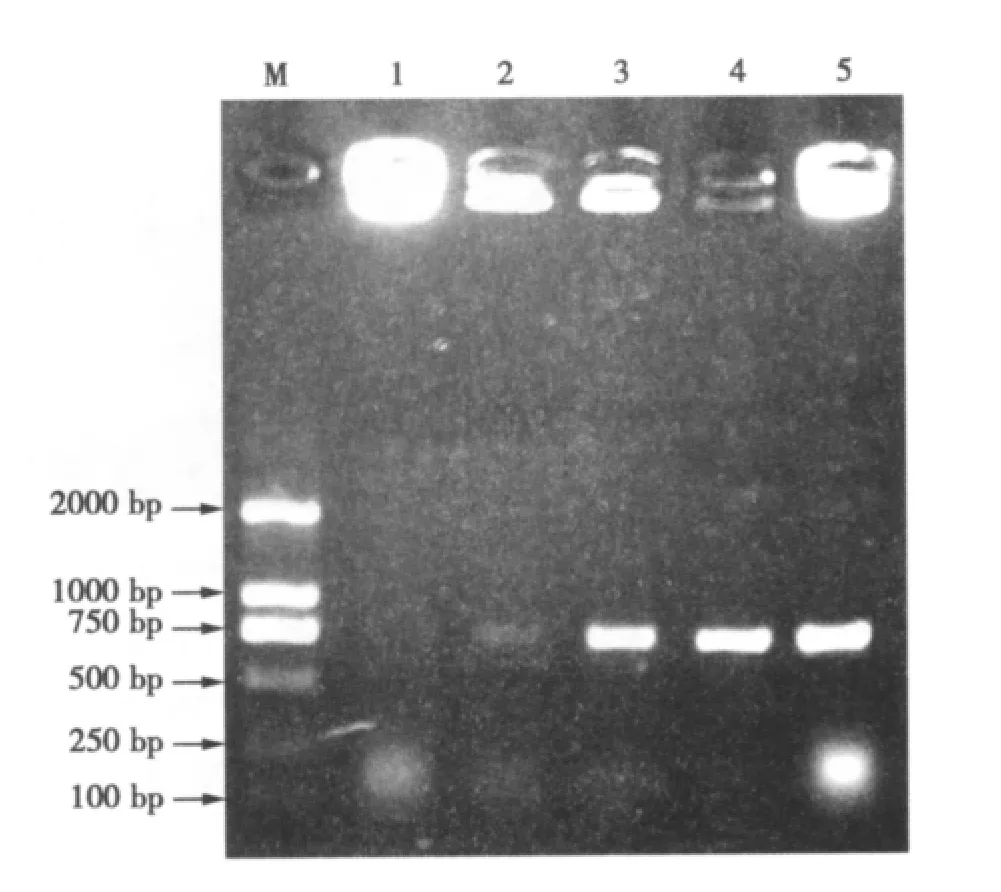
圖7 pEMT-pyrA轉入農桿菌的鑒定結果Fig.7 Characterization of pEMT-pyrA transfer in Agrobacterium
2.6 里氏木霉ura3基因缺失突變體的回復
采用農桿菌介導法將pyrA基因轉化里氏木霉QM9414-△ura3突變株,在不含有尿嘧啶核苷酸的基本培養基平板上篩選回復突變株。然后應用PCR篩選整合有pyrA基因的轉化子,在隨機挑取的30個轉化子中,有3個擴增出目的基因(見圖9)。
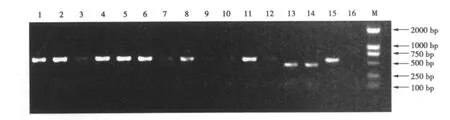
圖8 △ura3基因缺失突變株的PCR檢測Fig.8 PCR detection of△ura3 gene disruptant
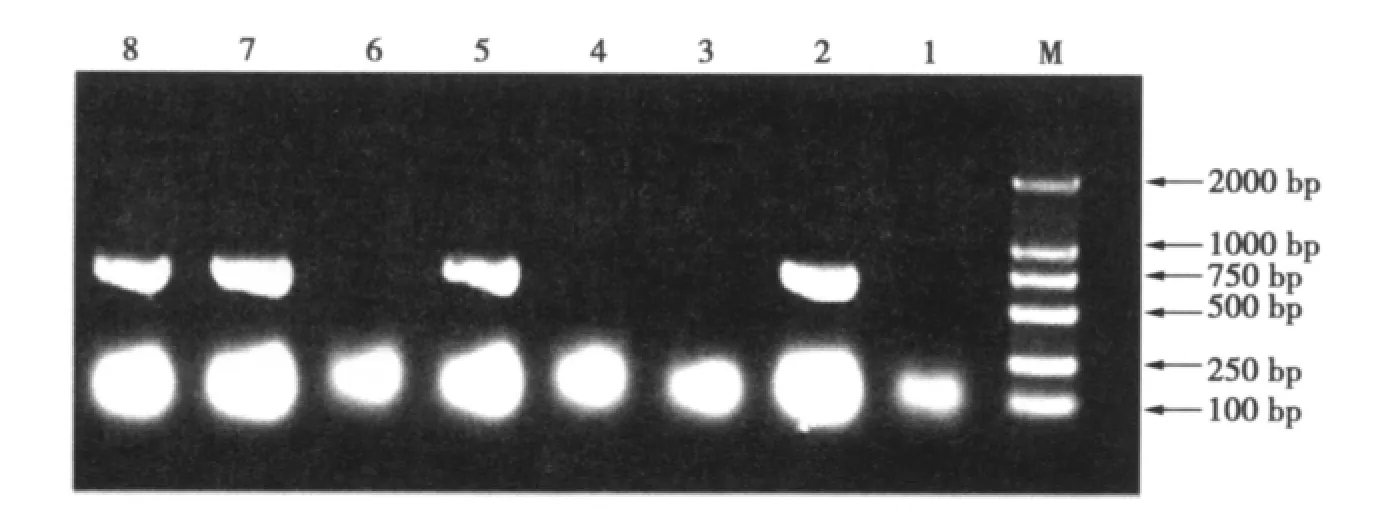
圖9 pyrA轉化子的PCR檢測Fig.9 PCR detection of pyrA gene
3 討論
目前,常用紫外誘變的方法對絲狀真菌的菌株進行改造,但是不易控制誘變的位點。而同源重組利用DNA轉化技術,通過載體DNA序列與靶細胞內染色體上同源DNA序列間的重組從而定向改變細胞遺傳特性,具有明顯的優勢[8]。
由于絲狀真菌遺傳轉化系統和選擇標記被成功的應用于許多的真菌轉化中,這就為人們在基因水平上研究真菌的遺傳背景和菌株的改造提供了有利的條件,其中以根癌農桿菌介導的遺傳轉化體系備受關注[11-12]。根癌農桿菌介導的絲狀真菌轉化與傳統的轉化方法相比具有以下優點[13-16]:①轉化效率高;②可導入大片段的DNA;③導入基因拷貝數低,表達效果好,穩定遺傳;④農桿菌轉化方法使用的技術、儀器簡單;⑤同源重組效率高。Lima等利用傳統的PEG介導的方法對T.atroviride進行轉化,同源重組的頻率很低,而利用農桿菌介導的方法獲得了60%的同源重組的轉化子,使得其敲除基因的效率可達14%~75%,成功實現了對tmk1和tga3基因非編碼區的基因破壞[17]。
本試驗全面考慮了高效基因修飾和置換技術的要點,有機整合了一些相關技術,通過農桿菌介導法提高里氏木霉的轉化效率,通過以ura3作為篩選標記降低了轉化子的假陽性,提高導入DNA片段的同源重組效率。所建立的高效基因修飾和置換系統符合食品級表達系統的要求,由此所研制的工程菌和重組蛋白具有高度的安全性,可用于食品加工,應用領域更為廣闊。
本研究利用農桿菌介導方法將帶有△ura3基因的載體導入到里氏木霉宿主細胞內,使載體上的△ura3基因與宿主細胞的染色體DNA發生同源重組,從而獲得以ura3作為篩選標記的營養缺陷菌株,同時證實農桿菌介導的方法能高效的敲除里氏木霉基因,為研究里氏木霉的功能基因提供了高效的、快速的方法。
4 結論
本研究從里氏木霉QM9414中克隆了ura3基因,構建了ura3基因敲除載體pEMT-△ura3。從黑曲霉中克隆了pyrA基因,構建了pyrA基因載體pEMT-pyrA。采用農桿菌介導法將ura3缺失基因轉化里氏木霉QM9414,篩選獲得QM9414-△ura3突變株,其同源重組率為16.6%。將pyrA基因轉化QM9414-△ura3突變株,篩選獲得回復突變株。
[1] Nevalainena K M H,Teoa V S J.Enzyme production in industrial fungi:molecular genetic strategies for integrated strain improvement[J].Applied Mycology and Biotechnology,2003(3):241-259.
[2] Punt P J,van Biezen N,Conesa A,et al.Filamentous fungi as cell factories for heterologous protein production[J].Trends Biotechnol,2002,20(5):19-33.
[3] 李銳,賀亮,鄧旭明.絲狀真菌蛋白質組分析[J].東北農業大學學報,2008,39(5):58-61.
[4] Levin A M,de Vries R P,W sten H A B.Localization of protein secretion in fungal coloies using a novel culturing technique;thering-plate system[J].Journal of Microbiogical Methods,2007,69(2):399-401.
[5] Wang T H,Wu J,Zong Y X.Advances of molecular biology of Trichoderma reesei[J].Mycosystema,2000,19(1):147-152.
[6] Nevalainen K M H,Valentino S J T,Bergquist P L.Heterologous protein expression in filamentous fungi[J].Trends Biotechnol,2005,23(9):468.
[7] Nayak T,Szewczyk E,Oaklay C E,et al.A versatile and efficient gene targeting system for Aspergillus nidulans[J].Genetics,2005(12):30.
[8] Degefu Y,Hanif M.Agrobacterium tumefaciens-mediated transformation of Helminthosporium turcicum,the maize leaf blight fungus[J].Arch Microbiol,2003,108:279-284.
[9] 薩姆布魯克等.分子克隆實驗指南[M].2版.北京:科學出版社,1992.
[10] 李杰,李晶,朱延明,等.DREB1A基因植物表達載體的構建[J].東北農業大學學報,2003,34(2):199-204.
[11] Ninomiya Y,Suzuki K,Ishii C,et al.Highly efficient gene replacements in Neurospora strains deficient for nonhomologous end-joining[J].Proc Natl Acad Sci,2004,101:12248-12253.
[12] Ruiz-Diez B.Strategies for the transformation of filamentous fungi[J].J APPl Microbiol,2002,92:189-195.
[13] Gouka R J,Gerk C,Hooykaas P J,et al.Transformation of Aspergillus awamori by Agrobacterium tumefaciens-mediated homologous recombination[J].Nat Biotechnol,1999,17:598-601.
[14] Fang W,Pei Y,Bidochka M J.Transformation of Metarhizium anisopliae mediated by Agrobacterium tumefaciens[J].Can J Microbiol,2006,52:623-626.
[15] Godio R P,Fouces R,Gudina E J,et al.Agrobacterium tumefaciens-mediatedtransformationoftheantitumorclavaricacidproducing basidiomycete Hypholoma sublateritium[J].Curr Genet,2004,46:287-294.
[16] 王敬國,張衛國,夏林,等.黑曲霉原生質體轉化效率影響因子研究[J].東北農業大學學報,2007,38(4):20-25.
[17] Lima I G,Duarte R T,Furlaneto L,et al.Transformation of the entomopathogenic fungus Paecilomyces fumosoroseus with Agrobacterium tumefaciens[J].Lett Appl Microbiol,2006,42:631-636.

