低溫弱光脅迫下茄子幼苗實時熒光定量PCR內參基因的篩選與評價











摘要:為了探索茄子Solanum melongena L.的基因表達模式,利用qRT-PCR方法分析了茄子ACT、EF1α、TUA、TUB、GAPDH、eIF、UBQ、UBI3、PP2A、CYP、RPL28、L25、SAND、TBP、DNAJ、APRT、EXP、CAC、HSP20和CysPro 20個候選內參基因mRNA的表達差異情況,并運用GeNorm、NormFinder和BestKeeper 3種計算方法評價茄子20個候選內參基因在低溫、弱光、低溫弱光葉片樣品中的表達穩定性。結果表明:茄子20個候選內參基因熒光定量引物擴增效率(E)數值在96.8%~117.6%,相關系數(R2)介于0.968 8~0.999 9,擴增效率良好,擴增反應具有高度的專一性,引物能特異性擴增,特異性好,均為單峰,樣品間擴增曲線重復性強,可用于qRT-PCR擴增。茄子20個候選內參基因CT值表達豐度分析,發現茄子20個候選內參基因平均CT值在19.76~31.20。3種計算方法的綜合評價分析結果顯示,TBP基因在所有樣本中為最穩定的內參基因。研究可為茄子低溫、弱光和低溫弱光脅迫下基因特異性表達研究提供了合適的內參基因。
關鍵詞:茄子;內參基因;實時熒光定量PCR;標準化;基因表達
中圖分類號:S641.1文獻標志碼:A文章編號:0253?2301(2024)08?0001?15
DOI:10.13651/j.cnki.fjnykj.2024.08.001
林琿,裘波音,朱海生,等.低溫弱光脅迫下茄子幼苗實時熒光定量PCR內參基因的篩選與評價[J].福建農業科技,2024,55(8):01?15.
優秀學者論壇
溫慶放,1965年生,二級研究員,福建省百千萬人才,福建省農業科學院作物研究所黨支部書記、所長,主要從事蔬菜育種與栽培研究。主持或參與了多項國家科技計劃、福建省重大科技專項、福建省種業產業化工程項目等蔬菜重大科技項目;獲福建省科技進步獎二等獎2項(排名第1)、三等獎1項(排名第1),參與獲得福建省科技進步獎一等獎1項(排名第5)、二等獎3項(排名分列為2、3、6名);主持或參與育成蔬菜品種20多個通過國家品種登記或省認(鑒)定;授權國家發明專利5件;發表論文130多篇,其中SCI收錄8篇;編著出版蔬菜專著7部。現任或兼任國家大宗蔬菜產業技術體系福州綜合試驗站站長、福建省蔬菜遺傳育種重點實驗室主任、福建省蔬菜工程技術研究中心主任、福建省園藝學會副理事長。
Screening and Evaluation of Reference Genes in Eggplant Seedlings by Real-time Fluorescence Quantitative PCR Under Low Temperature and Weak Light Stress
LIN Hui,QIU Bo-yin,ZHU Hai-sheng*,WEN Qing-fang*
(Fujian Key Laboratory of Vegetable Genetics and Breeding/Fujian Engineering Research Center for Vegetables/Crops Research Institute,Fujian Academy of Agricultural Sciences,Fuzhou,Fujian 350013,China)
Abstract:In order to explore the gene expression pattern of Solanum melongena L.,the qRT-PCR method was used to analyze the expression differences of mRNA in the 20 candidate reference genes of eggplant,including ACT,EF1α,TUA,TUB,GAPDH,eIF,UBQ,UBI3,PP2A,CYP,RPL28,L25,SAND,TBP,DNAJ,APRT,EXP,CAC,HSP20 and CysPro.The three calculation methods,including GeNorm,NormFinder and BestKeeper,were used to evaluate the expression stability of 20 candidate reference genes in eggplant samples at low temperature,weak light and low temperature and weak light stress.The results showed that the fluorescence quantitative primer amplification efficiency(E)values of 20 candidate reference genes in eggplant ranged from 96.8%to 117.6%,and the correlation coefficient(R2)was between 0.968 8?0.999 9,indicating that the amplification efficiency was good,and the amplified reaction was highly specific.The primers could be specifically amplified,with good specificity,all of which were unimodal.The amplification curves among the samples had strong repeatability,which could be used for qRT-PCR amplification.By analyzing the expression abundance of CT values of 20 candidate reference genes in eggplant,it was found that the average CT values of 20 candidate reference genes in eggplant ranged from 19.76 to 31.20.The comprehensive evaluation results of the three calculation methods showed that the TBP gene was the most stable reference gene in all the samples.The study could provide a suitable reference gene for the study of gene-specific expression in eggplant under low temperature stress,weak light stress and low temperature and weak light stress.
Key words:Eggplant(Solanum melongena L.);Reference gene;Quantitative real-time PCR;Standardization;Gene expression
茄子作為茄科茄屬的一種重要蔬菜,深受消費者喜愛,在我國各地廣泛種植[1]。它不僅富含多種膳食纖維、礦物質、蛋白質、酚類等營養物質,還含有豐富的維生素P和高水平抗氧化的有效成分[2?3]。茄子是喜溫蔬菜,而福建地處亞熱帶地區,熱量豐富,氣候溫暖,十分適宜茄子生長。近年來,隨著交通運輸和設施大棚的發展,茄子憑借其高經濟價值和簡單的栽培管理,已經成為福建省蔬菜的支柱產業之一[4]。溫度和光照是綠色植物生長發育中重要的環境因素[5]。然而,福建冬季和初春,茄子在生產過程中極易受到低溫和連續陰雨天氣的影響,導致落花、落果、畸形果等現象頻發,造成大面積減產,經濟損失巨大[1]。因此,深入探究茄子對低溫弱光脅迫的響應,制定一套適用于低溫弱光環境的茄子栽培管理技術,并培育耐低溫弱光的茄子品種,對福建茄子產業的發展具有重大意義。目前,盡管學者對茄子低溫弱光脅迫的研究已取得一定進展,但大多停留在生理水平,其在分子層面上的發生機理仍有待進一步闡明。挖掘耐寒關鍵基因,闡明其調控機制,對于茄子的安全高效生產以及耐寒種質創制具有重要意義。因此,準確檢測茄子在低溫弱光脅迫下的基因表達量,成為當下亟待開展的研究。
實時熒光定量PCR(Quantitative Real-Time PCR,qRT-PCR)是分析茄子幼苗低溫、弱光、低溫弱光非生物脅迫下功能基因表達量的一種有效的試驗方法,具有重復性好、靈敏度高、特異性強、對靶基因有高通量等特性[6?9]。為了獲得準確的基因表達數據,在試驗過程中要盡量避免由于引物設計、cDNA轉錄、PCR擴增等因素造成表達水平的偏差[10?13]。使用內參基因對目的基因表達數據進行標準化,是矯正偏差的重要手段[14]。理想的內參基因是在不同試驗處理、不同組織、不同發育階段等條件下都能一致表達的基因。
看家基因在維持細胞基本生命活動過程中保守性高,受到環境的影響較小[15]。因此,看家基因GAPDH[16]、UBQ[17]、TUB[18]、ACT[19]等被廣泛的應用于不同植物在各種試驗條件下的基因表達分析。然而,這些基因作為內參基因的適用性并非絕對,其穩定性在不同植物和試驗條件下有所差異。在單子葉植物短柄厥內參基因篩選中,GAPDH基因被認為不適合作為內參基因[20],但在虎耳草激素處理的試驗中,GAPDH基因卻是表達最為穩定的基因[16];在雙子葉植物中,UBQ10基因在珊瑚菜中表達最為活躍[21],而在大青楊中卻被鑒定為最佳內參基因[17];在管狀花目植物中,TUB基因在芝麻不同發育階段的營養組織中表達穩定性最高[18],但在番茄的TOCMOV致病系統研究中則不建議被作為內參基因使用[22];在茄科植物夜香樹(夜來香)中,ACT基因在花和葉的內參基因的評價中表達最為穩定[19],但在辣椒的非生物脅迫和激素處理試驗中,ACT則為最不穩定的基因之一[23]。此外,一些編碼細胞分化的基因,如PP2A[24]、CYP[25]、EXP[21]等,也常常被作為內參基因使用[26]。在茄屬植物的內參基因評價中,PP2A基因在番茄的氮脅迫、低溫和光脅迫下是最穩定基因[24],而在病毒侵染煙草基因表達變化的研究中,PP2A基因的表達穩定性是最差的基因之一[27]。因此,針對不同植物的不同試驗條件,篩選合適的內參基因尤為重要。
茄子內參基因的篩選已取得了一些初步的進展[28?32]。在茄子花芽、花、成熟葉、嫩葉、莖和根等組織的內參基因的篩選研究中,18S rRNA被鑒定為最適合內參基因[28];在茄子不同組織以及不同植物生長調節劑、生物脅迫、干旱、鹽處理下,TUA基因表達穩定性最好[29];在茄子果實的不同發育階段及不同品種研究中,SAND基因被認為是最佳內參基因[30];在茄子高溫脅迫下,EF1α基因表達穩定性最高[29]。可見,內參基因的表達水平在不同的試驗條件下不是穩定不變的。茄子在低溫、弱光、低溫弱光脅迫下內參基因的研究尚待深入。在特定的試驗條件下,篩選合適的內參基因是試驗順利進行的先決條件。為了在低溫、弱光、低溫弱光處理下研究參與3個脅迫的功能基因及轉錄因子,有必要篩選對各個具體試驗條件下最佳的內參基因來對熒光定量PCR試驗數據進行校正,這將促進茄子在基因表達水平的研究。
本研究選擇了茄子在低溫脅迫、弱光脅迫、低溫弱光脅迫下的樣品,利用qRT-PCR方法分析了ACT、EF1α、TUA、TUB、GAPDH、eIF、UBQ、UBI3、PP2A、CYP、RPL28、L25、SAND、TBP、DNAJ、APRT、EXP、CAC、HSP20和CysPro這20個植物常用的候選基因mRNA的表達差異情況,并運用GeNorm[31]、NormFinder[32]和Best-Keeper[33]3種計算方法對檢測結果中候選內參基因表達穩定性進行評價,旨在篩選適宜低溫弱光脅迫下茄子基因表達分析的內參基因,為茄子相關基因表達研究奠定基礎。
1材料與方法
1.1試驗材料
選用對低溫弱光反應不同的兩個茄子材料:耐低溫弱光品系epl008和低溫弱光敏感品系eplck。
1.2試驗設計
試驗在福建省蔬菜遺傳育種重點實驗室中進行。飽滿的種子用3%次氯酸鈉表面滅菌10 min,用蒸餾水清洗3次,再用蒸餾水浸泡12 h,用濕紗布包起然后放入帶濕濾紙的培養皿中,在28℃黑暗中發芽。3 d后,將萌發的種子播種在裝有育苗基質的干凈穴盤中,轉移到16h光照(28℃)/8 h暗周期、光合光子通量密度為300μmol·m?2·s?1、相對濕度為80%的條件下的氣候室中[30,35]。
本試驗共設置6個處理:(1)采集耐低溫弱光材料epl008在低溫處理下0、3、6、12、24和48 h的葉片。(2)采集耐低溫弱光材料epl008在弱光處理下0、3、6、12、24和48 h的葉片。(3)采集耐低溫弱光材料epl008在低溫弱光處理下0、3、6、12、24和48 h的葉片。(4)采集低溫弱光敏感材料eplck在低溫處理下0、3、6、12、24和48 h的葉片。(5)采集低溫弱光敏感材料eplck在弱光處理下0、3、6、12、24和48 h的葉片。(6)采集低溫弱光敏感材料eplck在低溫弱光處理下0、3、6、12、24和48 h的葉片。收集的茄子樣品用錫箔紙包裹液氮速凍置于超低溫冰箱存放,備用。每個樣品來自3株植物處理作為生物復制處理,試驗設計3個生物學重復。
1.3總RNA提取和cDNA的獲得
RNA提取按照通用植物總RNA提取試劑盒中的試驗手冊進行(中國北京,百泰克)。用1%(w/v)瓊脂糖凝膠電泳檢測所提RNA的完整性,用美國Thermo Fisher Science公司的Nano-Drop?8 000分光光度計測定RNA濃度和純度。A260/A280比值在1.9~2.1和A260/A230大于1.80的RNA樣品才用于cDNA合成。cDNA根據制造商(Takara)的說明,在20μL的反應系統中,使用HiScript II Q RT SuperMix for qPCR(+gDNA wiper)(Takara)合成cDNA。每個樣品所需的RNA用量,根據所提取的RNA濃度確定其比例,以確保每個組織樣品的mRNA含量保持一致。
1.4內參基因篩選和引物設計
茄子20個候選內參基因ACT、EF1-α、TUA、TUB、GAPDH、eIF、UBQ、UBI3、PP2A、CYP、RPL28、L25、SAND、TBP、DNAJ、APRT、EXP、CAC、HSP20.2和CysPro的篩選,參考前人在茄科茄屬蔬菜和其他作物研究成果[28?49](表1)。根據已有的茄子全基因組RNA-seq數據庫(http://eggplant.kazusa.or.jp/index.html),篩選得到20條基因全長序列。根據引物設計原則,利用軟件Premier 5.0設計熒光定量試驗所需引物(表1)。引物檢測反應程序為:94℃預變性3 min;94℃變性30 s,52℃退火30 s,2℃延伸30 s,35個循環;72℃延伸7 min。反應體系20.0μL:包括10×PCR buffer(含Mg2+)2.0μL、Primer F(10μmol·L?1)1.0μL、Primer R(10μmol·L?1)1.0μL、dNTP(10 mmol·L?1)1.0μL、Taq(5 U)0.2μL、cDNA 2.0μL、ddH2O 12.8μL。
1.5內參基因熒光定量PCR擴增
用稀釋10倍的cDNA模板生成標準曲線斜率(slope),用E=(10-1/slope–1)×100%計算每個候選內參基因的擴增效率,以R2預測標準曲線回歸方程的可靠性,進行3次重復。采用SYBR Green染料法,熒光定量PCR試驗按照SYBR Premix Ex Taq TM試劑盒TaKaRa公司操作說明書進行,在7 500 Real Time System儀[ABI(美國)公司]上完成,每個樣品設3次重復。反應體系10μL,包括2×SYBR Premix EXTaqTM 5μL、Primer F(10μmol·L?1)0.4μL、Primer R(10μmol·L?1)0.4μL、ROX Reference Dye(10μmol·L?1)0.2μL、cDNA 2.0μL、ddH2O 2.0μL。PCR擴增反應程序:95℃預變性30 s;95℃變性5 s,60℃退火34 s,40個循環。擴增完成后進行溶解曲線分析,溶解曲線程序:95℃15 s,60℃1 min,95℃15 s。分析溶解曲線確定每對擴增引物的特異性。
1.6數據分析
利用GeNorm、NormFinder和BestKeeper法分析20個候選內參基因穩定性。將qRT-PCR獲得數據導出至Microsoft Excel 2003中,并根據軟件要求對循環閾值(cycle threshold,CT)進行轉換。每個軟件產生候選內參基因穩定性的度量值,可用于對內參基因穩定性排序。最后,用幾何平均值綜合評價3種計算方法,生成最終排序表。
2結果與分析
2.1內參基因引物檢測
茄子20個候選內參基因熒光定量引物擴增效率(E)數值在96.8%~117.6%,相關系數(R2)介于0.968 8~0.999 9(表2),表明引物擴增效率良好,可用于qRT-PCR試驗。利用普通PCR對內參基因特異性進行檢測(圖1),結果表明20個內參基因擴增產物在100~250 bp,條帶單一,大小與目標片段相同,說明擴增反應具有高度的專一性,引物能特異性擴增,未出現引物二聚體,適用于qRT-PCR研究。以茄子葉片cDNA為模板,經qRT-PCR擴增,對候選內參基因的引物溶解曲線分析(圖2),結果表明20個候選內參基因引物特異性好,均為單峰,樣品間擴增曲線重復性強,說明CDNA模板可進行差異性擴增,qRT-PCR結果可信度高。
2.2茄子20個候選內參基因CT值表達豐度分析
茄子20個候選內參基因平均CT值在19.76~31.20(圖3)。根據CT值計算候選內參基因表達水平,候選內參基因在耐低溫弱光品系epl008和低溫弱光敏感品系eplck低溫脅迫、弱光脅迫和低溫弱光脅迫處理條件下基因表達水平不同。內參基因的表達豐度通過CT值來體現,一般來說,CT值越低表達豐度越高。由圖3可知,內參基因CYP的表達豐度(19.76±4.02)較其他內參基因高,其次是TUA基因的表達豐度,為20.46±3.53。20個內參基因中,GAPDH基因在耐低溫弱光品系epl008和低溫弱光敏感品系eplck低溫脅迫、弱光脅迫和低溫弱光脅迫處理條件下的變化范圍最大為31.20±4.07,SAND基因變化范圍最小,范圍為25.61±0.12。
2.3茄子內參基因的篩選
2.3.1 GeNorm軟件分析在GeNorm軟件分析中,通常以平均變異度(M)作為衡量內參基因穩定與否的標準,M越小,穩定性越好;反之,M越大,穩定性越差。學者們在分析中默認的M閾值為1.5。GeNorm軟件的分析結果(表3)表明,20個內參基因不同處理中的表達穩定性不同。以低溫脅迫的茄子epl008葉片為材料時,內參基因的M由低到高依次為PP2A=CysPr<APRT<EXP<CAC<DNAJ<L25<EF-1α<UBQ<GAPDH<ACT<TBP<TUA<CYP<RPL8<HSP20<UBI3<SAND<eIF<TUB;以低溫脅迫的茄子eplck葉片為供試材料時,內參基因的M由低到高依次為RPL8=TBP<UBI3<CAC<UBQ<PP2A<L25<APRT<DNAJ<SAND<GAPDH<TUA<EXP<EF-1α<eIF<CysPr<HSP20<TUB;以弱光脅迫的茄子epl008葉片為材料時,內參基因的M由低到高依次為APRT=CAC<CysPr<UBQ<PP2A<TBP<RPL8<UBI3<DNAJ<TUB<TUA<EXP<L25<HSP20<SAND<CYP<eIF<ACT<GAPAH<EF-1α;以弱光脅迫的茄子eplck葉片為材料時,內參基因的M由低到高依次為RPL8=SAND<TBP<UBI3<CYP<CAC<EXP<TUA<PP2A<TUB<APRT<DNAJ<L25<ACT<eIF<CysPr<EF-1α<GAPAH<UBQ<HSP20。以低溫弱光雙重脅迫的茄子epl008葉片為材料時,內參基因的M由低到高依次為DNAJ=APRT<CysPr<PP2A<L25<ACT<UBQ<EF-1α<EXP<CAC<HSP20<TUB<CYP<TUA<RPL8<TBP<eIF<UBI3<GAPDH<SAND;以低溫弱光雙重脅迫的茄子eplck葉片為材料時,內參基因的M由低到高依次為EF-1α=DNAJ<HSP20<UBQ<L25<APRT<UBI3<eIF<ACT<SAND<PP2A<CAC<TBP<CYP<TUA<CysPr<EXP<R PL8<TUB<GAPDH;在茄子所有樣本脅迫中,內參基因的M由低到高依次為UBI3=SAND<TBP<TUA<RPL8<eIF<ACT<PP2A<APRT<EXP<L25<DNAJ<UBQ<CysPr<TUB<CAC<G APDH<HSP20<CYP<EF-1α。茄子epl008葉片在低溫脅迫下,PP2A和CysPr的M最小,穩定性最高,TUB的M最大,穩定性最差;茄子eplck葉片在低溫脅迫下,RPL8和TBP的M最小,穩定性最高,TUB的M最大,穩定性最差;茄子epl008葉片在弱光脅迫下,APRT和CAC的M最小,穩定性最好,其次是CysPr,EF-1α的M最大,穩定性最差;茄子eplck葉片在弱光脅迫下,RPL8和SAND的M最小,穩定性最高,其次是TBP,HSP20的M最大,穩定性最差;茄子epl008葉片在低溫弱光雙重脅迫下,DNAJ和APRT的M最小,穩定性最高,SAND的M最大,穩定性最差;茄子eplck葉片在低溫弱光雙重脅迫下,EF-1α和DNAJ的M最小,穩定性最高,GAPDH的M最大,穩定性最差;在所有樣品脅迫下,UBI3和SAND的M最小,穩定性最高,EF-1α的M最大,穩定性最差。
根據GeNorm軟件得到的默認變異系數(VN/VN+1)確認最適內參基因數目。0.15為程序默認的閾值,VN/VN+1<0.15時,則認為最佳的內參基因數為N個。茄子epl008葉片在低溫脅迫,茄子eplck葉片在低溫脅迫,茄子epl008葉片在弱光脅迫,茄子eplck葉片在弱光脅迫,茄子epl008葉片在低溫弱光脅迫、茄子eplck葉片在低溫弱光脅迫和在所有樣本處理下,V2/V3均低于0.15,表明在這些處理中,2個參考基因即可獲得準確的結果(圖4)。
2.3.2 NormFinder軟件分析NormFinder軟件是通過計算基因表達穩定性(S)來分析內參基因的表達穩定性,S越大穩定性越差,S越小穩定性越好,穩定性和S呈現負相關。從NormFinder軟件分析結果(表4)發現,茄子epl008葉片在低溫脅迫下,UBQ的S最小,為0.056,穩定性最好,其次是EF1α,為0.133,TUB的S最大為0.924,穩定性最差;茄子eplck葉片在低溫脅迫下,UBQ的S最小,穩定性最好,其次是PP2A,TUB的S最大,穩定性最差;茄子epl008葉片在弱光脅迫下,APRT的S最好,其次是CAC,EF1α的S最大,穩定性最差;茄子eplck葉片在弱光脅迫下,TBP的S最小,穩定性最好,HSP20的S最大,穩定性最差;茄子epl008葉片在低溫弱光脅迫下,EF1α的S最小,穩定性最好,其次是ACT,SAND的S最大,穩定性最差;茄子eplck葉片在低溫弱光脅迫下,UBQ在試驗中波動不明顯,表達穩定性最高,其次是HSP20,GAPDH的S最大,說明它在試驗中表現很活躍。在所有樣本處理下,DNAJ的S最小,穩定性最好,其次是PP2A,EF1α的S最大,穩定性最差;這和GeNorm軟件分析的結果基本一致。
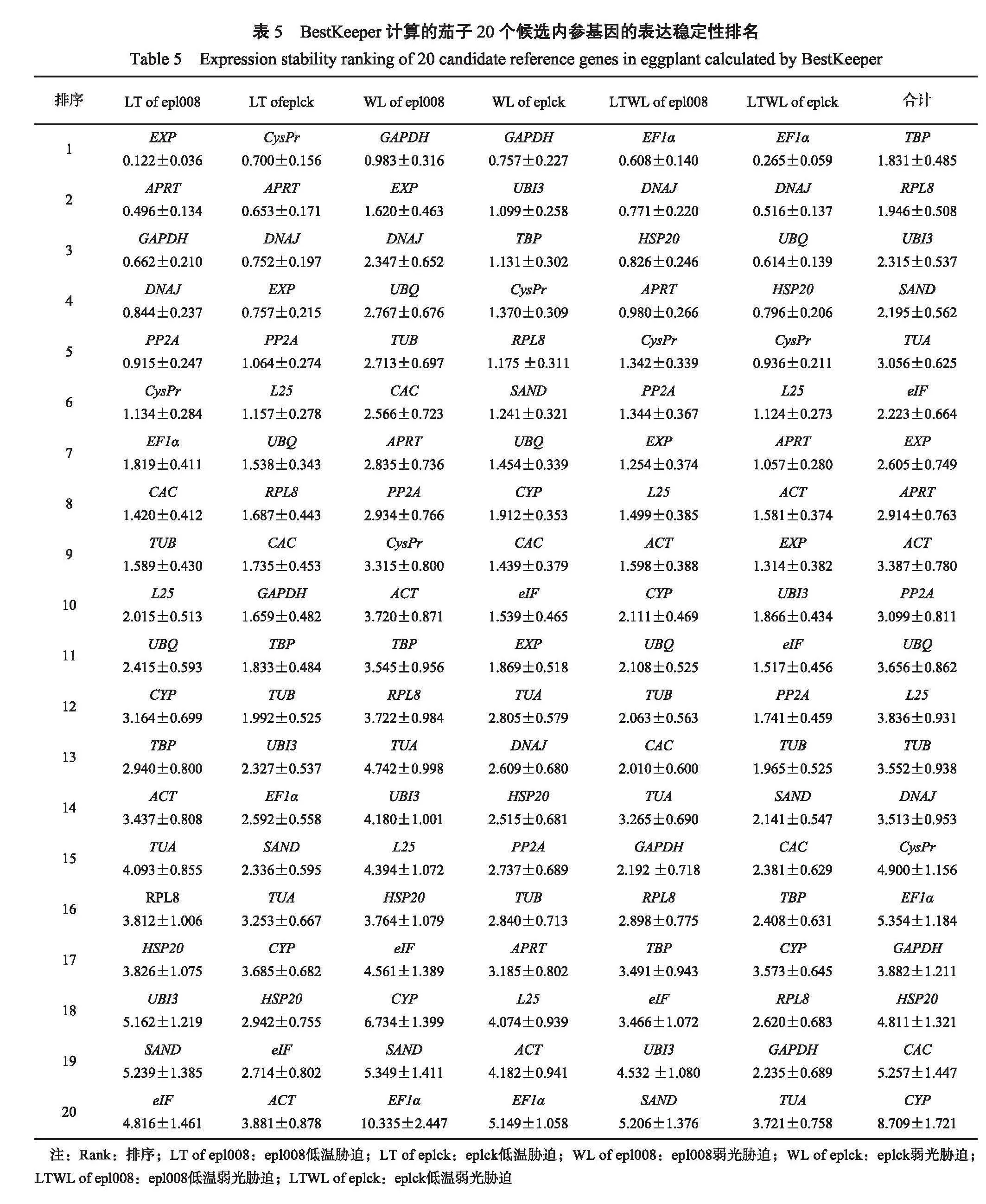
2.3.3 BestKeeper軟件分析BestKeeper軟件主要通過運算出標準偏差(standard deviation,SD)和變異系數(coefficients of variance,CV)通過比較各個值的大小,來確定20個候選內參基因的穩定性。標準偏差和變異系數越小,內參基因越穩定。運用BestKeeper軟件進行運算結果表明(表5),當以低溫脅迫的茄子epl008葉片為材料時,EXP的SD為0.122,CV為0.036,均小于1,穩定性最好,eIF的SD最大,為4.816,CV為1.461,穩定性最差。當以低溫脅迫的茄子eplck葉片為材料時,CysPr的SD為0.700,CV為0.156,均小于1,穩定性好,ACT的SD最大,且SD為3.881,CV為0.878,穩定性最差;當以弱光脅迫的茄子epl008葉片為材料時,GAPDH的SD為0.983,CV為0.316,穩定性最好,EF1α的SD大于1,為10.335,CV為2.447,穩定性最差;當以弱光脅迫的茄子eplck葉片為材料時,GAPDH的SD為0.757,CV為0.227,穩定性最好,EF1α的SD大于1,穩定性最差;當以低溫弱光脅迫的茄子epl008葉片為材料時,EF1α的SD為0.608,CV為0.140時,穩定性最好,SAND的SD為5.206,CV為1.376,穩定性最差。在所有樣品處理下,TBP的SD為1.83,CV為0.485,穩定性最好,CYP的SD為8.709,CV為1.721,穩定性最差。這與GeNorm和NormFinder軟件的分析結果基本一致。
2.3.4綜合評價茄子20個內參基因在所有樣品處理下,軟件GeNorm的分析結果為,UBI3和SAND為表達最穩定的內參基因,EF1α為表達最不穩定的內參基因;軟件NormFinder計算結果如下,DNAJ為表達最穩定的內參基因,EF1α為表達最不穩定的內參基因;軟件BestKeeper給出的結果表明,TBP為表達最穩定的內參基因,CYP為表達最不穩定的內參基因;3種不同的統計方法得到的結果各不相同,通過幾何均值統計法綜合分析GeNorm、NormFinder和BestKeeper軟件3種計算方法的結果,并結合geNorm軟件的對不同處理下最佳內參基因數量的分析結果(表6):茄子epl008葉片在低溫脅迫下,PP2A和APRT表達穩定性最好,eIF表達穩定性最差;茄子eplck葉片在低溫脅迫下,RPL8和TBP表達穩定性最好,HSP20表達穩定性最差;茄子epl008葉片在弱光脅迫下,APRT和CAC表達穩定性最好,EF1α表達穩定性最差;茄子eplck葉片在弱光脅迫下,TBP和UBI3表達穩定性最好,EF1α表達穩定性最差;茄子epl008葉片在低溫弱光脅迫下,EF1α和DNAJ表達穩定性最好,SAND穩定性最差;茄子eplck葉片在低溫弱光脅迫下,EF1α和DNAJ表達穩定性最好,GAPDH穩定性最差。在所有的樣品處理下,TBP和UBI3基因表達穩定性最好,CYP表達穩定性最差。
3討論與結論
茄子富含對人體有益的維生素P,是全球排名前十的健康蔬菜之一[50]。南方冬季初春,茄子的生長常常受到低溫弱光等惡劣環境條件的影響,茄子抗逆基因的挖掘、抗逆品種的培育等已成為科研工作者們的研究熱點[51?55]。qRT-PCR是確定抗逆基因表達最常用的手段之一,而表達量數據的精確性取決814bd4b4b5ebc8561f8a0037d0c7e00e于使用內參基因的穩定性。在針對茄子不同抗性品種的研究中,篩選出在低溫、弱光及低溫弱光等脅迫下表達穩定的內參基因,對于深入探究茄子應對低溫弱光逆境的機制具有重大意義[51]。本研究總結前人內參基因研究結果,結合茄子轉錄組測序數據挖掘候選內參基因,qRT-PCR數據顯示每個候選內參基因表達量都存在變化,這個結論和Wu等[56]的研究結果類似。在特定條件下看家基因表達量變化較小,但如果將之作為內參基因,還需要進一步對這些候選內參基因的穩定性進行評估。
不同的統計方法可能獲得截然相反的實驗數據,究竟哪個軟件評價的結果更可靠,目前沒有定論[57]。根據前人的經驗,本研究使用了GeNorm、NormFinder和BestKeeper3個軟件統計計算,分析20個候選內參基因的穩定性[58],發現每種軟件的候選內參基因穩定性排序存在差異。3種計算方法的分析結果相似,但由于分析方式和計算方法不同,候選內參基因穩定性排序上略有不同。GeNorm和NormFinder統計方法獲得相同的最活躍基因EF1α。BestKeeper軟件和獲得和綜合3種統計方法排序獲得了相同最穩定基因TBP和最不穩定基因CYP。這樣的結果是隨機的呢,還是說明BestKeeper軟件的統計方法優于其他兩種方法還有待進一步確定。本研究采用多種算法進行內參基因表達穩定性統計,最大程度上降低因不同的計算方法而造成的數據偏差,并根據幾何平均值綜合評價分析,結果表明,在這項研究中TBP在所有樣本中為最穩定的內參基因,而鄧嘉碩等[51]在冷脅迫等多種逆境處理下,篩選出的ACT基因則表現相對活躍。
肌動蛋白ACT作為細胞骨架形成的重要蛋白,常常被認為是表現最穩定的內參基因之一[59?60]。鄧嘉碩等[51]對不同組織與冷脅迫等逆境下茄子qRT-PCR內參基因篩選,ACT均為最穩定內參基因。為驗證ACT基因的穩定性,本試驗設計在低溫脅迫、弱光脅迫、低溫弱光脅迫處理下,采集低溫弱光敏感eplck和耐低溫弱光epl008品系0、3、6、12、24和48h的葉片為樣本,從轉錄組數據中挖掘包括ACT基因的20個候選內參基因中,篩選茄子在這3種非生物因素下的最佳內參基因。本研究對篩選的數據進行綜合評價分析,TBP在所有樣本中表達穩定性最佳。但在特定的試驗條件下,例如epl008品系低溫脅迫下PP2A基因穩定性最好,ACT穩定性排名第8;eplck品系低溫脅迫下,RPL8基因表達穩定性最佳,ACT排名第19。epl008品系弱光脅迫下,ACT基因穩定性排名第16;eplck品系低溫脅迫下,ACT基因穩定性排名第18。在低溫弱光脅迫下,epl008品系的樣本,ACT基因穩定性排名第5,在epl008品系樣本中,ACT基因穩定性排名第8。在所有的茄子樣本中,ACT基因穩定性排名第9。即使在相同的冷脅迫條件下,針對不同茄子品系,使用不同試驗設備,也可能得出不同的結果,ACT基因在茄子研究中沒有通用性。為了順利研究低溫弱光等脅迫下功能基因的表達水平,篩選茄子內參基因是關鍵步驟。
參考文獻:
[1]李彩霞,林碧英,申寶營,等.低溫對茄子幼苗生理特性的影響及耐冷性指標的篩選[J].福建農業學報,2018,33(9):930?936.
[2]DONG R Y,YU B W,YAN S S,et al.Analysis of vitamin P content and inheritance models in eggplant[J].Horticultural Plant Journal,2020,6(4):240?246.
[3]PHAM T N M,SHIN C Y,PARK S H,et al.Solanum melongena L.extract protects retinal pigment epithelial cells from blue light-induced phototoxicity in in vitro and in vivo models[J].Nutrients,2021,13:359.
[4]姜濤.福建省茄子生產調查與耐低溫資源篩選及生理響應研究[D].福州:福建農林大學,2017.
[5]楊玉凱,陳藝群,林碧英,等.光質對茄子幼苗光合特性及葉綠素熒光參數的影響[J].福建農林大學學報(自然科學版),2018,47(6):673?680.
[6]ZHANG Y,HAN X,CHEN S,et al.Selection of suitablereference genes for quantitative real-time PCR gene expression analysis in Salix matsudana under different abiotic stresses[J].Scientific Reports,2017,7:40290.
[7]WANG Z,XU J,LIU Y,et al.Selection and validation of appropriate reference genes for real-time quantitative PCR analysis in Momordica charantia[J].Phytochemistry.,2019,164:1?11.
[8]SUN H,JING X,SUN M,et al.Evaluation of reference genes for normalizing RT-qPCR in leaves and suspension cells of Cephalotaxus hainanensis under various stimuli[J].Plant Methods,2019,15:31.
[9]NGUYEN D,EAMENS A,GROF C.Reference gene identification for reliable normalisation of quantitative RT-PCR data in Setaria viridis[J].Plant Methods.,2018,14:24.
[10]EXP?SITORODR?GUEZ M,BORGES A A,BORGESP?REZ,A,et al.Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development processs[J].BMC Plant Biology,2008,8:131.
[11]LIU Q,QI X,YAN H,et al.Reference gene selection for quantitative real-time reverse-transcriptase PCR in annual ryegrass(Lolium multiflorum)subjected to various abiotic stresses[J].Molecules,2018,23:172.
[12]NOLAN T,HANDS R,BUSTIN S.Quantification of mRNA using real-time rt-PCR[J].Nature Protocols,2006,1(3):1559?1582.
[13]HUGGETT J,DHEDA K,BUSTIN S,et al,A.Real-time RT-PCR normalisation;strategies and considerations[J].Genes Immun,2005,6:279?284.
[14]BUSTIN S,BEAULIEU J F,HUGGETT J,et al.Practical implementation of minimum standard guidelines for fluorescence-based quantitative real-time PCR experiments[J].BMC Molecular Biology,2010,11:74.
[15]LI M Y,XIONG A S,WANG F,et al.Suitable Reference genes for Accurate gene expression analysis in parsley(Petroselinum crispum)for abiotic stresses and hormone stimuli[J].Frontiers In Plant Science,2016,7:1263.
[16]XIN LIU X,GUAN HR,SONG M,et al.Reference gene selection for qRT-PCR assays in Stellerachamaejasme subjected toabiotic stresses and hormone treatments based on transcriptome datasets[J].PeerJ,2018,6:e4535.
[17]WEI M,CHEN Y X,ZHANG M Q,et al.Selection and validation of reference genes for the qRT-PCR assays of populus ussuriensis gene expression under abiotic stresses and related ABA treatment[J].Forests,2020,11:476.
[18]WEI L B,MIAO H M,Ruihong ZHAO R H,et al.Identification and testing of reference genes for Sesame gene expression analysis by quantitative real-time PCR[J].Planta,2013,237:873?889.
[19]劉濤,熊青,許穎妍,等.夜香樹花期熒光定量PCR內參基因的篩選[J].植物科學學報,2017,35(4):534?542.
[20]HONG S Y,SEO,P J,YANG M S,et al.Exploring valid reference genes for gene expression studies in Brachypodium distachyon by real-time PCR[J].BMC Plant Biology,2008,8:112.
[21]LI L,LI N.FANG H,et al.Selection and validation of reference genes for normalisation of gene expre-ssion in glehnia littoralis[J].Scientific Rorports,2020,10:7374.
[22]LACERDA A L M,FONSECA L N,BLAWID R,et al.Reference gene selection for qPCR analysis in tomato-bipartite begomovirus interaction and validation in additional tomato-virus pathosystems[J].PLoS ONE,2015,10(8):e0136820.
[23]WAN H J,YUAN W RUAN M Y,et al.Identification of reference genes for reverse transcription quantitative real-time PCR normalization in pepper(Capsicum annuum L.)[J].Biochemical and Biophysical Research Communications,2011,416:24?30.
[24]L?VDAL T,LILLO C.Reference gene selection for quantitative real-time PCR normalization in tomato subjected to nitrogen,cold,and light stress[J].Analytical Biochemistry,2009,387(2):238?242.
[25]QU R J,MIAO Y J,CUI Y J,et al.Selection of reference genes for the quantitative real-time PCR normalization of gene expression in Isatis indigotica fortune[J].BMC Molecular and Cell Biology,2019,20:9.
[26]YIN X J,HE T T,YI K,et al.Comprehensive evaluation of candidate reference genes for quantitative real-time PCR-based analysis in Caucasian clover[J].Scientifc Reports,2021,11:3269.
[27]BAEKA E,YOON J Y,PALUKAITIS P.Validation of reference genes for quantifying changes in gene expression in virus-infected tobacco[J].Virology,2017,510:29?39.
[28]GANTASALA N P,PAPOLU P K,THAKUR P K,et al.Selection and validation of reference genes for quantitative gene expression studies by real-time PCR in eggplant(Solanum melongena L.)[J].BMC Research Notes,2013,6(1):1?11.
[29]KANAKACHARI M,SOLANKE A U,PRABHAKARAN N,etal.Evaluation of suitable reference genes for normalization of qPCR gene expression studies in brinjal(Solanum melongena L.)during fruit developmental stages[J].Applied Biochemistry And Biotechnology,2016,178(3):433?50.
[30]周曉慧,劉軍,莊勇.喀西茄內參基因實時熒光定量PCR表達穩定性評價[J].園藝學報,2014,41(8):1731?1738.
[31]龐強強,李植良,羅少波,等.高溫脅迫下茄子qRT-PCR內參基因篩選及穩定性分析[J].園藝學報,2017,44(3):475?486.
[32]VANDESOMPELE J,DE PRETER K,PATTYN F,et al.Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J].Genome Biology,2002,3(7):RESEARCH0034.
[33]ANDERSEN C L,JENSEN J L,?MTOFT T F.Normalization of real-time quantitative reverse transcription-PCR data:a model-based variance estimation approach to identify genes suited for normalization,applied to bladder and colon cancer data sets[J].Cancer Research,2004,64(15):5245?5250.
[34]PFAFFL M W,TICHOPAD A,PRGOMET C,et al.Determination of stable housekeeping genes,differentially regulated target genes and sample integrity:BestKeeper—Excel-based tool using pair-wise correlations[J].Biotechnology Letters,2004,26(6):509?515.
[35]WANG S B,LIU K W,ZHI L,et al.Evaluation of appropriate reference genes for gene expression studies in pepper by quantitative real-time PCR[J].Molecular Breeding,2012,30(3):1393?1400.
[36]SCHMIDT GW,DELANEY S K.Stable internal reference genes for normalization of real-time RT-PCR in tobacco(Nicotiana tabacum)during development and abiotic stress[J].Molecular Genetics And Genomics,2010,283:233?241.
[37]YU J J,SU Y,SUN J Y,et al.Selection of stable reference genes for gene expression analysis in sweet potato(Ipomoea batatas L.)[J].Molecular and Cellular Probes,2020,53:101610.
[38]TANG X,ZHANG N,SI H J,et al.Selection and validation of reference genes for RT-qPCR analysis in potato under abiotic stress[J].Plant Methods,2017,13:85.
[39]TANG F,CHU L W,SHU W B,et al.Selection and validation of reference genes for quantitative expression analysis of miRNAs and mRNAs in Poplar[J].Plant Methods,2019,15:35.
[40]HOSSAIN M S,AHMED R,HAQUE M S,et al.Identifcation and validation of reference genes for real-time quantitative RT-PCR analysis in jute[J].BMC Molecular and Cell Biology,2019,20:13.
[41]SU W B,YUAN Y,ZHANG L,et al.Selection of the optimal reference genes for expression analyses in diferent materials of Eriobotrya japonica[J].Plant Methods,2019,15:7.
[42]LIU B,JIANG Y T,LIN R Q,et al.Selection and Validation of Reference Genes 1 Desirable for Gene Expression Analysis by qRT-PCR on Seed Germination of Castanea henryi,2021:https://doi.org/10.1101/2021.01.27.428382.
[43]REN R,HUANG F Z,GAO R X,et al.Selection and validation of suitable reference genes for RT-qPCR analysis in dove tree(Davidia involucrata Baill.)[J].Trees,2019,33:837?849.
[44]ZHANG J,ZHU C S,HUO Y B,et al.Validation of suitable reference genes for quantitative gene expression analysis in Tripterygium wilfordii[J].Molecular Biology Reports,2019,46:4161?4174
[45]WANG C,CUI H M,HUANG T H,et al.Identification and validation of reference genes for qRT-PCR analysis in non-heading chinese cabbage flowers[J].Frontiers in Plant Science,2016,7:811.
[46]MARIOT R F,DE OLIVEIRA L A,VOORHUIJZEN M M,et al.Selection of reference genes for transcriptional analysis of edible tubers of potato(Solanum tuberosum L.)[J].PLoS ONE,2015,10:e120854.
[47]李飛,徐建飛,劉杰,等.冷馴化前后野生馬鈴薯Solanum acaule內參基因的篩選[J].西南農業學報,2012,25(5):1592?1595.
[48]LOPEZ-PARDO R,GALARRETA J,RITTER E.Selection of housekeeping genes for q RT-PCR analysis in potato tubers under cold stress[J].Molecular Breeding,2013(31):39?45.
[49]ZHAO Z,ZHOU H,NIE Z,et al.Appropriate Reference Genes for RT‐qPCR Normalization in Various Organs of Anemone flaccida Fr.Schmidt at Different Growing Stages[J].Genes,2021,12:459.
[50]陳錦溶,呂紫見,范麗莎,等.上位基因控制的茄子果色遺傳效應解析[J].中國農業科學,2023,56(23):4729?4741.
[51]鄧嘉碩,馮程,張瀧昊,等.不同組織與逆境脅迫下茄子qRT-PCR內參基因篩選[J].中國蔬菜,2024(8):37?46.
[52]WANG Y,BAN Q Y,LIU T J,et al.Genome-wide identification and expression analysis of Autophagy-Related Genes in eggplant(Solanum melongena L.)[J].Scientia Horticulturae,2024,330:113085.
[53]LI D,TANG X,DONG Y,et al.Comparative genomic investigation of TCP gene family in eggplant(Solanum melongena L.)and expression analysis under divergent treatments[J].Plant Cell Reports,2022,41(11):2213?2228.
[54]LI B,CHEN X,WU Y,et al.Gene characterization and molecular pathway analysis of reverse thermosensitive genic male sterility in eggplant(Solanum melongena L.)[J].Horticulture Research,2019,1(6):118.
[55]SAINI D K and KAUSHIK P.Visiting eggplant from a biotechnological perspective:A review Visiting eggplant from a biotechnological perspective:A review[J].Scientia Horticulturae,2019,253:327?340.
[56]WU J Y,ZHANG H G,LIU L Q,et al.Validation of reference genes for RT-qPCR studies of gene expression in preharvest and postharvest longan fruits under different experimental conditions[J].Frontiers in Plant Science,2016,7(439):780.
[57]QI S A,YANG L W,WEN X H,et al.Reference gene selection for RT-qPCR analysis of flower development in Chrysanthemum morifolium and Chrysanthemum lavandulifolium[J].Frontiers in Plant Science,2016,7:287.
[58]ZHANG J C,XIE W G,YU X X,et al.Selection of suitable reference genes for RT-qPCR gene expression analysis in Siberian wild rye(Elymus sibiricus)under different experimental conditions[J].Genes,2019,10(6):451.
[59]LIN H,ZHANG Q R,CAO J S,et al.Selection of suitable reference genes for real-time qPCR gene expression in cauliflower under abiotic stress and methyl jasmonate treatment[J].Biologia Plantarum,2022,66:46?55.
[60]YOU S,CAO K,CHEN C,et al.Selection and validation reference genes for qRT-PCR normalization in different cultivars during fruit ripening and softening of peach(Prunus persica)[J].Scientific Reports,2021,11(1):7302.
(責任編輯:柯文輝)

