云算GPM 混合圖譜分析系統在混合樣本檢驗中的應用
陳安琪
(1.復旦大學 基礎醫學院 法醫系,上海 200032; 2.司法鑒定科學研究院 上海市法醫學重點實驗室上海市司法鑒定專業技術服務平臺 司法部司法鑒定重點實驗室,上海 200063)
混合樣本鑒定是法醫物證領域最為常見的難題之一[1-2]。 源于犯罪現場或涉及刑事糾紛(例如性侵、人身攻擊與謀殺等)的生物學檢材,其樣本均存在DNA 混合的情況[3-4]。 該類樣本在進行基因分型時,可出現兩個或多個同性或異性貢獻者的DNA圖譜,給其中貢獻者的個體識別帶來了極大的挑戰。 為克服混合樣本的鑒定困難,提高法醫工作者對混合樣本的檢測能力,越來越多的分子標記借助二代測序(next-generation sequencing,NGS)技術手段來解決混合樣本的鑒定問題[5-6]。 雖然這些方法能夠在一定程度上解決混合樣本的拆分問題,但其應用依舊面臨兩大弊端:一方面,基于NGS 的方案將產出數量巨大的原始數據,其分析需要依賴強大的生信團隊,故并不適用于普通法醫學實驗室;另一方面,這些依賴新技術的新型分子標記沒有可供比對的人群數據庫,意味著在缺乏嫌疑人基因分型數據的情況下將很難鎖定罪犯,無法對案件偵破提供更具指向性的線索。
基于毛細管電泳(capillary electrophoresis,CE)的短串聯重復序列(short tandem repeat,STR)分型方法(CE-STR)是當今世界范圍內公認的法醫物證鑒定方法[7],STR 具有高度多態性和顯著的個體間差異,許多國家都建立了基于STR 的人群數據庫[8]。 因此,開發一款以法醫常用STR 分子標記為目標的窗口化分析系統極具價值。 云算GPM 混合圖譜分析系統是一項基于全連續法概率分型的軟件[9],相較于其他常用概率模型(如:二進制法、半連續法),其更為全面地考量了影響STR 準確檢出的各種因素(如:峰值變異性、混合比例及stutter 峰等),通過分析圖譜中的全部信息,以概率的形式給出可能的分型結果。 全連續法概率模型是目前領先的混合圖譜拆分方法,該分析系統或許是一個可靠的混合樣本分析系統。 為探究該分析系統對混合樣本的拆分能力,本研究應用Power-Plex21R○基因分型試劑盒對13 例2~3 人混合樣本進行了STR 分型,觀察了其在預設閾值條件下的分型情況,探究了云算GPM 混合圖譜分析系統對模擬混合樣本的基因型拆分情況。
1 材料與方法
1.1 材料
本研究所用DNA 樣本源于5 名已知基因型的無關個體,模擬混合樣本按以下比例(表1)進行混合。
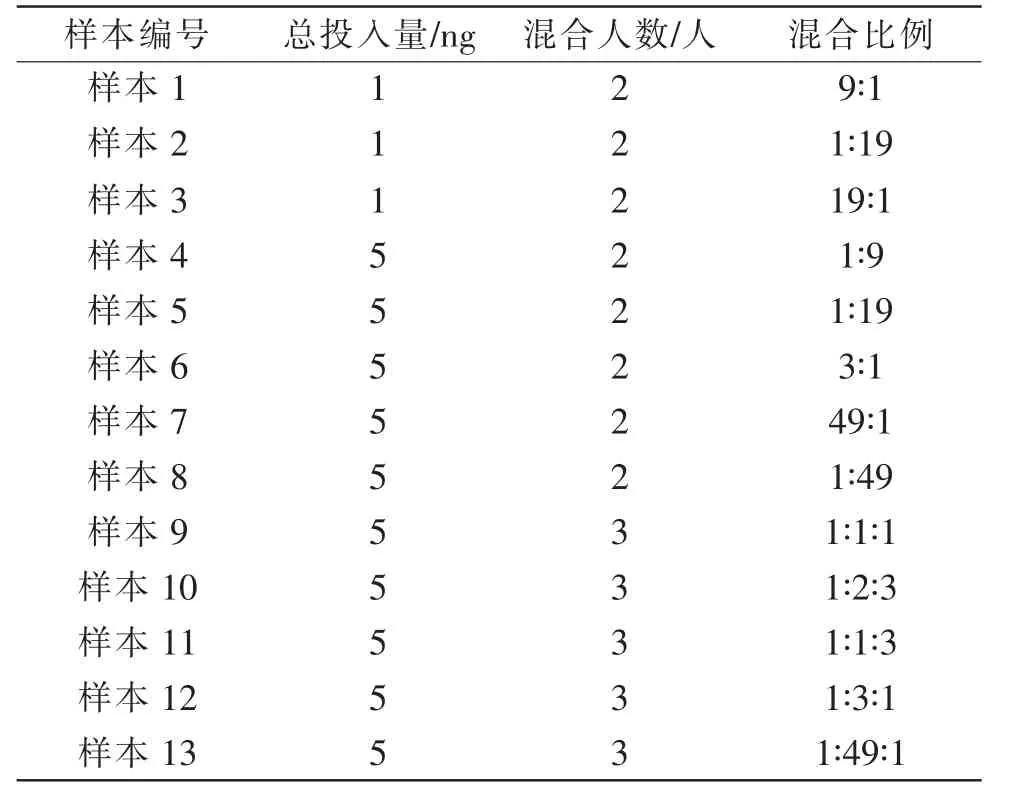
表1 混合樣本概況
1.2 方法
1.2.1 STR 分型
采用Power-Plex21R○試劑盒(美國Promega 公司)對13 例混合DNA 樣本進行復合擴增,擴增體系及程序嚴格遵照試劑盒說明書進行。 PCR 擴增產物用3130XL 型遺傳分析儀(美國Applied Biosystems 公司)進行毛細管電泳分型檢測,STR 基因座分型結果用GeneMapper ID-X 軟件(美國Applied Biosystems公司)在試劑盒預設的默認閾值下進行分析。
1.2.2 基于云算GPM 分析系統的混合圖譜分析
將毛細管電泳輸出的“FSA”格式原始文件導入云算GPM 混合圖譜分析系統(北京瑞源文德科技有限公司),并在軟件內部完成STR 分型分析。為減少人工判讀誤差對后續軟件分析的影響,本研究以混合樣本理論上的分型結果作為參考,對混合圖譜上的等位基因應標盡標,繼而進行下游的混合圖譜拆分分析。
2 結果
2.1 混合樣本的基因型檢出情況
本研究利用Power-Plex21R○試劑盒檢測了13例混合樣本的基因分型情況。 在系統預設的默認閾值條件下,其基因型檢出情況與預期結果存在較大差異。 如圖1 所示,絕大多數基因座的分型結果均不符合預期。 在基因座水平上,Amel 的基因型一致性最高,高達100%(13/13),TH01 次之(92.31%,12/13),D3S1358 位列第三(69.23 %,9/13)。 D13S317 與D16S539 是檢出一致性最低的基因座,僅為7.68%(1/13)。 在樣本水平上,多數樣本僅有1/4~1/5 的等位基因分型結果與預期相符, 樣本9 的分型結果一致性最高,約為76.19%(16/21)。 此外,樣本12、樣本10 和樣本6 的分型一致性同樣相對較高,分別為52.38%(11/21)、47.62%(10/21)和38.10%(8/21)。
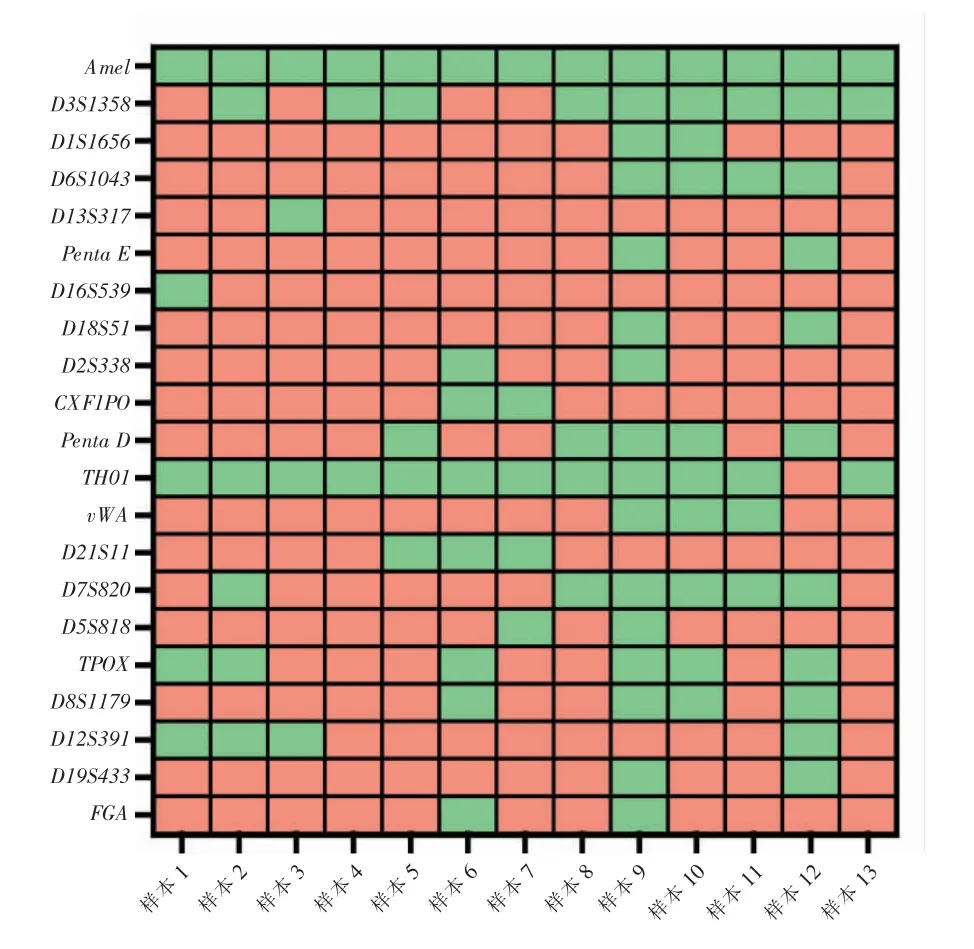
圖1 混合樣本基因型的預期分型與實際檢出情況的比較
2.2 基于云算GPM 的混合圖譜拆分
云算GPM 分析系統可對混合圖譜的基因型進行拆分,結果如圖2 所示(紅色表示分型結果完全不符合預期;橙色表示分型結果有部分符合預期;綠色表示分型結果與預期完全相同)。 13 例混合圖譜均可被成功拆分,除樣本1 的主要貢獻者與次要貢獻者的基因型拆分結果完全正確外,其他樣本的基因型結果均存在一定的誤差。 其中,主要貢獻者的檢出情況較為準確,僅樣本3 存在一個基因座(D12S391)的分型錯誤,約6.54%(18/273)的基因座分型存在拆分結果錯誤的情況,絕大多數基因座(93.04%,254/273)的分型結果完全正確[圖2(a)]。在次要貢獻者的等位基因檢出方面,其總體分型準確率遠不如主要貢獻者。 15 個次要貢獻者共產生315 個基因座分型結果,約50.48%(159/315)的次要貢獻者的基因型完全正確,約41.59%(131/315)的基因型僅有部分滿足預期,約7.94%(25/315)的分型結果是完全錯誤的[圖2(b)]。
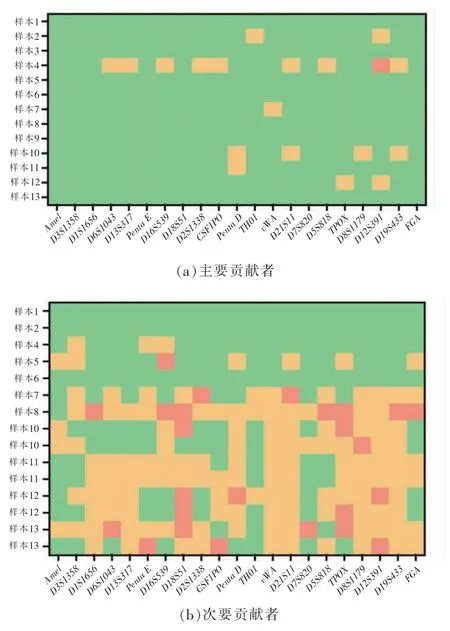
圖2 基于云算GPM 混合圖譜分析系統的混合樣本基因型拆分結果
總體來看,雖然各基因座的分型正確的個數均占多數,但不同基因座水平間的拆分結果準確性仍存在差異。 D7S820 是基因型拆分準確率最高的基因座(89.29%,25/28),Amel 次之(85.71%,24/28),TH01 位列第三(85.71%,24/28)。 除以上3 個基因座之外,還有其他9 個基因座(D3S1358、D1S1656、D6S1043、D13S317、Penta E、D2S1338、CSF1PO、D5S818和FGA)的準確率均在70%以上。 其中,Amel、D3S1358、D13S317、TH01 和vWA 沒有分型完全錯誤的情況發生。與此同時,基因型拆分結果較差的基因座分別為D18S51、TPOX 和D12S391(圖3)。
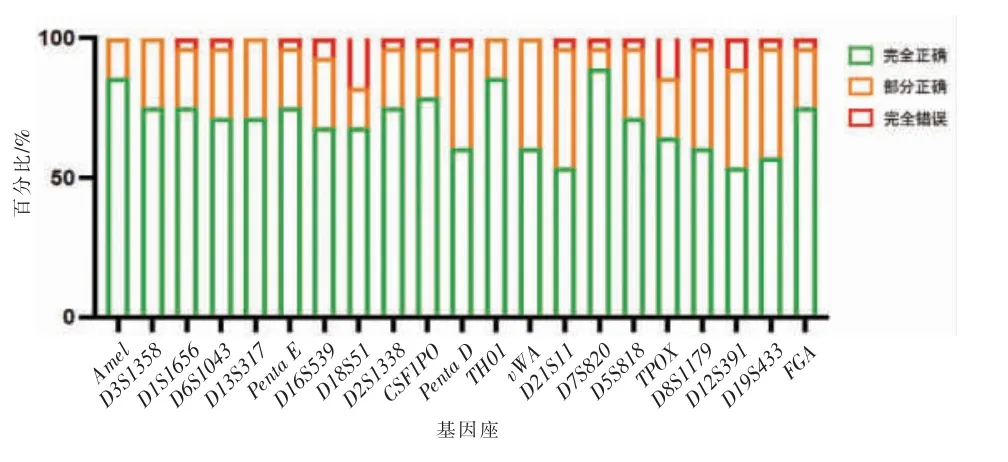
圖3 混合圖譜基因型拆分結果在基因座水平的表現
2.3 weight 值與分型結果的可靠性分析
為確保拆分結果的準確性,云算GPM 分析系統采用預設weight 值(權重)衡量所得基因型的可靠性。 本研究結果中,weight 值在90%以上的基因座有267 個,占總數的41.01%(267/651),其中,分型完全正確的基因座、部分正確的基因座和完全錯誤的基因座所占比例分別為99.63%(266/267)、0.37%(1/267)和0%(0/267)。值得注意的是,并非所有正確分型的基因座weight 值均大于90%。基因座分型完全正確、部分正確和完全錯誤這3 種情況所對應的weight 平均值分別為82.36 %±24.68 %、40.22%±16.56%和35.12%±18.24%[圖4(a)]。為進一步分析各分型結果下的weight 值分布情況,本研究對各分型結果下的基因座數進行了基于該頻率的擬合分析[圖4(b)]。 結果顯示,3 種分型結果在weight 值低于90 %時,均存在一定的交疊,雖然weight 值越低,其歸屬于錯誤分型的可能性越高,但無法完全根據某一weight 值作出結果是完全正確、部分正確或是完全錯誤的推斷。
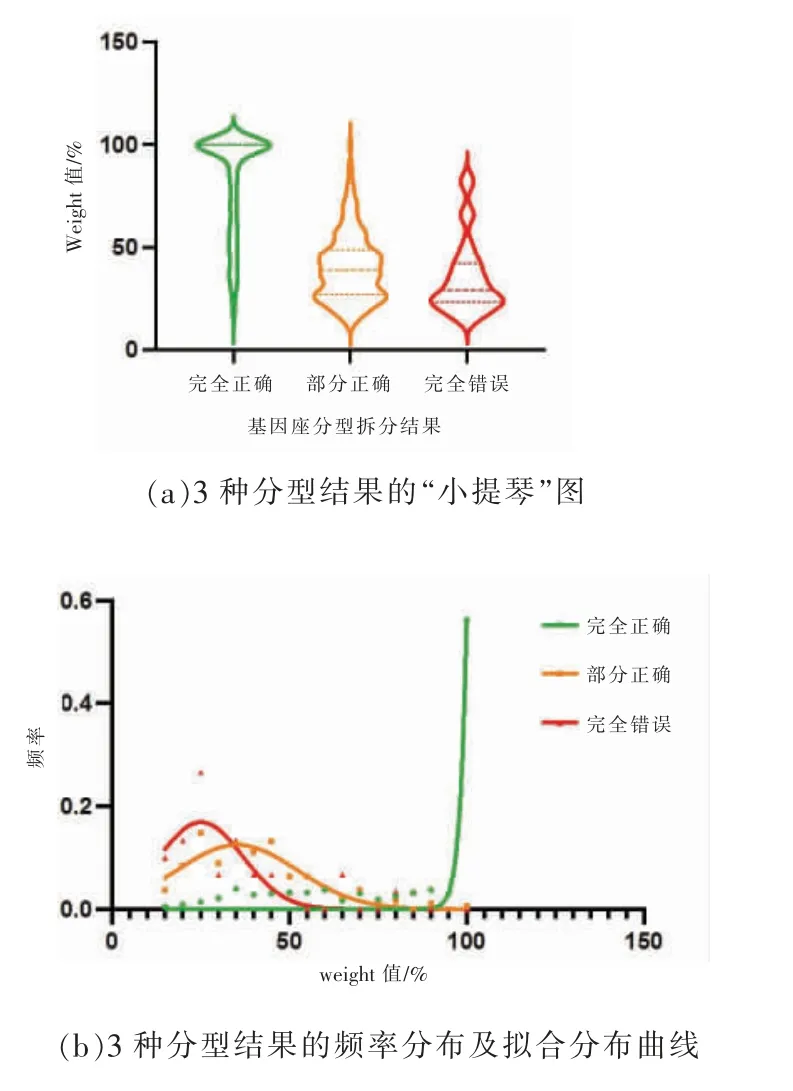
圖4 混合圖譜基因型拆分結果與weight 值的關系
3 討論
混合樣本的鑒定一直以來都是司法鑒定的重點與難點,其基因分型的成功拆分將為后續的案件偵破提供有效證據與線索[2]。 雖然用于混合樣本基因型檢測的方案層出不窮,但基于STR 分子標記的窗口化分析系統卻較為少見。 STRmix 和云算GPM分析系統是目前較為成熟的混合樣本拆分系統,李甫等[9]曾對這兩種分析系統進行了比較,認為兩者均可用于混合樣本的拆分,但其結果存在一定差異。 上述研究的樣本來源多限于真實案件樣本,缺乏對已知組分的模擬混合DNA 研究,因而難以確定檢測系統的正確性。 為進一步探究云算GPM 混合圖譜分析系統在混合樣本檢驗中的表現,本研究模擬了13 例2~3 人DNA 混合樣本,并用經過廣泛驗證的Power-Plex21R○試劑盒[10-14]對其進行檢測。為確保分型數據的有效性,避免因DNA 不足而導致的等位基因丟失,本研究中所有次要貢獻者的投入量均高于其最低檢出限(50 pg[11])。 由于該試劑盒的主要檢測對象為單一來源樣本,因此其系統內置的分析閾值在混合樣本的結果輸出上或許并不準確。 不出所料,在Power-Plex21R○試劑盒預設的默認閾值條件下,絕大多數的基因型均與預期不同(圖1)。 在對這21 個基因座的分型分析中發現,不同等位基因間的分型一致性存在差異。 所有Amel的基因型均符合預期,TH01 與D3S1358 的分型一致性同樣較高,分別為92.31 %和69.23 %,而D13S317 與D16S539 的一致性卻僅為7.68%。 基于人類遺傳基本規律,正常人的Amel 分型結果僅可能是XX、XY 的一種,故而推測本研究中STR 分型的一致性差異或與基因座的遺傳多態性相關。 有研究[15]表明,D13S317 與D16S539 的多態信息量(polymorphism information content,PIC)分別為0.794 3 和0.791 7,約是TH01(PIC 為0.603 6)與D3S1358(PIC為0.649 1)的1.22~1.32 倍,提示TH01 與D3S1358的高一致性極有可能是由該基因座有限的基因型組合所致。 對于多數混合樣本而言,僅有約20%的基因座分型結果一致,然而樣本9、樣本12、樣本10和樣本6 中分型一致的基因座數卻遠高于平均水平。 由于以上4 例樣本的投入量均為5 ng,因此可排除因投入量差異而帶來的分型差異。 在混合比例方面,由于以上3 個混合樣本的混合比例相對均衡,故而其主要貢獻者與次要貢獻者的信號也相對均衡,不易被占比高的組分所掩蓋,這可能是造成其在默認閾值下一致性相對較高的原因。 用于CESTR 分型檢測的試劑盒有很多,絕大多數的CESTR 檢測均是服務于單一來源DNA,而非混合樣本。使用默認閾值下的分析結果,必然會存在次要等位基因被覆蓋的偏差。 因此,混合圖譜的基因型認定依舊非常依賴法醫工作者的個人經驗。
由于云算GPM 分析系統主要依賴于研究人員對混合圖譜的基因型認定,而混合DNA 的等位基因認定對于結果拆分的正確性起著至關重要的作用。 為排除人工誤差,并最大限度地測試云算GPM分析系統的去卷積能力,本研究根據預期的DNA混合圖譜結果對電泳圖譜進行注釋,以期探究其對主要貢獻者和次要貢獻者的基因型識別情況。 結果顯示,混合圖譜的拆分結果并不能確保100%的準確性,在13 例混合樣本中,除樣本1 的拆分結果完全正確外,其他混合樣本或多或少均存在差錯(圖3)。主要貢獻者的總體分型準確率(93.04%)高于次要貢獻者(50.48%),該結果與常識相符,主要貢獻者因其投入量高的原因,信號也往往更強,能提供更為確切的信息[16-17]。
在法醫實踐中,拆分所得的基因型往往有同罪犯數據庫比對的需求,因此,用于比對的基因座分型正確性對于嫌疑目標的篩選至關重要。基因座在群體遺傳學中的多態性及其在特定檢測試劑盒中的性能表現,造成了其拆分的難易度存在差異。本研究中,各基因座拆分后的準確率顯示出了明顯的差異,基因型拆分準確性最高的基因座是D7S820,其準確率(89.29%)約為最低者(D21S11 和D12S391,53.57%)的1.67 倍(圖3),這提示某些基因座或許不適用于混合樣本的檢測。 如法醫工作人員有進行數據庫比對的必要,則可優先選擇weight 值較高的基因型,避免因基因型的拆分差錯而導致嫌疑人篩查錯誤。 就如何判斷已拆分基因分型的正確與否,云算GPM 混合圖譜分析系統內置的weight 值可用于判斷結果的可靠性,一般而言,weight 值大于90%被認為是分型結果可靠的指標[18]。 本研究中,各分型結果的weight 值存在差異,分型完全正確的基因座weight 值最高,部分正確與完全錯誤的基因座weight 值均較低,且這兩者間的差異也相對較小[圖4(a)]。 與此同時,基于weight值的頻率分布圖也顯示絕大多數weight 值大于90%的分型結果是完全正確的[圖4(b)]。 以上結果表明,區分基因座分型結果完全正確者相對較易,weight 值大于90%是基因分型正確的既不充分也不必要條件。
4 結論
服務于單一來源樣本的STR 分型檢測試劑盒對混合樣本的分型幫助有限,現有的混合樣本拆分軟件也不能解決混合圖譜中的基因型認定問題,混合樣本中的等位基因識別仍舊高度依賴法醫工作者的個人經驗。 基因分型的準確與否與該貢獻者的占比存在相關性,weight 值大于90%是一個相對可靠的分型結果評價指標。 由于多數樣本并未達到100%的分型一致性, 因此在有參考數據庫的情況下,選擇weight 值較高的基因座進行嫌疑人的篩選或許是最佳選擇。 綜上,本研究測試了云算GPM 混合圖譜分析系統對混合樣本基因型拆分的結果,探討了該系統在混合樣本基因型拆分上的可靠性,可為云算GPM 混合圖譜分析系統的使用及未來優化提供參考數據。

