近端與遠端胃癌患者胃內微生態結構和多樣性差異的對比研究
白艷麗 鄭玉峰 田笑笑
胃癌為常見惡性疾病。隨著人們的生活水平的提高,該病的發病率與死亡率呈上升趨勢,嚴重威脅人們生命健康[1]。日本胃癌協會根據胃彎大小的分點連線,將胃癌分為近端胃癌、中段胃癌、遠端胃癌,其中近端與遠端胃癌更常見[2]。多數胃癌的發生由多因素導致,如環境、基因、吸煙、不良飲食習慣等飲食。但隨著基因擴增測序技術的發展,大量研究表明胃內微生態與胃癌的發生發展關系密切[3]。為此,分析近端、遠端胃癌患者的胃內微生態結構與多樣性的特征,探尋近端、遠端胃癌患者胃內微生態特異菌種,為臨床治療提供參考信息,現報告如下。
1 資料與方法
1.1 一般資料
選擇2019年7月至2019年12月經我院收治的胃癌患者83例,其近端胃癌患者55例,遠端胃癌患者28例。本文經醫學倫理委員會批準開展。納入標準:①符合中華消化雜志《中國早期胃癌篩查流程專家共識意見(草案2017年,上海)》中相關胃癌診斷標準,且經病理學證實為近端、遠端胃癌者[4];②無消化系統疾病或相關胃癌癥狀史;③無心血管、內分泌、呼吸道等系統性疾病者;④入組前30 d內未服用抑酸、微生態制劑、抗生素等藥物者;⑤患者在知曉研究目的的情況下,自愿加入本研究,簽署同意書。排除標準:①呼氣試驗結果顯示為幽門螺旋桿菌感染為陽性者;②有家族腫瘤史。55例近端胃癌患者中,男性39例,女性16例;年齡54~68歲,平均(60.21±6.79)歲;28例遠端胃癌患者中,男性20例,女性8例;年齡51~66歲,平均(57.23±7.65)歲;兩組患者一般資料(性別、年齡等)比較無顯著差異(P>0.05),有可比性。
1.2 方法
1.2.1 采集樣本及處理 取樣前囑咐患者行胃鏡檢查前8 h禁飲禁食,早晨不可刷牙。將胃鏡頭完全置入胃內,用150 mL無菌生理鹽水沖洗胃壁,再將沖洗液全部吸入無菌吸引瓶內,取出吸引瓶,放入冰盒內,將收集好樣本送于實驗室,將樣本在生物安全柜中分為3部分,并做標記,將其中2份放入冰箱中保存備用,另一份待用。采用差速離心和超濾法將樣本細胞與細菌分離。將50 mL無菌離心管裝滿樣本混勻,用4 ℃低溫低速離心機,離心10 min,除沉淀,在生物安全柜內將上清液移入新50 mL離心管內,將上清液在5 μm無菌濾膜上濾過,僅留濾液,再將濾液濾過0.22 μm無菌濾膜,保留濾膜,濾膜上有富集的樣本細菌,于生物安全柜內,用剪刀將處理好后的濾膜剪碎為0.25 cm2碎片,裝入無菌培養皿中,便于樣本微生物提取DNA。
1.2.2 DNA提取 用Mo-BIO Power-Soil DNA isolation kit試劑盒提前DNA。將處理后的濾膜碎片用無菌鑷子放入PowerBead Tubes中混勻,在65 ℃條件下10 min金屬浴后渦旋PowerBead Tubes 2 min;再將70 μL Solution C1加入PowerBead Tubes中,充分混勻后,再渦旋振蕩15 min。將PowerBead Tubes室溫高速離心30 s,移500 μL上清液至2 mL收集管內,并加入250 μL Solution C2,振蕩5 s后,在4 ℃條件下孵育5 min,離心1 min,取600 μL上清液于收集管內,加入200 μL Solution C3,振蕩5 s后,在4 ℃條件下孵育5 min,離心2 min,取約740 μL上清液至2 ml收集管內;加1200 μL Solution C4,振蕩5 s后;將670 μL上述混合液移至Spin Filter中,室溫高速離心1min,去除流過液,DNA結合于柱上;再重復3次DNA上柱步驟,則混合液所有DNA上柱;加500 μL Solution C5至SpinFilter內,離心1 min,去除流過液,再空離心2 min,完全分離SolutionC5;將Spin Filter 置入1.5 mL DNA LoBind tube 上,將30 μL UltraPure DNase/RNase-free ditlledl water加入到SpinFilter白色濾膜中心,孵育2 min后離心1 min,DNA樣本洗脫于DNA Lo-Bind tube 溶液內備用。將提取的1 μL樣本基因組DNA經Qubit dsDNA HS Assay kits試劑盒處理后,采用Qubit熒光定量儀檢測其濃度;再取樣本DNA 3 μL,使用0.8%瓊脂糖凝膠電泳,25 min,檢驗提取DNA的完整性。
1.2.3 基因擴增 將16S rRNA基因V3-V4可變區視為PCR擴增靶標。引物:338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R (5'-GGACTACHVGGGTWTCTAAT-3')。反應體系(30 μL):PCR MasterMix 15 μL;Primer 3 μL;DNA 10 μL;ddH2O 2 μL。反應程序為:95 ℃預變性3 min;95 ℃變性30 s;55 ℃退火30 s;72 ℃延伸45 s;執行27個循環,72 ℃維持10 min。
1.2.4 高通測序和生信分析 用Qubit 2.0 DNA Assay Kit對處理后的DNA進行定量的PCR擴增,按1∶1混合體積上機開展測序。在核酸多聚酶作用下對產物突出端修為鈍端。將3'端腺苷酰作用后的DNA片段與PE頭相連。連接后的產物經核酸純化柱、瓊脂電泳進行分離,并取其適中段,用illumina 二代測序系統對兩端有連接頭的DNA段經10循環PCR開展富集,組成為300~400 bp插入小片段庫,再完成后續上機測序操作。利用系統將圖像轉換成數據,并識別分析。在Miseq序列標記上標簽、引物、接頭。用軟件對引物接頭序列進行去除后,根據具有重疊關系的PE序列特性,把成雙序列拼為一條完整序列,再對拼接后的序列進行識別、區分樣本數據,再將得到的數據進行質控過濾留其有效數據。在PCR反應過程中,一些不完整的擴增產物經循環擴增,與序列相近的同源板一同退火,繼續延伸得更雜合DNA片段稱為“嵌合體”;同時會產生非特異性擴增序列。為此,用USEARCH軟件剔除非特異性序列,并校正測序,檢測嵌合體。刪除嵌合體后序列可用BLAST法與參考數據庫中有代表性序列對比。剔除低于閾值的靶區外序列。
操作分類單元(operational taxonomic units,OTUs),將所有序列聚類后,按照序列與序列的相似性分為不同OTUs。并將相似性閾值設為97%進行分化。用Bergey分類法將菌群分為界、門、綱、目、科、屬水平。
1.2.5 多樣性分析 微生物多樣性通過單樣本多樣性分析完成,其中常用方法包括Alpha多樣性,α-多樣性,用兩者對菌落中物種的豐富度與多樣性進行分析。校正OTU數目,評估菌落的豐富度指數(包括Chaol、ACE指數)與多樣性指數(包括Shannon、Simpson指數)。再用QIIME軟件內的PCoA作圖,分析樣本見差距。
2 結果
2.1 兩組樣本α-多樣性參數比較
兩組樣本經過濾后平均共獲得序列30256條,用于構建OTUs,取得分類Tags數平均為24201條。兩組患者總數 Tags、分類單元tags、Total OTUS數目、Species OTUS數目、Genus OTUS數目、Species Chao 1、Genus Chao 1、Species Shannon指數均無顯著差異(P>0.05),近端胃癌組Genus Shannon指數水平高于遠端胃癌組(P<0.05),見表1。
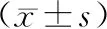
表1 兩組樣本α-多樣性參數比較
2.2 胃癌患者胃內相關細菌群落分類水平分析
近端胃癌組胃內相對豐度前8位菌門依次為:變形菌門為57.10%,泉古菌門為14.27%,擬桿菌門為8.29%,厚壁菌門為7.86%,放線菌門為5.29%,疣微菌門為3.00%,酸桿菌門為2.25%,芽單胞菌門為0.86%。遠端胃癌組胃內相對豐度前8位菌門依次為:變形菌門為56.42%,泉古菌門為14.11%,擬桿菌門為8.12%,厚壁菌門為7.79%,放線菌門為5.24%,疣微菌門為3.03%,酸桿菌門為2.27%,芽單胞菌門為0.84%。再分析近、遠端胃癌組樣本胃內相關細菌群落門水平構成情況,基于相對豐度在前10位的菌門,構建了樣本門水平的物種相對豐度柱形圖,兩組胃內細菌群落中,變形菌門豐度比例最高。分析近、遠端胃癌患者胃內細菌群落在屬水平的構成情況,近端胃癌豐度較高的菌屬依次為:不動桿菌屬占5.22%,斯瓦尼菌屬占3.74%,普里沃菌屬占2.43%,嗜鹽菌屬占1.78%,擬桿菌屬占1.61%,乳桿菌屬占1.52%,韋榮球菌占1.23%,螺桿菌屬占1.24%,棒桿菌屬占0.75%;遠端胃癌:不動桿菌屬占5.34%,斯瓦尼菌屬占3.73%,普里沃菌屬占2.41%,嗜鹽菌屬占1.75%,擬桿菌屬占1.63%,乳桿菌屬占1.50%,韋榮球菌占1.24%,螺桿菌屬占1.25%,棒桿菌屬占0.81%;兩組胃內細菌屬水平相對豐度無明顯差異。
2.3 兩組樣本細菌群落PCoA分析
近端、遠端胃癌組胃內相關細菌群落構成的PCoA分析顯示,橫坐標PC1變化率解釋為12.83%,縱坐標PC2變化率解釋為8.01%;經PCoA分析無法將近端、遠端胃癌組相關細菌群落結構有效區分,見圖1。

圖1 近端、遠端胃癌組患者胃內細菌群落結構的PCoA分析
3 討論
3.1 本研究意義
近年來,隨著胃內幽門螺旋桿菌和其他螺桿菌屬的發現,及隨后進行一系列人體與胃內幽門螺旋桿菌的深入研究證實,人體內胃內環境與腸道環境具有較強的相似性,均有特殊微生物生存環境[5]。目前多數研究對胃內菌群分析,并證實機體胃內除幽門螺旋桿菌外,還有其他大量復雜的菌群結構存在[6]。胃癌的發生發展是由多因素共同作用下導致,如遺傳、環境因素等。近端、遠端胃癌患者胃內真實的菌群構成情況并不清楚。此外,目前對近端、遠端胃癌的系統性研究較少見,多集中于口腔、胃內微生物對胃癌的影響。目前已有研究在正常人群與胃癌患者的胃內微生物進行分析證實,胃癌患者與正常受試者胃內菌落結構、多樣性均有明顯差異[7]。表明,胃癌患者胃內菌群結果與多樣性正發生改變,且與病情發展有關[8]。故本研究設計本課題,通過16SrDNA高通量測序和生物信息學分析方法分析近端、遠端胃癌患者胃內微生物群落構成和多樣性差異。
3.2 近端與遠端胃癌患者胃內微生態結構與多樣性分析
采用16SrDNA高通量測序進行胃內相關細菌群落的高通量測序,質控所得數據,對合格序列行OTUs注釋與樣本間α多樣性分析,總得來說,胃內共有鑒定多個菌門的細菌種系型[9]。α多樣性分析結果顯示,兩組患者總數 Tags、分類單元tags、OTUS數目、 Chao 1、Species Shannon指數均無顯著差異,近端胃癌組Genus Shannon指數水平高于遠端胃癌組(P<0.05);表明近端、遠端胃癌組患者菌群總數、種類多樣性無明顯差異,且具有較高的豐富度[10]。近端胃癌組相對豐度大于1%細菌門共7種,包括變形菌門為57.10%,泉古菌門為14.27%,擬桿菌門為8.29%,厚壁菌門為7.86%,放線菌門為5.29%,疣微菌門為3.00%,酸桿菌門為2.25%。遠端胃癌組相對豐度大于1%細菌門共7種,包括變形菌門為56.42%,泉古菌門為14.11%,擬桿菌門為8.12%,厚壁菌門為7.79%,放線菌門為5.24%,疣微菌門為3.03%,酸桿菌門為2.27%。兩組相對豐度大于1%細菌門均為共同擁有菌。此外對兩組患者胃內菌落屬水平相對豐度分析,顯示相對豐度前5位為近端胃癌組:不動桿菌屬占5.22%,斯瓦尼菌屬占3.74%,普里沃菌屬占2.43%,嗜鹽菌屬占1.78%,擬桿菌屬占1.61%。遠端胃癌:不動桿菌屬占5.34%,斯瓦尼菌屬占3.73%,普里沃菌屬占2.41%,嗜鹽菌屬占1.75%,擬桿菌屬占1.63%。兩組胃內細菌屬水平相對豐度無明顯差異。本研究對胃內細菌種屬的分類后發現患者胃內有大量細菌種型無法歸類于目前已知細菌序列,對于這些未知細菌種屬的生物學特性與臨床作用目前尚不清楚。此外,近端胃癌與遠端胃癌者胃內菌群測序結果總體對比,胃內菌落在物種門、屬等水平相對豐度值無顯著差異。有研究對不同地域、種族的人群胃內菌群進行分析,顯示胃內菌群構成極其相似,其中分別77.4%、79.8%的克隆片段可共享的[11]。還有學者對10例胃癌患者胃內菌群構成行多態技術與16SrDNA測序技術聯合分析,認為胃癌患者與消化不良胃黏膜正常患者胃內菌群構成比較結果顯示,其無明顯差異[12]。我們利用PCoA聚類分析方法對近端、遠端胃癌患者胃內總體細菌群落進行比對分析后顯示兩組間的胃內細菌群落構成和多樣性較相似。進一步說明近端、遠端胃內菌群構成和多樣性相似。
研究為樣本量小的單中心研究,需進一步擴大樣本量證實本研究結論。
綜上所述,近端與遠端胃癌患者胃內微生態結構和多樣性無顯著差異,且據有較高相似。

