PAIP1結合RNA測序及整合分析
鄭劍鋒 ,楊峻 ,李金平 ,楊小潔 ,邵文華 ,秘思思
(1.深圳市寶安區中心醫院,深圳大學第五附屬醫院,廣東 深圳 518102;2.桂林醫學院,廣西 桂林541004)
真核生物的蛋白質翻譯調控過程在基因表達調控中發揮重要作用,而其中翻譯起始階段的調控是限速過程,在蛋白質翻譯過程中的作用更加重要。翻譯起始過程的異常會導致失常的蛋白質組,表現為蛋白質的種類和相對表達量的異常,該過程的異常會導致疾病,包括腫瘤的發生[1-3],因此,了解真核生物翻譯起始階段的調控機理對疾病的靶向治療具有重要的指導意義。真核生物翻譯起始是一個多因素參與、多步驟、動態的反應過程[4]。所有這些步驟涉及翻譯起始因子與核糖體和mRNA的相互作用,除翻譯起始因子外,Poly(A)結合蛋白相互作用蛋白1(Poly(A)binding protein interacting protein 1,PAIP1)的作用尤為特殊,PAIP1能與真核翻譯起始因子3(Eukaryotic translation initiation factor 3,eIF3)和真核翻譯起始因子 4A(Eukaryotic translation initiation factor 4A,eIF4A)相互作用,促進了mRNA環形結構的形成,迄今為止,人類中只發現PAIP1參與mRNA環化銜接,因此,PAIP1是蛋白翻譯起始必須因子[5]。Sonenberg,N等研究發現,PAIP1可通過結合eIF4A和PABP促進蛋白質翻譯,并提出PAIP1可能具有結合mRNA分子的功能[6],然而,該研究沒有證實PAIP1能否結合mRNA分子。為了進一步研究PAIP1的功能,尤其證實PAIP1能否結合mRNA分子,即PAIP1的RNA結合功能,本研究通過RNA結合蛋白富集測序分析(RNA Binding Protein Immunoprecipitation sequencing,RIP-Seq)PAIP1 結合 RNA 的 分子 特征并進行富集分析,探討PAIP1的新功能。
1 資料與方法
1.1 細胞培養與質粒構建 設計PAIP1基因PCR擴增引物,F引物:agcccgggcggatccgaattcATGTCGGACGGTTTCGATC;R 引物:gtcatccttgtagtcctcgagC TGTTTTCGCTTACGCTCTG。依照Invitrogen公司的Trizol試劑操作說明書提取HeLa細胞RNA,根據Promega公司M-MLV操作說明書將RNA逆轉錄成cDNA,對PAIP1基因進行PCR擴增,用QIAGE N公司的QIAquick PCR Purification Kit純化PCR產物。使用Vazyme公司的ClonExpressII One Step Cloning Kit將PAIP1-Flag克隆基因連接到pIRES-hrGFP-1a載體上,構建PAIP1過表達和空載體質粒,將構建好的質粒通過脂質體2000轉染HeLa細胞,37℃培養48h。使用熒光顯微鏡觀察GFP熒光信號強度來判斷質粒轉染效率。
1.2 PAIP1蛋白質表達檢測 質粒轉染成功的HeLa細胞用預冷的1X PBS漂洗3次,棄掉PBS,向細胞培養皿中加入預冷的細胞裂解液 (1×PBS,0.5%脫氧膽酸鈉,0.1%SDS,0.5%NP40)對細胞樣本進行裂解,收集細胞裂解液,4℃,13000 g離心20min,去除細胞碎片。將5X SDS樣品緩沖液進入至細胞裂解液中,在沸水中煮沸10min,10%SDSPAGE分離復合物。用含5%脫脂乳粉的TBST緩沖液(20 mm tris緩沖鹽水和0.1%tween-20)在室溫下孵育 1h,一抗:Flag 抗體(1:2000)、GAPDH(1:2000)和HRP結合二抗孵膜,ECL發光顯影檢測相應蛋白水平的表達情況。
1.3 免疫共沉淀 HeLa細胞在含RNA酶抑制劑和蛋白酶抑制劑的預冷細胞裂解緩沖液中裂解5min, 然后劇烈振蕩,4℃,13000 g離心 20min,去除細胞碎片。用MNase酶部分降解細胞裂解液中的PAIP1-RNA復合物。之后加入A/G-DynaBeads,anti-flag抗體,或對照IgG抗體4℃孵育過夜。分別用低鹽洗滌液、高鹽洗滌液和1X PNK緩沖液各洗滌磁珠2次。用50μl洗脫緩沖液 (50mm Tris-Cl(PH=8.0),10mm EDTA(PH=8.0),1%SDS)對樣品進行重懸,70℃,1400r/min離心,孵育20min。置磁珠3min,將上清轉移至新的EP管中。取40μl樣本消解蛋白質,從PAIP1-RNA復合物中提取RNA。剩余10μl樣本用于免疫印跡 (western blotting,WB)檢測IP效率。
1.4 iRIP-seq文庫的制備及序列分析 將1.3得到PAIP1結合 RNA,用 KAPA-RNA Hyper Prep Kit試劑盒(KAPA,KK8541)制備 cDNA 文庫,在 illumina XTen平臺上對構建的cDNA文庫進行150bp雙端高通量測序。
1.5 數據分析 用TopHat 2[7]將讀段(reads)比對到參考基因組上,將唯一映射讀段用于后續分析。Piranha分析策略用于鑒定PAIP1在基因組上的結合區域[8]。根據測序深度和覆蓋度選取某一固定長度(xx nt)為單位(bin)將基因組等分,統計每個bin中的reads數目。模擬數據中reads的分布情況,來作為背景噪音,然后基于zero truncated negative binomial(ZTNB)來尋找reads分布顯著高于背景的位置。在此過程中每個bin都會獲得一個p-value,根據p-value進行顯著性篩選,得到真實的結合峰。Input組和IP組樣本分別進行模擬分析,去除IP組中與Input組重疊峰。通過peak關聯分析PAIP1結合的靶基因,靶基因結合基序用HOMER軟件[9]計算獲得。
1.6 功能富集分析 利用KOBAS 2.0server對基因進行KEGG聚類分析[10],以確定峰值相關基因(靶基因)的功能類別。采用超幾何試驗和Benjamini-Hochberg-FDR控制程序確定各項的富集程度[11]。
1.7 統計學分析 采用GraphPad Prism6.0版軟件進行數據分析。定量數據以平均數±標準差()記錄,采用t檢驗,定性數據采用Mann-Whitney U非參數檢驗,以P<0.05為差異有統計學差異意義。
2 結果
2.1 PAIP1-Flag過表達HeLa細胞系構建 使用pIRES-hrGFP-1a載體連接PAIP1-Flag基因構建過表達和空載體質粒,將構建好的質粒通過脂質體2000轉染HeLa細胞中。通過熒光顯微鏡照射觀察GFP熒光信號強度來判斷質粒轉染效率,見圖1A。質粒轉染成功的細胞用Western blot檢測PAIP1蛋白表達量,見圖1B。
2.2 免疫共沉淀捕獲PAIP1結合的RNA 使用Flag抗體對PAIP1過表達HeLa細胞進行RIP實驗,以IgG作為陰性對照。Western blot檢測結果分析見圖2A,實驗所用的Flag抗體能特異性地免疫沉積富集PAIP1-Flag蛋白。對免疫沉淀富集的PAIP1-Flag-RNA復合物和Input樣本提取RNA,RNA分子3’端以堿基互補配的方式結合illumina接頭,反轉錄形成cDNA第一鏈,以鏈置換的方法連接另一端illumina接頭,再使用illumina建庫和測序兼容引物PCR擴增,擴增后的cDNA文庫使用DNA Clean磁珠純化,在illumina測序平臺上進行雙端150bp測序反應,測序cDNA文庫片段大小,見圖 2B。
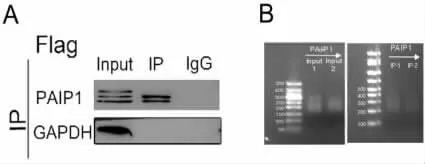
圖2 免疫共沉淀捕獲PAIP1結合的RNA
2.3 高通量測序及數據基礎分析 在illumina-Xten平臺中對文庫進行雙端150bp的測序,獲得2千萬左右的原始reads數據,見表1A。使用TopHat 2軟件將有效測序數據比對到人的參考基因組上,見表1B。在參考基因組上具有唯一定位的非冗余數據(rmdup reads)統計其在基因組各個分區部分情況,見表1C。
2.4 數據高級分析 使用Piranha軟件以Input樣本為背景對PAIP1-Flag實驗樣本rmdup reads進行Peak Calling,2次實驗樣本各分別得到peak數為4096和3308。對實驗樣本特異性的peak寬度進行統計,80%的peak寬度在200bp左右,見圖3A。同時對基因功能區peak分布進行分析,發現PAIP1主要結合位點位于 3’UTR、CDS和 Introns區,見圖3B。進一步對peak關聯的gene用京都基因和基因組百科全書(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析,以確定與 PAIP1 蛋白結合的RNA參與的主要生化代謝途徑、信號傳導途徑、分子功能、生物過程和細胞組分。分析結果顯示,見圖3C,PAIP1結合的mRNA分子主要參與了焦點粘連、剪切體、RNA轉運、蛋白多糖在癌癥中的作用、HIF-1信號通路、PI3K-Akt信號通路等功能。
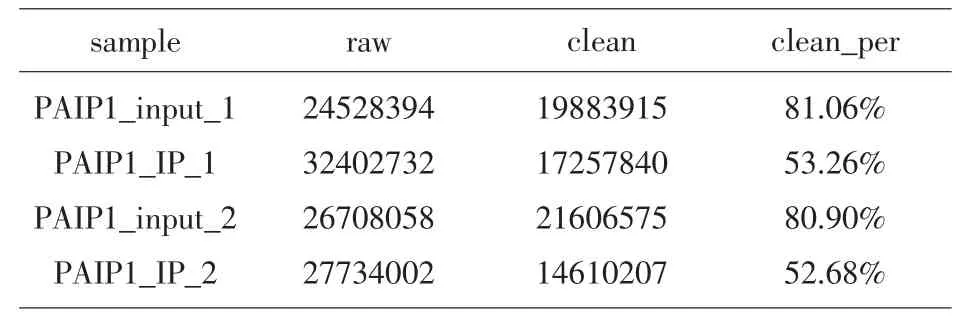
表1 A原始reads數據統計
3 討論
RNA 結合蛋白 (RNA binding proteins,RBPs)的定義為能結合RNA的蛋白質,與基因轉錄后調控有關,包括參與RNA的可變剪接,RNA的空間折疊等,從而導致RNA的表達量的變化,因此,精確確定RBPs在RNA上的結合位置和結合特點至關重要[12,13]。目前研究RBP結合位點的主要方法有CLIP-seq,但是CLIP-seq也存在有缺陷,如實驗過程復雜,成功率較低,文庫豐富度不夠等。與CLIP-seq類似的方法RIP-seq,實驗步驟相對簡單,而且能獲得和CLIP-seq比較類似的實驗結果[14]。最近有研究報道,使用DO-RIP-seq技術,也可以成功地獲得RBPs的結合位點,這為RIP-seq技術分析RBPs的結合位點提供了方案和依據[15]。

表1 B有效reads數據比對統計
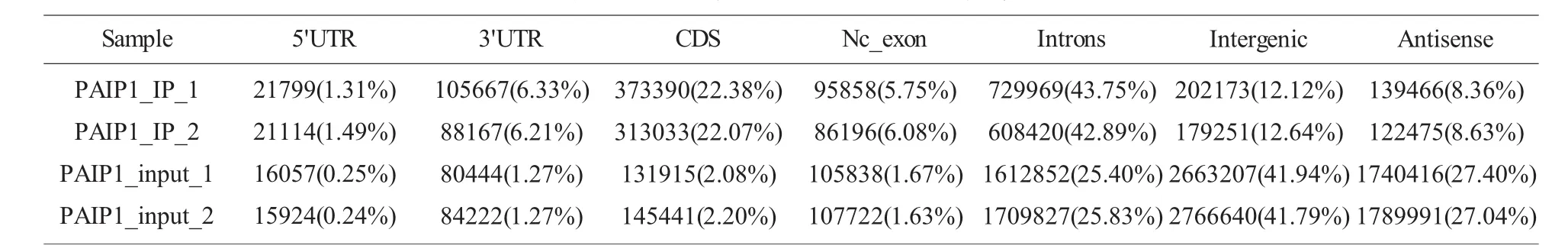
表1 C reads在基因組不同區域上的分布

圖3 結合峰相關基因聚類分析
本研究在Hela細胞中過表達PAIP1進行iRIP-seq,發現PAIP1具有廣泛的RNA結合活性,結合顯著富集在CDS和Intron區域,表明PAIP1可能具有調控mRNA可變剪接功能。iRIP-seq數據綜合KEGG分析表明,PAIP1結合的RNA分子主要參與了焦點粘連、剪切體、RNA轉運、蛋白多糖在癌癥中的作用、HIF-1信號通路、PI3K-Akt信號通路。本研究對于PAIP1蛋白質的功能和在癌癥中的作用提供了基礎大數據和新的研究方向。
PAIP1以前被報道的功能是調控蛋白質翻譯[6,16],我們的研究結果顯示PAIP1結合的基因和蛋白質翻譯起始、延伸、終止相關,表明PAIP1具有新的調控功能,通過調控翻譯相關基因的表達從而進一步調控蛋白質翻譯。PAIP1作為RNA結合蛋白的功能未見報道,本研究也明確了PAIP1的新功能,即RNA結合功能,進一步證實了Sonenberg,N等的設想[6]。本研究將為PAIP1蛋白質的功能及機制研究提供新的研究方向。
本研究通過高通量測序手段尋找PAIP1結合靶標,發現PAIP1結合的分子參與了腫瘤相關分子通路,但是,這些結果是通過生物信息學分析得出的,其具體功能還未得到驗證,為了進一步明確PAIP1的作用,下一步將開展功能驗證實驗從而驗證這些結果。
文章分析得出:PAIP1結合的mRNA分子主要參與了焦點粘連、剪切體、RNA轉運、蛋白多糖在癌癥中的作用、HIF-1信號通路、PI3K-Akt信號通路等功能。這些猜想是基于信息學分析結果推論得到的,需在討論中提及后續的驗證實驗,如果條件允許,請進行下一步的驗證實驗已驗證該猜想。

