苯丙氨酸羥化酶基因突變的生物信息學分析*
來怡君,陳 紅,章印紅,張 杰,朱寶生,唐新華
(1.昆明理工大學醫學院,昆明 650500;2.昆明理工大學附屬醫院/云南省 第一人民醫院遺傳診斷中心,昆明 650032)
苯丙酮尿癥(phenylketonuria,PKU)是一種常見的氨基酸代謝障礙性疾病,屬常染色體隱性遺傳病,臨床表現為智力低下、自閉癥、運動障礙等[1]。該病主要因肝臟中的苯丙氨酸羥化酶(phenylalanine hydroxylase,PAH)缺乏或者活性不足導致苯丙氨酸不能正常代謝為酪氨酸而產生[2]。早在1983年PAH基因就被成功分離和克隆,并被公認為是PKU的致病基因[3]。近幾年,隨著基因組和外顯子組測序的發展,很多新發突變,特別是一些癥狀相對較輕的突變也逐漸被檢測出來[4]。截至目前,已經在患有PKU的患者中鑒定了約1 000種不同的PAH基因突變體[5]。并且PAH基因具有突變異質性,不同種族與地區的突變位點及分布有較大差異[6]。本研究采用云南省漢族PKU患者中發現的6例PAH基因新發突變為材料,從生物信息學角度分析PAH基因的錯義突變與蛋白結構,以及進一步可能導致的功能改變的關系,并利用生物信息學方法嘗試探討新發剪接突變的發生機制,為以后關于PKU的研究提供有價值的信息。
1 材料與方法
1.1材料 (1)6例數據庫中未收錄的PAH基因新突變見表1。(2)使用的網絡資源,包括美國國立生物技術信息中心(NCBI,網址:https://www.ncbi.nlm.nih.gov/)、Swiss-Model軟件(https://www.swissmodel.expasy.org/)、CRYP-SKIP(http://cryp-skip.img.cas.cz/)、BDGP軟件(http://www.fruitfly.org/seq_tools/splice.html)、HSF(http://www.umd.be/HSF/)等。
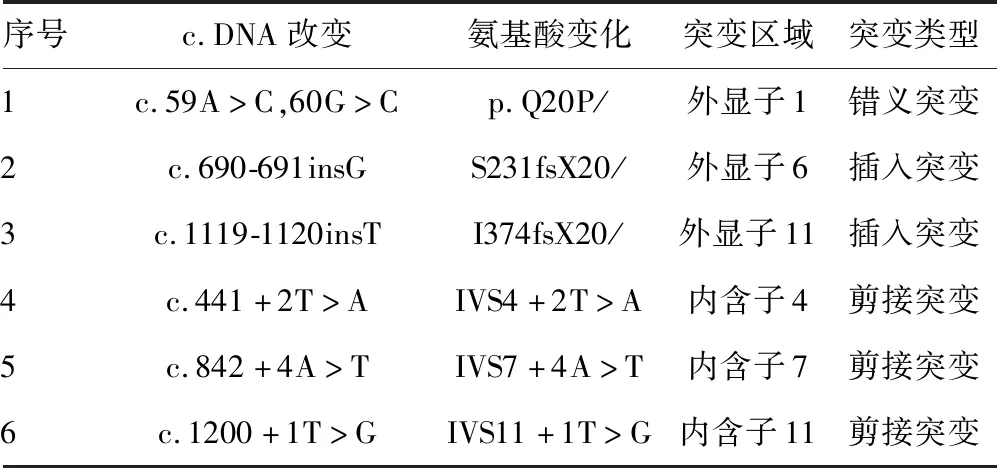
表1 云南地區PKU患者PAH基因新突變[7]
1.2方法
1.2.1獲取人類PAH基因全序列及其分析 從NCBI的GenBank中基因數據庫獲取人類PAH基因組定位和其編碼的蛋白質序列信息。
1.2.2PAH新發錯義突變及插入突變位點所在外顯子的同源建模及功能分析 利用Swiss-model在線工具對1例錯義突變和2例插入突變所在外顯子區域進行外顯子同源建模,分析基因突變對其所在外顯子的蛋白結構和功能的影響。
1.2.3PAH新發剪接突變的生物學效應分析 采用3種基于不同網絡算法的生物信息學工具分析新發現的剪接變異。其中,在CRYP-SKIP界面上直接將變異序列按其要求的格式輸入即可獲得預測結果,包括因剪接變異導致的隱蔽剪接位點激活(PCR-E)和外顯子跳躍(1-PCR-E)的可能性大小兩個結果[8]。柏克萊果蠅基因組項目(Berkeley Drosophila Genome Project,BDGP)和人類剪接查找服務器(the Human Splicing Finder,HSF)可預測可能存在的隱蔽剪接位點及基因變異對剪接位點的改變,使用時分別將野生型基因序列和變異型基因序列進行在線分析,即可獲得變異前后目標基因序列所有可能的剪接位點及分值。
2 結 果
2.1PAH結構 在GenBank中基因數據庫查找PAH基因可知:PAH基因全長79 278 bp,位于人類12號染色體長臂上(12q23.2),包含13個外顯子,12個內含子,CDS編碼452個氨基酸,在66~1 421 bp見圖1。

圖1 人類PAH基因結構
2.2PAH突變位點外顯子的同源建模及功能分析 由于本研究中的3例突變分別發生在外顯子1、外顯子6和外顯子11上,而外顯子1因其序列太短未找到同源序列,因此將外顯子1,2結合起來對外顯子1處發生的點突變(c.59A>C,60G>C)進行同源建模,并對突變后的外顯子6和外顯子11分別進行同源建模,見圖2。其中,圖2A是正常的外顯子1,2結構,圖2B是點突變(c.59A>C,60G>C)突變后的外顯子1,2預測結構,可以看出突變導致外顯子的一處β片層消失。圖3A是正常的外顯子6的空間結構,圖3B中由于(c.690-691insG),導致移碼突變S231fsX51,即該三聯密碼子下游第51位氨基酸處蛋白合成終止,致使外顯子6的空間構象發生明顯改變。外顯子11上的第1 119位和1 120位插入堿基T(c.1119-1120insG),導致移碼突變I374fsX,即374位后第20位氨基酸處蛋白合成終止,由于此處突變可能會影響外顯子12和13的合成,因此選擇從正常的外顯子10開始到外顯子13進行同源建模,同時對(c.1119-1120insG)突變后的外顯子10之后的部分進行同源建模。圖4A是正常的外顯子10,11,12,13的結構;圖4B是正常的外顯子10,11的結構;圖4C是(c.1119-1120insG)突變后外顯子10,11,12,13的結構,可以看出(c.1119-1120insG)的突變導致外顯子11三級結構部分缺失。
2.3PAH新發剪接突變的生物學效應分析 經3種生物信息學軟件綜合分析上述突變,CRYP-SKIP預測值中,PCR-E(在右邊的刻度盤上用紅色指針表示)取值介于0和1之間,較高的值表示支持隱蔽剪接位點激活,較低值則表示支持外顯子跳躍。突變預測結果顯示c.441+2T>A突變后第4外顯子5′端產生隱蔽剪接位點(PCR-E)的可能性為0.35,3′端的值接近于0,c.842+4A>T突變后第7外顯子5′端產生隱蔽剪接位點(PCR-E)的可能性為0.39,3′端的值接近于0。c.1200+1T>G突變后外顯子11中5′端產生隱蔽剪接位點(PCR-E)的可能性為0.66,3′端的值接近于0。
BDGP顯示正常PAH基因第4內含子5′端剪接位點(AGgt,g為突變處)的預測值為0.94(預測值為0~1),c.441+2T>A突變后第4內含子5′端剪接位點(AGgt,g為突變處)的預測值則為0.96(預測值為0~1),而IVS4+2T>A和IVS7+4A>T突變后BDGP預測值均消失(預測值為0)。
HSF預測中CV值高于80者具有較強剪接信號,CV值為70~80的剪接位點剪接信號較弱,少部分剪接位點的CV值為65~70[9](預測值為0~100)。預測結果顯示c.441+2T>A使第4內含子的5′剪接供體共識值(CV值)由正常的83.22變為56.39。c.842+4A>T使第7內含子的5′剪接供體CV值為由82.11變為73.32,而3′剪接受體CV值由68.35變為39.4。c.1200+1T>G突變使第11內含子的5′剪接供體CV值為由95.95變為69.12,而3′剪接受體CV值并無明顯變化。
A:正常的外顯子1,2結構;B:外顯子1處點突變突變后的預測結構
圖2外顯子1,2的同源建模結果
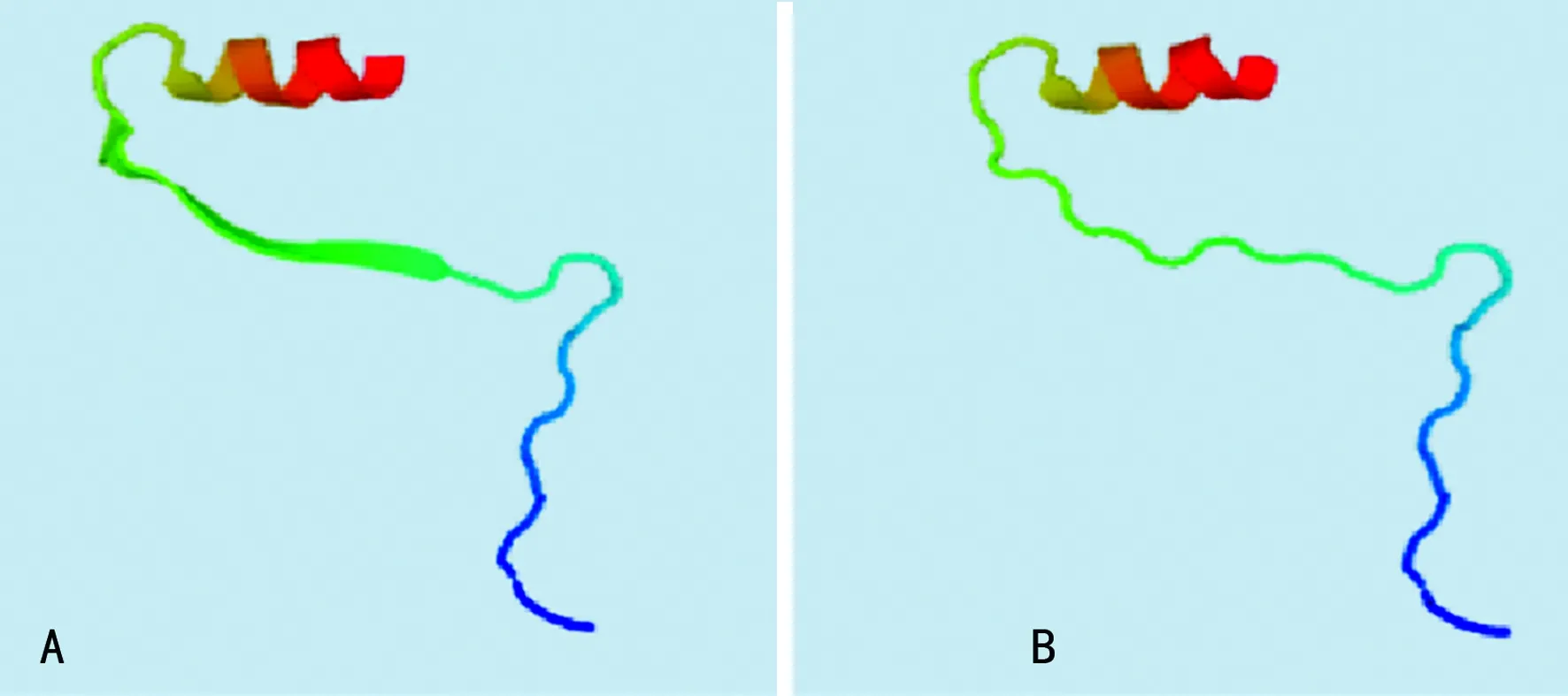
A:正常外顯子6的空間結構;B:外顯子6空間構象發生改變
圖3外顯子6的同源建模結果
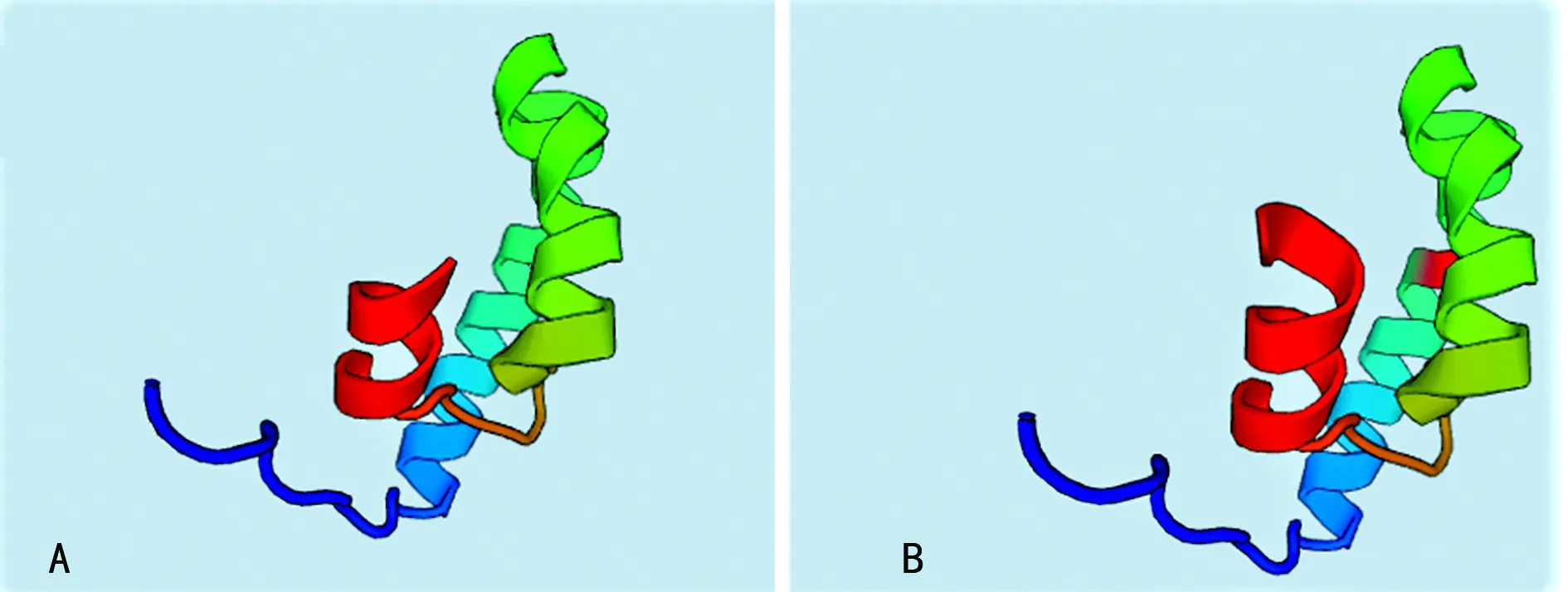
A:正常的外顯子10,11,12,13的結構;B:正常的外顯子10,11的結構;C:突變后外顯子10,11,12,13的結構
圖4外顯子10-13的同源建模
3 討 論
一般認為,PKU是由于PAH基因突變引起PAH蛋白功能損壞或喪失而導致的,PAH基因中細微的改變都會引起酶蛋白三維結構的改變,并可造成PAH活性降低甚至完全喪失,導致在臨床上產生PKU病患。而PAH蛋白的三級結構是一個同源四聚體。此前對于PKU基因型與表型關系的研究多是利用質譜技術和 X射線衍射技術構建PAH的3D晶體結構模型來進行的[10],而最新研究發現了全長哺乳動物(大鼠)PAH的第1個X射線晶體結構[11]。本研究中使用Swiss-model對1例錯義突變和2例插入突變所在的第1、第6和第11外顯子的同源建模分析中發現(c.59A>C,60G>C)突變導致外顯子1,2區域的片層結構消失,另外此突變位于外顯子與內含子的交界處,可能會嚴重影響PAH基因后續的轉錄、翻譯和蛋白折疊等[6]。(c.690-691insG)突變導致外顯子6的空間構象發生改變;而外顯子11上的(c.1119-1120insG)突變不僅影響了外顯子11的空間構象,并且嚴重影響了后續外顯子12和外顯子13的構象,這3例突變預測的結果顯示突變會直接影響PAH單體的結構完整性。而最近有研究揭露PKU相關的基因突變會對PAH同源四聚體的穩定性產生影響,造成PAH蛋白不同程度的降解,從而導致嚴重的PKU表型[12]。例如PAH基因R408W突變后導致翻譯的蛋白質極易降解,最終在患者體內或體外測不出PAH,從而導致嚴重的PKU表型,而R413P突變則導致PAH四聚體結構的高度不穩定,影響正常的單體狀態和蛋白酶的功能,臨床表現為嚴重代謝障礙型PKU。所以作者大膽推測,這3例突變甚至有可能導致突變體的穩定性受到影響,致使PAH蛋白降解,從而影響其功能。
目前,在PKU相關的PAH基因缺陷中,約13%的突變影響保守的3′和5′剪接位點,而大多數報道的剪接突變都是通過影響5′端剪接供體位點從而引起疾病的[13]。本研究中對于3種剪接突變的多種預測結果顯示,第7內含子處的c.842+4A>T突變可能導致外顯子的3′端發生了跳躍,第4內含子的c.441+2T>A突變和第11內含子處c.1200+1T>G的突變都有可能是由于外顯子的5′端影響剪接,其中第4內含子的c.441+2T>A突變可能原因是其5′端發生了跳躍。在最近一項PAH基因外顯子11處兩個致病剪接突變(c.1199+17G>A和c.1199+20G>C)的研究中發現,與5′剪接位點相結合的小核糖核蛋白顆粒U1 snRNA在與外顯子11的剪接過程中起著重要作用,而突變則會破壞U1 snRNA和內含子5′剪接位點的結合,從而影響剪接。此外還證明了通過修飾U1 snRNA可以實現對剪接突變的校正[14]。本研究中第11內含子處c.1200+1T>G突變的預測結果說明,突變可能降低了11內含子5′端隱蔽剪接位點的活性,但c.1200+1T>G突變是否同樣破壞U1 snRNA和內含子5′剪接位點的結合還不得而知,需要進一步的實驗驗證。
盡管本研究采用生物信息學方法對PAH基因突變進行了分析,但基因突變對功能的影響是一個復雜的過程,且生物信息學方法還有待提升,具體的基因突變與功能的關系及突變后的機制還要以實驗結果為準,這需要后續進一步的系統研究。

