亞硝酸還原酶基因克隆、表達與純化
魏計東,張慶芳,竇少華,于 爽,遲乃玉,王曉輝,* (1.大連大學生命科學與技術學院,遼寧大連 116622; 2.遼寧省海洋微生物工程技術研究中心,遼寧大連 116622)
?
亞硝酸還原酶基因克隆、表達與純化
魏計東1,2,張慶芳1,2,竇少華1,2,于 爽1,2,遲乃玉1,2,王曉輝1,2,*
(1.大連大學生命科學與技術學院,遼寧大連 116622; 2.遼寧省海洋微生物工程技術研究中心,遼寧大連 116622)
該研究通過聚合酶鏈反應(PCR)方法從木糖氧化產堿菌(AchromobacterxylosoxidansDL-1)基因組DNA中成功克隆含銅亞硝酸還原酶基因。PCR測序表明該基因全長1083個核苷酸,編碼360個氨基酸,預測其理論分子質量約38.924 kDa,等電點(pI)4.83,命名為CuNiR(Genbank登錄號KX674378)。結構域分析該蛋白編碼區包含信號肽,1個銅離子結合位點,1個氧化還原酶催化域。將CuNiR基因構建到pET22b載體,并轉化至大腸桿菌(Escherichiacoli,E.coli)BL21(DE3)中誘導表達,十二烷基硫酸鈉-聚丙烯酰胺凝膠電泳(SDS-PAGE)檢測顯示目的蛋白可溶表達。利用鎳柱親和層析純化重組蛋白,酶活檢測顯示比活力為123.82 U/mg,為后期含銅亞硝酸還原酶(CuNiR)的生化性質表征奠定理論基礎。
木糖氧化產堿菌DL-1,含銅亞硝酸還原酶(CuNiR),克隆,表達,純化
自然界中氮素循環是各個元素循環的中心,幾乎所有有機體都需要有機氮作為細胞的必需成分,氮元素成為地球表面生物量增長的限制性因素[1]。在氮素循環中,固氮作用與脫氮作用是兩個十分重要的環節,生物圈中氮素的引入主要是通過固氮作用,而最終實現對大氣中蘊藏豐富的氮素的利用。與此同時,生物圈中氮素的釋放則離不開脫氮作用。脫氮作用是指通過一系列的厭氧電子傳遞反應把硝酸經由亞硝酸、一氧化氮、氧化亞氮等中間體還原為氮氣或氨的過程[2]。反硝化作用主要由反硝化細菌[3]在微氧或厭氧條件下完成。
亞硝酸還原酶是催化亞硝酸鹽還原為一氧化氮(NO)的酶。反硝化途徑[4]由一系列的還原反應組成,每一步都有特定的酶來催化完成。而亞硝酸還原酶最終實現了將溶液態的亞硝酸向氣態NO的相變,直接導致了氮素從陸表環境中的流失,可以認為是反硝化途徑中最為關鍵的一步[5];其次,利用亞硝酸還原酶降解發酵食品中的亞硝酸鹽是應對亞硝酸鹽污染的根本對策。對許多細菌中的亞硝酸還原酶分析表明它有兩種類型:以細胞色素cd1(cd1NiR)和金屬Cu(CuNiR)為輔基,其中細胞色素cd1NiR更為經常的發生[6]。兩種類型的亞硝酸還原酶在結構和氧化還原中心的差異很大,但催化相同的單電子亞硝酸還原反應。第一個含銅亞硝酸還原酶是在1955年,由Iwasaki[7]等人于自名古屋大學的土壤中分離的Alcaligenes(現在稱為Achromobacter)xylosoxidans NCIB11015菌株中發現的。亞硝酸還原酶中CuNiR含量相對較少,但是表現了更廣泛的環境、地域、種群等分布上的多樣性。迄今為止,已在十幾種具有反硝化作用的微生物中明確鑒定出CuNiR的存在[8]。最新研究熱點主要集中于亞硝酸還原酶催化作用的分子機理,具體涉及電子在兩個類銅位點之間的傳遞,以及CuNiR結合于類型2銅位點(T2Cu)的亞硝酸還原反應發生情況[9];而關于該酶的基因水平研究較少[10]。
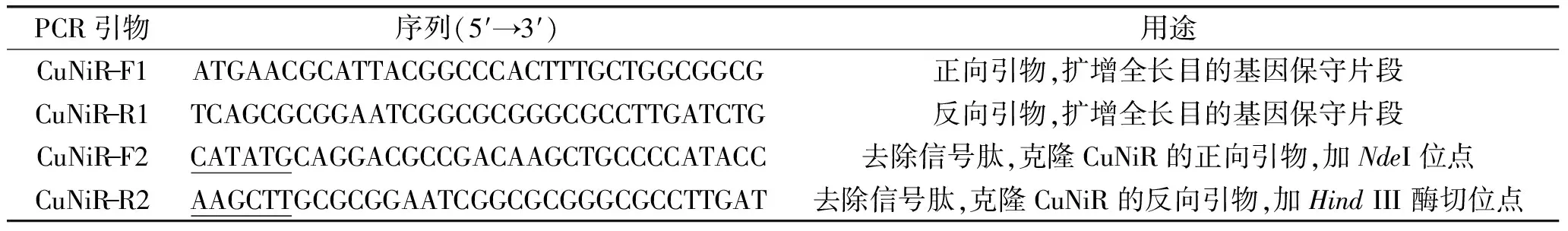
表1 PCR引物序列Table 1 PCR primer sequences
本文在分子水平上實現編碼含銅亞硝酸還原酶基因的克隆、表達及純化。含銅亞硝酸還原酶基因CuNiR的克隆、表達以及酶的純化是重組基因工程菌生產亞硝酸還原酶(NiR)產業化應用的重要研究基礎。通過對CuNiR基因克隆和表達的研究,有助于我們了解亞硝酸還原酶(NiR)基因結構與表達產物之間的關系,并為根據不同應用目的進行亞硝酸還原酶(NiR)分子工程化的改造奠定基礎。
1 材料與方法
1.1 材料與儀器
木糖氧化產堿菌(AchromobacterxylosoxidansDL-1) 由本實驗室分離并保存;大腸埃希氏菌(Escherichiacoli,E.coli)JM109和BL21(DE3) 為本實驗室保存,分別為基因克隆和表達宿主菌;表達載體pET-22b(+) 購自Novagen公司;引物合成和基因測序 由寶生物工程(大連)有限公司完成;十二烷基硫酸鈉(SDS)、瓊脂糖、Tris Promega公司;LA Taq DNA聚合酶、DNA凝膠回收試劑盒、質粒純化試劑盒、低分子量蛋白Marker 寶生物(大連)工程有限公司;蛋白濃度測定試劑盒 碧云天公司;LB培養基(1000 mL):胰蛋白胨10 g,酵母提取物 5 g,NaCl 10 g,pH7.0;LB固體培養基需向液體培養基內加入2%(w/v)瓊脂;LA培養基:LB液體或固體培養基中加入終濃度100 μg/mL氨芐青霉素。
PCR擴增儀 TaKaRa公司;GS-158低溫臺式離心機 BECKMAN公司;J21-M高速冷凍離心機 BECKMAN公司;臺式冷凍離心機 Eppendorf公司;CH1015超級恒溫水浴槽 上海恒平儀器廠;Inazge MlasterRVDS電泳成像系統 Parmacia Biotech公司;DYY-III形電泳槽 北京六一儀器廠;Milli-Q Academic超純水器;pH計 BECKMAN公司。
1.2 實驗方法
1.2.1CuNiR基因的克隆與序列分析 從平板上挑取菌株AchromobacterxylosoxidansDL-1單菌落,接種于50 mL LB液體培養基,37 ℃、160 r/min振蕩培養12 h,12000 r/min離心10 min收集菌體,按細菌基因組DNA提取試劑盒操作步驟提取基因組DNA。
以基因組DNA為模板,根據Kataoka K等[11]的方法設計含銅亞硝酸還原酶基因序列(AB013078)的兩對引物,見表1。PCR反應條件為:94 ℃ 2 min,1個循環;94 ℃ 30 s,55 ℃ 45 s,68 ℃ 7 min,30個循環;68 ℃ 15 min,1個循環。PCR擴增產物經1%(w/v)瓊脂糖電泳檢測,將PCR產物大小正確的克隆送寶生物工程(大連)有限公司進行測序。采用Vector NTI Suite軟件進行開放閱讀框(open reading frames,ORF)分析。通過NCBI上的本地序列基本搜索工具(basic local alignment search tool,BLAST)分析序列同源性。采用在線的簡單模塊構架搜索工具(simple modular architecture research tool,SMART)進行序列結構域分析。
1.2.2CuNiR基因的表達與純化 按實驗方法構建CuNiR基因的表達載體,熱轉化至E.coliCompetent Cell JM109中,涂布平板培養菌體。隨機挑取的12個單菌落進行PCR鑒定,鑒定正確的質粒分別命名為CuNiR-P-1和CuNiR-P-2,1%瓊脂糖凝膠電泳檢測。CuNiR基因(去除信號肽)和原核表達載體pET22b(+)的PCR產物經NdeI/HindⅢ雙酶切后用T4 DNA 連接酶連接,構建重組原核表達載體pET22b-CuNiR,并轉化至大腸桿菌感受態細胞E.coliBL21(DE3)中。雙酶切鑒定正確的陽性克隆接入含氨芐的LB培養基中,37 ℃培養12 h。按1%的接種量接種到含氨芐的LB培養基中。當OD600 nm達到0.6~0.8左右,加入1.0 mmol/L的IPTG,37 ℃,100 r/min誘導2 h。4 ℃,6000×g離心10 min,收集菌體,用適量PBS緩沖液(pH7.0)洗滌菌體并重懸。離心管置于冰上超聲(400 W)破碎三次,每次10 s,間隔1 min,直至菌體透明。4 ℃,8000×g離心20 min,收集上清液,即為粗蛋白。粗蛋白用Ni2+-NTA柱(Novagen)進行親和層析純化[12],洗脫的蛋白液4 ℃保存。蛋白樣品經變性聚丙烯酰胺凝膠電泳(SDS-PAGE)檢測,其中濃縮膠濃度5%,分離膠濃度12%,用考馬斯亮藍R-250染色。低分子量蛋白Marker用于SDS-PAGE。
1.2.3 蛋白質濃度測定 蛋白濃度通過Bradford蛋白濃度測定試劑盒(Bradford Protein Assay Kit)測定。
1.2.4CuNiR酶活性的測定CuNiR活性測定采用Na2S2O4-MV法[13]。測定酶活反應體系250 μL:0.1 mol/L磷酸鹽緩沖液(pH7.5)125 μL,0.1 mol/L NaNO212.5 μL,0.1 mol/L MV 7.5 μL,0.1 mol/L Na2S2O440 μL,酶液50 μL。30 ℃水浴中反應10 min,劇烈振蕩終止反應(以磷酸緩沖液為空白)。取10 μL加格利斯試劑顯色,在波長540 nm處比色法測定亞硝酸鹽的變化。
硝酸還原酶活力單位通過在25 ℃下,每分鐘還原1 μmol亞硝酸鹽所消耗的酶量來表示。比活力用1 mg蛋白質中酶的活力單位數來表示。
2 結果與分析
2.1CuNiR基因的擴增與序列分析
以AchromobacterxylosoxidansDL-1基因組DNA為模板,PCR擴增得到一條長度約1083 bp的DNA片段(圖1)。DNA測序結果表明:該基因包含1083個核苷酸構成的開放閱讀框架,命名為CuNiR,該基因編碼360個氨基酸,理論分子量38.924 kDa,等電點為4.83。本文首次報道從AchromobacterxylosoxidansDL-1中克隆亞硝酸還原酶基因。Blast比對分析該序列同已發表的A.xylosoxidansAU1011亞硝酸還原酶(GeneBank No. AB013078)氨基酸同源性達96%。但未見比對菌株中該蛋白表達純化研究。結構域分析該基因包含一段24個氨基酸構成的信號肽,1個銅離子結合結構域和1個氧化還原酶催化域。

圖1 含銅亞硝酸還原酶基因的PCR擴增Fig.1 Agarose gel electrophoresis of the CuNiR gene through PCR注:M:DNA Marker DL2000;1:CuNiR gene。
2.2CuNiR基因測序載體的鑒定
將以CuNiR-F2/R2為引物PCR擴增得到的目的片斷CuNiR基因克隆至pMD19-T simple載體中,構建了重組質粒pMD19-T simple/CuNiR,通過1%瓊脂糖凝膠電泳檢測。由圖2可見在約3.7 kbp處得到4條明亮清晰DNA條帶,其中去除信號肽CuNiR基因片段大小約1011 bp,pMD19-T simple載體大小為2692 bp,結果顯示目的片斷已經成功克隆至質粒載體中,成功構建重組質粒pMD19-T simple/CuNiR,進一步測序驗證正確的目的基因進行后續分析。

圖2 重組質粒pMD19-T simple/CuNiR的PCR電泳Fig.2 Agarose gel electrophoresis of recombinant plasmid pMD19-T simple/CuNiR注:M:Lambda DNA/Hind III Marker; 1~4:重組質粒pMD19-T simple/CuNiR。
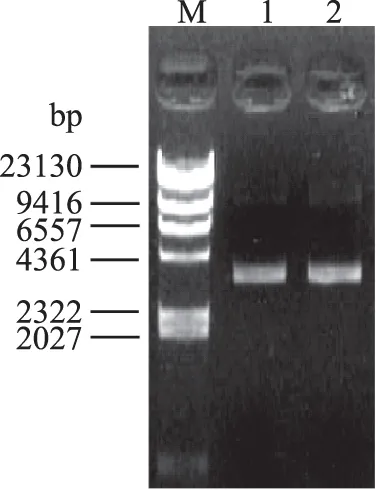
圖3 重組質粒pET-22b-CuNiR瓊脂糖凝膠電泳Fig.3 Agarose gel electrophoresis of pET-22b/CuNiR plasmid注:M:λ-Hind Ⅲ DNA Marker;1:CuNiR-P-1;2:CuNiR-P-2。
2.3CuNiR基因表達載體的構建與鑒定
提取重組表達質粒CuNiR-P-1和CuNiR-P-2進行1%瓊脂糖凝膠電泳檢測。結果如圖3所示,重組表達質粒與預期大小一致。
將質粒CuNiR-P-1和CuNiR-P-2分別用限制性內切酶NdeⅠ/HindⅢ進行雙酶切,取20 μL酶解產物上樣進行1%瓊脂糖凝膠電泳檢測,結果如圖4所示。從圖4中可以看出,兩個重組質粒酶解產物片段大小分別約為1 kbp和5.3 kbp左右,說明目的CuNiR基因已成功的亞克隆至表達載體pET-22b(+)中。
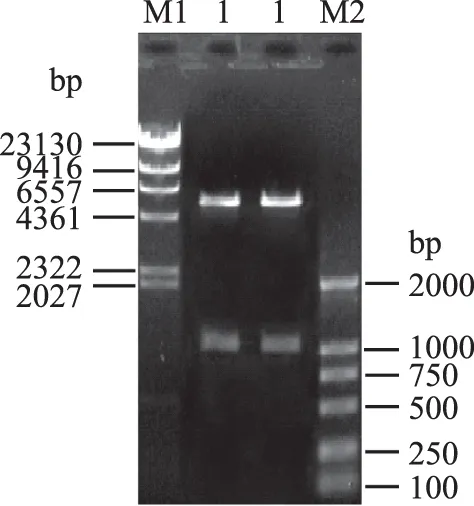
圖4 質粒CuNiR-P-1和CuNiR-P-2酶切產物回收Fig. 4 Agarose gel electrophoresis of recombinant expression plasmid pET-22b(+)/CuNiR digested with restriction enzyme注:M1:λ-Hind Ⅲ DNA Marker; 1:CuNiR-P-1 Nde I/HindⅢ酶切; 2:CuNiR-P-2 Nde I/HindⅢ酶切; M2:DNA Marker DL2000。
2.4CuNiR目的蛋白誘導表達與檢測
將經鑒定正確的原核重組表達質粒載體pET-22b(+)/CuNiR(CuNiR-P-1)轉化至表達宿主菌E.coliBL21(DE3)中誘導表達。菌體裂解物進行SDS-PAGE電泳,由圖5可知,泳道1~3空表達載體pET-22b(+)宿主菌BL21(DE3)未見目的蛋白,而泳道4和5約36 kDa位置處出現特異條帶,與預期表達的相對分子質量相符,說明目的蛋白CuNiR在宿主菌E.coliBL21(DE3)中成功表達,且在全細胞和細胞漿的可溶性蛋白中檢測到目的蛋白。由此可知,目的蛋白CuNiR在大腸桿菌中可溶表達。
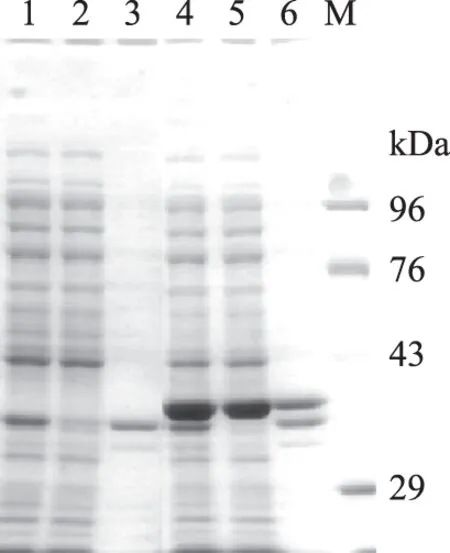
圖5 目的蛋白CuNiR的SDS-PAGE檢測結果Fig.5 SDS-PAGE analysis of CuNiR protein注:1:pET-22b(+)全細胞;2:pET-22b(+)上清; 3:pET-22b(+)沉淀;4:pET-22b(+)/CuNiR全細胞; 5:pET-22b(+)/CuNiR上清;6:pET-22b(+)/CuNiR沉淀; M:TakaRa Protein Marker(Low)。
2.5CuNiR的純化與酶活檢測
在搖瓶水平,1 L發酵液經1 mmol/L IPTG誘導2 h離心收集菌體,超聲破碎后得到CuNiR上清液蛋白。破碎上清液經Ni-NTA純化重組蛋白,SDS-PAGE檢測洗脫收集液,如圖6所示。目標蛋白被120、300 mmol/L咪唑溶液洗下收集,獲取純度較高且濃度較大的目標蛋白。純化后CuNiR總蛋白含量為96 mg,比活力為123.82 U/mg,總活力為11887.08 U。
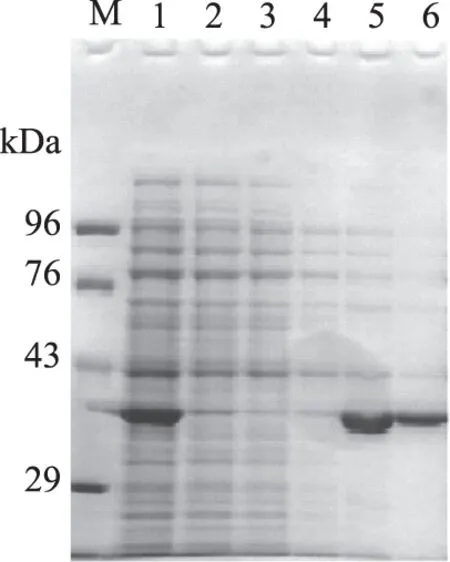
圖6 重組含銅亞硝酸還原酶CuNiR的分離純化Fig.6 Purification of the recomninant CuNiR注:M:低分子量蛋白Marker; 1:E.coli BL21(DE3)/pET22b-CuNiR細胞裂解液; 2:Ni-NTA純化后CuNiR過柱后洗脫液; 3:磷酸緩沖液(pH7.5)洗脫液;4:20 mmol/L咪唑洗脫液; 5:120 mmol/L咪唑洗脫液;6:300 mmol/L咪唑洗脫液。
3 結論
亞硝酸還原酶大多數為胞內酶,受分離純化技術的影響,該酶的應用受到了極大限制[14]。本研究所用的pET系列表達載體是有史以來在E.coli中克隆表達重組蛋白功能最強大的系統,保證外源目的基因成功表達與純化。本實驗中我們選擇了帶有NdeⅠ酶切位點的表達載體pET-22b(+),該表達載體多克隆位點C端有一個能編碼6個組氨酸的融合標簽His-Tag,6×His通常不影響表達產物的生物學活性,因而不必通過酶水解獲得目的蛋白,使表達的整個過程更為簡單,同時該融合標簽可用于目的蛋白的檢測和純化。在融合標簽His-Tag后還相連一個終止密碼子TGA,它保證了目的蛋白和His-Tag的完整表達[15-16]。截至目前文獻報道亞硝酸還原酶主要集中于甜菜、小麥、菠菜等植物中[17-18],而微生物亞硝酸還原酶報道較少[19-20]。本研究將含銅亞硝酸還原酶基因成功克隆并構建表達載體pET22b,再轉化至大腸桿菌(Escherichiacoli,E.coli)BL21(DE3)中誘導表達,目的蛋白經SDS-PAGE檢測顯示為可溶表達,隨后利用鎳柱親和層析純化蛋白,酶比活可達123.82 U/mg,為下一步酶學性質研究及工業化生產奠定基礎。
[1]Orellana L H,Rodriguez-R L M,Higgins S,et al. Detecting nitrous oxide reductase(NosZ)genes in soil metagenomes:method development and implications for the nitrogen cycle[J]. Mbio,2014,5(3):191-194.
[2]Allen J W,Barker P D,Daltrop O,et al. Why isn’t ‘standard’ heme good enough for c-type and d1-type cytochromes[J]. Dalton Transactions,2005,21(21):3410-3418.
[3]Sharma S,Szele Z,Schilling R,et al. Influence of freeze-thaw stress on the structure and function of microbial communities and denitrifying populations in soil[J]. Journal of Experimental Medicine,2006,72(3):2148-2154.
[4]Antonyuk S V,Strange R W,Sawers G,et al. Atomic resolution structures of resting-state,substrate-and product-complexed Cu-nitrite reductase provide insight into catalytic mechanism[J]. Proceedings of the National Academy of Sciences,2005,102(34):41-46.
[5]Hallin S,Lindgren P E. PCR Detection of genes encoding nitrite reductase in denitrifying bacteria PCR detection of genes encoding nitrite reductase in denitrifying bacteria[J]. Applied and Environmental Microbiology,1999,65(4):1652-1657.
[6]Tsuneda S,Miyauchi R,Ohno T,et al. Characterization of denitrifying polyphosphate-accumulating organisms in activated sludge based on nitrite reductase gene[J]. Journal of Bioscience and Bioengineering,2005,99(4):403-408.
[7]Yamaguchi K,Deligeer,Nakamura N,et al. Isolation and characterization of two distinct azurins from Alcaligenes xylosoxidans subsp. xylosoxidans NCIB11015 or GIFU1051[J]. Chemistry Letters,1995,89(5):353-354.
[8]Moura I,Moura J J. Structural aspects of denitrifying enzymes[J]. Current Opinion in Chemical Biology,2001,5(2):168-175.
[9]Bykov D,Neese F. Six-electron reduction of nitrite to ammonia by cytochrome c nitrite reductase:insights from density functional theory studies[J]. Inorganic Chemistry,2015,54(19):3-16.
[10]Suzuki M,Hirai T,Arai H,et al. Purification,characterization,and gene cloning of thermophilic cytochrome cd1 nitrite reductase from Hydrogenobacter thermophilus TK-6[J]. Journal of Bioscience & Bioengineering,2006,101(5):391-397.
[11]Kataoka K,Furusawa H,Takagi K,et al. Functional analysis of conserved aspartate and histidine residues located around the type 2 copper site of copper-containing nitrite reductase[J]. Journal of Biochemistry,2000,127(2):345-348.
[12]李美玉,張慶芳,王曉輝. 幾丁質結合蛋白基因克隆、表達與純化[J]. 中國釀造,2015(11):41-46.
[13]Rosa M. Purification and characterisation of a possible assimilatory nitrite reductase from the halophile archaeon Haloferax mediterranei[J]. FEMS Microbiology Letters,2001,196(2):113-118.
[14]Treusch A H,Leininger S,Kletzin A,et al. Novel genes for nitrite reductase and Amo-related proteins indicate a role of uncultivated mesophilic crenarchaeota in nitrogen cycling[J]. Environmental Microbiology,2005,7(12):85-95.
[15]Ai-Jia J I,Ning X B. Construction and expression of Prokaryotic expression Vector pET28a-EGFP[J]. Journal of Microbiology,2011,31(4):69-73.
[16]Yuan Z G,Zhang J P,Chu Y W,et al. Expression of target gene in eukaryotic cells driven by prokaryotic T7 promoter and its RNA polymerase][J]. Chinese Journal of Biotechnology,2005,21(2):182-186.
[17]杜永成,王玉波,范文婷,等. 不同氮素水平對甜菜硝酸還原酶和亞硝酸還原酶活性的影響[J]. 植物營養與肥料學報,2012,18(3):717-723.
[18]Rajasekhar V K,Oelmüller R. Regulation of induction of nitrate reductase and nitrite reductase in higher plants[J]. Physiologia Plantarum,2006,71(4):517-521.
[19]Prudêncio M,Eady R R,Sawers G. The Blue Copper-Containing Nitrite Reductase from Alcaligenes xylosoxidans:Cloning of the nirA Gene and Characterization,of the Recombinant Enzyme[J]. Journal of Bacteriology,1999,181(8):2323-2329.
[20]Ho W H,Ooi B L. Cytoplasmic expression of the Achromobacter xylosoxidans blue copper nitrite reductase in Escherichia coli and characterisation of the recombinant protein[J]. Protein Expression & Purification,2003,32(2):288-292.
Cloning,expression and purification of copper nitrite reductase
WEI Ji-dong1,2,ZHANG Qing-fang1,2,DOU Shao-hua1,2,YU Shuang1,2,CHI Nai-yu1,2,WANG Xiao-hui1,2,*
(1.School of Life Science and Biotechnology,Dalian University,Dalian 116622,China; 2.Liaoning Technology of Marine Microbiological Engineering Research Center,Dalian 116622,China)
In this study,a novel copper nitrite reductase gene was cloned fromAchromobacterxylosoxidansDL-1 by using a PCR protocol. The gene contained a 1083 bp open reading frame encoding a 360 amino acid protein with an estimated molecular mass of 38.924 kDa and isoelectronic point(pI)of 4.83,namedCuNiR(Genebank no. KX674378). Based on domains analysis,CuNiRhad a signal peptide,a copper binding site classified as Cu-oxidase-3,and one nitrite reductase catalytic domain(Cu-oxidase). A recombinant plasmid,pET22b-CuNiRwas constructed and transformed intoE.coliBL21(DE3). The cells were induced by addition of IPTG.CuNiRhad been successfully expressed by the analysis of sodium dodecyl sulphate-polyacrylamide gel electrophoresis(SDS-PAGE). The recombinant protein was purified by Ni-NTA resin,and the specific activity was 123.82 U/mg,which lay the theoretical basis for biochemical characterization of the copper nitrite reductase.
AlcaligenesxylosoxidansDL-1;copper nitrite reductase;cloning;expression;purification
2016-12-14
魏計東(1991-),男,碩士研究生,研究方向:微生物與酶工程,E-mail:weijidongsk@163.com。
*通訊作者:王曉輝(1981-),女,博士,副教授,研究方向:微生物與酶工程,E-mail:wangxiaohui@dlu.edu.cn。
國家自然科學基金項目(31500039)。
TS201.2
A
1002-0306(2017)14-0101-05
10.13386/j.issn1002-0306.2017.14.020

