16S核糖體DNA Amplicon測序法分析2型糖尿病患者口腔微生物多樣性
李宗澤,趙 澤,張文菲,陶 榮,孫雪飛,田 宇
16S核糖體DNA Amplicon測序法分析2型糖尿病患者口腔微生物多樣性
李宗澤,趙 澤,張文菲,陶 榮,孫雪飛,田 宇
目的 探討2型糖尿病患者口腔微生物多樣性及結構的差異。方法 采集23例2型糖尿病患者與23名正常人的唾液與齦上菌斑樣本,提取口腔菌群的總DNA,利用16S核糖體DNA(16S ribosome DNA,16SrDNA)Amplicon測序法進行測序,使用Pandaseq、Qiime等軟件分析菌群多樣性及結構。結果 2型糖尿病患者和正常人口腔菌群豐度及優勢菌屬差異無統計學意義(P>0.05)。2型糖尿病患者樣本中布雷德菌屬、瘤胃菌科、幽門螺桿菌科豐度較高,正常人樣本中韋榮菌科、Selenomonadales目、Negativicutes綱、月形單胞菌屬、丁酸弧菌屬、Anaeroglobus屬、Candidatus-Saccharibacteria門、Saccharibacteria_genera_incertae_sedis屬、諾卡菌科、氣微菌屬豐度較高,差異有統計學意義(P<0.05)。結論 用16SrDNA Amplicon測序法分析2型糖尿病患者與正常人口腔菌群結構具有相似性,幽門螺桿菌與2型糖尿病之間的關系還有待于進一步的研究。
2型糖尿病;口腔菌群;16S核糖體DNA
2型糖尿病又稱非胰島素依賴型糖尿病,占糖尿病患者90%左右。近幾十年來,在飲食結構上的變化及運動量的減少等多種因素影響下,2型糖尿病的發病率呈上升趨勢,發病年齡呈年輕化趨勢[1]。2型糖尿病作為與遺傳因素和環境因素有關的一種影響全身多器官的慢性內分泌代謝疾病,常潛在發病,長期血糖升高,機體代謝紊亂,導致全身微循環障礙,使得包括口腔在內的全身器官發生并發癥。而口腔作為人體的器官之一,在發生疾病時,會影響整個人體。口腔疾病與全身疾病兩者相互影響,而口腔微生物的失調可誘發多種口腔疾病,且口腔微生物與全身健康關系密切[2]。
口腔微生物多樣性研究,初期多采取人工培養的方法,但是許多口腔微生物不能采取原有的方法進行培養,能夠培養的微生物種類可能不足一半[3],致使對口腔中的微生物多樣性認識不足。隨著測序技術的快速發展,出現了16S核糖體DNA(16S ribosome DNA,16SrDNA)分析技術。測序技術是對口腔微生物群體進行高通量測序,分析序列的構成來確定特定環境中口腔微生物的構成情況、基因的組成以及功能。通過不同環境下口腔微生物的構成差異分析口腔微生物與環境因素或宿主之間的關系。
本研究采用Illumina測序平臺對來自第四軍醫大學唐都醫院內分泌科的2型糖尿病患者及其家屬(與患者均為夫妻關系)口腔微生物樣本進行多樣性分析。
1 資料與方法
1.1 資料
1.1.1 研究對象 第四軍醫大學唐都醫院內分泌科的2型糖尿病患者23例及其家屬(與患者均為夫妻關系)23名,年齡30~65歲。23對受試者分為2組,2型糖尿病組與正常人組。2型糖尿病組唾液樣本與齦上菌斑樣本分別標注為A與a,正常人組唾液樣本與齦上菌斑樣本分別標注為B與b。
1.1.2 納入標準
1.1.2.1 正常人 樣本的選擇需要滿足:①近6個月未使用抗生素、可的松類激素、可刺激機體免疫系統的細胞因子、甲氨蝶呤等免疫抑制劑;②近6個月未大劑量使用益生菌(每日攝入量>108 cfu);③近7天內未使用局部抗生素(如漱口水);④人類免疫缺陷病毒、乙型肝炎病毒、丙型肝炎病毒陰性;⑤未在孕期及哺乳期的婦女;⑥無臨床長期用藥治療的慢性病,如肺、心血管、胃腸道、肝臟和腎臟的慢性功能性疾病以及腫瘤等。
1.1.2.2 2型糖尿病患者 樣本的選擇需要滿足:①近6個月未使用抗生素、可的松類激素、可刺激機體免疫系統的細胞因子、甲氨蝶呤等免疫抑制劑;②近6個月未大劑量使用益生菌(每日攝入量>108 cfu);③近7天內未使用局部抗生素(如漱口水);④人類免疫缺陷病毒、乙型肝炎病毒、丙型肝炎病毒陰性;⑤未在孕期及哺乳期的婦女;⑥無除2型糖尿病以外的需要長期用藥治療的其他慢性疾病。
1.2 方法
1.2.1 樣本采集 對所有受試者進行全口檢查。受試者采集樣本前12 h應避免飲食、刷牙及使用牙線。臨床樣本采集方法:①唾液樣本采集,本實驗所采集的樣本全部為非刺激性唾液。指導受試者讓唾液自行流入50 mL離心管內,不能咳痰,采集量為10 mL。②齦上菌斑采集,使用滅菌刮匙刮取牙齒頰、舌側齦上菌斑,置于1.5 mL的離心管內。所有樣本均在冰浴下2 h內送至實驗室,-80℃保存備用。1.2.2 DNA提取 樣本4℃冰箱解凍,加入三羥甲基氨基甲烷-乙二胺四乙酸緩沖液(pH 8.0),震動5 min使樣本充分混合于緩沖液內。使用快速純化高品質DNA提取盒(QIAamp DNA Micro Kit,GER,德國Qiagen公司)提取細菌全基因組DNA。
1.2.3 引物設計并合成 16SrDNA擴增選擇區域為V3~V4區,使用的通用引物為F341和R806。在通用引物的5′端加上適合測序的索引序列和接頭序列,完成特異性引物的設計(圖1)。
1.2.4 聚合酶鏈反應擴增、產物純化及測序 以稀釋后的基因組DNA為模板,使用預混熱啟動高保真酶試劑盒(KAPA HiFi Hotstart ReadyMix PCRkit,中國艾德科技公司)進行聚合酶鏈反應(polymerase chain reaction,PCR),確保擴增的準確性和高效性。用2%瓊脂糖凝膠電泳檢測PCR產物,并用Axy-Prep DNA凝膠回收試劑盒(美國Axygen公司)切膠回收PCR產物。回收后,利用紫外微量分光光度計(Thermo NanoDrop 2000,美國賽默飛公司)和2%瓊脂糖凝膠電泳進行質檢。測序由上海銳翌生物科技有限公司Illumina平臺來完成。通過16SrRNA Amplicon測序法進行雙端(paired-end)測序。雙端測序序列通過序列之間的重疊關系拼接成長序列,并對拼接后的序列進行質控,得到有效序列。對16SrDNA高變區序列進行測序,測序為V3~V4區;通過Pandaseq軟件[4]利用重疊關系將雙末端測序得到的成對序列拼接成一條序列,得到高變區的長序列。去除平均質量值低于20的序列;去除序列中含N堿基數超過3個的序列。
1.2.5 分析方法
1.2.5.1 可操作分類單元分析 將所有序列完全一樣的有效序列根據其豐度大小進行排序,過濾掉其中的單獨序列,在0.97相似度下進行聚類;對聚類后的序列進行嵌合體過濾,得到用于物種分類的可操作分類單元(operational taxonomic units,OTUs);單個有效序列在OTUs序列上進行比對,將能比對上的序列提取出來,得到最終比對到的序列(mapped reads)[5-6]。根據比對到的序列繪制聚類Venn圖,分析2型糖尿病患者與正常人口腔菌群的多樣性。
1.2.5.2 物種豐度分析 根據物種注釋結果,分別在門、屬分類等級對各個樣本作物種相應的柱狀圖,形成物種相對豐度柱狀圖,查看各樣本在不同分類等級上相對豐度較高的物種和比例。
1.2.5.3 單個樣本多樣性分析 單個樣本多樣性是對樣本內單個樣本中物種多樣性的分析,包括觀察到的物種指數、豐度估計值指數、香農指數、辛普森多樣性指數及系統發育多樣性指數。利用Qiime軟件[7-8]計算樣本內單個樣本物種多樣性指數值,并作出相應的稀釋曲線[9]。分別對單個樣本多樣性各個指數進行分析,2組樣本比較使用R語言wilcox.test函數,篩選不同條件下差異有統計學意義的單個樣本多樣性分析指數。
1.2.5.4 樣本間多樣性分析 樣本間多樣性分析是用來比較一對樣本在物種多樣性方面存在的差異大小。
1.2.5.4.1 UniFrac熱圖分析 UniFrac是利用系統進化的信息來比較樣本間的物種群落差異。其計算結果可以作為一種衡量樣本間多樣性的指數,它考慮了物種間的進化距離,該指數越大表示樣本間的差異越大。本研究中UniFrac結果為加權UniFrac,加權UniFrac考慮了序列的豐度。UniFrac距離分布在熱點圖上,通過對UniFrac結果的聚類,具有相似的樣本聚類在一起,反應樣本間的相似性。根據各樣本差異性的統計結果,對樣本進行聚類分析并計算樣本間距離,以判斷各樣本物種組成的相似性。
1.2.5.4.2 主坐標分析 為進一步展示樣本間物種多樣性差異,使用主坐標分析(principal coordinates analysis,PCoA)的方法展示各個樣本間的差異大小[10-12]。橫坐標表示第一主坐標,縱坐標表示第二主坐標,如果2個樣本距離較近,則表示這2個樣本的物種組成較相近。
1.2.5.4.3 多響應置換過程分析 多響應置換過程分析(multi-response permutation procedures,MRPP)是用于分析組間微生物群落結構差異的一種方法,統計量值大于0表明組間差異大于組內差異,觀察值越小表明組內差異越小,預期值越小表明組間差異越小;P<0.05為差異有統計學意義。
1.2.6 物種分析 在所有水平上通過R語言stats包kruskal.test函數找出2型糖尿病患者與正常人口腔唾液及齦上菌斑差異有統計學意義(P<0.05)的物種。
2 結果
2.1 測序數據概況與OUTs聚類 23對樣本16SrDNA高通量測序數據得到有效序列5 447 331條,平均長度419 bp,A、B、a、b樣本對應的序列條數分別為1 372 856條、1 279 076條、1 361 548條、1 433 851條。A、B、a、b樣本的測序深度為1.0,覆蓋100%樣本的微生物組。在97%相似性水平上進行聚類,共得到825個OTUs。A、B、a、b樣本包含的OTUs數目分別為599、562、727、614個;其中,A、B樣本共有的OTUs數目為519個,a、b樣本共有的OTUs數目為557個,a樣本特有的OTUs數目最多(170個)。見圖2。

圖2 OTUs Venn圖分析
2.2 2型糖尿病患者與正常人之間口腔菌群結構
2.2.1 口腔菌群結構門水平 2型糖尿病患者與正常人的口腔唾液及齦上菌斑的菌群中,擬桿菌門(Bacteroidetes)、厚壁菌門(Firmicutes)、變形菌門(Proteobacteria)、梭桿菌門(Fusobacteria)、放線菌門(Actinobacteria)為主要菌群,多達95%以上(圖3),2型糖尿病患者與正常人的口腔唾液與齦上菌斑菌群優勢物種極其相似;唾液和齦上菌斑樣本中優勢物種所占比例不相同,唾液樣本中優勢菌群所占比例由大到小為擬桿菌門>厚壁菌門>變形菌門>梭桿菌門>放線菌門,齦上菌斑樣本中優勢菌群所占比例由大到小為梭桿菌門>擬桿菌門>厚壁菌門>變形菌門>放線菌門。

圖3 2型糖尿病患者與正常人口腔菌群門水平柱狀圖
2.2.2 口腔菌群結構屬水平 2型糖尿病患者與正常人的口腔菌群中唾液樣本和齦上菌斑樣本屬水平均差異不大(圖4)。
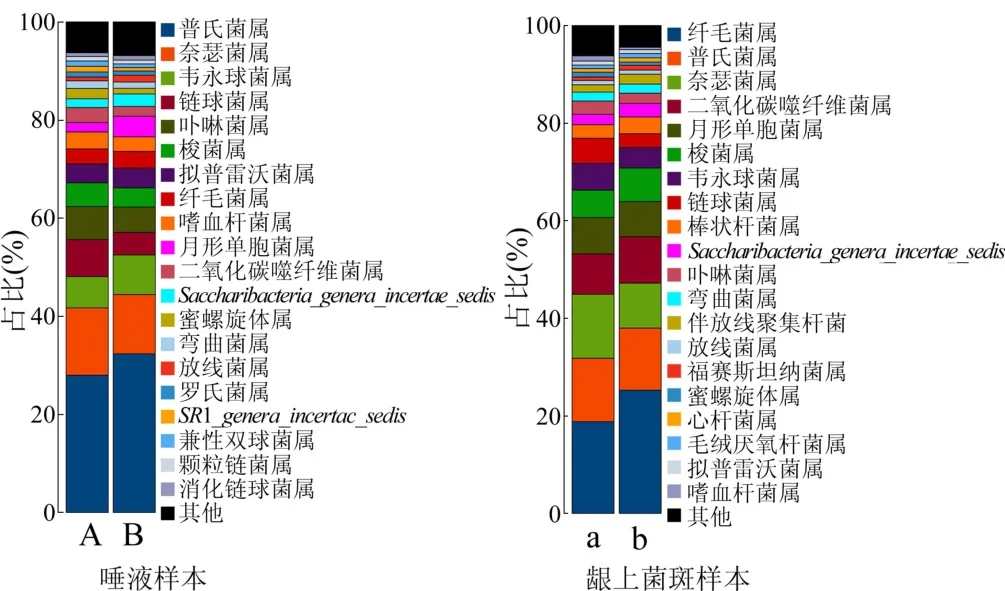
圖4 2型糖尿病患者與正常人口腔菌群屬水平柱狀圖
2型糖尿病患者與正常人的口腔菌群中唾液樣本普氏菌屬(Prevotella)、奈瑟菌屬(Neisseria)、韋永球菌屬(Veillonella)、鏈球菌屬(Streptococcus)、卟啉菌屬(Porphyromonas)、梭菌屬(Fusobacterium)、擬普雷沃菌屬(Alloprevotella)為優勢菌屬,多達80%以上,而齦上菌斑樣本纖毛菌屬(Leptortrichia)、普氏菌屬(Prevotella)、奈瑟氏菌屬(Neisseria)、二氧化碳噬纖維菌屬(Capnocytophaga)、月形單胞菌屬(Selenomonas)、梭菌屬(Fusobacterium)、韋永球菌屬(Veillonella)、鏈球菌屬(Streptococcus)為優勢菌屬。樣本優勢菌屬中,唾液樣本最多的菌屬為普氏菌屬(Prevotella),齦上菌斑樣本最多的菌屬為纖毛菌屬(Leptortrichia)。
2.2.3 單個樣本多樣性 單個樣本多樣性分析見圖5。2型糖尿病患者與正常人口腔菌群各指數差異無統計學意義(P>0.05,表1)。
2.2.4 樣本間多樣性分析
2.2.4.1 UniFrac熱圖 2型糖尿病患者與正常人口腔菌群的聚類分析見圖6。
2.2.4.2 PCoA 2型糖尿病患者與正常人唾液樣本、齦上樣本PCoA見圖7。
2.2.4.3 MRPP 2型糖尿病患者與正常人唾液樣本及齦上樣本菌斑組間MRPP差異無統計學意義(P>0.05,表2)。
2.2.5 物種分析 2型糖尿病患者樣本中布雷德菌屬(Bulleidia)、瘤胃菌科(Ruminococcaceae)、幽門螺桿菌科(Helicobacteraceae)豐度較高,正常人樣本中韋榮菌科(Veillonellaceae)、Selenomonadales目、Negativicutes綱、月形單胞菌屬(Selenomonas)、丁酸弧菌屬(Butyrivibrio)、Anaeroglobus屬、Candidatus-Saccharibacteria門、Saccharibacteria_genera_incertae_ sedis屬、諾卡菌科(Nocardioidaceae)、氣微菌屬(Aeromicrobium)豐度較高,2組物種差異有統計學意義(P<0.05,表3。)

圖5 樣本物種豐度以及多樣性指數稀釋曲線圖
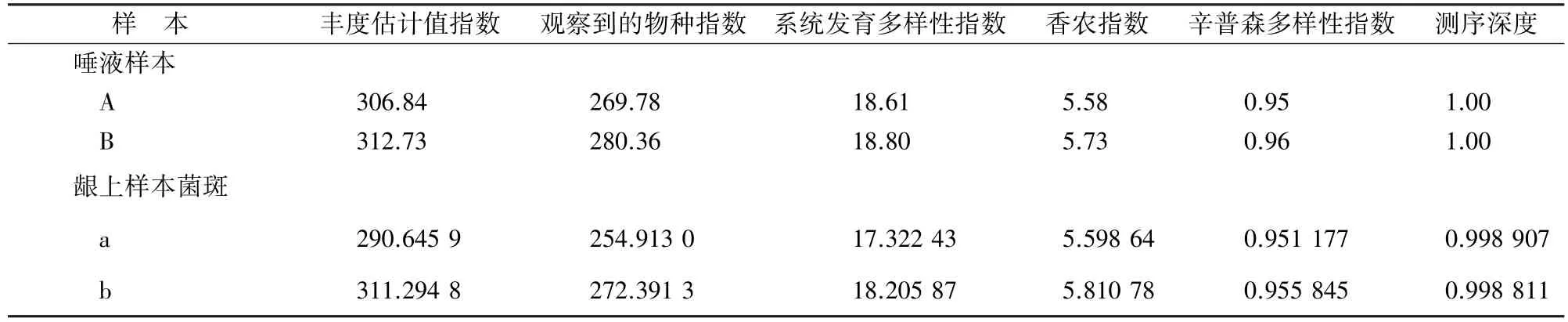
表1 2型糖尿病患者與正常人唾液樣本、齦上樣本菌斑菌群單個樣本多樣性指數檢驗

圖6 2型糖尿病患者與正常人唾液樣本、齦上樣本間多樣性聚類分析UniFrac熱圖

圖7 2型糖尿病患者與正常人唾液樣本、齦上樣本主坐標分析
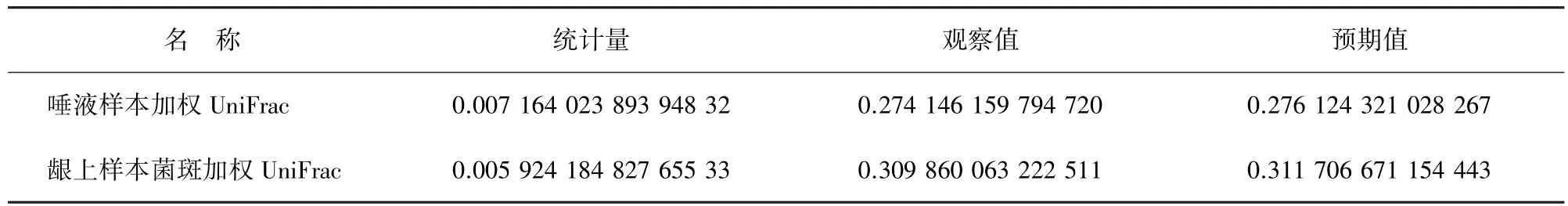
表2 2型糖尿病患者與正常人唾液及齦上菌斑組間MRPP

表3 2型糖尿病患者與正常人口腔菌群所有水平上的主要物種
3 討論
通過香農指數與辛普森多樣性指數用來估算微生物群落的單個樣本多樣性,當指數曲線趨于平緩時表示此時的測序數據量比較合理,香農指數與辛普森指多樣性指數值越大,多樣性越高。本研究在對單個樣本多樣性各個指數分析表明,2型糖尿病患者與正常人無論是在唾液還是在齦上菌斑多樣性差異均無統計學意義(P>0.05,表1)。
在樣本間多樣性分析中,熱圖分析能夠反應樣本之間的相似性,藍色越深表示物種的相似性越近,反之顏色越紅表示物種的相似性越遠。但本研究中,2型糖尿病患者的口腔菌群與正常人的口腔菌群混在其中,無法將聚類分開(圖6);組間MRPP從另外一個角度說明2型糖尿病患者的口腔菌群與正常人的口腔菌群組間差異無統計學意義(P>0.05,表2)。
本研究結論可能導致的原因:①測序方法不同,不同的測序方法會導致獲得信息的不同;②選擇樣本不同,口腔微生物是一個極其復雜的生態系統,不同個體之間的差異巨大;③在選取2型糖尿病患者的對照組時,特意選取的正常人與2型糖尿病患者為夫妻關系。做這樣選擇的目的就是為了觀察共同的生活飲食習慣對口腔菌群變化是否存在影響。本研究顯示夫妻間口腔菌群差異性較小,具有相似性。2型糖尿病患者與正常人的口腔菌群中,擬桿菌門、厚壁菌門、變形菌門、梭桿菌門、放線菌門為主要菌群,高達95%以上。其中梭桿菌門是導致人類口腔疾病的重要菌門[13];變形菌門被證實與口腔感染密切相關[14];梭菌屬易與螺旋體混合感染,會引起急性潰瘍性齦炎、急性壞死性齦炎等,當牙周炎癥加重時此菌數會大為增加、炎癥減輕時數目又會減少,是牙周疾病治療效果觀察的指標之一;卟啉菌屬是口腔菌屬又一個與牙周病相關的菌屬[15],它可侵入牙齦成纖維細胞,并在相當濃度的抗生素作用下存活[16]。
本研究中,幽門螺桿菌在2型糖尿病患者中豐度較高,與正常人比較差異有統計學意義(P<0.05,表3)。幽門螺桿菌是否與2型糖尿病存在相關性?目前,幽門螺桿菌感染與2型糖尿病之間的關系存在爭議。Jeon等[16]對782例60歲以上既往無糖尿病史、且具有致病菌數據的薩克拉門托地區人群進行為期10年的回顧性研究發現,幽門螺桿菌感染與2型糖尿病之間存在顯著關聯。Chen和Blaser[17]分析了來自美國第3次(1988—1994年)和第4次(1999—2004年)全國健康與營養調查的數據,發現幽門螺桿菌與糖化血紅蛋白水平呈正相關(P<0.01),但幽門螺桿菌感染與2型糖尿病之間無明顯關聯,提示幽門螺桿菌可能參與糖耐量受損的發生。需進一步研究口腔微生物的多樣性與2型糖尿病之間的關系,深入研究口腔微生物的作用將有助于口腔疾病與系統性疾病的預防與治療。
[1]潘長玉,金文勝.2型糖尿病流行病學[J].中華內分泌代謝雜志,2005,21(5):5S-1-5S-5.
[2]徐欣,何金枝,周學東.口腔微生物群落在口腔與全身疾病預警中的作用[J].華西口腔醫學雜志,2015,33(6): 555-560.
[3]Aas JA,Paster BJ,Stokes LN,et al.Defining the normal bacterial flora of the oral cavity[J].J Clin Microbiol,2005,43(11):5721-5732.
[4]Masella AP,Bartram AK,Truszkowski JM,et al.PANDA-seq:paired-end assembler for illumina sequences[J].BMC Bioinformatics,2012,13:31.
[5]Edgar RC,Haas BJ,Clemente JC,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[6]Edgar RC.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nat Methods,2013,10(10): 996-998.
[7]Caporaso JG,Kuczynski J,Stombaugh J,et al.QIIME allows analysis of high-throughput community sequencing data [J].Nat Methods,2010,7(5):335-336.
[8]Kemp PF,Aller JY.Bacterial diversity in aquatic and other environments:what 16S rDNA libraries can tell us[J]. FEMS Microbiol Ecol,2004,47(2):161-177.
[9]Yu Z,Yang J,Liu L,et al.Bacterioplankton community shifts associated with epipelagic and mesopelagic waters in the Southern Ocean[J].Sci Rep,2015,5:12897.
[10]Xu J,Lian F,Zhao L,et al.Structural modulation of gut microbiota during alleviation of type 2 diabetes with a Chinese herbal formula[J].ISME J,2015,9(3):552-562.
[11]Yasuda K,Oh K,Ren B,et al.Biogeography of the intestinal mucosal and lumenal microbiome in the rhesus macaque[J].Cell Host Microbe,2015,17(3):385-391.
[12]Mohd Shaufi MA,Sieo CC,Chong CW,et al.Deciphering chicken gut microbial dynamics based on high-throughput 16S rRNA metagenomics analyses[J].Gut Pathog,2015,7:4.
[13]周典蓉,張澤,趙曉航.應用二代測序方法研究長遠航作業人員口腔細菌變化[J].中國衛生檢驗雜志,2016,26(10):1369-1372.
[14]Naito M,Hirakawa H,Yamashita A,et al.Determination of the genome sequence of Porphyromonas gingivalis strain ATCC 33277 and genomic comparison with strain W83 revealed extensive genome rearrangements in P.gingivalis [J].DNA Res,2008,15(4):215-225.
[15]Irshad M,van der Reijden WA,Crielaard W,et al.In vitro invasion and survival of Porphyromonas gingivalis,in gingival fibroblasts;role of the capsule[J].Arch Immunol Ther Exp(Warsz),2012,60(6):469-476.
[16]Jeon CY,Haan MN,Cheng C,et al.Helicobacter pylori infection is associated with an increased rate of diabetes[J]. Diabetes Care,2012,35(3):520-525.
[17]Chen Y,Blaser MJ.Association between gastric Helicobacter pylori colonization and glycated hemoglobin levels[J].J Infect Dis,2012,205(8):1195-1202.
Analysis of oral microbial diversity in patients with type 2 diabetes mellitus by 16S ribosome DNA Amplicon sequencing
LI Zongze,ZHAO Ze,ZHANG Wenfei,TAO Rong,SUN Xuefei,TIAN Yu
(Department of Endodontics,School of Stomatology,the Fourth Military Medical University,Xi’an Shaanxi 710032,China)
Objective To discuss the differences in microbial diversity and structure of oral type 2 diabetes mellitus.Methods The total DNA of saliva and gingival plaques were collected from 23 patients with type 2 diabetes mellitus and normal subjects.The total DNA of oral flora was extracted and sequenced by 16S ribosome DNA(16SrDNA)Amplicon sequencing.The diversity of flora was analyzed by Pandaseq and Qiime softuare.Results There was no significant difference in abundance and dominant bacteria between type 2 diabetes mellitus and normal population(P>0.05).However,the prevalence of type 2 diabetes mellitus in the samples of Bulleidia,Ruminococcaceae,and Helicobacter pylori were rich and higher than normal(P<0.05).While the genus Velvetiaceae,Selenomonadales,Negativicutes,Selenomonas,Butyrivibrio,Anaeroglobus,Candidatus-Saccharibacteria,Saccharibacteria_genera_incertae_sedis,Nocardioidaceae,and Aeromicrobium were rich in normal subjects(P<0.05).Conclusion There was a significant difference in the structure of oral cavity flora between type 2 diabetes mellitus and normal people by 16SrDNA Amplicon sequencing,but the difference was not significant.The relationship between Helicobacter pylori and type 2 diabetes mellitus remains to be further studied in flora differences.
Type 2 diabetes mellitus;Oral flora;16S ribosome DNA(16SrDNA)
R781
B
2095-3097(2017)03-0151-06
10.3969/j.issn.2095-3097.2017.03.006
2017-02-23 本文編輯:徐海琴)
國家自然科學基金面上項目(81371140,81670974);陜西省自然科學基礎研究計劃面上項目(2013JM4037)
710032陜西西安,第四軍醫大學口腔醫院牙體牙髓病科(李宗澤,趙 澤,張文菲,陶 榮,孫雪飛,田 宇)
田 宇,E-mail:tianyu@fmmu.edu.cn

