用于未純化蛋白樣品核酸適配體篩選的Western 印跡-SELEX篩選技術的建立
李慧 ,趙強,梁超,丁紅梅,李潔,黃皚雪,蘇雪婷,張令強,李少華,邵寧生
軍事醫學科學院a.基礎醫學研究所;b.放射與輻射醫學研究所;北京 100850
指數富集的配體系統進化(systematic evolution of ligands by exponential enrichment,SELEX)技術是一種新型的高通量生物文庫篩選技術[1-2]。經SELEX技術獲得基于空間結構,與靶分子高特異性、高親和力結合的寡核苷酸配基被稱為適配體(aptamer)。由于隨機文庫中的寡核苷酸序列能形成多樣的空間結構,因此SELEX 技術可篩選的靶分子范圍十分廣泛,包括金屬離子、蛋白多肽、有機染料等,其中蛋白多肽類靶分子最多,包括酶、生長因子、抗體、轉錄因子、細胞黏附分子和選擇素等[3-7]。完整的病毒顆粒、細菌病原體,甚至完整的哺乳動物細胞也可以通過完整細胞消減SELEX 技術篩選出其高親和力的寡核苷酸配基[8-11]。SELEX 技術作為一種極其有用的分子生物學工具日益受到重視,并在基礎醫學、臨床診斷和疾病治療中都顯示了廣闊的應用前景。
傳統SELEX 技術中以蛋白多肽類物質為靶標進行篩選時,首先需要獲得單一的純化蛋白,而很多蛋白、多肽類物質并不是單一的純蛋白,從而使其無法作為靶標進行篩選。因此,我們嘗試將Western印跡和SELEX 技術交叉結合,建立基于免疫印跡的蛋白篩選技術,即Western 印跡-SELEX 技術。利用該方法對蛋白、多肽類靶物質進行篩選時,不要求靶蛋白是純化的、單一的,大大降低了篩選靶標的標準,提高了篩選效率。
1 材料與方法
1.1 隨機單鏈DNA文庫和引物
文庫兩端為固定的引物退火序列,中間為含45個堿基的隨機序列,Gp45 文庫序列為5'-GCAATG GTACGGTACTTCC(45N)CAAAAGTGCACGCTACT TTGCTAA-3'。正向引物Plong-1[5'-GCAATGGTA CGGTACTTCC-3',5'端進行生物素(Bio)修飾],反向引物Pstem-loop(5'-GCTAAGCGGGTGGGACTTCC TAGTCCCACCCGCTTAGCAAAGTAGCGTGCACTTT TG-3'),反向引物Pll(5'-TTAGCAAAGTAGCGTGC ACTTTTG-3')。Pll 用于擴增雙鏈產物;Pstem-loop除含有與模板互補的序列外,5'端還含有33 個富含GC堿基的反向重復序列(下劃線部分),用于阻斷正鏈的延伸,PCR產生不同長度的單鏈DNA(ssDNA)。
1.2 Western印跡-SELEX篩選流程
取約1200 pmol 的Bio-Gp45 文庫,100℃加熱5 min 后迅速置于冰上冷卻10 min,加入結合緩沖液(1×PBS,1 mmol/L MgCl2,1 μg/μL ytRNA,1 μg/μL wDNA,0.1% BSA)中,孵育體系為100 μL。文庫先與轉有消減蛋白的PVDF 膜于37℃孵育60 min,回收未結合的ssDNA,即消減文庫,再與轉有靶蛋白的PVDF膜于37℃孵育60 min,將PVDF膜放入含5 mL 洗滌緩沖液(1×PBS,1 mmol/L MgCl2)的平皿中洗滌3 次,每次5 min,再將PVDF 膜剪碎放入EP 管中,加入200 μL ddH2O,95℃煮10 min,離心收集液體,取5 μL做模板進行PCR擴增。重復上述步驟,共進行4 輪篩選。隨著篩選輪次的增加,逐漸增加洗滌次數,縮短文庫與靶蛋白孵育的時間,增加SELEX篩選的特異性。
1.3 次級文庫的生成和回收
采用不對稱引物PCR 方法獲得次級ssDNA 文庫,以Bio-Plong-1 和Pstem-loop 為引物,按100 μL體系進行PCR 反應(95℃預變性5 min;94℃變性1 min,37℃退火80 s,58℃延伸1 min;最后58℃終延伸5 min)。為減少非特異性擴增,提高文庫質量,須優化PCR 擴增的循環數,9 個循環開始,每隔3 個循環取一次樣,共擴增30 個循環,上樣7 mol/L 尿素8%變性PAGE 膠分析,選擇目的條帶清晰的擴增循環數進行大量PCR 擴增,經7 mol/L 尿素8%變性凝膠分離,切膠回收目的單鏈,經洗脫、乙醇沉淀和離心,最終獲得Bio標記的次級篩選文庫。
1.4 富集文庫的克隆、測序與分析
將富集文庫用對稱PCR 方法擴增成雙鏈DNA(dsDNA),切膠純化后連接到pUC18T 載體中,轉化大腸桿菌DH5α,隨機挑選50 個陽性克隆進行序列測定,采用RNAstructure 4.6軟件和MEME在線軟件(httt://meme.sdsc.edu/meme/cgi-bin/meme.cgi)分析適配體的一、二級結構。
1.5 適配體的特異性檢測
將一定濃度的Bio 標記的適配體溶解于合適體積的緩沖液(1×PBS,1 mmol/L MgCl2)中,100℃變性5 min 后立即置于冰上充分冷卻10 min,與轉有靶蛋白(或其他無關蛋白,作為陰性對照)的PVDF 膜于37℃共孵育60 min,用洗滌緩沖液(1×PBS,1 mmol/L MgCl2)洗膜3 次,每次3 min,再將膜放到按1∶300 稀釋的辣根過氧化物酶(HRP)反應液中室溫孵育40 min,用同上洗滌緩沖液洗膜3 次,每次3 min,室溫避光加顯色液顯色3 min,暗室壓片顯影。
在96 孔ELISA 板中每孔加入含2 μg 靶蛋白的蛋白包被液100 μL,4℃包被過夜,次日棄包被液,每孔加入100 μL 含1%酪蛋白(casein)的馬來酸封閉液,室溫封閉60 min,將不同濃度Bio 標記的適配體溶解于100 μL 孵育緩沖液(1×PBS,1 mmol/L MgCl2)中,100℃變性5 min 后立即置于冰上充分冷卻10 min,再加到包被有靶蛋白的酶聯條中,37℃孵育3 h,棄去孔內液體,每孔用350 μL 洗滌液洗滌3次,最后一次洗滌后把孔內液體完全甩干,每孔加入100 μL 按1∶100 稀釋的HRP,室溫孵育40 min,棄去孔內液體,洗板5 次,每孔加入100 μL TMB 顯色底物37℃避光顯色,當有明顯顏色變化時,加10 μL終止液,酶聯儀讀取D450nm值。
2 結果
2.1 Western印跡-SELEX技術的建立
利用Western 印跡將相對分子質量不同的蛋白質通過電泳區分開,并轉移至PVDF 膜上,通過BiossDNA 文庫先與消減靶標孵育后,回收未結合的Bio-ssDNA 文庫,將其與篩選靶標共同孵育,進一步通過洗滌、PCR 擴增、切膠回收獲得次級文庫,其流程如圖1。
2.2 特異識別靶蛋白的ssDNA適配體的富集
采用Western 印跡-SELEX 技術進行循環篩選,利用生物素-鏈霉親和素-辣根過氧化物酶系統檢測每輪次級文庫的富集情況。顯影結果顯示,隨著篩選輪次的增加,與靶蛋白特異結合的適配體逐漸得到富集,與前3 輪富集文庫相比,第4 輪文庫中適配體的富集量得到大幅提升,暗室壓片結果可見明顯條帶(圖2),提示經過4輪篩選得到的寡核苷酸配基特異識別靶蛋白。
2.3 適配體一級結構同源性及二級結構分析
將第4 輪富集文庫進行克隆測序,得到50 個適配體的序列,經MEME 在線軟件對序列的同源性進行比較,共分為6 個家族(圖3);進一步利用RNA structure 軟件對每個家族中所包含序列的二級結構進行分析,初步挑選出6條適配體。
2.4 適配體與靶蛋白結合的特異性分析
Western 印跡分析表明,同原始文庫Gp45 相比,挑選出的6 條適配體均與靶蛋白有一定的結合,其中6#適配體與靶蛋白的結合最強,只特異結合靶蛋白而不結合無關對照蛋白,而原始文庫Gp45則與上述蛋白均無結合(圖4)。
ELISA 分析表明,經生物素-鏈霉親和素-辣根過氧化物酶系統檢測后,6#適配體與靶蛋白孵育后的D450nm值最大,遠高于原庫Gp45,這一結果與Western 印跡檢測結果一致。提示6#適配體不僅能特異識別經SDS 變性處理的靶蛋白,還能特異識別天然結構的靶蛋白(圖4)。
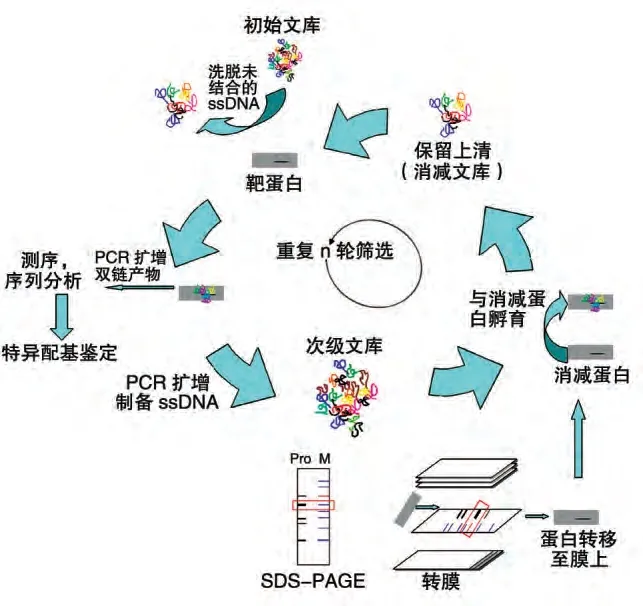
圖1 Western印跡-SELEX篩選技術路線

圖2 富集文庫與靶蛋白結合的鑒定
3 討論
SELEX技術作為一種極其有用的分子生物學工具而日益受到重視,并在基礎醫學、臨床診斷和疾病治療中顯示了廣闊的應用前景。SELEX技術具有很多優點:①經SELEX 技術篩選出的寡核苷酸配基具有很強的特異性。茶堿與其他黃嘌呤類似物如咖啡因、可可堿的結構非常相似,而通過SELEX 方法篩選到的寡核苷酸配基只特異性地結合茶堿,與其他類似物無反應,與茶堿的親和力比與咖啡因的親和力高10 000倍[12]。由于寡核苷酸配基與蛋白質的結合力高于抗原與抗體之間的結合,因此,將生物素、熒光素、活性基團偶聯到寡核苷酸配基上,可對新蛋白質進行細胞定位,也可用于分離純化蛋白質。②由于寡核苷酸配基的高特異性和高親和力,將其作為診斷試劑比抗體具有更高的靈敏度和準確性,而且這種特異性和親和力還不受組織或樣品中非靶蛋白的干擾。單獨或與抗體組合應用于多種診斷模式已顯示出其獨特的優越性,特別是可以彌補抗體在診斷領域中應用的不足。③SELEX技術篩選還具有周期短的優點,并有望實現高通量篩選[13]。經SELEX技術篩選出的適配體不受免疫條件和免疫原性限制,可體外人工合成,變性與復性可逆,可修飾,并有利于長期保存和室溫運輸。經過近20 年的數據累積不難發現,人們在寡核苷酸配基和抗體方面所做的努力是朝著一個相同的方向邁進的。鑒于SELEX技術的優越性,也許在不久的將來,寡核苷酸配基很可能成為被動免疫中抗體的替代物,實驗室里的單克隆抗體制備技術將會被一種費用不高、較為方便的SELEX技術所替代。
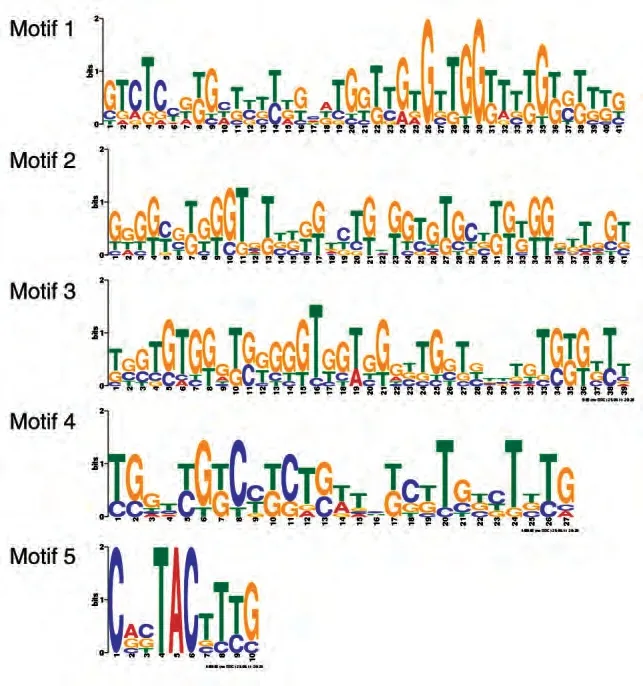
圖3 MEME在線軟件分析序列的同源性
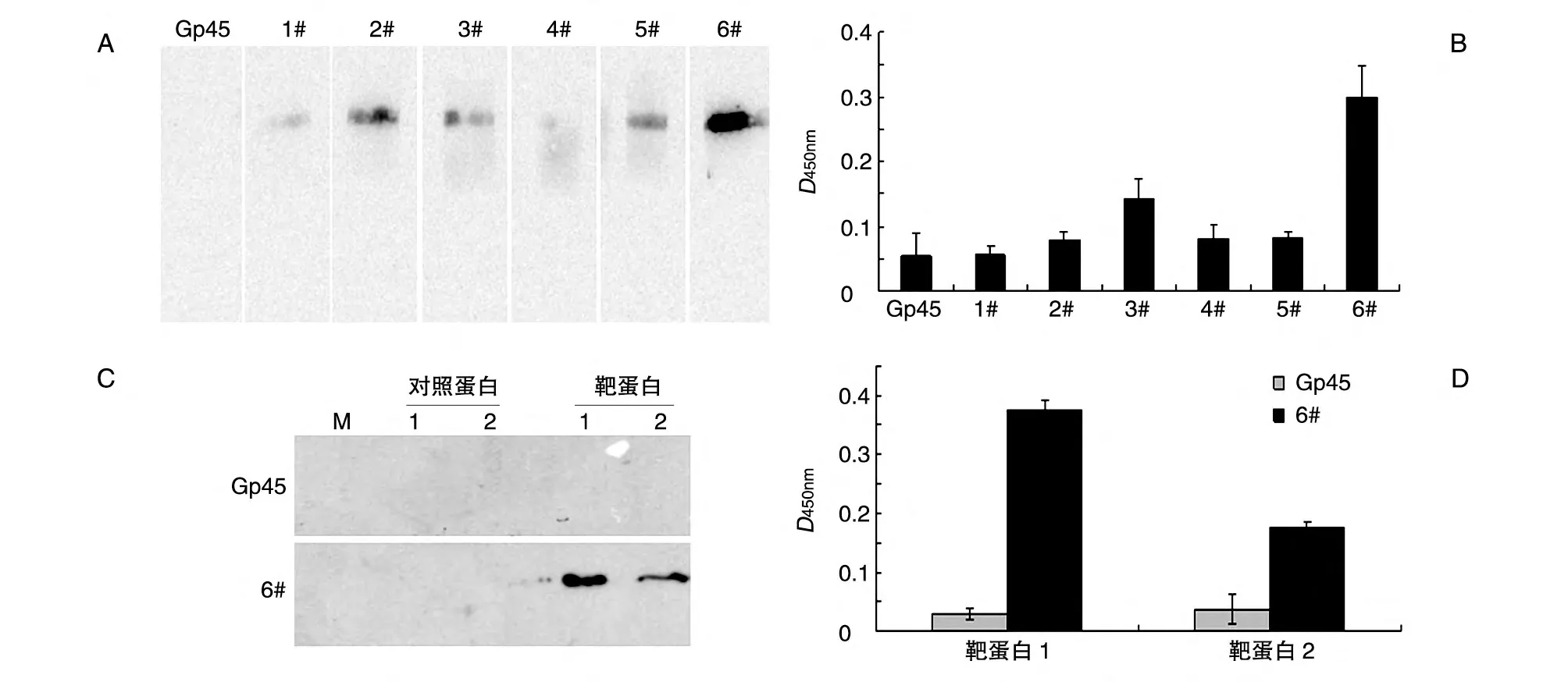
圖4 挑選出的6條適配體與靶蛋白特異結合的鑒定
文庫是否富集是SELEX 篩選成敗的重要指標。經過4 輪篩選,我們獲得了能夠與靶蛋白特異結合的富集文庫,分析、純化這些差異適配體并對其進行DNA 序列測定,根據序列同源性及二級結構得到6 條有代表性、結構穩定的適配體,檢測發現2#、3#、5#和6#適配體均能與靶蛋白特異結合,其中6#適配體與靶蛋白的結合最明顯,只特異結合靶蛋白而不結合無關對照蛋白。這一結果一方面說明我們建立的Western 印跡-SELEX 技術是可信穩定的,另一方面也表明適配體可以像抗體一樣作為診斷試劑,或經過改造用于預防與治療疾病。
綜上,我們建立了一種新的SELEX 篩選方法,即Western 印跡-SELEX。蛋白質的Western 印跡技術結合了凝膠電泳的高分辨率和固相免疫測定的特異敏感等多種特點,可檢測到低至1~5 ng 的靶蛋白。將Western 印跡和SELEX 技術結合起來,可以實現非純化蛋白的篩選,大大提高了篩選速度和效率,為SELEX 技術在基礎醫學、臨床診斷和疾病治療等方面開辟了一條新的途徑,有良好的應用前景。
[1]Ellingtion A D,Szostak J W.In vitro selection of RNA molecules that bind specifical[J].Nature,1990,346(6287):818-822.
[2]Tuerk C,Gold L.Systematic evolution of ligands by exponential enrichment:RNA ligands to bacteriophage T4 DNA polymerase[J].Science,1990,249(4968):505-510.
[3]Tang Jijun,Xie Jianwei,Shao Ningsheng,et al.The DNA aptamers that specifically recognize ricin toxin are selected by two in vitro selection methods[J].Electrophoresis,2006,27(7):1303-1311.
[4]Williams B A R,Lin L,Lindsay S M,et al.Evolution of a histone H4-K16 acetyl-specific DNA aptamer[J].J Am Chem Soc,2009,131(18):6330-6331.
[5]Cella L N,Sanchez P,Zhong W,et al.Nano aptasensor for protective antigen toxin of anthrax[J].Anal Chem,2010,82(5):2042-2047.
[6]Tran D T,Janssen K P F,Pollet J,et al.Selection and characterization of DNA aptamers for egg white lysozyme[J].Molecules,2010,15(3):1127-1140.
[7]Tok J,Lai J,Leung T,et al.Selection of aptamers for signal transduction proteins by capillary electrophoresis[J].Electrophoresis,2010,31(12):2055-2062.
[8]Mosing R K,Mendonsa S D B,Bowser M T,et al.Capillary electrophoresis-SELEX selection of aptamers with affinity for HIV-1 reverse transcriptase[J].Anal Chem,2005,77(19):6107-6112.
[9]Chen Fan,Zhou Jing,Luo Fengling,et al.Aptamer from whole-bacterium SELEX as new therapeutic reagent against virulent Mycobacterium tuberculosis[J].Biochem Biophys Res Commum,2007,357:743-748.
[10]Wang Chenglong,Zhang Ming,Yang Guang,et al.Singlestranded DNA aptamers that bind differentiated but not parental cells:subtractive systematic evolution of ligands by exponential enrichment[J].J Biotechnol,2003,102:15-22.
[11]Shangguan D,Cao Z C,Li Y,et al.Aptamers evolved from cultured cancer cells reveal molecular differences of cancer cells in patient samples[J].Clin Chem,2007,53:1153-1155.
[12]Jenison R D,Gill S C,Pardi A,et al.High-resolution molecular discrimination by RNA[J].Science,1994,263(5152):1425-1429.
[13]邵寧生,楊光,房濤,等.生物文庫技術-噬菌體展示與SELEX技術[M].北京:軍事醫學科學院出版社,2011.

