BRCA1基因甲基化及甲硫氨酸合成酶與乳腺癌發病的關系
馬文慧 侯琳 韓琳琳
青島大學醫學院生物化學與分子生物學教研室,山東 青島 266021
D NA甲基化是腫瘤發生的一個早期特征,通過基因組總體水平低甲基化和某些啟動子區域高甲基化造成基因功能的失活,從而導致腫瘤的形成[1]。S-腺苷甲硫氨酸(S-adenosyl-L-methionine,SAM)是體內甲基的直接供體,通過甲硫氨酸循環及葉酸代謝得到補充,故推測此循環障礙可能導致甲基化異常。甲硫氨酸合成酶(methionine synthase,MS)是參與甲基供體生成的關鍵酶,已發現MS基因多態性與乳腺癌發病相關[2],其多態性可能通過影響基因的表達,使酶水平或活性改變,導致DNA合成紊亂和甲基化異常[3]。BRCA1基因是非常重要的乳腺癌易感基因和抑癌基因,散發性乳腺癌罕見BRCA1基因突變,但癌組織中該基因表達卻顯著下降,目前研究認為這與BRCA1啟動子甲基化有關,但對BRCA1基 因甲基化機制的報道尚顯不足。散發性乳腺癌BRCA1甲基化與腫瘤分級[4]、癌組織分型[5]、激素受體[6-7]相關,但其與雌激素受體(estrogen receptor,ER)陰性/陽性相關尚無定論。本研究首先檢測乳腺癌組織及相應癌旁組織、乳腺良性病變組織中BRCA1 mRNA的表達水平及甲基化狀態,同時檢測葉酸代謝過程中關鍵酶MS基因的表達,探討該酶與BRCA1基因甲基化及其與乳腺癌發生的關系,從而為腫瘤甲基化機制及乳腺癌的發病機制提供新的理論依據。
1 資料和方法
1.1 資料
1.1.1 乳腺癌組織標本
病例組選取2009年8月—2009年11月在青島大學醫學院附屬醫院行外科 手術切除新鮮組織標本71例,其中良性病變組織9例、乳腺癌及癌旁組織(距癌>3 cm)各31例(獲知情同意),標本切除后迅速放入-70 ℃冰箱保存。術前所有患者皆未接受治療,術后均經病理組織學確診。全部乳腺癌病例戶籍均為山東地區,無家族乳腺癌發病史。
1.1.2 主要試劑
MMLV逆轉錄試劑盒和Wizard DNA clean up純化試 劑盒購自美國Promega公司,對苯二酚和亞硫酸氫鈉購自美國Sigma公司,耐熱DNA
聚合酶購自大連寶生物公司,飽和酚購自北京索萊寶科技有限公司。
1.1.3 主要儀器
GeneAmp2400PCR反應儀(PE公司),紫外分光光度計(Eppendorf公司),5415D高速離心機(Eppendorf公司),5417R高速冷凍離心機(Eppendorf公司)
1.2 方法
1.2.1 組織DNA、RNA提取
采用酚-氯仿抽提法提取組織DNA,DNA經紫外分光光度計定量,-20 ℃保存備用。參照FASTAGEN-RNAfast200試劑盒(上海飛捷生物技術公司產品)操作說明提取組織總RNA。RNA經瓊脂糖凝膠電泳鑒定其完整性,通過紫外分光光度計測定D260(260 nm光密度值)與D280(280 nm光密度值),計算D260/D280比值,計算RNA的純度與濃度。用MMLV逆轉錄酶進行逆轉錄反應,合成cDNA。
1.2.2BRCA1基因的反轉錄PCR (RT-PCR)
用2 μg總RNA進行逆轉錄。cDNA經半定量PCR進行擴增,BRCA1及內參照β-actin特異性引物的序列和擴增片段長度見表1。PCR為25 μL反應體系,反應條件: 94 ℃預變性5 min;94 ℃變性30 s,55 ℃退火30 s,72 ℃延伸30 s,30個循環;72 ℃終延伸7 min。PCR產物在含溴化乙錠的2%瓊脂糖凝膠中電泳,由Quantity One 4.6.6 (BIO-RAD)分析條帶D值。
1.2.3 組織DNA的亞硫酸氫鹽處理
在50 μL反應體系中加入2 μg DNA和3 mol/L NaOH 5.5 μL,42 ℃水浴30 min,再加入新鮮配制的對苯二酚30 μL,3.6 mol/L亞硫酸氫鈉520 μL,50 ℃水浴16 h。采用Wizard DNA clean-up system (Promega公司)純化試劑盒純化修飾過的DNA。純化后的DNA加入3 mol/L NaOH 5.5 μL,37 ℃溫育15 min,加入10 mol/L乙酸銨33 μL和冰無水乙醇270 μL于-20 ℃沉淀過夜,然后4 ℃,12 000 r/min離心棄上清液(半徑6.5 cm),用70%乙醇洗滌后將沉淀溶于20 μL去離子水中,于-20 ℃冰箱中保存待用。
1.2.4BRCA1的甲基化特異性聚合酶鏈反應(methylation specific PCR, MSP)
用亞硫酸氫鹽修飾 的DNA作模板進行PCR擴增,BRCA1引物參照文獻[8]設計(表1)。PCR反應條件: 95 ℃預變性10 min;94 ℃變性1 min,60 ℃退火30 s,72 ℃延伸30 s,35個循環;72 ℃終延伸10 min。擴增結果用含溴化乙錠的2%瓊脂糖凝膠電泳檢測。用凝膠成像自動分析系統觀察結果。判定標準:觀察純化后的DNA以不同引物擴增的產物,若用甲基化引物可擴出產物,而未甲基化引物不能擴出者,計為“+”,即完全甲基化;反之計為“-”,為完全未甲基化;均有產物者計為“±”,即部分甲基化。
1.2.5MS基因的檢測
方法同1.2.2,MS基因反應條件:94 ℃預變性5 min;94 ℃變性30 s,57 ℃退火30 s,72 ℃延伸30 s,35個循環;72 ℃終延伸7 min。
1.3 統計學處理
采用GraphPad Prism 5和SPSS 11.0統計軟件進行統計學數據分析,數據用均數±標準差()表示。計數資料采用χ2檢驗,兩個獨立樣本采用t檢驗,MS基因表達與BRCA1甲基化的相關性用Spearman相關分析,P<0.05為差異有統計學意義。
2 結 果
2.1 組織中BRCA1 mRNA表達
乳腺良性病變組織BRCA1 mRNA均有表達,乳腺癌組織、癌旁組織BRCA1 mRNA出現低表達或缺失,31例乳腺癌組織中,17例BRCA1 mRNA表達下降或缺失。其mRNA的表達量用組織中的mRNA的D值與內參D值的比值表示,結果顯示乳腺癌組織BRCA1 mRNA表達量(0.16±0.08)明顯低于乳腺癌旁組織表達量(0.41±0.09)及乳腺良性病變組織表達量(0.69±0.14),差異均有統計學意義(P<0.05,圖1)。
2.2 BRCA1基因啟動子甲基化狀態
乳腺良性病變組織中未發現BRCA1甲基化,BRCA1基因在31例乳腺癌及癌旁組織中甲基化檢出率分別為29.0%(9/31)和3.2%(1/31),經統計學分析,差異有統計學意義(χ2=7.631,P<0.05,圖2)。BRCA1甲基化與散發性乳腺癌組織學分級、E R陰性有關(P<0.05),但與發病年齡、組織分型和淋巴結轉移無關(P>0.05,表2)。

2.3 BRCA1 mRNA表達與其甲基化的關系
在18例BRCA1 mRNA表達陰性的乳腺癌組織中,有8例BRCA1基因發生了甲基化,在13例BRCA1 mRNA表達陽性的乳腺癌組織中有1例BRC A1基因發生甲基化,兩者相比,差異有統計學意義(χ2=4.949,P<0.05)。
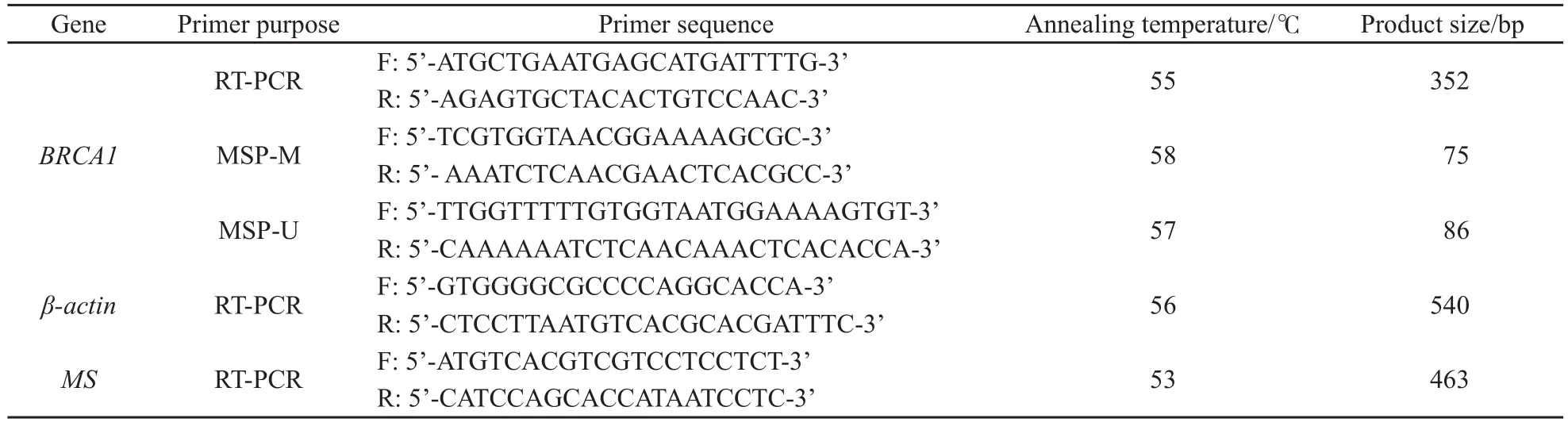
表 1 所用引物序列Tab.1 Primer sequences and PCR conditions

2.4 MS mRNA的表達與乳腺癌的關系
乳腺良性病變組織中MS mRNA有表 達,乳腺癌組織及癌旁組織中MS mRNA低表達或缺失(圖3),其mRNA的表達量用 組 織中的mRNA的D值與內參D值的比值表示。結果顯示,乳腺癌組織的表達量(0.26±0.06)明顯低于乳腺良性病變組織的MS mRNA表達量(0.60±0.21)及癌旁組織的表達量(0.40±0.13),差異均有統計學意義(P<0.05,圖3)。
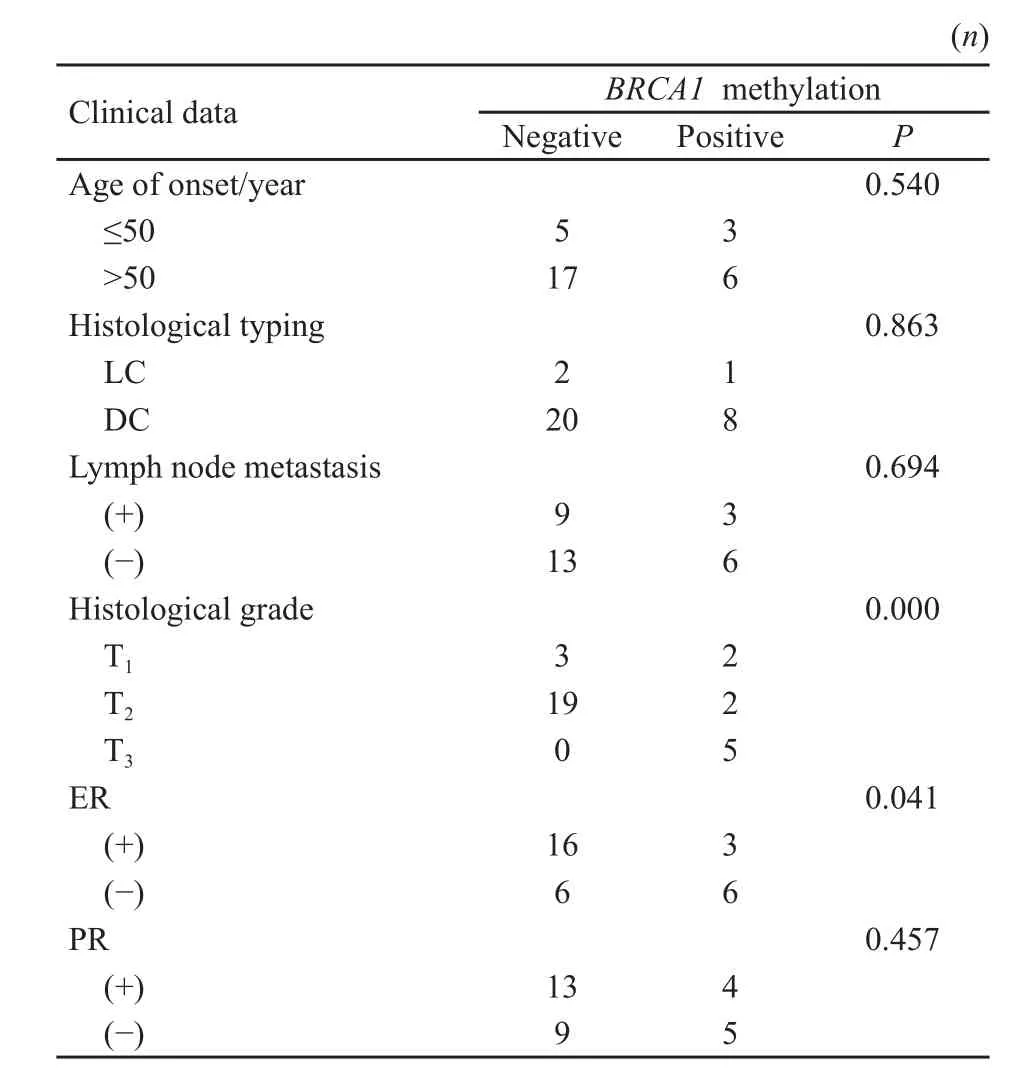
表 2 乳腺癌組織BRCA1甲基化與臨床資料的關系Tab.2 Relationship between BRCA1 methylation in breast cancer tissues and clinical features

2.5 MS基因表達與BRCA1基因甲基化的關系
MS基因表達陰性的14例乳腺癌組織中,有7例BRCA1基因發生了甲基化;MS基因表達陽性的17例乳腺癌組織中,有2例其BRCA1發生甲基化,經Spearman相關分析結果顯示,乳腺癌組織中MS mRNA的表達與BRCA1基因甲基化有相關性(r=0.419,P<0.05)。
3 討 論
乳腺癌是女性最常見的惡性腫瘤之一,嚴重威脅著女性的健康。BRCA1基因主要功能是在細胞增殖周期的整個環節對細胞分裂和生長進行調控,使細胞按照正常程序周期性分 裂、生長和凋亡[9-10],是重要的乳腺癌抑癌基因。抑癌基因失活可以導致細胞周期失控,細胞發生異常增殖而癌變。本實驗檢測到BRCA1基因在乳腺癌組織中表達明顯低于癌旁組織及乳腺良性病變組織,提示該基因的失表達與乳腺癌患病密切相關。大量研究已證實甲基化是除基因突變和缺失以外導致抑癌基因失活的第三種機制,在腫瘤的發生、發展中起重要的作用。研究已發現許多抑癌基因通過DNA異常甲基化引起表達抑制甚至關閉而失活,如Rb、APC、MGMT、P16等。我們檢測到BRCA1基因乳腺癌組織甲基化檢出率為29%,這與之前報道的用不同方法檢測到9%~44%的乳腺癌樣本中BRCA1基因啟動子甲基化結果較一致,癌組織甲基化檢出率顯著高于相應癌旁組織(3.2%)及乳腺良性病變組織(0)[11-12]。曾有報道提出,BRCA1基因甲基化的腫瘤可能表型模擬家族BRCA1基因腫瘤[13]。我們觀察到BRCA1甲基化與散發性乳腺癌組織學分級、ER負性有關,這與Birgisdottir等[5]報道的結果相一致。然而Matros等[6]報道BRCA1基因在高級別ER陽性腫瘤的啟動子甲基化頻率高,這意味著其表型的關聯可能更加復雜。經統計,BRCA1基因的表達與該基因發生甲基化密切相關,DNA甲基化機制可以較合理地解釋散發性乳腺癌的BRCA1基因轉錄水平異常。
癌癥基因組學的顯著特點是全基因組的低甲基化,和特定位點的高甲基化并存。DNA低甲基化可以提高特定原癌基因(如與增殖和轉移相關基因)的表達,而特定位點高甲基化可以使抑癌基因(如DNA修復基因)的表達降低,從而增加患癌癥的風險[14],而啟動這些變化的機制尚待研究。基因啟動子區甲基化模式的改變,是在轉錄水平調節基因表達的途徑之一[15]。大量研究表明,正常細胞中抑癌基因啟動子是低甲基化甚至非甲基化的。然而,腫瘤細胞中抑癌基因卻常發生甲基化,從而導致抑癌基因低表達和癌細胞增殖能力提高[16-18]。MS主要生化功能是催化同型半胱氨酸復甲基為甲硫氨酸,甲硫氨酸在轉甲基之前,與ATP作用生成SAM,為DNA的甲基化提供甲基,是參與甲基供體生成的關鍵酶。大量實驗已證明MS多態性與乳腺癌患病有關,本實驗室也發現MS2756等 位基因G型突變能夠增加乳腺癌的患病風險,并推測這可能與MS多態性的改變影響了酶的活性,從而影響了甲基供體生成,導致甲基化異常有關[19]。而本 實驗進一步檢測到乳腺良性病變組織中MS mRNA均有表達,乳腺癌患者的癌組織及癌旁組織中MS低表達或缺失,提示MS基因低表達能增加乳腺癌患病風險 。本實驗結果顯示,乳腺癌組織中MS mRNA的表達與BRCA1基因甲基化有相關性,這表明MS基因的表達可能影響某些基因的甲基化狀態。我們推測MS基因低表達,會影響體內甲基供體生成,導致甲基化異常,從而引起BRCA1基因低甲基化。但我們卻發現MS基因表達陰性癌組織中BRCA1基因發生甲基化的頻率較表達陽性的顯著升高,表明MS基因低表達與抑癌基因BRCA1甲基化有關,符合腫瘤細胞抑癌基因常常發生甲基化這一表觀遺傳學特征。Stempak等[20]、Kim 等[21]、Gaia等[22]研究表明,甲基供體缺乏對甲基化的影響是位點和基因特異性的,此外,甲基化的變化方向可能是細胞、靶器官、轉化具體階段特異性的,而且未必 是基因組和基因之間或位點的DNA甲基化相同。具體機制還有待于進一步研究。近年來,DNA甲基化模式異常已被確立為人類癌癥的標志之一。甲硫氨酸循環關鍵酶低表達,導致體內甲基化水 平異常,抑癌基因CpG島高甲基化而失活,從而引發癌癥,我們得到的結果初步證實了這一假設。我們的研究結果強調了甲硫氨酸循環對乳腺癌發病的重要性。雖然該循環酶網絡十分復雜,但評估限速酶及其與環境的相互作用對于評估乳腺癌風險是十分關鍵的。
[1]Ehrlich M.DNA methylation in cancer: Too much, but also too little [J].Oncogene, 2002, 21(35): 5400-5413.
[2]Lu M, Wang F, Qiu J.Methionine synthase A2756G polymorphism and breast cancer risk: a meta-analysis involving 18, 953 subjects [J].Breast Cancer Res Treat,2010, 123: 213-217.
[3]Xinran X, Jia C.One-carbon metabolism and breast cancer:an epidemiological perspective [J].J Genet Genomics,2009, 36: 203-214.
[4]Niwa Y, Ciyama T, Nakajima T.BRCAl expression status in relation to DNA methylation of BRCA1 promoter region in sporadic breast cancers [J].Jpn J Cancer Res, 2000, 91(5):519-526.
[5]Birgisdottir V, Stefansson OA, Bodvarsdottir SK, et al.Epigenetic silencing and deletion of the BRCA1 gene in sporadic breast cancer [J].Breast Cancer Res, 2006, 8:R38.
[6]Matros E, Wang ZC, Lodeiro G, et al.BRCA1 promoter methylation in sporadic breast tumors: relationship to gene expression profiles [J].Breast Cancer Res Treat, 2005, 91:179-186.
[7]Esteller M, Corn PG, Baylin SB, et al.A gene hypermethylation profile of human cancer [J].Cancer Res, 2001, 61(8):3225-3229.
[8]Esteller M, Silva JM, Dominguez G, et al.Promoter hypermethylation and BRCA1 inactivation in sporadic breast cancer and ovarian tumors [J].J Natl Cancer Inst(Bethesda), 2000, 92(7): 564-569.
[9]Venkitaraman AR.Cancer susceptibility and the functions of BRCA1 and BRCA2 [J].Cell, 2002, 108: 171-182.
[10]Paull TT, Cortez D, Bowers B, et al.Direct DNA binding by Brca1 [J].Proc Natl Acad Sci U S A, 2001, 98: 6086-6091.
[11]Birgisdottir V, Stefansson OA, Bodvarsdottir SK, et al.Epigenetic silencing and deletion of the BRCA1 gene in sporadic breast cancer [J].Breast Cancer Res, 2006, 8(4):R38.
[12]Butcher DT, Rodenhiser DI.Epigenetic inactivation of BRCA1 is associated with aberrant expression of CTCF and DNA methyltransferase (DNMT3B) in some sporadic breast tumours[J].Eur J Cancer, 2007, 43(1): 210-219.
[13]Turner N, Tutt A, Ashworth A.Hallmarks of 'BRCAness' in sporadic cancers [J].Nat Rev Cancer, 2004, 4: 814-819.
[14]Duthie SJ.Epigenetic modifications and human pathologies:cancer and CVD [J].Proc Nutr Soc, 2011, 70(1): 47-56.
[15]Turner JD, Pelascini LP, Macedo JA.et al.Highly individual methylation patterns of alternative glucocorticoid receptor promoters suggest individualized epigenetic regulatory mechanisms [J].Nucleic Acids Res, 2008, 36: 7207-7218.
[16]Taghavi N, Biramijamal F, Sotoudeh M.et al.p16INK4a hypermethylation and p53, p16 and MDM2 protein expression in esophageal squamous cell carcinoma [J].BMC Cancer,2010, 10: 138.
[17]Gonzalo V, Lozano JJ, Mu?oz J.et al.Aberrant gene promoter methylation associated with sporadic multiple colorectal cancer [J].PLoS One, 2010, 5: e8777.
[18]Wang X, Lau KK, So LK, et al.CHD5 is down-regulated through promoter hypermethylation in gastric cancer [J].J Biomed Sci, 2009, 16: 95.
[19]韓琳琳, 侯琳, 宋金蓮.甲硫氨酸合成酶多態性及CHD5基因甲基化與乳腺癌發病的關系[J].中國癌癥雜志,2010, 20(4): 251-255.
[20]Stempak JM, Sohn KJ, Chiang EP, et al.Cell and stage of transformation-specific effects of folate deficiency on methionine cycle intermediates and DNA methylation in an in vitro model [J].Carcinogenesis, 2005, 26: 981-990.
[21]Kim YI.Nutritional epigenetics: impact of folate deficiency on DNA methylation and colon cancer susceptibility [J].J Nutr, 2005, 135(11): 2703-2709.
[22]Gaia B, Erika V, Sei-Ichi M, et al.Mild folate deficiency induces genetic and epigenetic instability and phenotype changes in prostate cancer cells [J].BMC Biol, 2010, 8: 6.

