代謝工程改造野生耐酸酵母生產L-乳酸
張勤,張梁,丁重陽,王正祥,石貴陽
1 江南大學 工業生物技術教育部重點實驗室,無錫 214122
2 江南大學生物工程學院 生物資源與生物能源研究中心,無錫 214122
乳酸因其安全性、光學特性及其特殊的分子結構被廣泛應用于食品、醫藥和化妝品等工業。由于人體只能利用L(+)-乳酸,世界衛生組織提倡在食品及醫藥行業使用 L(+)-乳酸取代目前普遍使用的DL(+)-乳酸[1-2]。另外,L-乳酸的一個重要用途為合成聚乳酸,聚乳酸因可以由可再生資源生產并能被生物降解而被認為是最有發展前景的聚合物之一[3]。2009年世界L-乳酸需求量為13~l5萬t,主要集中在美國、西歐和日本,且需求量將以年均 5%~8 %的速度持續增長,加之聚乳酸的逐步推廣使用,L(+)-乳酸產業規模將急劇擴大[4]。
目前,L-乳酸的生產方法主要為微生物發酵法。現國內外研究主要集中在乳桿菌和米根霉[5-6]。在乳酸發酵過程中,pH值會隨著乳酸的生成而逐漸降低,而低pH值會抑制細菌和根霉菌的生長和發酵能力[7]。雖然 CaCO3作為中和劑,可以維持最適 pH值。但是乳酸鈣結晶細小,且結晶過程不易控制,另外 30%乳酸鈣殘留在結晶母液中,不能結晶出來,大量的副產物 (石膏) 會造成環境污染。另外NaOH或NH4OH可也作為中和劑,但用量大、成本高[8-9]。
相對于細菌和霉菌,酵母菌具備更好的耐酸特性,可在強度更高的酸性條件下生存和生長。早在1994年Dequin和Barr就報道了代謝工程酵母菌株異源表達L-乳酸脫氫酶,其L-乳酸產量為12 g/L[10]。之后也有學者研究將來源于乳酸菌、米根霉和其他來源的乳酸脫氫酶在釀酒酵母中表達,其L-乳酸產量也提高至25.7 g/L和38.0 g/L[11-12]。
然而有文獻顯示,L-乳酸比其他大多數有機酸和無機酸引起的細胞內酸化速度更快,對細胞造成的損傷更為嚴重[13]。因此,一般酵母和以 HCl或H2SO4等無機酸為篩選壓力得到的耐酸菌在 L-乳酸生產上并不能表現出其在耐酸方面的優越性。本研究則以自然界中篩選得到的一株耐高濃度乳酸的酵母菌為出發菌株,在國內首次采用基因工程技術,通過表達米根霉乳酸脫氫酶基因,改造野生耐酸酵母菌株,增加其乳酸代謝途徑,代謝葡萄糖生成乳酸。可以降低傳統乳酸發酵過程中因添加鈣所產生的污染,也可降低氨水等中和劑的用量并簡化操作過程,可大幅度降低生產成本,為以耐酸酵母菌高產L-乳酸進行了有益的探索。
1 材料與方法
1.1 材料
1.1.1 樣品
水果皮、土壤、酒曲等。
1.1.2 菌株與來源
本研究所用菌株及質粒如表1所示。

表1 本研究中所用菌株及質粒Table 1 Strains and plasmids used in this study
1.1.3 培養基
YEPD培養基:葡萄糖20 g/L;蛋白胨20 g/L;酵母膏10 g/L。用于固體培養基時添加20 g/L瓊脂粉;挑選轉化子時添加800 mg/L的G418抗生素。
篩選培養基 (Selection medium):葡萄糖20 g/L;蛋白胨 20 g/L;酵母膏10 g/L;以乳酸調節pH值至2.5。
單碳源培養基 (Solo carbon medium):乳酸17 g/L;(NH4)2SO47.5 g/L;KH2PO43.5 g/L;MgSO4·7H2O 0.75 g/L;酵母膏0.5 g/L。
PDA培養基:葡萄糖20 g/L;馬鈴薯200 g/L,瓊脂粉20 g/L,用于米根霉的培養。
LB培養基:NaCl 10 g/L;蛋白胨10 g/L;酵母膏5 g/L。用于固體培養基時添加20 g/L瓊脂粉;挑選轉化子時添加終濃度為30 mg/L的卡那霉素。
發酵培養基:葡萄糖 100 g/L,蛋白胨20 g/L;酵母膏10 g/L。
1.2 方法
1.2.1 采樣與分離
稱取5 g樣品,加至含100 mL無菌生理鹽水及玻璃珠的三角瓶中,30 ℃、150 r/min振蕩打散30 min;樣品懸液用無菌生理鹽水稀釋,選擇適合的稀釋度,分別涂布YEPD平板,30 ℃培養5~7 d,觀察菌落生長情況,并將新長出的形態上有差異的單菌落挑出。
1.2.2 耐酸酵母的篩選
從分離得到的單菌落中挑取典型的酵母菌落分別接種于以乳酸調節pH值至4的YEPD液體培養基中,30 ℃培養48 h。選擇生長較好的酵母菌株依次接入篩選培養基中,培養48 h,選擇長勢最好的菌株在YEPD試管斜面上劃線,4 ℃保存。
1.2.3 分類鑒定
形態學鑒定:按照文獻[14]的方法進行。
分子生物學鑒定:酵母DNA的提取方法參照《精偏分子生物學實驗指南》[15],所用引物為真菌ITS通用引物 (上游 ITS1:5′-TCCGTAGGTGAA CCTGCGG-3′;下游ITS4:5′-TCCTCCGCTTATTGA TATGC-3′),PCR擴增產物經電泳檢測后測序,在核酸序列數據 (GenBank) 中進行同源序列搜索,根據同源序列搜索結果,確定該菌株所屬的最近屬種。
生理生化鑒定:按照文獻[14]的方法進行。
1.2.4 分析方法
生長量測定方法:取不同發酵時間的發酵液,用721型分光光度計于600 nm處測其吸光值。
殘糖測定方法:生物傳感儀測定發酵液內殘糖含量。
HPLC法測定有機酸:發酵液經8 000 r/min離心5 min,取300 μL上清液加入700 μL無水乙醇沉淀蛋白質3 h以上后,12 000 r/min離心15 min,0.45 μm有機微孔濾膜后,通過HPLC進行測定。分析條件:色譜柱SH1011;流動相0.01 mol/L H2SO4;流速0.8 mL/min;進樣量5 μL;柱溫50 ℃;檢測器為紫外檢測器,檢測波長為210 nm。
1.2.5 常規基因克隆操作方法
大腸桿菌感受態制備,外源基因片段與載體連接,簡易轉化操作,大腸桿菌質粒快速提取,PCR擴增,絲狀真菌基因組DNA制備等操作參見文獻[15]。
1.2.6 目的基因的PCR擴增及克隆
根據米根霉 L-乳酸脫氫酶基因 (ldhA) 序列(GenBank Accession No. EF15228.1),設計引物如下:Roldh1:5′-CGCGGATCC ATGGTATTACACTCA-3′,Roldh2:5′-CCGAAGCTT TCAACAGCTACTTTTA-3′(下劃線所示分別為BamHⅠ、HindⅢ酶切位點)。 PCR反應體系體積為50 μL,擴增條件:95 ℃預變性5 min;94 ℃變性30 s,55 ℃退火60 s,72 ℃延伸60 s,共30個循環;最后72 ℃延伸10 min。
1.2.7 質粒的構建
將PCR獲得的ldhA基因用BamHⅠ和HindⅢ雙酶切,經純化后與經同樣酶切的質粒 pYX212-kanMX連接,構建重組質粒pYX212-kanMX-ldhA。
1.2.8 G418的敏感測定
收集耐酸酵母菌體,ddH2O洗滌2次后懸浮,30 ℃進行饑餓培養2~3 h。培養后的細胞經適當稀釋后涂布于含不同濃度G418抗生素的YEPD平板,30 ℃培養3~4 d,觀察生長情況。
1.2.9 轉化方法
酵母轉化采用電穿孔轉化法,轉化后涂布于含G418抗性的YEPD平板,挑選轉化子。
電穿孔轉化條件:1 500 V,5 ms,電擊2次;電脈沖儀的型號為ECM399。
1.2.10 轉化子驗證
菌落PCR驗證和SDS-PAGE電泳驗證等操作均參見文獻[15]。
1.2.11 重組菌發酵實驗
種子液制備:將重組菌從YEPD抗性平板上刮取一單菌落至YEPD液體培養基 (250 mL三角瓶,裝液量為30 mL) 中,30 ℃、150 r/min培養24 h,即為種子液,轉接發酵液時接種量為10%。
重組菌發酵產乳酸驗證實驗:將不同重組菌的種子液分別接種至發酵培養基 (250 mL三角瓶,裝液量為50 mL),30 ℃、150 r/min培養48 h。
發酵液初始pH值對發酵產乳酸的影響:分別在發酵培養基中滴加乳酸,使其培養基pH值分別為 2.5、3.0、3.5、4.0,并以未調節 pH值的培養基 (pH 7.0) 作空白對照。將種子液分別轉接至已調節pH值的發酵培養基中 (250 mL三角瓶,裝液量為50 mL),30 ℃、150 r/min培養48 h。
2 結果與分析
2.1 耐酸酵母的分離篩選
從自然界96個樣品中篩選出55株能在乳酸調節pH值達2.5的YEPD培養基中存活或生長的酵母菌株,其中17株酵母能夠生長。逐一經YEPD培養基、乳酸篩選培養基和乳酸單碳源培養基中搖瓶發酵初篩,以生長量為指標進一步復篩。對多個批次的酵母反復比較之后,確定一株編號為No.4的菌株能在pH值2.5 YEPD培養基中生長良好。
2.2 分類鑒定
2.2.1 生理生化與分子生物學鑒定
菌株No.4有典型的酵母菌株形態,細胞呈卵形或球形,多邊芽殖。對此菌進行分子生物學鑒定,將此菌株的ITS序列與NCBI中同源性較高的幾株菌進行比對和同源性分析,同源樹 (圖1) 清楚地顯示出以分子生物學特征為基礎,菌株No.4與NCBI中其他一些參考酵母菌株的親緣關系。由實驗獲得的結果可以推斷此耐酸酵母的分類地位應屬于假絲酵母屬Candida sp.。同時對此菌進行生理生化鑒定,結果如表 2所示,可初步推測該耐酸酵母應屬于木蘭假絲酵母Candida magnolia。

圖1 DNAman軟件繪制菌株No.4的同源樹Fig. 1 Homology tree of strain No.4 drawn by DNAman.

表2 菌株No.4的生理生化特征Table 2 Physiological and biochemical characteristics of strain No.4
2.2.2 C. magnolia的乳酸耐受性
對耐酸酵母C. magnolia 進行乳酸耐受性實驗,將其分別接種至添加不同量乳酸的 YEPD培養基中,觀察其乳酸含量對此耐酸菌生長的影響,其結果如圖2所示,在乳酸濃度低于68 g/L (pH值約為2.5) 時,培養基中的乳酸對C. magnolia的生長影響較小,高于68 g/L時,菌體生長逐漸受到抑制,但此耐酸菌在乳酸濃度高達93.5 g/L時,仍能夠生長,其OD可達到6.5,說明此菌對乳酸具有極高的耐受性,為耐酸微生物。

圖2 乳酸含量對培養基pH值 (A) 及C. magnolia生長(B) 的影響Fig. 2 Effect of concentration of lactic acid on the pH of medium (A) and the growth of C. magnolia (B).
2.2.3 C. magnolia的有機酸利用分析
對耐酸酵母C. magnolia進行有機酸利用實驗,將其分別接種至篩選培養基和單碳源培養基中,發現在篩選培養基中生長旺盛,而在單碳源培養基中基本不能生長,且C. magnolia 對乳酸的利用率均低于5%。說明C. magnolia 能在營養豐富的高濃度乳酸條件正常生長,但不能在以乳酸為唯一碳源的培養基中生長。
2.3 產乳酸重組菌的構建
因為 C. magnolia 的耐乳酸而不利用乳酸的特性,本實驗設計以此菌為研究對象,通過表達米根霉乳酸脫氫酶基因,試圖將代謝流引向乳酸形成方向,以達到生產乳酸的目的。
2.3.1 質粒構建及酶切
按照 1.2.7構建質粒,得到表達載體 pYX212-kanMX-ldhA,將該質粒進行酶切驗證,結果如圖 3所示,證明質粒構建成功。

圖3 重組表達載體pYX212-kanMX-ldhA的鑒定Fig. 3 Identification of recombinant expression vector pYX212-kanMX-ldhA. 1: plasmid pYX212-kanMX digested with BamH I digestion; 2: identification of recombinant plasmid pYX212-kanMX-ldhA by PCR; 3: recombinant plasmid pYX212-kanMX-ldhA digested with BamH I and Hind III digestion; 4: recombinant plasmid pYX212-kanMX-ldhA digested with BamH I digestion; 5: λDNA/Pst I marker.
2.3.2 G418的敏感測定
重組菌的篩選要以G418抗生素為篩選壓力,因此需要測定原始耐酸酵母對G418抗生素的敏感性,按照 1.2.8進行實驗,發現能夠完全抑制原始耐酸酵母菌株C. magnolia生長的最低G418濃度為800 mg/L。因此在后續篩選轉化子實驗中,均在培養基中添加800 mg/L的G418來抑制原始菌的生長。
2.3.3 質粒pYX212-kanMX-ldhA的電轉化和篩選重組酵母轉化子
將質粒 pYX212-kanMX-ldhA電擊轉化進入感受態酵母C. magnolia 細胞中,轉化后的酵母涂布在含800 mg/L G418抗生素的YEPD平板上,按照1.2.9挑選轉化子進行菌落 PCR擴增,電泳驗證(PCR擴增ldhA片段)。結果發現菌株C. magnolia-2和C. magnolia-3均有一條大小約為1.0 kb的片段,與ldhA片段大小基本一致,而對照原始菌株則沒有,因此推測此條帶即為 ldhA片段。即 C. magnolia-2和 C. magnolia-3已成功轉入重組質粒 pYX212-kanMX-ldhA。
將經 PCR驗證的轉化子 C. magnolia-2、C. magnolia-3、及原始菌株 C. magnolia 進行細胞破碎,細胞破碎液稀釋適當濃度后進行SDS-PAGE電泳驗證,結果如圖4所示,轉化子C. magnolia-2和C. magnolia-3均在相對分子量約為43 kDa附近出現了明顯的蛋白表達條帶,與預期大小相一致,而原始菌C. magnolia 則未見此蛋白的表達。進一步證明了C. magnolia-2和C. magnolia-3為陽性轉化子,外源乳酸脫氫酶基因已得到表達。
2.4 產乳酸酵母發酵性能的研究
2.4.1 重組菌發酵產乳酸驗證實驗
為測定上述實驗所得重組菌的發酵能力,進行通風發酵產乳酸實驗。將重組菌C. magnolia-2和C. magnolia-3及原始菌株C. magnolia接種于發酵培養基中,其結果如圖 5所示,原始菌株不能利用葡萄糖進行發酵生產乳酸,而重組菌在發酵過程中明顯有乳酸生成,且隨著發酵過程的進行,乳酸逐漸得到積累。對C. magnolia-2和C. magnolia-3兩株重組菌的最終乳酸含量進行比較,發現C. magnolia-2最終乳酸含量略高于C. magnolia-3,因此在后續發酵試驗中選擇C. magnolia-2進一步研究。
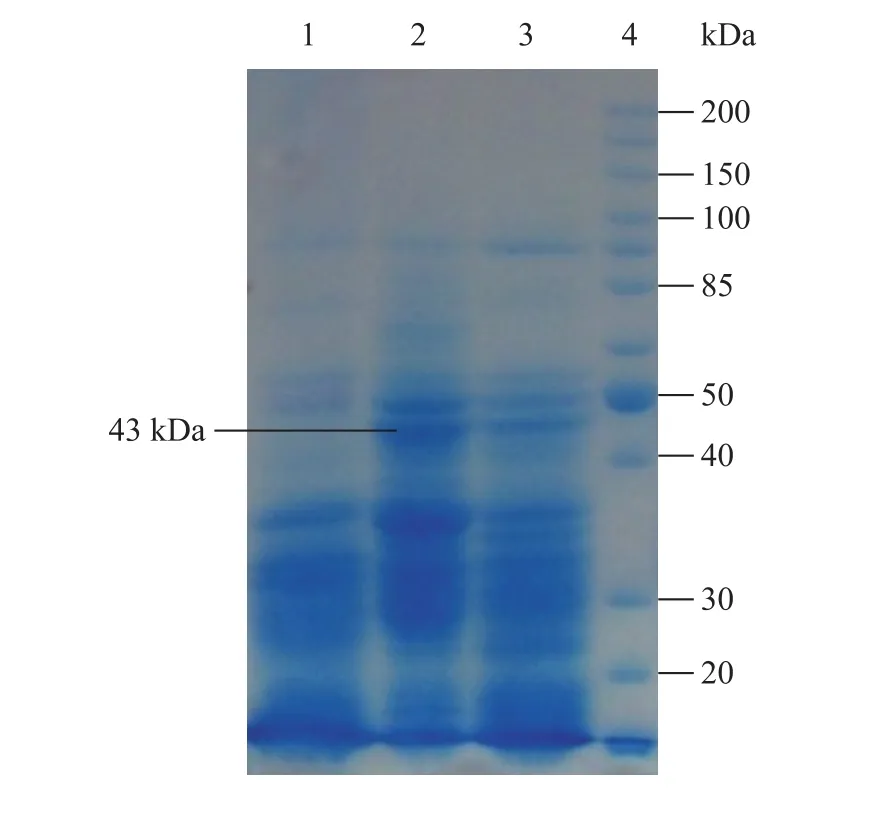
圖4 C. magnolia及其重組菌SDS-PAGE電泳驗證結果Fig. 4 SDS-PAGE of C. magnolia and its recombinant cells. 1: C. magnolia; 2: C. magnolia-2; 3: C. magnolia-3; 4: protein marker.
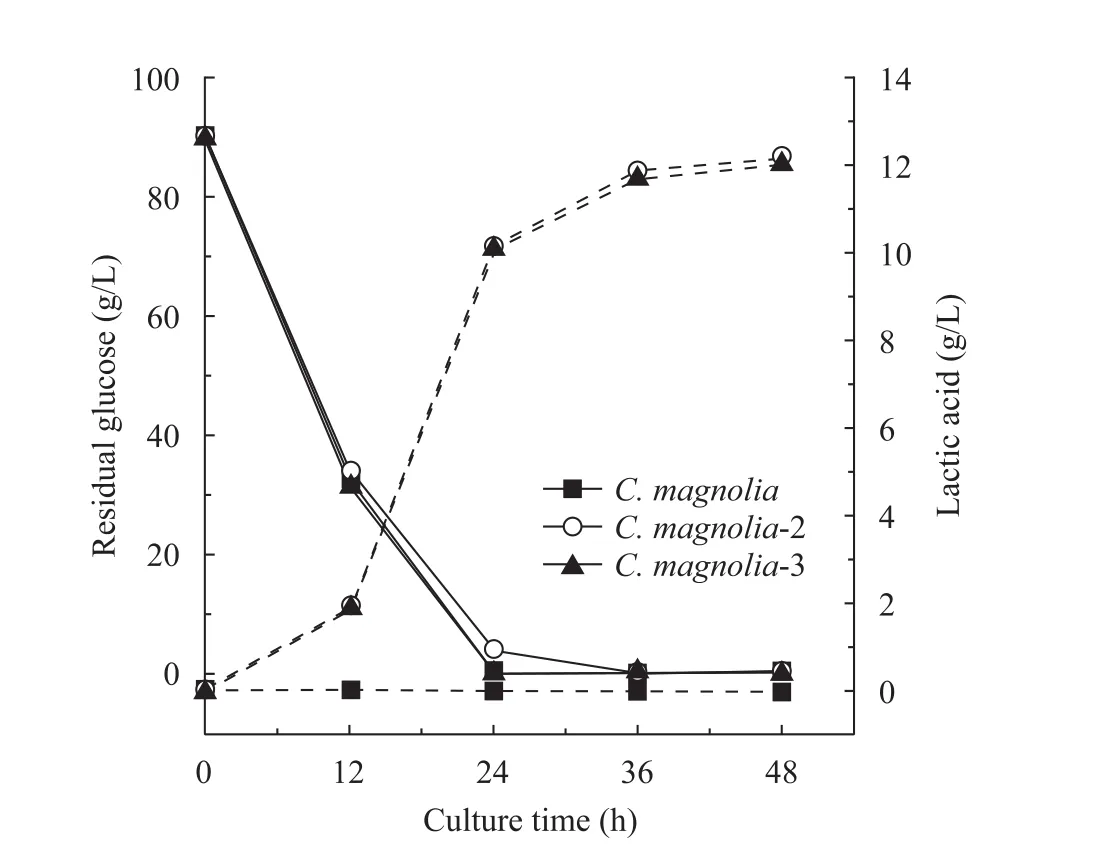
圖5 重組菌產乳酸性能的驗證Fig. 5 Different recombinants fermented in fermentation medium. Solid line represents the concentration of glucose, and dash line represents the concentration of lactic acid.
2.4.2 發酵液初始pH值對發酵產乳酸的影響
因宿主菌為耐酸菌,為研究此重組菌是否同樣可在酸性條件下具有發酵生成乳酸的能力,設計實驗將C. magnolia-2分別接種于pH值不同的發酵培養基。其結果如圖6所示,重組菌C. magnolia-2于培養基pH值范圍在2.5~7.0范圍內進行發酵,均能轉化葡萄糖生成乳酸,且在pH為3.5時糖酸轉化率最高,達到16%以上。

圖6 培養基初始pH值對發酵產乳酸的影響Fig. 6 Effect of initial pH on lactic acid production. LAC: lactic acid.
3 討論
在酵母產乳酸工程菌研究方面,國外已有報道。Skory等將米根霉的 L-乳酸脫氫酶基因轉入釀酒酵母中,在pH 5.5時糖酸轉化率最高時,在pH 3.5時,糖酸轉化率降低了約10%,產酸明顯受到了抑制[6];其他關于應用酵母生產L-乳酸的研究,絕大多數依舊需要以 CaCO3、NaOH或氨水調節,將發酵過程的pH值控制在4~6.6,才能使發酵正常進行[10-12],說明這些工程菌株在乳酸耐受性方面并沒有很大的優勢。
本研究首次從自然界中篩選得到一株耐高濃乳酸 (以乳酸調節pH值至2.5) 的耐酸酵母菌株,并且此耐酸菌株不能利用乳酸。對該菌進行分子生物學和生理生化鑒定后推斷其分類地位為木蘭假絲酵母C. magnolia。并首次以此為出發菌株,采用基因工程育種技術,在耐酸酵母中表達米根霉乳酸脫氫酶基因,構建乳酸代謝途徑,代謝葡萄糖生成乳酸,證明了利用基因工程技術使此耐酸酵母生產乳酸的可行性。重組菌在pH 2.5~7.0之間均能有效生成乳酸,在pH值為3.5時,糖酸轉化率最高,并且在發酵過程中pH值降低至2.5時,仍有乳酸繼續生成。因此,該重組菌在乳酸發酵過程中大幅減少碳酸鈣等中和劑用量方面表現出較好的應用前景;同時,在低 pH條件下可以一定程度上減少雜菌的污染幾率。
將本實驗所得的重組菌應用于乳酸發酵時,初步優化其培養條件,即可使乳酸產量達到40 g/L以上 (數據另發),表明重組菌株通過進一步的發酵工藝優化具有較好的提升空間。下一步工作中,為提高乳酸產量和轉化率,可在本文構建的菌株基礎上深入研究中間代謝產物積累與乳酸積累之間的關系,并確定其他可能的限速因素,如乙醇、甘油和NADH等對酵母乳酸代謝的影響,最終解決由于中間代謝產物的積累而導致乳酸產量較低的問題;對發酵條件如 pH值和溶氧等進行精確調節控制也可進一步提高發酵水平。同時,基于本文的研究策略,通過更換乳酸脫氫酶基因的啟動子等調控序列或通過增加拷貝數以增強其乳酸脫氫酶的酶活,將有可能進一步較大幅度提高乳酸發酵產率。
REFERENCES
[1] Maas RHW, Bakker RR, Jansen MLA, et al. Lactic acid production from lime-treated wheat straw by Bacillus coagulans: neutralization of acid by fed-batch addition of alkaline substrate. Appl Microbiol Biotechnol, 2008, 78(5): 751?758.
[2] Yun JS, Ryu HW. Lactic acid production and carbon catabolite repression from single and mixed sugars using Enterococcus faecalis RKY1. Proc Biochem, 2001, 37(3): 235?240.
[3] Wang LM, Zhao B, Liu B, et al. Efficient production of l-lactic acid from corncob molasses, a waste by-product in xylitol production, by a newly isolated xylose utilizing Bacillus sp. strain. Bioresour Technol, 2010, 101(20): 7908?7915.
[4] Wang DR. Development and use of lactic acid. Grain Proc,
2009, 34(1): 44?47.
汪多仁. L-乳酸的開發與應用進展. 糧食加工, 2009, 34(1): 44?47.
[5] Li J, Tang Y, Liang FL, et al. Cloning and function
analysis of L-lactate dehydrogenase gene from Lactobacillus sp. MD-1. Chin J Biotech, 2004, 20(5): 725?729.李劍, 赟
唐 , 梁鳳來, 等. L-乳酸脫氫酶基因克隆及功能分析. 生物工程學報, 2004, 20(5): 725?729.
[6] Jiang ST, Zheng Z, Zhu Y, et al. Repeated intermittent
L-lactic acid fermentation technology by self-immobilized Rhizopus oryzae. Chin J Biotech, 2008, 24(10): 1729?1733.
姜紹通, 鄭志, 朱羽, 等. 無載體固定化米根霉重復間歇發酵生產 L-乳酸. 生物工程學報, 2008, 24(10): 1729?1733.
[7] Cohn GC. Lactic acid producing yeast cells having nonfunctional L- or D-lactate: ferricytochrome c oxidoreductase gene: US, WO/2007/117282 A2. 2007-10-18.
[8] Yu L, Pei XL, Lei T. Study on L-lactic acid production by glucose-tolerant Lactobacillus rhamnosus GS2. Food Res Des, 2007, 28(11): 84?87.
于雷, 裴曉林, 雷霆. 耐糖鼠李糖乳桿菌發酵生產 L-乳酸的研究. 食品研究與開發, 2007, 28(11): 84?87.
[9] Xu GQ, Chu J, Wang YH, et al. Effects of different neutralizers on L(+)-lactic acid fermentation. Ind Microbiol, 2007, 37(4): 1?5.徐國謙, 儲炬, 王永紅, 等. 不同的中和劑對 L(+)-乳酸發酵的影響. 工業微生物, 2007, 37(4): 1?5.
[10] Dequin S, Barre P. Mixed lactic acid-alcoholic fermentation by Saccharomyes cerevisiae expressing the Lactobacillus casei L(+)-LDH. Biotechnology, 1994, 12(2): 173?177.
[11] Skory CD. Lactic acid production by Saccharomyces cerevisiae expressing a Rhizopus oryzae lactate dehydrogenase gene. J Ind Microbiol Biotechnol, 2003, 30(1): 22?27.
[12] Ishida N, Saitoh S, Tokuhiro K, et al. Efficient production of L-lactic acid by metabolically engineered Saccharomyces cerevisiae with a genome-integrated L-lactate dehydrogenase gene. Appl Environ Microbiol, 2005, 71(4): 1964?1970.
[13] De Hemptinne A, Marrannes R, Vanheel B. Influence of organic acids on intracellular pH. Am J Physiol, 1983, 245(3): 178?183.
[14] Barnett JA, Payne RW. Characteristics and Identification. Qingdao: Qingdao Ocean University Press, 1991.巴尼特JA, 佩恩RW. 酵母菌的特征與鑒定手冊. 青島:青島海洋大學出版社, 1991.
[15] Ausubel FM, Brent E, Kingston RE, et al. Short Protocol in Molecular Biology. Yan ZY, Wang HL, Translated. Beijing: Science Press, 1998.奧斯伯 FM, 布倫特 E, 金士頓 RE, 等. 精編分子生物學實驗指南. 顏子穎, 王海林, 譯. 北京: 科學出版社, 1998.

