差向異構結構域在非核糖體肽合成中的作用
薛永常 吳欣園 朱晨陽
摘要:差向異構結構域作為非核糖體肽合成酶體系中重要組分,將L型氨基酸異構化為D型氨基酸,賦予了非核糖體肽獨特的生物活性和對蛋白酶的抵抗力。本文結合近期研究闡述了差向異構結構域的蛋白結構特征和異構化過程中的去質子化/再質子化機制,此外還對差向異構結構域在非核糖肽合成中的調控作用進行了總結,展望了非核糖體肽合成酶的未來發展趨勢,希望為非核糖體肽合成酶的工程改造和新藥的研發提供理論基礎和依據。
關鍵詞:非核糖體肽合成酶;差向異構結構域;非核糖體肽;立體化學轉化;構象
中圖分類號:Q71文獻標志碼:A
The role of epimerization domain in the synthesis of nonribosomal peptides
Xue Yongchang, Wu Xinyuan, and Zhu Chenyang
(School of Biological Engineering, Dalian Polytechnic University, Dalian 116034)
Abstract As an important component in the system of nonribosomal peptide synthetase, the epimerization domain isomerizes L-amino acids into D-amino acids, giving nonribosomal peptides unique biological activity and resistance to protease. Combined with recent research, this paper describes the protein structural characteristics of the epimerization domain and the mechanism of deprotonation/re-protonation during isomerization. In addition, the regulatory role of epimerization domains in nonribosomal peptide synthesis is summarized, and the future development trend of nonribosomal peptide synthase is prospected, in order to provide a theoretical basis for the engineering modification of nonribosomal peptide synthase and development of new medicines.
Key words Nonribosomal peptide synthetases; Epimerization domain; Nonribosomal peptide; Stereochemical transformation; Conformation
自然界中微生物產生大量小分子生物活性肽和次級代謝物,其中大部分屬于非核糖體肽(nonribosomal peptide, NRP)類化合物。NRPs是由非核糖體肽合成酶(nonribosomal peptide synthetase, NRPS)催化合成的,含有蛋白源性氨基酸、脂肪酸和羥基酸以及D型氨基酸(D-amino acid, D-AA)等[1],具有抗真菌、抗病毒、抗癌等重要的藥理活性[2-4]。NRPS是一種大型多模塊酶,由功能不同的結構域組成相應的模塊,并按特定順序排列而成[5-8]。作為NRPS的修飾性結構域,差向異構(epimerization, E)結構域負責把連接在肽基載體蛋白(peptidyl carrier protein domain, PCP)上的L型氨基酸(L-amino acid, L-AA)立體異構化為D-AA,在非核糖體多肽上摻入非蛋白源性氨基酸,如短桿菌肽(gramicidin)A[9]。D-AA的摻入使NRPs蛋白水解敏感性降低,增強了NRPs的生物學活性[10],提高了NRPs的結構多樣性,因此對NRPs合成中E結構域的研究具有重要的實用價值。本文對近年來在E結構域的結構及其作用機制的研究進行綜述,希望為尋找新非核糖體肽類化合物合成方式及其新藥研發提供理論依據。
1 E結構域的發現
1968年首次在短桿菌肽S的產生菌短芽胞桿菌(Bacillus brevis)中發現了E結構域的存在[11],隨后Stachelhaus等[12]發現它在NRPs合成中獨特的立體異構活性,才將其正式命名為差向異構結構域。E結構域和縮合(condensation, C)結構域、環化結構域、Dual E/C結構域均是C結構域超家族成員,E結構域和C結構域有一定的同源性,存在一段相同的保守基序HHxxxDG,但催化殘基有細微差別[13],E結構域一般作為一個完整模塊中的第四個結構域,與同模塊的PCP結構域相連,進行氨基酸或多肽的異構化,其下游通常緊隨一個DCL結構域負責肽鏈的合成。E結構域根據不同模塊對結合底物的傾向性,分別在起始模塊和延伸模塊對氨酰基或肽基底物異構化[14],豐富了NRPs結構的多樣性。
2 E結構域的結構
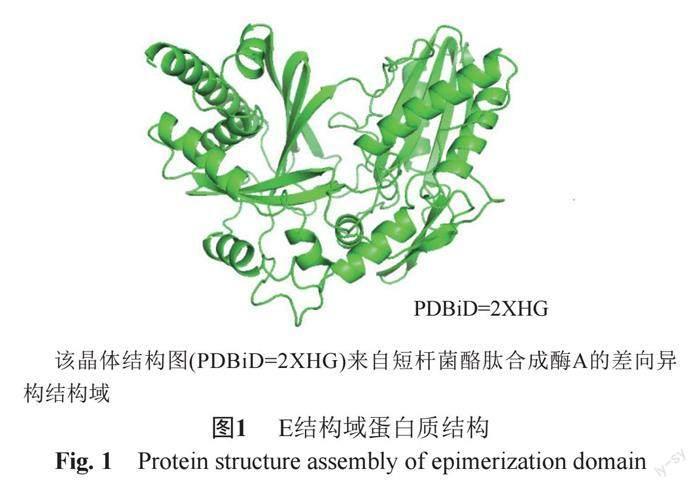
E結構域相對分子質量大約為50 ku[10],第一個E結構域晶體結構來自于短桿菌酪肽合成酶A(tyrocidine synthetase A, TycA),由結構相似、分別稱為N末端亞域和C末端亞域的兩部分組成(圖1),這兩個亞域都具有與氯霉素乙酰轉移酶(chloramphenicol acetyltransferase, CAT)類似的折疊結構。在每個亞域的中心都由一個大的β-折疊片層組成,兩側主要是兩側以α-螺旋序列為主,C末端亞域的中心由6個β-折疊組成,外側由9個α-螺旋依靠兩個延伸鏈覆蓋;N末端子域由5個β-折疊組成中心結構,并由5個α-螺旋和一個小β-折疊覆蓋,兩個亞域在連接處形成與PCP結合的口袋,整體結構表現為呈“V”形的偽二聚體,活性位點位于整個區域的中心,即兩個亞域的交界處[15]。比對GrsA的E結構域和弧菌素合成酶(vibriobactin synthetase)VibH的序列及其二級結構,發現二者存在極高的結構相似性,它們的活性位點表面都有一個相似的通道[16]。但E結構域和C結構域在底環區(floor loop)與橋區(bridge region)存在明顯差異 [15]。E結構域的底環區比C結構域多5個氨基酸殘基,在供體側呈一個鎖扣狀結構,與相鄰的PCP結構域相互作用。而E結構域橋區的這部分多肽鏈則對應于C結構域的受體位點,橋區在兩個類似CAT結構域形成的裂縫中扭曲構象,覆蓋了E結構域頂部的活性位點,阻斷其受體側,導致活性位點只能從對應于C結構域的供體側進入。因此,PCP結構域可與E結構域之間以一種類似于它與其下游C結構域供體側之間相互作用的方式互相影響,這反映了PCP結構域的多功能性,它不僅能與上游腺苷化(adenylation, A)結構域和下游C結構域相互作用,還能與E結構域相互作用,從而證實了PCP結構域在NRP合成過程中作為中間體穿梭的核心作用。
3 E結構域活性位點及作用機制
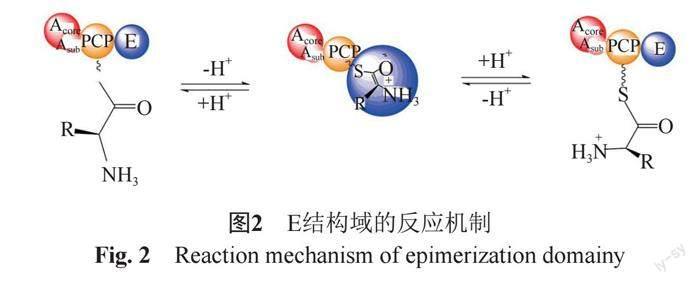
自E結構域被發現以來,對其生物化學研究較少,主要研究集中在探索其作用機制方面。Stachelhaus等[17]通過對短桿菌肽S合成酶(gramicidin S synthetase 1, GrsA)起始模塊中E結構域的保守氨基酸殘基誘變,發現其活性中心基序的H753A、D757S、D767S、E892A和Y976A對異構化的活性至關重要,其中H753A和D757S的突變致使E結構域失活,其余突變僅減緩了氨基酸異構化的速率。此外還在E結構域突變體上添加了放射性標記探索其質子轉移反應,發現了異構化過程中的去質子化/再質子化機制。E結構域活性表面存在一個與C結構域類似的通道,PCP的磷酸泛酰巰基乙胺(4-phos-phopantetheine, Ppant)臂能從E結構域的N端結合在此通道內,并在活性位點與L-氨酰基底物共價連接,催化氨基酸的Cα異構(圖2)。在GrsA的E結構域中,His753的咪唑側鏈與Ppant臂的硫原子相連,Tyr976、His894和Lys945為Ppant臂提供氫鍵型相互作用,殘基R896、D757和S760通過氫鍵相互作用,將Eα4固定在催化輔助位置,其結構指向Ppant臂的末端,通過α-螺旋的偶極矩為烯醇鹽中間體提供正靜電穩定性[18]。只有當E結構域與PCP的Ppant結合時,它才能夠催化氨基酸Cα消旋[19]。E結構域催化底物立體化學轉化,產生L型和D型異構體的平衡混合物,下游的DCL結構域特異性地識別D型異構體,促進其和下游受體氨酰基之間形成肽鏈[20-21]。
Samel等[15]和Chen等[18]均對異構化機理進行了討論,認為在異構化反應過程中L-AA的Cα質子與充當堿的殘基反應,產生一種烯醇中間體,充當酸的殘基為其提供質子形成D-AA。然而他們對組氨酸和谷氨酸的酸堿分類意見不一,因為這兩個殘基中的任何一個突變對異構化都有影響。Samel等[15]在研究TycA的PCP-E雙結構域時認為在沒有結合底物的生理條件下,His743殘基質子化,它的催化活性取決于與中間體的靜電相互作用,而不是酸堿催化。組氨酸不作為酸或堿,而是用來穩定瞬時出現的中間體,Glu882則更有可能是酸堿催化劑。但在突變實驗中發現谷氨酸突變能使Cα質子提取減少6倍,而組氨酸的突變卻能消除E結構域介導的Cα質子提取[17],所以Chen等[18]更傾向于認為組氨酸作為堿存在。
隨著探針技術日益成熟,其在研究NRPS各結構域間的相互作用方面發揮了巨大作用[22-23],Kim等[24]設計交聯探針探究GrsA中E結構域催化機制時發現了氯乙烯甘氨酸可作為PCP和E結構域交聯劑,但其易水解,僅具有適度的交聯性。隨后Kim等[25]又設計了磺酸鹽交聯探針,它可與谷氨酸、組氨酸、天冬氨酸等幾種氨基酸發生獨特的反應,可以輕松對其捕獲和檢測。研究發現,與組氨酸相比,谷氨酸更適合作為磺酰探針的主要親核試劑,即谷氨酸作為堿基催化異構化過程。
4 E結構域在NRPs生物合成中的作用
4.1 改變氨基酸構象
自然界中氨基酸存在L-AA和D-AA兩種不同的立體異構形式,L-AAs通常作為核糖體多肽合成的基本單元,D-AAs則多用于細胞壁肽聚糖的合成。與核糖體多肽不同,非核糖體多肽通常含有D-AAs, NRPS E結構域通過催化酶結合的氨酰基或肽基-S-Ppant的碳原子去質子化和再質子化,將L-AA轉化為D-異構體,參與非核糖體多肽生物合成,使得NRPS能合成含有D-AAs的新型抗癌抗菌類藥物,如來自海洋耐鹽發光細菌Staphylococcus aureus S2753生成的含D-Ala的新型NRP環縮肽Solonamide A和B對金黃色葡萄球菌毒力基因表達具較強抑制作用[26]。從海綿相關真菌Aspergillus similanensis KUFA 0013中分離出一種新的NRP環六肽similanamide (鄰氨基苯甲酸-L-Val-D-Leu-L-Ala-N-methyl-L-Leu-D-哌可酸)具有一定的抗癌活性[27]。Zheng等[28]在曲霉Aspergillus ustus 3.3904基因組中發現了一個oxepinamide類似物的生物合成基因簇,該基因簇合成的一種含有D-Phe、L-Ile、鄰氨基苯甲酰基的新型產物oxepinamide F可用于治療動脈粥樣硬化、糖尿病等疾病。E結構域的存在極大豐富了非核糖體多肽的種類,使其成為挖掘新抗生素類藥物的重要目標。
4.2 調節正確中間體的轉移
在細菌NRPS裝配系中,E結構域通常位于含有“通信”結構域(communication-mediating domain, COM)的C末端,它促進了酶分子間的有序相互作用和中間體的定向轉移。整合在延伸模塊中的E結構域僅在上游縮合作用完成后才異構肽基-S-Ppant底物的硫酯化殘基[29]。由于兩種異構體之間平衡的調節,在與下游結構域縮合之前,D-氨酰基或肽基-S-Ppant的排他性易位由下游C結構域的立體選擇性供體位點控制[30],因此差向異構化的時間是非常重要的。
在標準的NRPS延伸模塊中,氨酰基底物先與上游的C結構域縮合,然后才進行異構化。而起始模塊中由于缺乏上游的C結構域,氨酰基底物直接異構化,然后再進行縮合。延伸模塊中的E結構域具有一定的底物特異性,它優先作用于多肽[31],下游的C結構域會選擇含有D-AA的肽基作為其縮合反應的供體,這使得異構化反應的時間受到限制,從而確保了在延伸模塊中其過程不會發生異常[32]。
Stein等[14]在短桿菌酪肽合成酶B(tyrocidine synthetase B, TycB)重組雙模蛋白TycB2–3-AT·CATE/COMtycA的產物中發現E結構域更傾向于合成三肽L-Phe-D-Phe-L-Pro,TycB2–3-AT·CAT/EtycA與TycB1-CAT/TEsrf的相互作用,導致過量非必需二肽產物D-Phe-L-Pro-DKP的產生,說明了TycA的E結構域位于多模N-轉移酶的C-末端時會影響S-Ppant中間產物的有序轉移,且C-末端氨酰基和肽基-E結構域在相互作用和錯誤啟動上存在著顯著的功能差異。延伸模塊的E結構域(肽基-E結構域)在一定程度上優化肽鍵的形成、異構化以及調控中間轉移到下游模塊的進程,而起始模塊的E結構域(氨基酰-E結構域)則影響上游縮合并抑制多肽鏈的錯誤起始。因此E結構域的選擇對非核糖體肽生物組合工程的成功應用具有決定性意義。
4.3 調控PCP-E結構域連接子
在NRPS組裝的化學和動力學研究中,不同結構域間連接子在調控結構域間的相互作用上起著關鍵作用,PCP-E連接子區域便是如此。在Tyc6PCP-C雙結構域結構中,C結構域與PCP結構域之間的一個18個氨基酸組成的肽鏈顯示出相當大的構象靈活性,最后7個氨基酸從C結構域解離,并不與這兩個結構域相互作用[33]。而PCP-E結構域之間的連接子沿著E結構域的表面形成有序相互作用,其中兩個殘基對Arg613/Asp788和Arg614/Glu785對于連接子在E結構域上的定位以及PCP結構域在E結構域活性位點隧道的正確定位十分重要。E785R/D788R雙突變可以干擾連接子與E結構域表面的相互作用,證明了連接子區域在PCP-E雙結構域相互作用中的重要性[23]。關于PCP結構域與E/C結構域的供體位置相互作用以及E結構域的底物識別和催化等方面的研究尚不透徹,E結構域與連接區的具體作用還需進一步剖析。
5 展望
NRPs的發現和開發對制藥、食品、農業和環境科學領域產生了巨大影響。但NRPs不能完全滿足藥代動力學的需要,且隨著抗生素耐藥性的增強,NRPS的工程化改造,生產改性或新型化合物迫在眉睫。為滿足工業化需要,人們對NRPS進行了一系列改造。Zhang等[34]在利用定向進化策略改變A結構域的底物特異性的實驗中成功得到了比野生型催化效率高的突變體。Zobel等[35]重組白僵菌素合成酶、恩鐮孢菌素合成酶和環縮肽PF1022合成酶的激活模塊獲得了一個新型環六肽。Hacker等[36]重新調節NRPS通信結構域的相互作用豐富了其產物的長度多樣性。這些操作雖能生成一些預期的新產物,但由于酶的結構組織中的變化導致了其表達出現問題。即使成功表達,與野生型對應物相比,新型NRPs的產量也明顯降低或不具備可檢測的活性。因此提高重組NRPS生物合成效率成為NRPS改造工作首要問題。目前C結構域底物特異性和NRPS裝配線高級結構的研究受到廣泛關注,如何巧妙利用模塊特異性及模塊間相互作用來改造NRPS也是未來研究的重點。而鑒于NRPS系統和產品的多樣性,研究完整NRPS裝配線結構和功能表征亦是重中之重。在NRPs生物合成過程中,E結構域將L-AA異構化為D-AA,豐富了NRPs的功能和結構多樣性。近年來,對E結構域的催化機制和結構學方面的研究已有較好的進展,隨著晶體學的發展,利用化學探針和低溫電鏡等手段解析PCP結構域與E結構域供體位點相互作用及其催化狀態之間的動態轉變將是未來研究的熱點。通過對NRPs延伸過程的操控和對NRPS裝配線精細加工及模塊間相互作用的研究有助于揭示NRPs結構域間的作用機理,也對NRPs模塊操作和新型NRPs類藥物的研發有重要意義。
參 考 文 獻
Wheadon M J, Townsend C A. Evolutionary and functional analysis of an NRPS condensation domain integrates β-lactam, D-amino acid, and dehydroamino acid synthesis[J]. Proc Natl Acad Sci U S A, 2021, 118(17): e2026017118.
Zipperer A, Konnerth M C, Laux C, et al. Human commensals producing a novel antibiotic impair pathogen colonization[J]. Nature, 2016, 535(7613): 511-516.
Nikolouli K, Mossialos D. Bioactive compounds synthesized by nonribosomal peptide synthetases and type-I polyketide synthases discovered through genome-mining and metagenomics[J]. Biotechnol Lett, 2012, 34(8): 1393-1403.
Tavano R, Malachin G, De Zotti M, et al. The peculiar N- and C-termini of trichogin GA IV are needed for membrane interaction and human cell death induction at doses lacking antibiotic activity[J]. Biochim Biophys Acta, 2015, 1848(1): 134-144.
Jaremko M J, Davis T D, Corpuz J C, et al. Type II nonribosomal peptide synthetase proteins: Structure, mechanism, and protein-protein interactions[J]. Nat Prod Rep, 2020, 37(3): 355-379.
Bloudoff K, Fage C D, Marahiel M A, et al. Structural and mutational analysis of the nonribosomal peptide synthetase heterocyclization domain provides insight into catalysis[J]. Proc Natl Acad Sci U S A, 2017, 114(1): 95-100.
Duban M, Cociancich S, Leclère V. Nonribosomal peptide synthesis definitely working out of the rules[J]. Microorganisms, 2022, 10(3): 577.
Reimer J M, Haque A S, Tarry M J, et al. Piecing together nonribosomal peptide synthesis[J]. Curr Opin Struct Biol, 2018, 49: 104-113.
Allen T W, Andersen O S, Roux B. Structure of gramicidin A in a lipid bilayer environment determined using molecular dynamics simulations and solid-state NMR data[J]. J Am Chem Soc, 2003, 125(32): 9868-9877.
Süssmuth R D, Mainz A. Nonribosomal peptide synthesis-principles and prospects[J]. Angew Chem Int Ed Engl, 2017, 56(14): 3770-3821.
Yamada M, Kurahashi K. Adenosine triphosphate and pyrophosphate dependent phenylalanine racemase of Bacillus brevis Nagano[J]. J Biochem, 1968, 63(1): 59-69.
Stachelhaus T, Marahiel M A. Modular structure of peptide synthetases revealed by dissection of the multifunctional enzyme GrsA[J]. J Biol Chem, 1995, 270(11): 6163-6169.
Bloudoff K, Schmeing T M. Structural and functional aspects of the nonribosomal peptide synthetase condensation domain superfamily: Discovery, dissection and diversity[J]. Biochim Biophys Acta Proteins Proteom, 2017, 1865(11): 1587-1604.
Stein D B, Linne U, Hahn M, et al. Impact of epimerization domains on the intermodular transfer of enzyme-bound intermediates in nonribosomal peptide synthesis[J]. Chembiochem, 2006, 7(11): 1807-1814.
Samel S A, Czodrowski P, Essen L. Structure of the epimerization domain of tyrocidine synthetase A[J]. Acta Crystallogr D Biol Crystallogr, 2014, 70(5): 1442-1452.
Keating T A, Marshall C G, Walsh C T, et al. The structure of VibH represents nonribosomal peptide synthetase condensation, cyclization and epimerization domains[J]. Nat Struct Biol, 2002, 9(7): 522-526.
Stachelhaus T, Walsh C T. Mutational analysis of the epimerization domain in the initiation module PheATE of gramicidin S synthetase[J]. Biochemistry, 2000, 39(19): 5775-5787.
Chen W H, Li K, Guntaka N S, et al. Interdomain and intermodule organization in epimerization domain containing nonribosomal peptide synthetases[J]. ACS Chem Biol, 2016, 11(8): 2293-2303.
Stein T, Kluge B, Vater J, et al. Gramicidin S synthetase 1(phenylalanine racemase), a prototype of amino acid racemases containing the cofactor 4-phosphopantetheine[J]. Biochemistry, 1995, 34(14): 4633-4642.
Clugston S L, Sieber S A, Marahiel M A, et al. Chirality of peptide bond-forming condensation domains in nonribosomal peptide synthetases: The C5 domain of tyrocidine synthetase is a DCL catalyst[J]. Biochemistry, 2003, 42(41): 12095-12104.
Luo L, Burkart M D, Stachelhaus T, et al. Substrate recognition and selection by the initiation module PheATE of gramicidin S synthetase[J]. J Am Chem Soc, 2001, 123(45): 11208-11218.
Shi C, Miller B R, Alexander E M, et al. Design, synthesis, and biophysical evaluation of mechanism-based probes for condensation domains of nonribosomal peptide synthetases[J]. ACS Chem Biol, 2020, 15(7): 1813-1819.
Miyanaga A, Kurihara S, Chisuga T, et al. Structural characterization of complex of adenylation domain and carrier protein by using pantetheine cross-linking probe[J]. ACS Chem Biol, 2020, 15(7): 1808-1812.
Kim W E, Patel A, Hur G H, et al. Mechanistic probes for the epimerization domain of nonribosomal peptide synthetases[J]. Chembiochem, 2019, 20(2): 147-152.
Kim W E, Ishikawa F, Re R N, et al. Developing crosslinkers specific for epimerization domain in NRPS initiation modules to evaluate mechanism[J]. RSC Chem Biol, 2022, 3(3): 312-319.
Mansson M, Nielsen A, Kj?rulff L, et al. Inhibition of virulence gene expression in Staphylococcus aureus by novel depsipeptides from a marine Photobacterium[J]. Mar Drugs, 2011, 9(12): 2537-2552.
Prompanya C, Fernandes C, Cravo S, et al. A new cyclic hexapeptide and a new isocoumarin derivative from the marine sponge-associated fungus Aspergillus similanensis KUFA 0013[J]. Mar Drugs, 2015, 13(3): 1432-1450.
Zheng L, Wang H, Fan A, et al. Oxepinamide F biosynthesis involves enzymatic d-aminoacyl epimerization, 3H-oxepin formation, and hydroxylation induced double bond migration[J]. Nat Commun, 2020, 11(1): 4914.
Linne U, Marahiel M A. Control of directionality in nonribosomal peptide synthesis: Role of the condensation domain in preventing misinitiation and timing of epimerization[J]. Biochemistry, 2000, 39(34): 10439-10447.
Belshaw P J, Walsh C T, Stachelhaus T. Aminoacyl-CoAs as probes of condensation domain selectivity in nonribosomal peptide synthesis[J]. Science, 1999, 284(5413): 486-489.
Stein D B, Linne U, Marehiel M A. Utility of epimerization domains for the redesign of nonribosomal peptide synthetases[J]. FEBS J, 2005, 272(17): 4506-4520.
Luo L, Kohli R M, Onishi M, et al. Timing of epimerization and condensation reactions in nonribosomal peptide assembly lines: Kinetic analysis of phenylalanine activating elongation modules of tyrocidine synthetase B[J]. Biochemistry, 2002, 41(29): 9184-9196.
Samel S A, Schoenafinger G, Knappe T A, et al. Structural and functional insights into a peptide bond-forming bidomain from a nonribosomal peptide synthetase[J]. Structure, 2007, 15(7): 781-792.
Zhang K, Nelson K M, Bhuripanyo K, et al. Engineering the substrate specificity of the DhbE adenylation domain by yeast cell surface display[J]. Chem Biol, 2013, 20(1): 92-101.
Zobel S, Boecker S, Kulke D, et al. Reprogramming the biosynthesis of cyclodepsipeptide synthetases to obtain new enniatins and beauvericins[J]. Chembiochem, 2016, 17(4): 283-287.
Hacker C, Cai X, Kegler C, et al. Structure-based redesign of docking domain interactions modulates the product spectrum of a rhabdopeptide-synthesizing NRPS[J]. Nat Commun, 2018, 9(1): 4366.

