陸地棉動態株高與SSR標記的關聯分析
文 佳,黃陳玨,嵇子涵,李黎貝,馮 震,喻樹迅
(浙江農林大學現代農學院,浙江臨安 311300)
0 引 言
【研究意義】棉花是重要的經濟作物之一[1-3]。棉花株高與棉花產量、抗倒伏和機械化等密切相關[4]。株高過高會增加作物倒伏的風險,過低則影響生物量,降低光合產物的積累。因此,培育合適株高的棉花品種對棉花高產穩產具有重要意義。【前人研究進展】Wang等[5]較早利用重組自交系群體,對株高等性狀進行數量性狀位點(quantitative trait locus,QTL)定位,獲得了3個與株高性狀相關的QTL位點。王新坤等[6]以矮稈突變體為親本,構建分離群體,采用復合區間作圖法進行QTL定位,共檢測到4個與株高相關的QTL,其中qPH-LG6-1表型貢獻率最大,為47.98%,顯著大于前人所得株高QTL的貢獻率。何蕊等[7]通過對408個單株進行SSR鑒定,發現了16個與株高性狀相關的QTL,其中有7個在2個及以上環境中被檢測到,這些在多環境檢測到的QTL表現出很好的穩定性。Shang等[8]以重組自交系以及后代作為材料對兩個環境的棉花株高以及多個發育階段的雜種優勢進行了動態分析,在2個環境中分別獲得41和23個株高相關的QTL位點,可以更綜合、更全面地了解控制該性狀位點的動態表達。Ma等[9]探索了不同發育階段下棉花株高相關的動態QTL,通過部分顯性和超顯性及上位效應解釋了陸地棉株高的遺傳基礎。全基因組關聯分析(genome-wide association study,GWAS)是復雜數量性狀研究的主要方法,能以較高的分辨率和靈敏度識別出目標性狀關聯的候選基因[10-11]。相比于常規QTL,GWAS具有無需構建作圖群體、可以同時對多個等位基因進行分析、利用群體長期進化中的重組信息使定位分辨率更高等優點。Su等[12]對355份陸地棉組成的自然群體在6個環境下的株高性狀進行了調查,通過GWAS得到8個與株高性狀顯著相關的SNP位點,最終定位到與株高相關的候選基因Gh_D03G0922。Ji等[13]發現在棉花矮化突變體AS98中HDPA-D12位點拷貝變異編碼了一個反應元件結合蛋白轉錄因子GhDREB1B,研究表明GhDREB1B過表達會使棉花株高顯著降低。【本研究切入點】植物株高是一個動態發育性狀,受多個主效基因和微效基因控制[14]。雖然前人通過GWAS方法開展了棉花株高的遺傳研究已有較多報道,但關于研究棉花動態株高方面的報道較少[10-11,15]。需利用棉花自然群體對動態株高性狀進行關聯分析。【擬解決的關鍵問題】以185份陸地棉品種組成自然群體,對PH-ST1和PH-ST2進行表型鑒定,結合137對SSR多態性引物擴增出的355個等位變異,利用GLM和MLM模型對開花期和吐絮期的株高及標記進行關聯分析,發掘不同時期與株高顯著相關的SSR標記,為培育棉花株型新品種提供理論基礎。
1 材料與方法
1.1 材 料
選取185份陸地棉品種(系),其中170份為中國品種(分屬于我國長江流域棉區、黃河流域棉區、西北內陸棉區和北部特早熟棉區);15份材料來自于美國、阿塞拜疆等國家。
1.2 方 法
1.2.1 試驗設計
分別在中國海南三亞(18°29′N,109°52′E)(2020年和2021年)種植2個重復;中國湖北黃岡(30°57′N ,114°92′E)(2021年)、中國山東臨清(36°48′N ,115°41′E)(2021年)種植3個重復,每重復選取9株長勢一致的植株測量株高。田間試驗均采取隨機區組試驗設計,單行根據當地栽培方式種植。中國海南三亞:行長3 m, 行距為0.5 m,株距0.2 m ;中國湖北黃岡:行長6 m,行距為0.8 m, 株距 0.4 m ;中國山東臨清:行長5 m,行距為0.6 m ,株距0.3 m 。
1.2.2 SSR標記及基因型分型
采用改良CTAB法[16]提取棉花葉片基因組,所用試劑從南京諾唯贊生物科技有限公司購買。對185份陸地棉品種(系)自然群體進行基因型檢測,檢測到355個等位變異[17]。137對SSR引物由實驗室選取,均勻覆蓋陸地棉26條染色體上[17],所有SSR引物由生工生物工程(上海)股份有限公司合成。
1.2.3 關聯分析
群體結構分析參照文獻[19]的數據,用矩陣“Q”作為協變量來降低假陽性率。采用Tassel 3.0軟件計算親緣關系矩陣(K),通過一般線性模型(general linear model,Q)和混合線性模型(mixed linear model,Q+K)2種方法分別對5個環境下不同時期株高的表型數據與SSR標記進行關聯分析。參考Bresghellohe等[18]提出的無效等位變異(null allele)計算方法,分析棉花株高表型效應值。參照Evanno等[19]方法,計算ΔK來確定適宜的亞群數。
1.3 數據處理
使用軟件R對開花期和吐絮期株高數據進行描述性統計分析,包括平均值、極大值、極小值、標準差和變異系數。參照Knapp等[20]方法計算廣義遺傳力(h2)。采用R語言中lme4包[21]計算出多環境下不同時期株高的最佳線性無偏預測(best linear unbiased prediction,BLUP)分析4個環境下的表型數據,獲得2個時期BLUP值作為第5個環境下的陸地棉表型數據。
2 結果與分析
2.1 棉花動態株高表型差異
研究表明,4個環境中,PH-ST1平均值分別為58.45、46.93、60.02和59.92 cm,標準差分別為8.72 、5.84 、7.95 和7.49 ,變異系數介于12.45%~14.56%;PH-ST2平均值分別為66.21、98.05、71.66和88.30 cm,標準差分別為11.14、18.07 、10.61 和15.78 ,變異系數介于14.81%~18.43%。不同時期株高的基因型、基因型與環境互作變異方差均達到極顯著水平;PH-ST1廣義遺傳率為63.67%,PH-ST2廣義遺傳率為85.66%。PH-ST1和PH-ST2的遺傳力穩定且比較高,主要受基因型的影響,適合關聯分析。表1
2.2 群體結構差異
研究表明,K=2時,ΔK出現最大值,即185份材料從遺傳結構上可被分為2類亞群。其中,一個亞群包含了95份材料,約占總材料的51%;另一個亞群包含了90份材料,約占總材料的49%。在Structure軟件中,當K=2時,獲得了每個材料的Q值,將得到的Q矩陣作為關聯分析的協變量,消除群體結構對關聯分析結果的影響。圖1
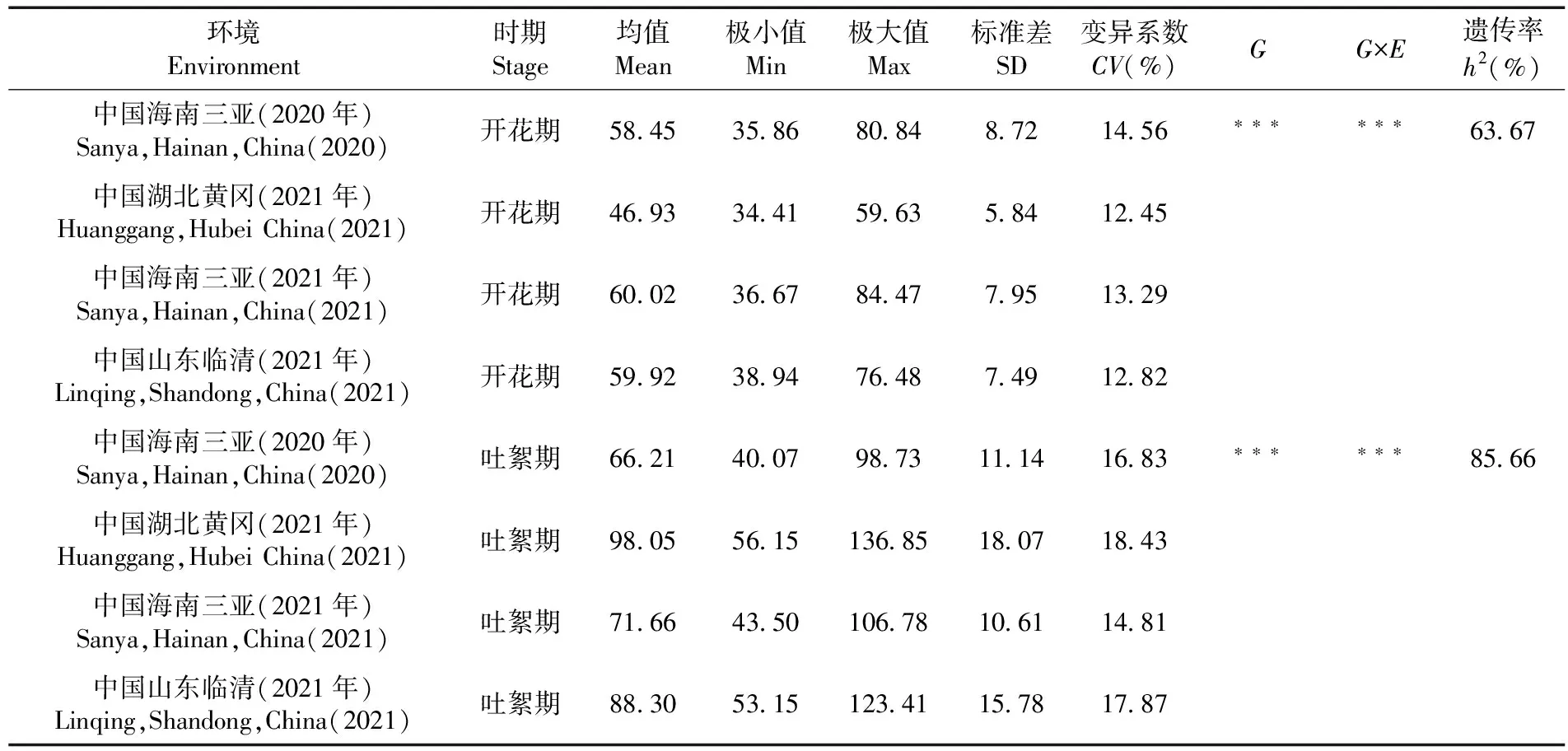
表1 185份陸地棉材料在不同環境下株高性狀
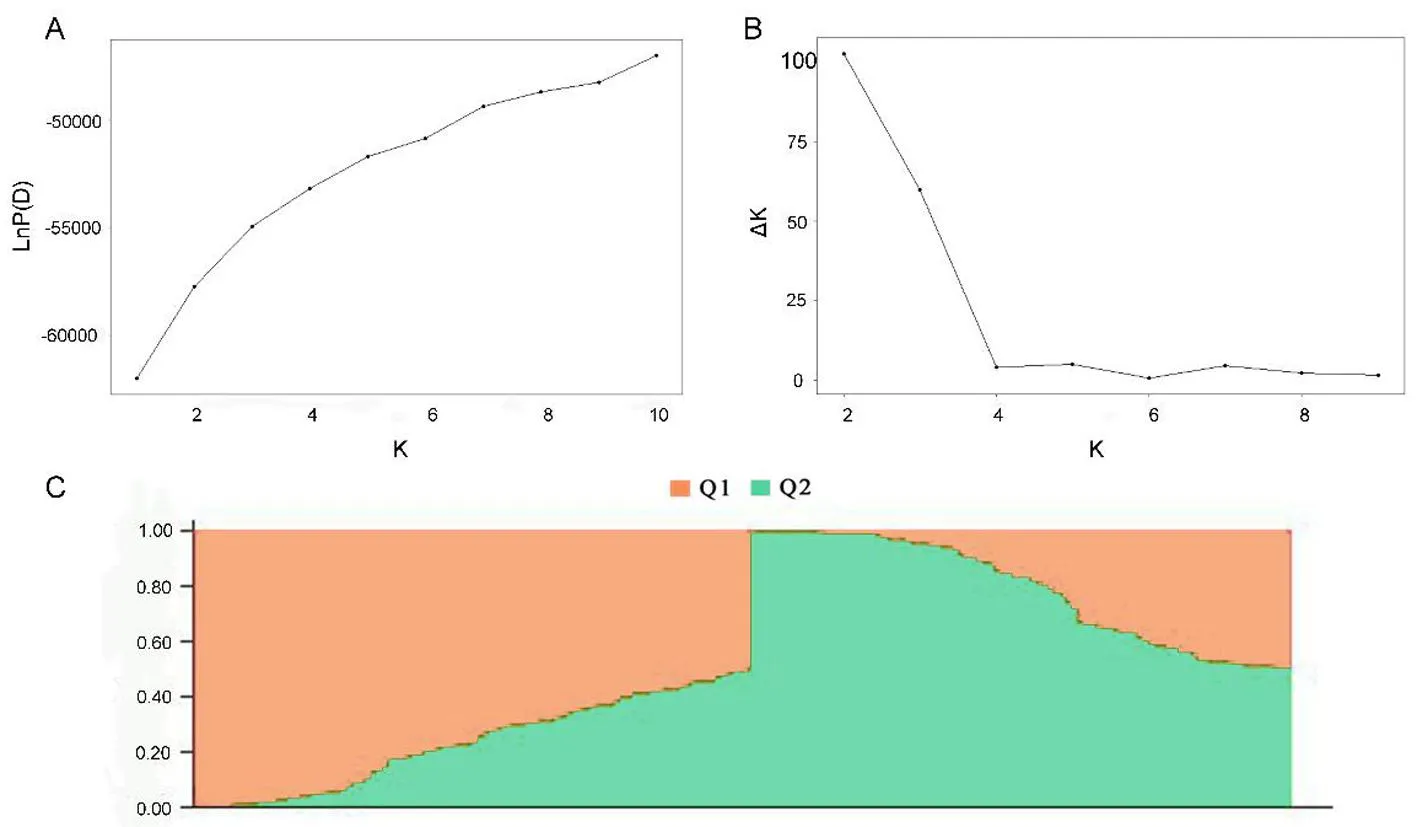
圖1 185份陸地棉材料的群體結構Fig.1 The population structure of 185 upland cotton accessions
2.3 基于GLM模型下動態株高的關聯性
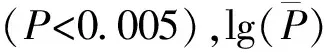
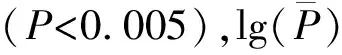
2.4 基于MLM模型下動態株高的關聯性
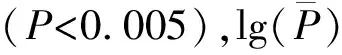
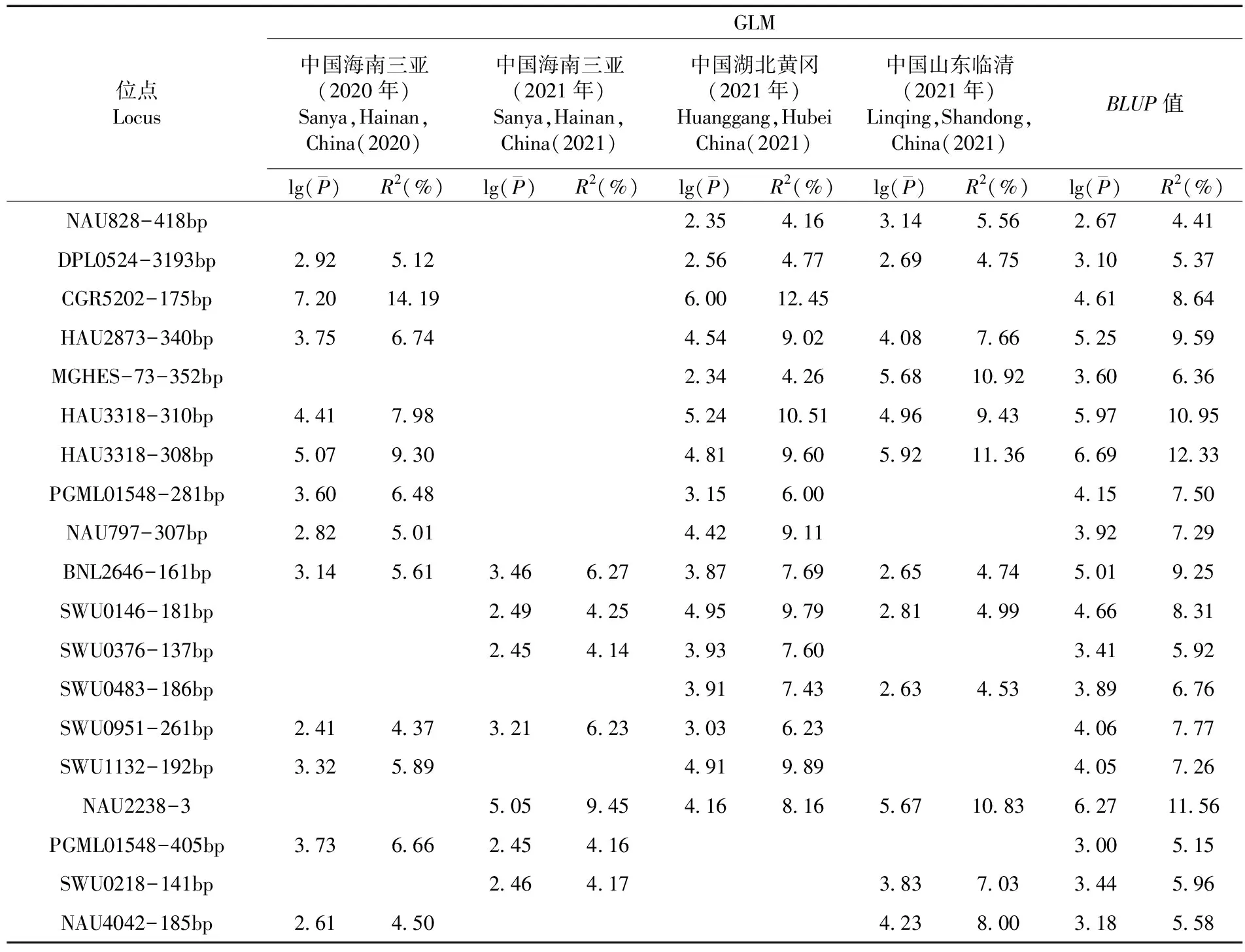
表2 基于GLM模型在3個及以上環境的PH-ST1關聯性
2.5 顯著位點表型貢獻率及效應
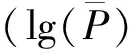
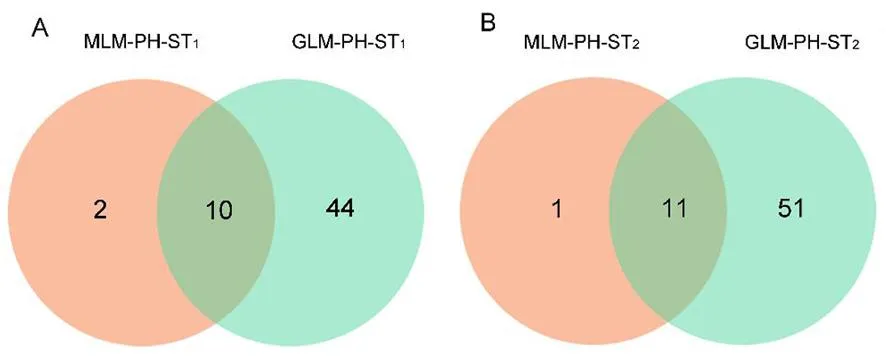
圖2 在GLM和MLM模型下不同時期株高的SSR位點韋恩圖Fig.2 Venn diagrams of SSR locus associated with plant height at different stages based on GLM and MLM model
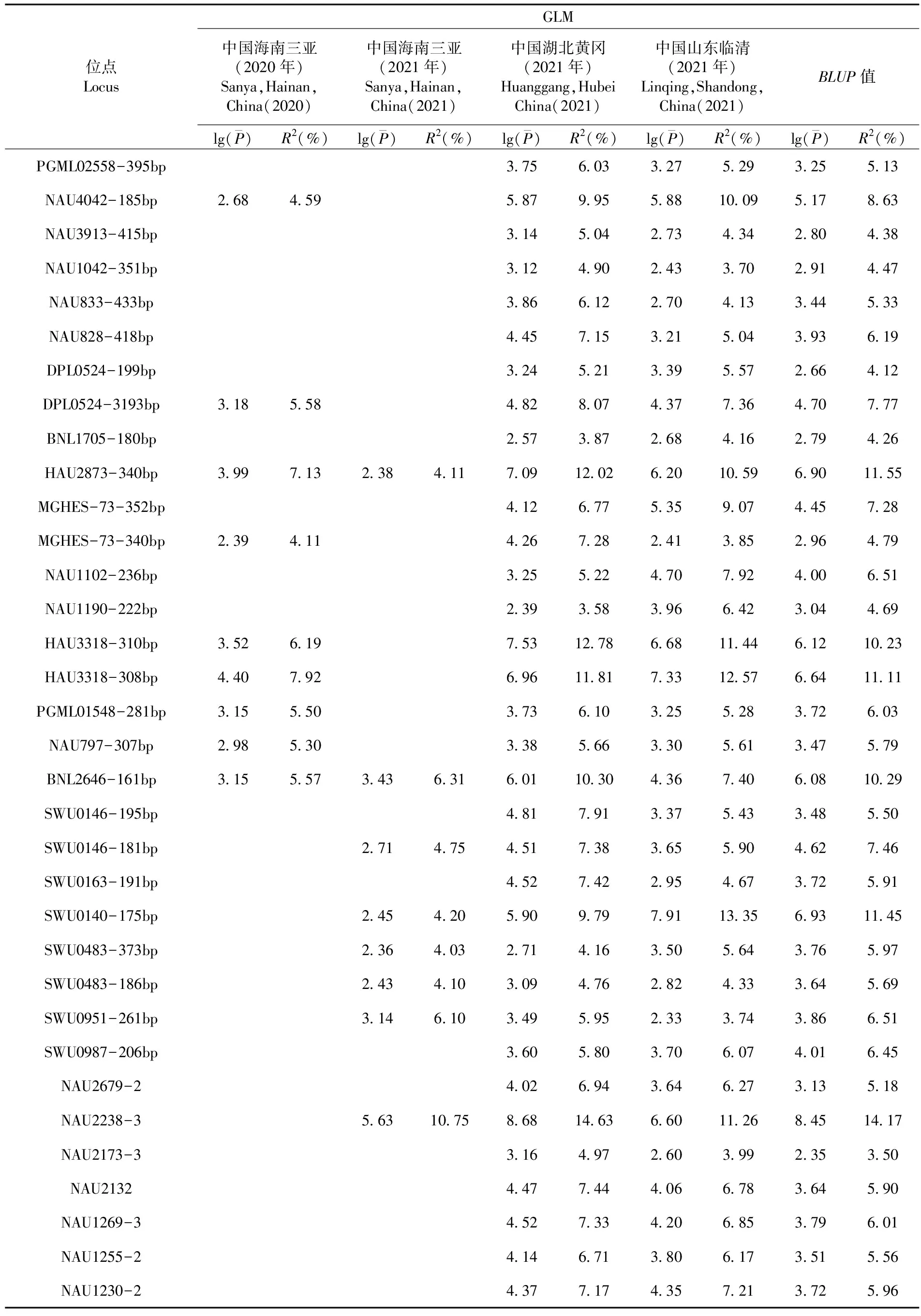
表3 基于GLM模型在3個及以上環境的PH-ST2的關聯性
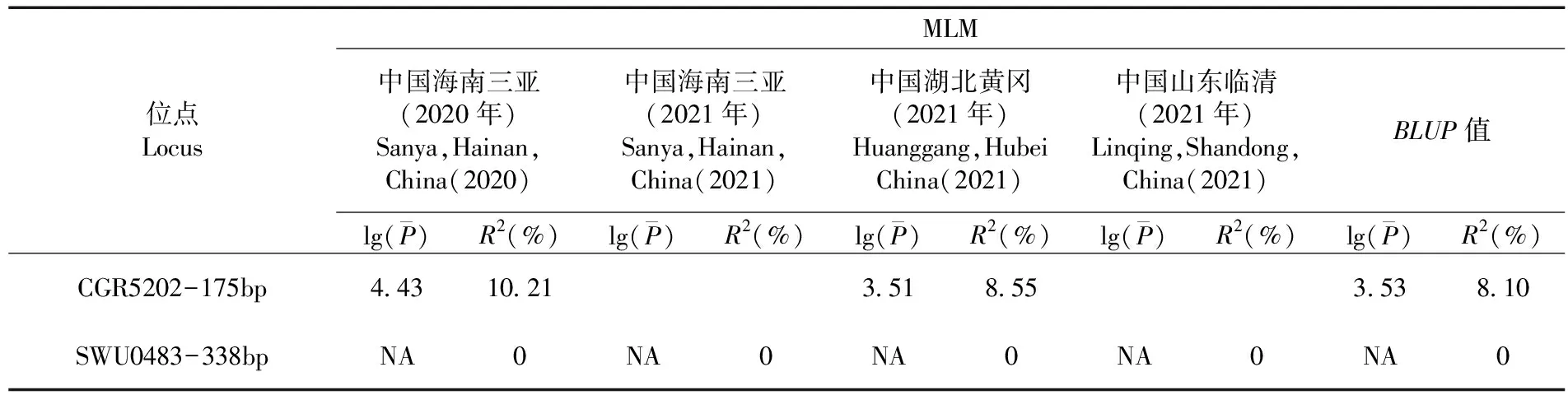
表4 基于MLM模型在3個及以上環境的PH-ST1關聯性
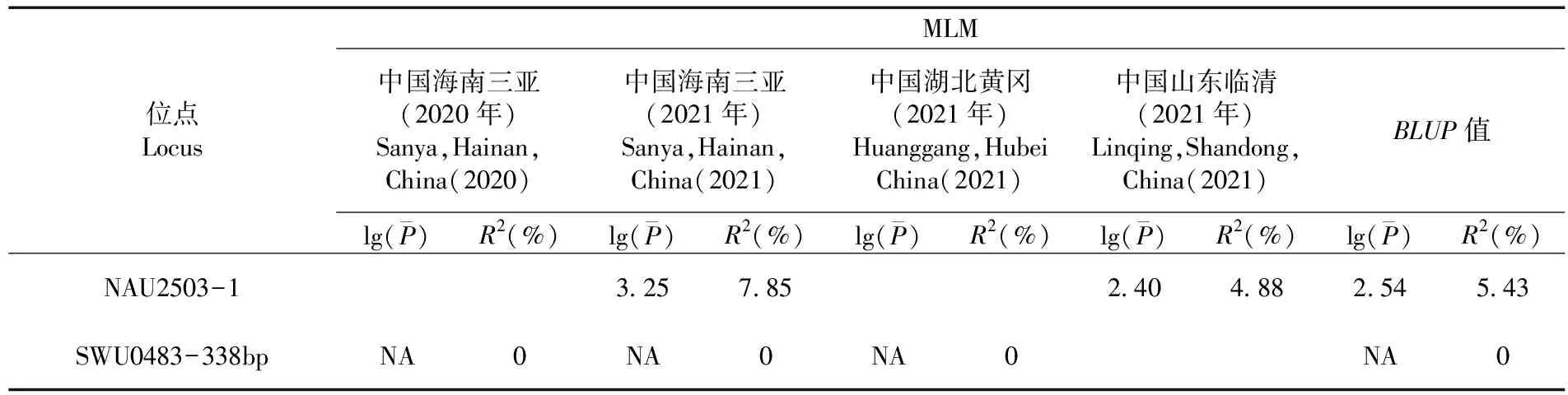
表5 基于MLM模型在3個及以上的環境的PH-ST2的關聯性
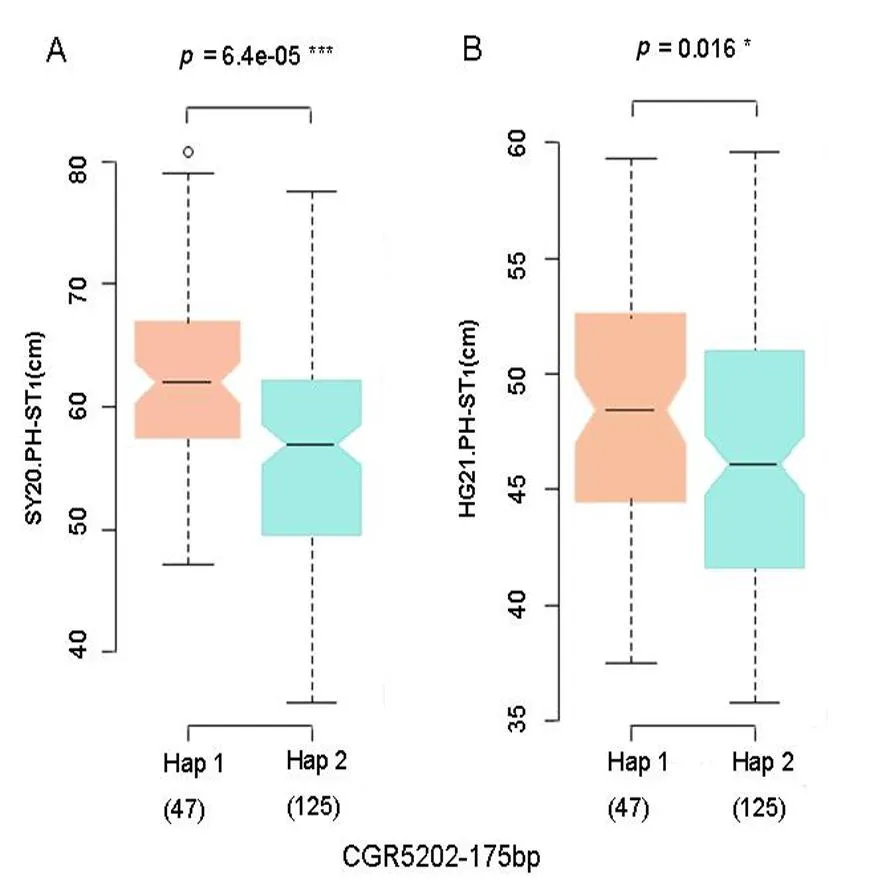
注:Hap 1:不含有CGR5202-175bp位點的材料;Hap 2:含CGR5202-175bp位點的材料
貢獻率最高的位點CGR5202-175bp在2020年中國海南三亞和2021年中國湖北黃岡環境下都能與PH-ST1表型顯著關聯到,其中125份材料含有該位點,47份材料不含該位點,含該位點的材料的株高平均值要顯著低于不含有該位點的材料的株高平均值。與PH-ST2相關的貢獻率最高的位點為NAU2238-3,且在2021年中國海南三亞、中國湖北黃岡、中國山東臨清環境下能顯著關聯到。其中,153份材料不含該位點,27份材料含該位點,含該位點的材料的株高平均值要顯著低于不含有該位點的材料的株高平均值。圖3,圖4,圖5
3 討 論
3.1 分子標記在作物育種中的作用
隨著分子標記技術的迅速發展,不僅加快了育種進程,同時也為復雜的數量性狀的遺傳分析提供了基礎[22]。傳統分子標記主要有以下幾種:限制性長度多態性(restriction fragment length polymorphism,RFLP)、擴增片段長度多態性 (amplified fragment length polymorphism,AFLP)、SSR等。
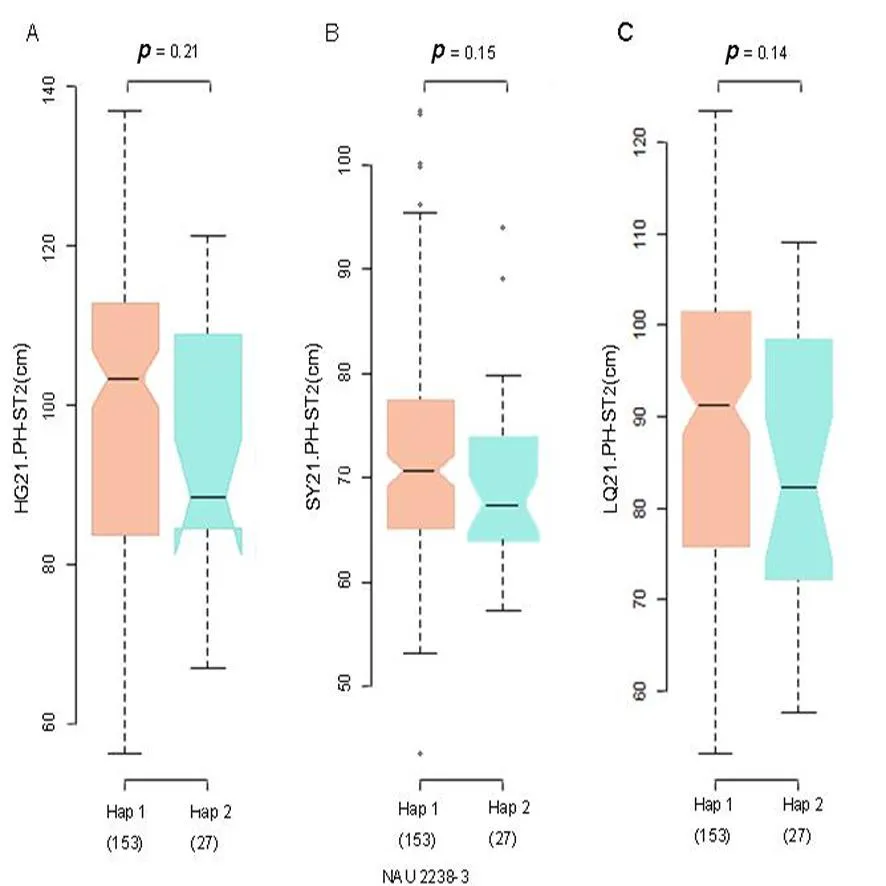
Hap 1:不含有NAU2238-3位點的材料;Hap 2:含NAU2238-3位點的材料
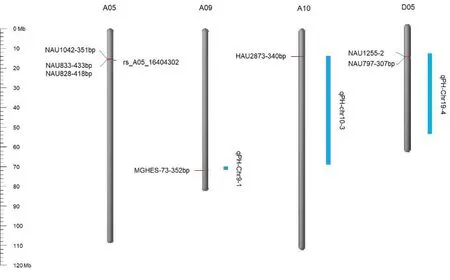
注:藍色代表前人定位到的QTL,紅色代表位于前人定位QTL 1Mb范圍內的SSR位點
RFLP和AFLP已成功應用于許多物種的遺傳多樣性分析,但對棉花多倍體基因組等大型基因組進行基因分型時效率較低[23]。相比于RFLP和AFLP,SSR標記具有多態性高、可重復性強、易于轉化和穩定可靠等優點[24-25]。與前人研究與株高性狀相近顯著關聯的位點:位點NAU833-433bp、染色體A05、物理位置15535921.15536120[26],位點NAU828-418bp、染色體A05、物理位置15535921.15536120[26],位點NAU1042-351bp、染色體A05、物理位置15535848.15536094[26],位點MGHES-73-352bp、染色體A09、物理位置72905477.72905691[9],位點HAU2873-340bp、染色體A10、物理位置12349682.12350027等[8],位點NAU797-307bp、染色體D05、物理位置14213616.14213809等[9],位點NAU1255-2、染色體D05、物理位置14213543.14213783[8,9]。研究利用185份陸地棉品種(系)構成的自然群體為材料,選取了137對SSR多態性引物,對5個環境的PH-ST1和PH-ST2進行關聯分析。由于研究篩選的SSR引物密度較低,導致標記之間的遺傳區域較大,難以快速挖掘目標位點和基因。因此,后續可根據結果劃定關聯區域,在關聯區域結合基因功能注釋開發高密度的單核苷酸多態性(single nucleotide polymorphism,SNP)標記,得到與目標性狀緊密連鎖的標記位點,并應用到相關性狀的輔助選擇中,加快育種進程。
3.2 動態株高的分析在作物育種中的作用
研究結果與前人研究共同定位到株高性狀相關的位點有7個,新定位到與株高性狀相關的位點有24個。shang等[8]通過構建遺傳圖譜并結合不同時期株高的表型數據進行QTL定位分析,檢測到的qPH-chr10-3和qPH-chr19-4分別與研究鑒定的顯著關聯位點HAU2873-340bp 和NAU1255-2位置相近;NAU797-307bp、MGHES-73-352bp 位點與Ma等[9]QTL定位得到的株高QTL位點qPH-Chr19-4、qPH-Chr9-1的位置相近,NAU1255-2位點與qPH-Chr19-4中NAU3437標記重合;Li等[26]研究發現了與赤霉素生物合成途徑相關的基因GhGA2Ox8,在改良植物株型方面顯示出良好的潛力,能夠使冠層更加致密,適合高密度種植,提高棉花產量,定位到該基因的 SNP位點與研究NAU833-433bp、NAU828-418bp、NAU1042-351bp位點物理位置小于1 Mb。該7個位點可能是調控棉花株高的關鍵位點,但還需后續進一步驗證。
3.3 利用GLM和MLM模型鑒定動態株高的主要位點
全基因組關聯分析是解析作物復雜數量性狀遺傳結構的一個重要的工具[27-30]。研究同時采用 GLM模型和MLM模型進行關聯分析[31-32]。研究結果表明無論是單個環境還是多個環境下所關聯出來的結果,MLM模型所關聯到的SSR位點始終少于GLM模型,劉其寶等[17]、王龍等[33]也得出相似的結論,MLM模型的假陽性率低于GLM模型。研究中發現與PH-ST1相關聯的CGR5202-175bp位點和與PH-ST2相關聯的NAU2238-3位點能在多個環境中同時被檢測到且貢獻率大于10%,且在前人研究結果中尚未出現。今后可通過構建分離群體對CGR5202-175bp和NAU2238-3位點進一步驗證,提高結論的準確性。
4 結 論