‘月月粉’月季NAC家族全基因組鑒定及皮刺發育相關成員的篩選
由玉婉,張雨,孫嘉毅,張蔚
‘月月粉’月季NAC家族全基因組鑒定及皮刺發育相關成員的篩選
由玉婉,張雨,孫嘉毅,張蔚
華中農業大學園藝林學學院/園藝植物生物學教育部重點實驗室/農業農村部華中都市農業重點實驗室, 武漢 430070
從‘月月粉’月季全基因組中鑒定NAC家族成員,并進行生物信息學和表達模式分析,為研究‘月月粉’的潛在功能提供理論基礎,篩選可能參與皮刺發育的候選基因。以擬南芥NAC氨基酸序列為參考,利用雙向Blast及HMM結構域檢索篩選RcNAC;對篩選成員進行理化性質、亞細胞定位、序列特征、順式元件和系統進化分析;基于已釋放轉錄組數據,分析在不同組織器官,和不同逆境處理條件下的表達特性;同時通過‘月月粉’不同發育階段皮刺組織轉錄組測序,篩選與皮刺發育可能相關的。共鑒定得到116個s,均含有完整典型的NAM保守結構域,其編碼蛋白包含69—713個氨基酸,等電點從4.43—9.54,分子質量從7.87—79.99 kD,81個RcNACs被預測定位于細胞核內;115個s在7條染色體上不均勻分布,1個未明確位置信息;系統進化樹分析將AtNACs、OsNACs和RcNACs聚為21類;在不同組織器官中,116個s表達模式各異,遭受水脅迫和感染灰霉病之后,31個s表達量發生變化;在衰老的花組織中,6個s表達量上升;皮刺轉錄組數據中檢測到53個s,其中26個s為差異表達基因。基于已有轉錄組數據分析,s協同調控植物組織發育及逆境脅迫響應;結合皮刺轉錄組數據,部分成員可能參與皮刺細胞增殖、次生細胞壁生物合成及細胞程序性死亡等過程,可作為皮刺發育相關候選基因進行深入研究。
‘月月粉’;NAC轉錄因子;轉錄組測序;皮刺發育
0 引言
【研究意義】月季(sp.)是薔薇科(Rosaceae)薔薇屬(L.)的一個重要類群,因其色彩絢麗、花型優美、花香馥郁,且連續開花,在世界各地被廣泛種植和栽培,具有極高的觀賞價值和經濟價值。然而,月季莖桿多著生皮刺,對月季的田間管理、采后運輸及園林應用等造成了極大不便,并帶來了安全隱患。因此,培育無刺或軟刺品種是月季育種的重要目標之一。對中國古老月季品種‘月月粉’(Old Blush)NAC家族基因進行全面的生物信息學分析和表達特性研究,有利于深入了解NAC家族基因的功能,并對月季品種的遺傳改良具有重要的參考意義。【前人研究進展】已有研究表明,薔薇屬植物皮刺起源于表皮下的原分生組織,分為腺體型皮刺與非腺體型皮刺,依據皮刺是否有毛附著及是否分叉可進一步劃分為5種亞類[1],‘月月粉’皮刺為典型的非腺體型皮刺。轉錄因子(transcription factor)在基因表達過程中起著重要的作用,其中最常見的一類轉錄因子就是轉錄激活子和轉錄抑制子,這類轉錄因子與特定的DNA序列結合,在轉錄水平調控基因的相關表達[2]。ZHANG等[3]利用高通量測序手段,分析了有刺野薔薇及無刺突變體間的差異表達基因,從而確定了可能與皮刺形成有關的關鍵基因和代謝途徑。在這些差異表達基因中,一些與發育和次生代謝相關的轉錄因子基因,包括、、和等,在著生皮刺的節間部位表達明顯上調。其中,NAC轉錄因子是植物特有的最大的轉錄因子家族之一,命名由(no apical meristem)、(transcription activation factor)、(cup-shaped cotyledon)3個基因的首字母縮寫組成[4],這3個基因在N端編碼高度保守的蛋白序列,與DNA的結合能力相關,包含A—E五個亞域,蛋白C端具有多樣性,其富含酸性氨基酸、脯氨酸、絲氨酸、蘇氨酸等簡單重復序列,與轉錄調控相關[5-6]。隨著植物各基因組數據的釋放,在擬南芥()、水稻()、棉花(spp.)等物種中都已鑒定出大量的[7-9]。研究表明,廣泛參與植物生長發育進程,在植物種子發育、分生組織生長、次生壁和木質部的形成、根的生長以及植物的衰老過程中都起著重要的作用[10-15],在黃瓜(L.)中,NAC轉錄因子還與黃瓜刺的形成及刺密度相關[16]。同時,在植物脅迫響應中發揮著重要的作用。的沉默增強了萵苣()對假單胞菌的抗性,并且降低了植株在干旱脅迫下的萎蔫效應[17];在煙草中表達歐洲葡萄‘粉紅亞都蜜’(Yatomo Rose),植株對干旱的耐受性降低[18];桃(L.)在低溫脅迫處理下,16個表達量顯著上調[19];番茄()在響應鋁脅迫時發揮重要作用[20]。以上研究表明,NAC家族成員廣泛參與植物生長發育及各種防御反應。【本研究切入點】已在多個物種中被鑒定,并發現其發生不同程度的功能分化,但在‘月月粉’中尚未有NAC家族相關研究報道。目前,高質量的‘月月粉’雙單倍體基因組信息已經公布[21-22],為研究相關基因的功能及關系奠定了堅實的基礎。【擬解決的關鍵問題】本研究利用生物信息學手段,鑒定‘月月粉’NAC基因家族,并系統分析‘月月粉’NAC家族的理化性質、基因結構、染色體定位及系統進化等,結合組織表達特異性、脅迫表達模式以及不同發育階段皮刺轉錄組數據,為進一步研究的生物學功能,尋找可能與皮刺發育相關的,并應用于月季品種的遺傳改良提供理論參考。
1 材料與方法
試驗于2021年7至2022年3月在華中農業大學進行。
1.1 ‘月月粉’NAC家族成員鑒定及理化性質分析
在NCBI數據庫(https://www.ncbi.nlm.nih.gov/)以及薔薇科基因組數據庫(https://www.rosaceae.org)中分別下載獲得擬南芥和‘月月粉’基因組序列及基因結構注釋文件。在PlantTFDB數據庫(http:// planttfdb.gao-lab.org/)[23]中下載擬南芥NAC蛋白ID,結合擬南芥基因組序列獲得擬南芥NAC家族成員基因序列,使用TBtools[24]和NCBI中的Blast工具進行雙向blast比對篩選,結合HMM結構域檢索,初步得到候選s,利用在線網站NCBI-Batch-CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)對候選RcNACs序列進行保守結構域分析,得到最終NAC家族成員。利用ExPASy(https://web. expasy.org/compute_pi/)分析RcNACs等電點和蛋白分子量等理化性質,使用WoLF PSORT(http://www.genscript.com/wolf-psort.html)對RcNACs進行亞細胞定位預測。
1.2 ‘月月粉’NAC家族成員系統進化分析
在PlantTFDB數據庫中下載水稻(subsp.)NAC氨基酸序列,使用MEGA X[25]對擬南芥、水稻和‘月月粉’中鑒定出的NAC蛋白的氨基酸序列進行多序列比對,并采用鄰接法(Neighbor-Joining)構建進化樹(bootstrap=1000)。利用iTOL[26]在線網站(https://itol.embl.de/)進行進化樹美化。
1.3 ‘月月粉’NAC家族染色體定位、共線性分析、基因結構及保守基序分析
利用在線網站MEME(http://meme-suit.org/index. html)對‘月月粉’NAC蛋白進行保守基序序列預測[27]。利用TBtools工具,基于‘月月粉’、擬南芥基因組序列進行共線性分析,并結合‘月月粉’基因組注釋文件信息以及NCBI-Batch-CDD保守結構域分析結果,對s染色體定位、共線性結果、基因結構以及保守基序進行可視化展示。
1.4 ‘月月粉’NAC家族啟動子順式元件分析
結合‘月月粉’基因組注釋文件,使用在線分析網站PlantCARE(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)對s順式作用元件進行分析,并利用TBtools進行可視化展示。
1.5 ‘月月粉’NAC家族成員表達分析
1.5.1 組織及誘導表達分析 利用RcNAC成員的氨
基酸序列在‘月月粉’參考轉錄組數據庫(https://lipm- browsers.toulouse.inra.fr/pub/RchiOBHm-V2/)進行blast比對,獲取并下載部分成員在幼葉和幼莖(FTN),水脅迫的植株葉片(FTS),感染灰霉病LR18的葉片(FTB),白色幼根(RAC),休眠的腋芽即營養分生組織(NDB),萌發的腋芽即營養分生組織(DBO),花分生組織轉變時期的花芽(IFL),花分生組織和早期花器官包括萼片、花瓣、雄蕊和心皮(IMO),閉合的花(BFL),開放的花(OFT),衰老的花(SEN),雄蕊(DET),從授粉到色素積累早期的果實(CYN)組織中的表達數據[28]。利用TBtools工具繪制不同組織表達熱圖。
1.5.2 皮刺相關的篩選及qRT-PCR分析 以華中農業大學薔薇科植物種質資源圃內保存的中國古老月季品種‘月月粉’為試材,采集其植株莖桿上4個不同發育階段的皮刺。Stage I為柔軟幼嫩、尚未木質化的皮刺,Stage Ⅱ為一定程度木質化、尖端開始向基部彎曲的皮刺,Stage Ⅲ為木質化較完全、形態基本成熟的皮刺,Stage IV為發育成熟、并初步開始衰老的皮刺(圖1)。每個階段的樣本設置3個生物學重復,樣品送至武漢博越致和生物科技有限公司進行轉錄組測序,建立皮刺不同發育時期基因數字表達譜,并根據TPM法計算基因表達量的結果。使用EASYspin植物RNA快速提取試劑盒(RN0902,艾德萊生物)提取4個階段樣本RNA,使用PrimeScript? RT Reagent Kit with gDNA Eraser (Perfect Real Time)(RR047A,TaKaRa)進行cDNA第一鏈的合成,供熒光定量使用。從轉錄組測序結果中選取10個基因,使用Primer-BLAST(https://www.ncbi.nlm.nih.gov/ tools/primer-blast/)設計定量引物,內參基因為‘月月粉’,在QuantStudio? 6 Flex熒光定量PCR儀(Thermo)上進行分析,每個階段設置3個生物學重復,擴增體系共10 μL,含1 μL cDNA,上、下游引物各0.4 μL,TB GreenII(RR820A,TaKaRa)5 μL,ROX Reference Dye II 0.2 μL,ddH2O 3 μL。反應程序為兩步法:95℃預變性30 s;95℃變性5 s,60℃退火/延伸34 s,40個循環。用參照基因的ΔCt法計算10個基因的表達量。
基于轉錄組數據,通過差異倍數(|log2(Fold change)|≥1)和顯著水平(-value<0.05)兩個水平挑選出樣品間的差異表達基因[29],并對其中的進行統計與分析,利用TBtools軟件繪制NAC家族基因表達熱圖,對在皮刺4個階段轉錄組數據中高表達的進行qRT-PCR分析(引物信息見表1)。
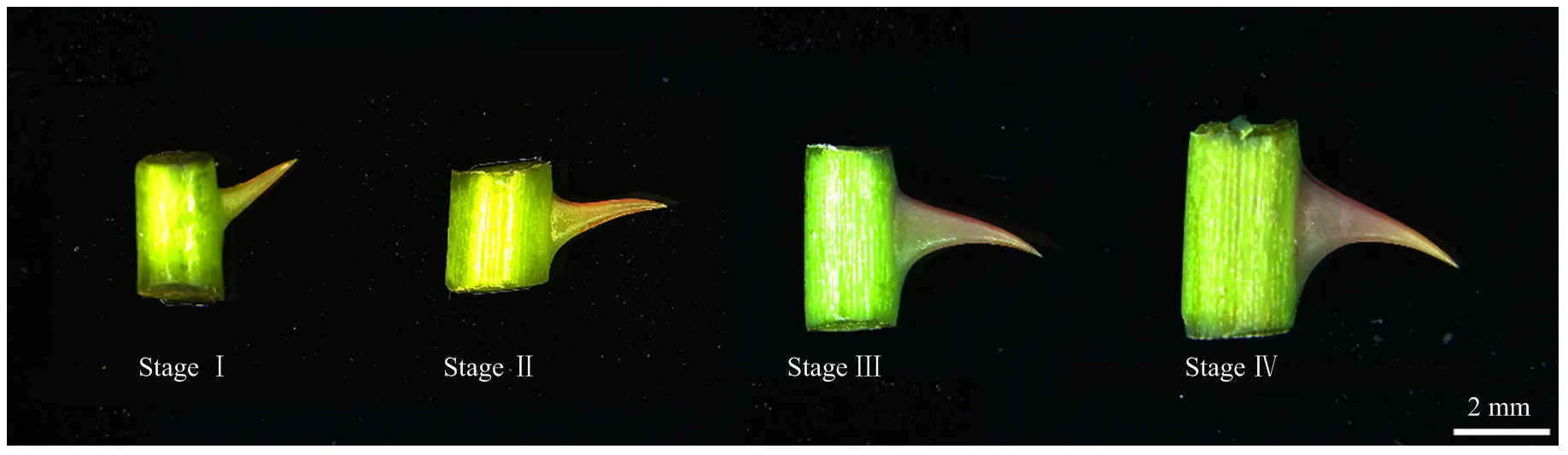
Stage I:柔軟幼嫩、尚未木質化的皮刺;Stage II:一定程度木質化、尖端開始向基部彎曲的皮刺;Stage III:木質化較完全、形態基本成熟的皮刺;Stage IV:發育成熟、并初步開始衰老的皮刺

表1 qRT-PCR引物序列
2 結果
2.1 RcNAC家族成員鑒定及進化分析
基于擬南芥基因組序列,從PlantTFDB數據庫中獲取138個擬南芥NAC家族成員氨基酸序列。根據‘月月粉’基因組序列文件以及基因結構注釋文件,利用TBtools工具,對擬南芥和‘月月粉’基因組進行雙向blast,并利用Pfam NAC家族結構域索取號進行HMM結構域檢索。經過保守結構域分析,最終鑒定得到116個s,依據其在染色體上的位置,命名為—。為了獲取RcNAC家族的系統發育關系,進一步探討NAC家族的進化歷史,利用MEGA X使用鄰接法對116個RcNACs與138個AtNACs、170個OsNACs氨基酸序列構建進化樹(圖2)。依據親緣關系遠近,將這424個NACs劃分為21個分支,依次命名為組A—U。AtNACs、OsNACs與RcNACs不均勻分布于進化樹各分支中,在B分支中,只含有AtNACs,C分支中,只含有RcNACs,但兩個分支親緣關系較近,推測這兩支NAC在擬南芥和‘月月粉’中可能參與調控不同的生物學進程。
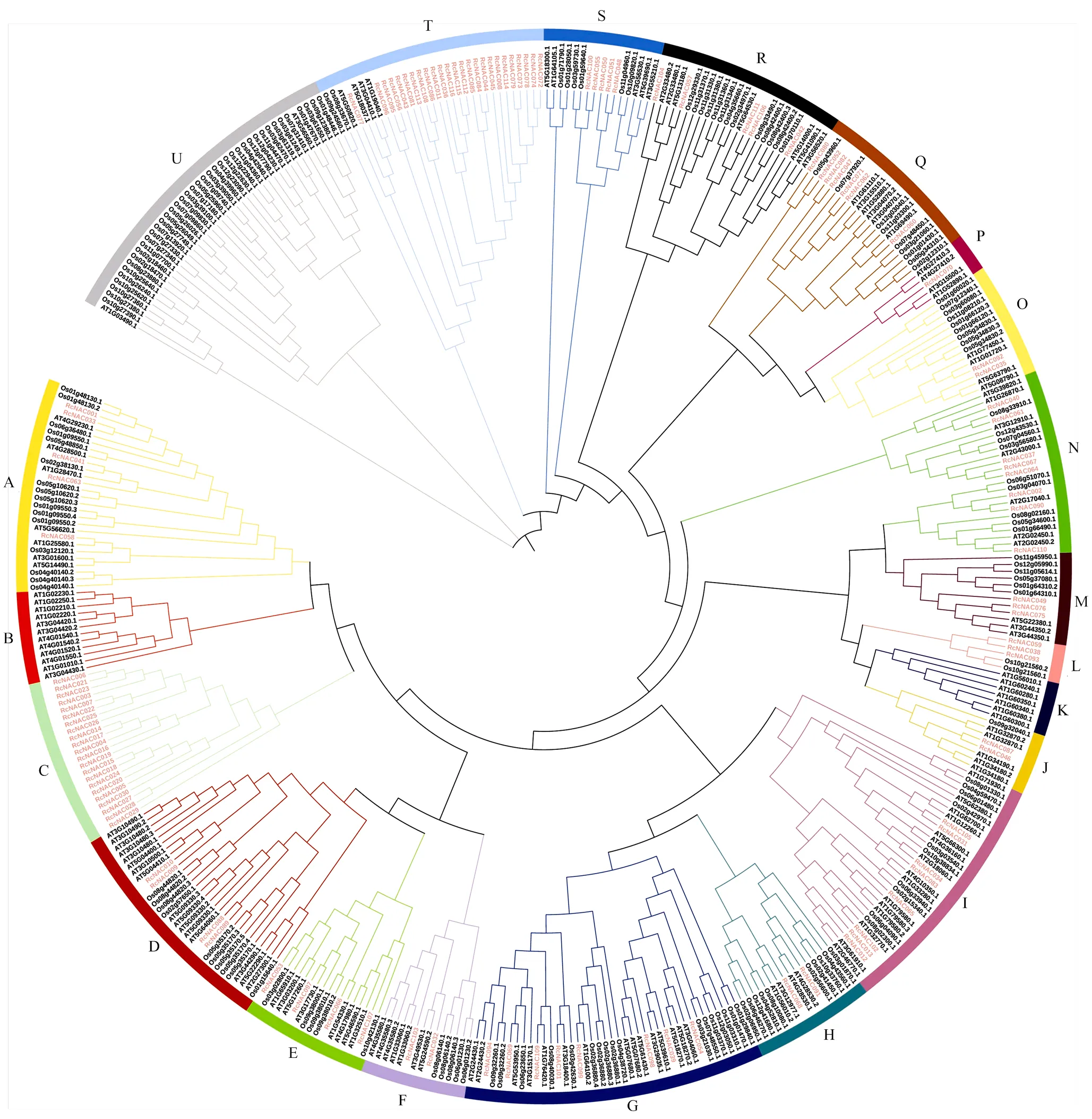
圖2 ‘月月粉’(紅)、擬南芥(黑)、水稻(黑)NAC蛋白系統進化樹
2.2 RcNAC家族基本信息
各進化樹分支包含NAC數量差距較大,為1—25個。對116個NAC家族基因蛋白序列進行基本信息分析,其蛋白長度為69—713個氨基酸,等電點為4.43—9.78,分子質量為7.87—79.99 kD。亞細胞定位預測結果顯示,多數RcNACs定位于細胞核中,還有部分定位于葉綠體、過氧化物酶體、高爾基體、內質網、液泡以及細胞膜上(表2)。

表2 ‘月月粉’NAC家族成員基本信息
2.3 RcNAC家族染色體定位、共線性分析與基因結構分析
‘月月粉’的116個s成員在7條染色體上不均勻分布(圖3),1號染色體上最多,有31個s;4號染色體上最少,僅有7個成員;未注釋到‘月月粉’7條染色體上,暫用Chr0表示。在116個s成員中,有6對串聯重復基因,其中有3對分布在1號染色體上,2對分布在6號染色體上,1對分布在3號染色體上。對擬南芥和‘月月粉’基因組進行共線性分析(圖4),結果表明,54個s與36個s存在共線性關系,其中,與、存在共線性關系,其余都只與一個存在共線性關系。整體上看,擬南芥5號染色體和‘月月粉’2號染色體含有最多數量的共線性基因,共有7組。
對s進行基因結構分析(圖5),同一亞群成員內含子外顯子模式大致相同,s成員內含子0—8個,所有的s成員都含有保守結構域NAM,分布在其蛋白序列N端。
研究表明,擬南芥中的NAC家族成員保守結構域中共包含A、B、C、D、E 5個亞域[5]。對鑒定出的RcNACs蛋白序列進行保守基序預測,分析并保留10個motif。同組RcNACs成員的motif在分布類型和分布順序上基本相同。其中,motif 1(GFRFHPTDEELVSYY)與亞域A,motif 4(IIPEIDLYKYEPWDLPEKAKI)與亞域C,motif 6(EWYFFTPRDRK)以及motif3(YPNGTRTNRATGSGYWKA)與亞域B,motif 5(ISGSKKVIGMKKTLVFYKGRA)以及motif2(PKGQKTBWVMHEYRL)與亞域D,motif 7(DDWVLCRLFKK)與亞域E序列相似,有62個RcNACs包含這全部7個motif,且在RcNAC050中有重復的NAC完整基序。motif 9與motif 10分布在序列C端,猜測與RcNAC的轉錄調控功能相關。
2.4 RcNAC啟動子順式作用元件分析
利用PlantCARE對s上游2 000 bp序列中的順式元件進行分析預測(圖6),結果表明,除了啟動子和增強子區域常見的順式作用元件外,s啟動子上還具有不同數量的光響應元件、厭氧誘導元件、干旱響應元件、低溫應答元件、分生組織調控元件,以及響應不同植物激素(脫落酸、赤霉素、生長素、茉莉酸甲酯、水楊酸)等多種特異性順式作用元件。

圖3 RcNACs在染色體上的分布
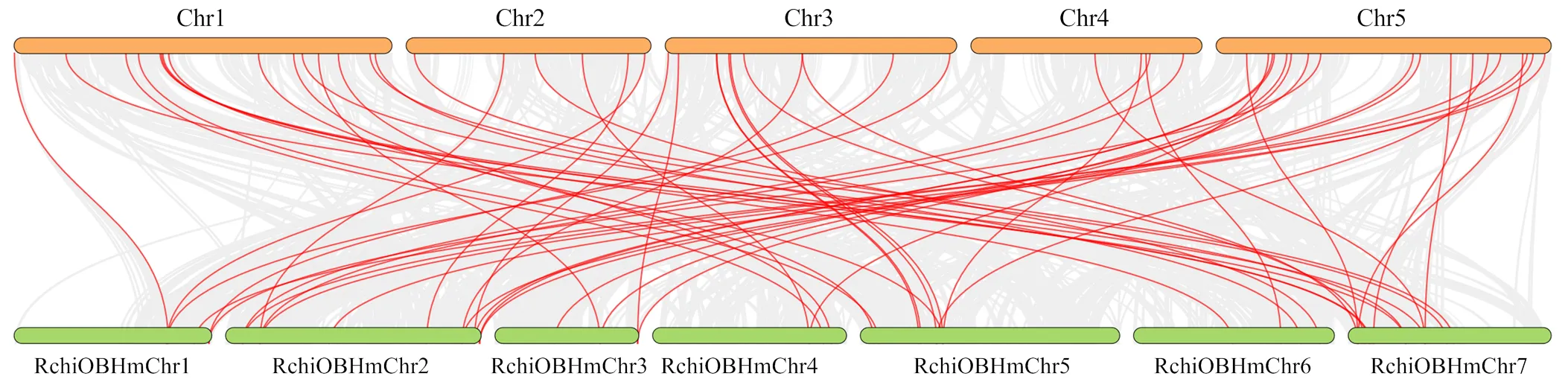
圖4 RcNACs 與AtNACs共線性分析

圖5 ‘月月粉’NAC蛋白保守基序預測及保守結構域分析

圖6 RcNACs啟動子順式作用元件分析
2.5 RcNAC家族成員組織及誘導表達分析
使用RcNACs氨基酸序列在月季轉錄組數據庫[28]中進行比對,得到其中42個成員在‘月月粉’13個不同組織樣本中的轉錄組RPKM數據,繪制熱圖并進行分析(圖7)。結果顯示,除外,其余40個成員均在一個或多個樣本中表達。
葉片經脅迫處理(FTS、FTB)后,相比于對照組(FTN),有31個NAC成員的表達量有明顯變化,且14個成員在FTS中的表達量高于它們在其他12個組織中的表達量。其中,在FTS、FTB組織中表達量上調變化最為顯著,在感染灰霉病的植株葉片中表達量下降明顯。

Rosa-FTS:水脅迫的植株葉片;Rosa-FTB:感染灰霉病LR18的葉片;Rosa-FTN:幼葉和幼莖;Rosa-RAC:白色幼根;Rosa-NDB:休眠的腋芽即營養分生組織;Rosa-DBO:萌發的腋芽即營養分生組織;Rosa-IFL:花分生組織轉變時期的花芽;Rosa-IMO:花分生組織和早期花器官包括萼片,花瓣,雄蕊和心皮;Rosa-BFL:閉合的花;Rosa-OFT:開放的花;Rosa-SEN:衰老的花;Rosa-DET:雄蕊;Rosa-CYN:從授粉到色素積累早期的果實
在不同組織和器官中,不同成員的表達水平有明顯差異。在FTN,在NDB、SEN、CYN,在RAC、IFL、BFL、OFT、DET,在DBO、IMO組織中相對于其他NAC成員高表達,其中,在FTN中表達量較高,其他成員表達量略低;在花器官的不同發育階段組織(IFL、IMO、BFL、OFT、SEN)中,檢測到35個NAC成員在一至多個階段表達,、、、、、在花器官發育后期高表達,整體表達量呈上調趨勢,在花發育5個組織中持續穩定高表達,這些成員可能參與調控花器官的生長發育進程;、只在早期花器官中表達,、只在閉合的花中表達,猜測其在花器官發育特定階段行使功能。
2.6 皮刺相關RcNAC篩選及表達分析
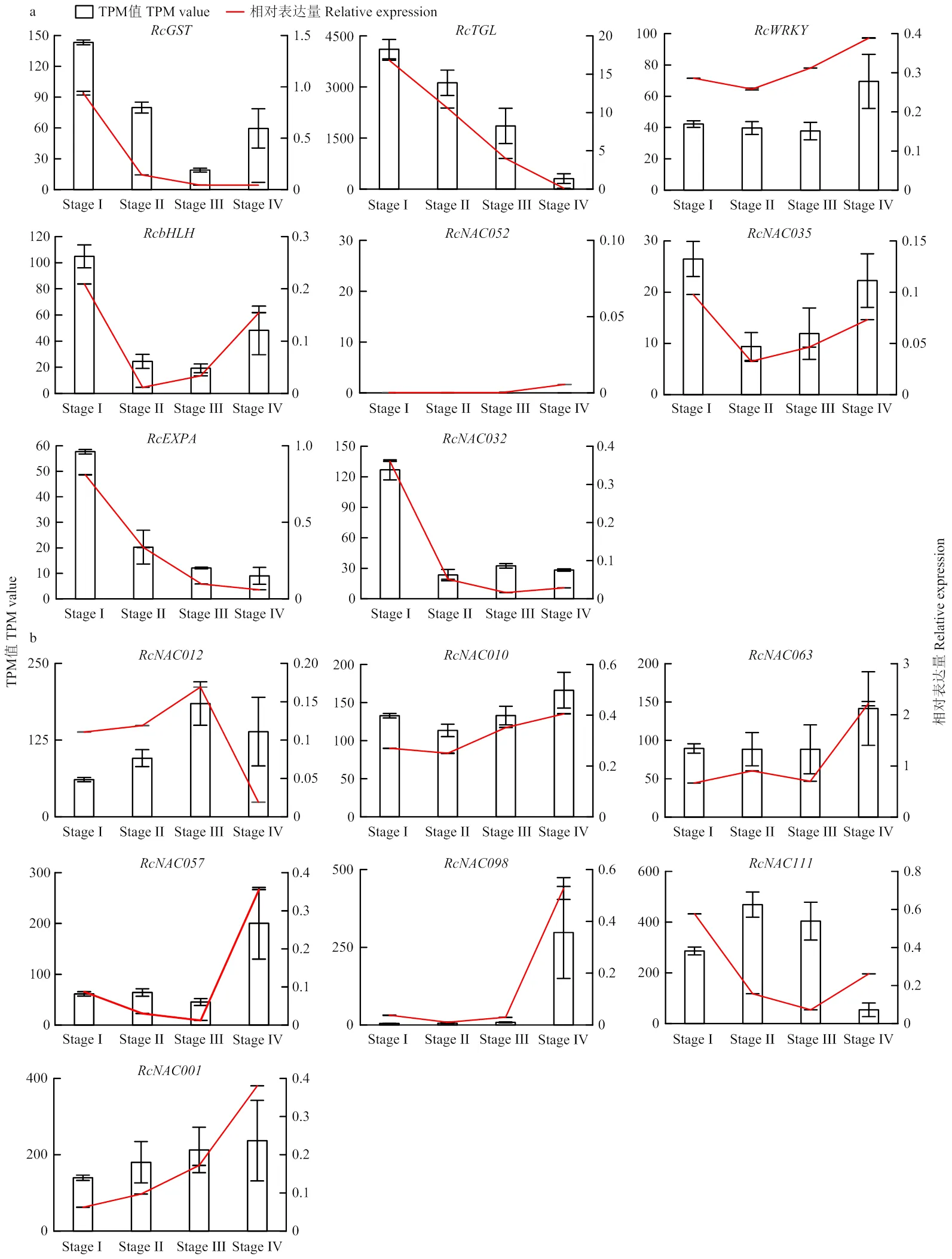
根據月月粉不同發育階段皮刺轉錄組中表達量繪制熱圖,其中63個成員因其表達量過低未納入分析,基于剩余53個成員在皮刺中的表達量的TPM值繪制熱圖(圖8-a),對各表達模式進行聚類分析,共聚為4組七7個亞組。其中,組只包含,其在Stage Ⅰ中高表達;組在各階段相對平穩表達,-1成員在各階段表達量在50—110,-2成員在各階段表達量在0—50,其中有少數基因在Stage Ⅰ或Stage Ⅳ中表達量相對Stage Ⅱ和Ⅲ較高;組成員在Stage Ⅳ中高表達,-1成員表達量高于200.00,-2表達量約100.00;組成員在皮刺多個發育階段高表達。
從中篩選不同發育階段皮刺差異表達,共有26個。Stage I—Ⅱ有11個基因差異表達,10個在Stage Ⅱ下調,1個上調;Stage Ⅱ—Ⅲ中有5個基因差異表達,全為上調;Stage Ⅲ—IV中有18個基因差異表達,全為上調(圖9),此18個上調基因包含Stage Ⅱ—Ⅲ中的5個差異基因。分析各差異基因在不同皮刺發育階段表達量趨勢變化(圖8-b),、、、整體呈上升趨勢,在Stage Ⅲ—Ⅳ階段上調趨勢最為明顯,猜測這些基因與皮刺的硬化與木質化程度可能息息相關;、、、、在4個階段表達較穩定,在Stage Ⅰ和Stage Ⅳ略高,各階段表達量未超過5.00;在Stage Ⅰ—Ⅱ呈下調趨勢,可能與Stage Ⅰ中皮刺細胞的增殖有關,在Stage Ⅱ—Ⅳ呈上升趨勢,猜測其也參與了皮刺后期木質化階段;、、、變化趨勢相同,在Stage Ⅰ高表達,Stage Ⅱ表達下降,之后穩定表達,其中在Stage Ⅰ表達量為126.82,在Stage Ⅱ下降為23.34,其在皮刺前期細胞增殖過程中可能行使一定功能;其余12個差異表達基因在Stage Ⅰ—Ⅲ表達較為平穩,在Stage Ⅲ—Ⅳ呈上調趨勢,推測這些基因可能參與調控皮刺木質化及細胞程序性死亡。
2.7 皮刺轉錄組相關基因的qRT-PCR驗證
在皮刺轉錄組數據隨機選取8個基因進行qRT-PCR分析,其中包含3個NAC家族基因,1個三酰甘油脂肪酶基因,1個谷胱甘肽轉移酶基因,以及WRKY、bHLH、Expansin家族基因各1個。結果顯示,各基因qRT-PCR結果與皮刺轉錄組數據計算結果相符,說明轉錄組數據較準確可靠(圖10-a)。此外,選取皮刺轉錄組中NAC家族及-1組中7個s進行qRT-PCR分析,結果如圖10-b所示,其中的qRT-PCR分析結果與轉錄組數據差異較大,其余6個s qRT-PCR分析結果與轉錄組數據相符。
3 討論
3.1 NAC家族基因在不同物種間具有一定保守性
本研究基于s對月月粉基因組數據進行分析,共鑒定出116個s,與蘋果()中鑒定得到的180個[30],沙梨()中185個[31],海濱木槿(Sieb. et Zucc)中182個[32]s相比,基因數目較少;與杉木()中鑒定得到的45個[33],毛竹()中94個[34]s相比,基因數目略多;與桃中119個[19]s數量相近。由此可見,在不同的物種之間的數量有明顯差異,同科物種數量差異也較大,推測這與物種進化和基因組復制相關。對RcNAC蛋白序列進行分析,有62個RcNACs蛋白包含完整的5個亞域(A—E),說明NAC在不同物種中的序列保守性較高。
3.2 RcNAC參與調控‘月月粉’脅迫應答及生長發育
有研究表明,在經過鹽、干旱和脫落酸處理后,表達量顯著上調[35];敲除后,降低了水稻對脫落酸的敏感性,但是提高了水稻對鹽脅迫的敏感性[36];在干旱脅迫和復水后,部分基因的表達量出現變化[37]。本研究對‘月月粉’不同組織中s表達量進行分析,在FTS組織中,14個s表達量上調,推測這些基因在‘月月粉’響應水分脅迫過程中發揮作用,這與前期的研究結果一致。以往研究顯示,NAC轉錄因子在花器官發育及開花調控中發揮重要作用[38],本研究中多個s在花器官的不同發育階段表達量發生顯著變化,推測這些基因在‘月月粉’花器官發育過程中發揮重要作用。另外,牛早柱等[39]研究發現,4個s在葡萄果實成熟過程中發揮重要作用;Martín-Pizarro等[40]研究表明,草莓NAC轉錄因子FaRIF是草莓果實成熟早期的關鍵調節因子。在‘月月粉’CYN組織中,和高表達,推測這些基因在‘月月粉’果實成熟過程中行使一定功能。

圖8 ‘月月粉’NAC基因在不同發育階段皮刺中的表達量熱圖

圖9 ‘月月粉’不同發育階段皮刺差異表達NAC數量
3.3 RcNAC參與調控‘月月粉’皮刺發育
在月季[41]和野薔薇[3]中,皮刺的發育伴隨著木質素、花青素、類黃酮等物質的積累。‘月月粉’在Stage Ⅱ—Ⅲ中木質素不斷積累至完全,結合進化樹與4個階段皮刺轉錄組數據,在‘月月粉’皮刺轉錄組Stage Ⅰ—Ⅲ階段高表達,qRT-PCR分析其在Stage Ⅰ和Stage Ⅳ相對表達量較高。與()的進化距離較近,通過負調節次生細胞壁纖維合成和細胞程序性死亡來影響管狀分子和木質部發育。過表達導致木質部導管缺失和管狀分子標記基因表達很少或不表達[42],推測在‘月月粉’皮刺發育過程中調節木質化的進程,在皮刺發育初期是否也發揮一定作用需要進一步驗證。與旁系同源的在皮刺轉錄組中未檢測到表達,其是否在皮刺發育中發揮作用有待進一步研究。在Stage Ⅳ中表達上調,與()為同源基因,在擬南芥次生細胞壁合成中發揮作用[43],且在毛果楊中的同源基因通過調節木質素、纖維素合成的關鍵基因來調控次生壁組織合成[44],是否在皮刺細胞的次生壁合成過程中發揮作用需要進一步驗證。在Stage Ⅰ—Ⅳ中整體表達呈上升趨勢,研究表明,其同源基因()在花瓣和雄蕊發育后期表達,在葉片衰老期間上調[45],且調控擬南芥角果衰老[46],這與‘月月粉’皮刺發育階段和SEN組織中的表達模式相同,猜測與皮刺衰老進程相關。在Stage Ⅰ高表達,為中極少數在皮刺發育前期高表達的基因,qRT-PCR分析進一步證實其在Stage Ⅰ相對表達量較高,其是否在皮刺發育早期行使重要功能?、、和在皮刺發育各個階段的表達量均較高,其相似的表達模式是否暗示其功能的冗余,這些問題還有待深入研究。
4 結論
本研究在‘月月粉’全基因組中鑒定出116個s,不均勻分布在7條染色體上,均具有NAM保守結構域,系統進化樹將RcNACs分為21類。多數s在遭受水分脅迫或灰霉病感染的葉片組織中表達量發生顯著變化;在皮刺發育進程中,絕大多數成員中后期表達顯著,這些基因可能主要參與皮刺木質化及細胞程序性死亡等相關進程。、、和在皮刺發育多個階段高表達,在皮刺StageⅠ中高表達,可作為皮刺形態發生和發育相關重點候選基因。
a:轉錄組隨機挑選基因的qRT-PCR分析;b:NAC家族δ及γ-1組中7個RcNAC的qRT-PCR分析
[1] ZHOU N N, SIMONNEAU F, THOUROUDE T, OYANT L H, FOUCHER F. Morphological studies of rose prickles provide new insights. Horticulture Research, 2021, 8(1): 221. doi: 10.1038/s41438- 021-00689-7.
[2] SINGH K B. Transcriptional regulation in plants: The importance of combinatorial control. Plant Physiology, 1998, 118(4): 1111-1120. doi: 10.1104/pp.118.4.1111.
[3] ZHANG Y, ZHAO M J, ZHU W, SHI C M, BAO M Z, ZHANG W. Nonglandular prickle formation is associated with development and secondary metabolism-related genes in Rosa multiflora. Physiologia Plantarum, 2021, 173(3): 1147-1162. doi: 10.1111/ppl.13510.
[4] SOUER E, VAN HOUWELINGEN A, KLOOS D, MOL J, KOES R. The no apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries. Cell, 1996, 85(2): 159-170. doi: 10.1016/ s0092-8674(00)81093-4.
[5] OLSEN A N, ERNST H A, LEGGIO L L, SKRIVER K. NAC transcription factors: Structurally distinct, functionally diverse. Trends in Plant Science, 2005, 10(2): 79-87. doi: 10.1016/j.tplants.2004. 12.010.
[6] 張慧珍, 白雪芹, 曾幼玲. 植物NAC轉錄因子的生物學功能. 植物生理學報, 2019, 55(7): 915-924. doi: 10.13592/j.cnki.ppj.2019. 0107.
ZHANG H Z, BAI X Q, ZENG Y L. Biological functions of plant NAC transcription factors. Plant Physiology Journal, 2019, 55(7): 915-924. doi: 10.13592/j.cnki.ppj.2019.0107. (in Chinese)
[7] JENSEN M K, KJAERSGAARD T, NIELSEN M M, GALBERG P, PETERSEN K, O'SHEA C, SKRIVER K. TheNAC transcription factor family: structure-function relationships and determinants of ANAC019 stress signalling. Biochemical Journal, 2010, 426(2): 183-196. doi: 10.1042/BJ20091234.
[8] OOKA H, SATOH K, DOI K, NAGATA T, OTOMO Y,MURAKAMI K, MATSUBARA K, OSATO N, KAWAI J,CARNINCI P, HAYASHIZAKI Y, SUZUKI K, KOJIMA K,TAKAHARA Y, YAMAMOTO K, KIKUCHI S. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana. DNA Research, 2003, 10(6): 239-247. doi: 10.1093/dnares/10.6.239.
[9] SUN H, HU M L, LI J Y, CHEN L, LI M, ZHANG S Q, ZHANG X L, YANG X Y. Comprehensive analysis of NAC transcription factors uncovers their roles during fiber development and stress response in cotton. BMC Plant Biology, 2018, 18(1): 150. doi: 10.1186/s12870- 018-1367-5.
[10] SUN L, LIU L P, WANG Y Z, YANG L, WANG M J, LIU J X. NAC103, a NAC family transcription factor, regulates ABA response during seed germination and seedling growth in. Planta, 2020, 252(6): 95. doi: 10.1007/s00425-020-03502-2.
[11] YANG J H, LEE K H, Du Q, YANG S, YUAN B J, QI L Y, WANG H Z. A membrane-associated NAC domain transcription factor XVP interacts with TDIF co-receptor and regulates vascular meristem activity. New Phytologist, 2020, 226(1): 59-74. doi: 10.1111/nph. 16289.
[12] ZHONG R O, LEE C, HAGHIGHAT M, YE Z H. Xylem vessel- specific SND5 and its homologs regulate secondary wall biosynthesis through activating secondary wall NAC binding elements. New Phytologist, 2021, 231(4): 1496-1509. doi: 10.1111/nph.17425.
[13] FANG S, SHANG X G, YAO Y, LI W X, GUO W Z. NST- and SND-subgroup NAC proteins coordinately act to regulate secondary cell wall formation in cotton. Plant Science, 2020, 301: 110657. doi: 10.1016/j.plantsci.2020.110657.
[14] 文靜, 王春濤, 楊永平. 植物木質部次生細胞壁加厚調控的研究進展. 西南林業大學學報(自然科學版), 2021, 41(2): 182-188.
WEN J, WANG C T, YANG Y P. Advances in regulation of xylem secondary cell wall thickening in plants. Journal of Southwest Forestry University (Natural Science Edition), 2021, 41(2): 182-188. (in Chinese)
[15] CHEN D D, CHAI S C, MCINTYRE C L, XUE G P. Overexpression of a predominantly root-expressed NAC transcription factor in wheat roots enhances root length, biomass and drought tolerance. Plant Cell Reports, 2018, 37(2): 225-237. doi: 10.1007/s00299-017-2224-y.
[16] LIU X W, WANG T, BARTHOLOMEW E, BLACK K, DONG M M, ZHANG Y Q, YANG S, CAI Y L, XUE S D, WENG Y Q, REN H Z. Comprehensive analysis of NAC transcription factors and their expression during fruit spine development in cucumber (L.). Horticulture Research, 2018, 5: 31. doi: 10.1038/s41438- 018-0036-z.
[17] MEISRIMLER C N, PELGROM A J E, OUD B, OUT S, Van den ACKERVEKEN G. Multiple downy mildew effectors target the stress-related NAC transcription factor LsNAC069 in lettuce. Plant Journal, 2019, 99(6): 1098-1115. doi: 10.1111/tpj.14383.
[18] 朱自果, 陰啟忠, 張慶田, 韓真, 張倩, 李勃. 歐洲葡萄‘粉紅亞都蜜’NAC基因DRL1負向調節植物抗旱性. 園藝學報, 2020, 47(12): 2290-2300. doi: 10.16420/j.issn.0513-353x.2020-0185.
ZHU Z G, YIN Q Z, ZHANG Q T, HAN Z, ZHANG Q, LI B. DRL1,a NAC gene fromYatomo Rose, negatively regulates the drought tolerance. Acta Horticulturae Sinica, 2020, 47(12): 2290-2300. doi: 10.16420/j.issn.0513-353x.2020-0185. (in Chinese)
[19] 李小蘭, 張瑞, 郝蘭蘭, 王鴻. 桃NAC家族基因生物信息學分析及其響應低溫脅迫的表達特征. 浙江農業學報, 2022, 34(4): 766-780.
LI X L, ZHANG R, HAO L L, WANG H. Bioinformatics analysis of peach NAC gene family and its expression characteristics in response to low temperature stress. Acta Agriculturae Zhejiangensis, 2022, 34(4): 766-780. (in Chinese)
[20] JIN J F, WANG Z Q, HE Q Y, WANG J Y, LI P F, XU J M, ZHENG S J, FAN W, YANG J L. Genome-wide identification and expression analysis of the NAC transcription factor family in tomato () during aluminum stress. BMC Genomics, 2020, 21(1): 288. doi: 10.1186/s12864-020-6689-7.
[21] RAYMOND O, GOUZY J, JUST J, BADOUIN H, VERDENAUD M, LEMAINQUE A, VERGNE P, MOJA S, CHOISNE N, PONT C, CARRERE S, CAISSARD J C, COULOUX A, COTTRET L, AURY J M, SZECSI J, LATRASSE D, MADOUI M A, FRANCOIS L, FU X P,. The Rosa genome provides new insights into the domestication of modern roses. Nature Genetics, 2018, 50(6): 772-777. doi: 10.1038/ s41588-018-0110-3.
[22] HIBRAND S L, RUTTINK T, HAMAMA L, KIROV I, LAKHWANI D, ZHOU N N, BOURKE P M, DACCORD N, LEUS L, SCHULZ D, Van de GEEST H, HESSELINK T, Van LAERE K, DEBRAY K, BALZERGUE S, THOUROUDE T, CHASTELLIER A, JEAUFFRE J, VOISINE L, GAILLARD S,. A high-quality genome sequence of Rosa chinensis to elucidate ornamental traits. Nature Plants, 2018, 4(7): 473-484. doi: 10.1038/s41477-018-0166-1.
[23] TIAN F, YANG D C, MENG Y Q, JIN J P, GAO G. PlantRegMap: charting functional regulatory maps in plants. Nucleic Acids Research, 2020, 48(D1): D1104-D1113. doi: 10.1093/nar/gkz1020.
[24] CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202. doi: 10.1016/j.molp.2020.06.009.
[25] KUMAR S, STECHER G, LI M, KNYAZ C, TAMURA K. MEGA X: Molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. doi: 10.1093/molbev/msy096.
[26] LETUNIC I, BORK P. Interactive Tree Of Life (iTOL) v5: An online tool for phylogenetic tree display and annotation. Nucleic Acids Research, 2021, 49(W1): W293-W296. doi: 10.1093/nar/gkab301.
[27] BAILEY T L, BODEN M, BUSKE F A, FRITH M, GRANT C E, CLEMENTI L, REN J, LI W W, NOBLE W S. MEME SUITE: tools for motif discovery and searching. Nucleic Acids Research, 2009, 37(Web Server issue): W202-W208. doi: 10.1093/nar/gkp335.
[28] DUBOIS A, CARRERE S, RAYMOND O, POUVREAU B, COTTRET L, ROCCIA A, ONESTO J P, SAKR S, ATANASSOVA R, BAUDINO S, FOUCHER F, Le BRIS M, GOUZY J, BENDAHMANE M. Transcriptome database resource and gene expression atlas for the rose. BMC Genomics, 2012, 13: 638. doi: 10.1186/1471-2164-13-638.
[29] LOVE M I, HUBER W, ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology, 2014, 15(12): 550. doi: 10.1186/s13059-014-0550-8.
[30] SU H Y, ZHANG S Z, YUAN X W, CHEN C T, WANG X F, HAO Y J. Genome-wide analysis and identification of stress-responsive genes of the NAM-ATAF1,2-CUC2 transcription factor family in apple. Plant Physiology Biochemistry, 2013, 71: 11-21. doi: 10.1016/ j.plaphy.2013.06.022.
[31] AHMAD M, YAN X H, LI J Z, YANG Q S, JAMIL W, TENG Y W, BAI S L. Genome wide identification and predicted functional analyses of NAC transcription factors in Asian pears. BMC Plant Biology, 2018, 18(1): 214. doi: 10.1186/s12870-018-1427-x.
[32] WANG Z Q, NI L J, LIU D N, FU Z K, HUA J F, LU Z G, LIU L Q, YIN Y L, LI H G, GU C S. Genome-wide identification and characterization of NAC family inSieb. et Zucc. under various abiotic stresses. International Journal of Molecular Sciences, 2022, 23(6): 3055. doi: 10.3390/ijms23063055.
[33] 高文杰, 劉嬌, 馬祥慶, 帥鵬. 杉木NAC基因家族基因的鑒定及生物信息學分析. 中南林業科技大學學報, 2022, 42(2): 108-118. doi: 10.14067/j.cnki.1673-923x.2022.02.012.
GAO W J, LIU J, MA X Q, SHUAI P. ldentification and bioinformatics analysis of Chinese fir NAC gene family. Journal of Central South University of Forestry & Technology, 2022, 42(2): 108-118. doi: 10.14067/j.cnki.1673-923x.2022.02.012. (in Chinese)
[34] SHAN X M, YANG K B, XU X R, ZHU C L, GAO Z M. Genome-wide investigation of the NAC gene family and its potential association with the secondary cell wall in Moso Bamboo. Biomolecules, 2019, 9(10): 609. doi: 10.3390/biom9100609.
[35] WANG Q, GUO C, LI Z Y, SUN J H, DENG Z C, WEN L C, LI X X, GUO Y F. Potato NAC transcription factor StNAC053 enhances salt and drought tolerance in transgenic. International Journal of Molecular Sciences, 2021, 22(5): 2568. doi: 10.3390/ ijms22052568.
[36] ZHANG X, LONG Y, CHEN X X, ZHANG B L, XIN Y F, LI L Y, CAO S L, LIU F H, WANG Z G, HUANG H, ZHOU D G, XIA J X. A NAC transcription factor OsNAC3 positively regulates ABA response and salt tolerance in rice. BMC Plant Biology, 2021, 21(1): 546. doi: 10.1186/s12870-021-03333-7.
[37]曹瑞蘭, 李知青, 歐陽雯婷, 胡冬南, 周增亮, 蘇文娟, 陳霞, 劉娟.油茶NAC基因鑒定及對干旱脅迫響應分析. 江西農業大學學報, 2021, 43(6): 1357-1370. doi: 10.13836/j.jjau.2021145.
CAO R L, LI Z Q, OUYANG W T, HU D N, ZHOU Z L, SU W J, CHEN X, LIU J. Identification of NAC Gene in Camellia oleifera and Analysis of Its Response to Drought Stress. Acta Agriculturae Universitatis Jiangxiensis, 2021, 43(6): 1357-1370. doi: 10.13836/j. jjau.2021145. (in Chinese)
[38] 王佳麗, 王鶴冰, 楊慧勤, 胡若琳, 魏大勇, 湯青林, 王志敏. NAC轉錄因子在植物花發育中的作用. 生物工程學報, 2022, 38(8): 2687-2699.
WANG J L, WANG H B, YANG H Q, HU R L, WEI D Y, TANG Q L, WANG Z M. The role of NAC transcription factors in flower development in plants. Chinese Journal of Biotechnology, 2022, 38(8): 2687-2699. (in Chinese)
[39] 牛早柱, 趙艷卓, 陳展, 宣立鋒, 牛帥科, 褚鳳杰, 楊麗麗. 葡萄果實成熟相關NAC轉錄因子的篩選、克隆及表達分析. 果樹學報, 2022, 39(7): 1137-1146.
NIU Z Z, ZHAO Y Z, CHEN Z, XUAN L F, NIU S K, CHU F J, YANG L L. Screening, Cloning and Expression Analysis of NAC TranscriptionFactors Related to Grape Fruit ripening. Journal of Fruit Science, 2022, 39(7): 1137-1146. (in Chinese)
[40] MARTIN-PIZARRO C, VALLARINO J G, OSORIO S, MECO V, URRUTIA M, PILLET J, CASANAL A, MERCHANTE C, AMAYA I, WILLMITZER L, FERNIE A R, GIOVANNONI J J, BOTELLA M A, VALPUESTA V, POSE D. The NAC transcription factor FaRIF controls fruit ripening in strawberry. Plant Cell, 2021, 33(5): 1574-1593. doi: 10.1093/plcell/koab070.
[41] SWARNKAR M K, KUMAR P, DOGRA V, KUMAR S. Prickle morphogenesis in rose is coupled with secondary metabolite accumulation and governed by canonical MBW transcriptional complex. Plant Direct, 2021, 5(6): e00325. doi: 10.1002/pld3.325.
[42] ZHAO C S, AVCI U, GRANT E H, HAIGLER C H, BEERS E P. XND1, a member of the NAC domain family in, negatively regulates lignocellulose synthesis and programmed cell death in xylem. Plant Journal, 2008, 53(3): 425-436. doi: 10.1111/j. 1365-313X.2007.03350.x.
[43] SAKAMOTO S, MITSUDA N. Reconstitution of a secondary cell wall in a secondary cell wall-deficientmutant. Plant & cell physiology, 2015, 56(2): 299-310.
[44] 李媛, 陳金煥, 金曌, 侯景丫, 姜玉松, 邢海濤. 毛果楊NAC128基因在次生壁形成中的功能. 林業科學, 2020, 56(11): 62-72.
LI Y, CHEN J H, JIN Z, HOU J Y, JIANG Y S, XING H T. Functions of NAC128 Gene from Populus trichocarpa in Secondary Cell Wall Formation. Scientia Silvae Sinicae, 2020, 56(11): 62-72. (in Chinese)
[45] GUO Y F, GAN S S. AtNAP, a NAC family transcription factor, has an important role in leaf senescence. Plant Journal, 2006, 46(4): 601-612. doi: 10.1111/j.1365-313X.2006.02723.x.
[46] KOU X H, WATKINS C B, GAN S S.AtNAP regulates fruit senescence. Journal of Experimental Botany, 2012, 63(17): 6139-6147. doi: 10.1093/jxb/ers266.
Genome-Wide Identification of NAC Family and Screening of Its Members Related to Prickle Development inOld Blush
YOU YuWan, ZHANG Yu, SUN JiaYi, ZHANG Wei
College of Horticulture & Forestry Sciences, Huazhong Agricultural University/Key Laboratory of Horticultural Plant Biology, Ministry of Education/Key Laboratory of Urban Agriculture in Central China, Ministry of Agriculture and Rural Affairs, Wuhan 430070
This study was designed to identify thegene family inOld Blush and to analyze the sequences characteristics and expression pattern ofs to reveal the biological functions ofs, which also provided an important foundation to explore the role ofs in prickles.The BLATP and HMMER search were conducted to identify NAC proteins inOld Blush using the sequences of NAC proteins of. Physical and chemical properties, subcellular location, structure and phylogenetic relationship of each gene were further analyzed. Based on the released transcriptome data, the expression characteristics ofs in different tissues and organs under different stress conditions were analyzed. What’s more, the technology of RNA-seq was used to screengenes that might be related to the prickle development.In this study, 116genes fromOld Blush genome were identified and characterized. Theses genes encoded proteins containing 69 to 713 amino acids, with the theoretical isoelectric points ranging from 4.43 to 9.54 and the molecular weight ranging from 7.87 to 79.99 kD. The prediction of subcellular localization showed that 81s were located in the nucleus. Moreover,s were unevenly distributed on 7 chromosomes. According to phylogenetic relationships, AtNACs, OsNACs and RcNACs were clustered into 21 groups. These 116s were differentially expressed in various tissues and organs, and the expression levels of 31 members changed in response to abiotic and biotic stresses. Furthermore, in the RNA-seq data of prickles, 53s were detected, among which 26 members were differentially expressed genes.This study demonstrated thats were involved in the regulation of plant development and stress responses. Some members might be involved in the processes of prickle cell proliferation, secondary cell wall biosynthesis, and programmed cell death, which could be selected as candidate genes related with prickle development for further study.
‘Old Blush’; NAC transcription factor; transcriptome sequencing; prickle development

2022-03-04;
2022-04-24
國家自然科學基金(32072617)
由玉婉,E-mail:1797435616@qq.com。通信作者張蔚,E-mail:zhangw@mail.hzau.edu.cn
(責任編輯 趙伶俐)

