羅非魚魚糜自然發酵過程中微生物群落結構對其品質形成的影響
趙 躍,李春生*,王悅齊,陳勝軍,李來好*,黃 卉
(1.上海海洋大學食品學院,上海 201306;2.中國水產科學研究院南海水產研究所,農業農村部水產品加工重點實驗室,廣東 廣州 510300)
羅非魚(Tilapia)是我國南方最主要的淡水養殖品種之一,憑借生長速度快、繁殖量大、容易養殖、產量高等特點,逐漸成為全球關注的淡水養殖魚類[1]。2019年我國羅非魚的總產量達到164.2萬 t,較2018年產量提高了1.05%[2]。近幾年,我國羅非魚養殖產量占全球羅非魚總產量的近50%[3]。目前,羅非魚加工產品形式單一,大部分加工成魚片出口,精深加工產品少,產品附加值低[4]。因此,合理利用羅非魚資源,開發切實有效的加工技術,已成為我國羅非魚產業持續健康發展的當務之急。魚糜加工是目前國內外發展較快的魚制品加工方式。魚糜制品具有高蛋白、低脂肪、口感嫩爽、食用方便等特點,并且產量逐年增加。由于海水魚魚糜具有良好的凝膠特性,市場上魚糜制品的生產原料多采用海水魚[5]。研究發現,羅非魚魚肉中鹽溶性蛋白含量低,應用傳統魚糜加工方式加工形成的魚糜制品凝膠強度差,極大地限制了羅非魚魚糜加工的應用[4]。因此提高羅非魚魚糜凝膠強度,開發高品質羅非魚魚糜制品,已成為羅非魚魚糜加工亟待解決的關鍵問題。
利用微生物發酵技術提高淡水魚魚糜品質是目前研究的熱點。發酵魚糜一般使用乳酸菌作為發酵劑,依靠快速降低的pH值促進魚肉凝膠化,抑制有害微生物的生長[6-7]。同時,乳酸菌發酵作用使魚糜的蛋白質、脂肪或碳水化合物發生不同變化,形成其特有的風味與口感[8]。國內外對發酵魚糜的研究主要集中于利用市售的乳酸菌等微生物菌種作為發酵劑,分析加菌發酵對魚糜品質和風味等相關理化指標的影響。楊方[9]利用Lactobacillus plantarum發酵鰱魚魚糜,建立發酵魚糜體系和酸性模擬體系,探討魚肉內源酶對發酵魚糜凝膠特性的影響。Liu Zhongyi等[6]利用L.casei、Streptococcuslactis、Saccharomyces cerevisiae和Monascus anka混合發酵鳙魚魚糜,結果發現加菌發酵后的魚糜在味道和外觀上都優于未加菌的對照組。王洋等[7]利用L.pentosus、L.plantarum、Staphylococcus sciuri、Staphylococcus xylosus等復合發酵劑發酵鱘魚魚糜,其產品具有優良的感官品質和理化特性。然而,目前發酵魚糜研究主要集中鰱魚、鳙魚、草魚、鱘魚等淡水魚,對羅非魚魚糜發酵過程品質變化的研究鮮見相關報道。
發酵魚糜一般采用添加單菌或復合菌劑的方式進行發酵生產,然而由于魚糜原料中微生物菌群比較復雜,實際的魚糜品質形成過程是由復雜的微生物菌群代謝活動引起。明確發酵魚糜復雜微生物群落的結構及其功能調控是實現發酵魚糜產業升級的重要環節。然而,目前絕大部分研究并未考慮微生物群落結構及其代謝活動對發酵魚糜品質的影響。而且,由于不同淡水魚魚糜中蛋白質、脂質等營養物質差異較大,盲目使用微生物發酵劑不僅不會改善魚糜品質,而且會與原料自身優勢菌群發生競爭性抑制,大大降低發酵魚糜的品質。因此,如何篩選適用于特定淡水魚魚糜的功能微生物是發酵魚糜研究的難點。目前,基于16S/18S/ITS的二代高通量測序技術已較為普遍地應用于傳統發酵食品微生物菌群多樣性研究。朱雯娟等[10]利用16S rRNA高通量測序技術對梅香魚樣品的微生物菌群進行分析,發現Lactobacillus、Staphylococcus和Tetragenococcus是參與梅香魚發酵過程的優勢菌屬。Wang Zongmin等[11-12]通過16S rRNA和ITS高通量測序技術,明確了Acetobacter、Lactobacillus、Enhydrobacter、Lactococcus、Gluconacetobacer、Bacillus、Staphylococcus7 個微生物菌屬是鎮江香醋中產生獨特風味的核心微生物菌群。利用16S rRNA高通量測序技術能夠解析羅非魚發酵魚糜中的微生物菌群,進一步利用Pearson、主成分分析(principal component analysis,PCA)等統計學分析方法能夠揭示微生物菌群對發酵魚糜品質形成的影響規律,有利于定向篩選適用于特定淡水魚魚糜的功能微生物,避免所用微生物發酵劑不合適或效果差,降低了發酵劑選擇盲目性,節省了發酵魚糜生產成本。
因此,本研究以羅非魚魚糜為研究對象,利用16S rRNA高通量測序技術揭示羅非魚魚糜自然發酵過程中微生物菌群變化情況,結合羅非魚魚糜發酵過程中質構、凝膠強度、色度等品質變化結果,借助Pearson、PCA等統計學分析方法揭示微生物菌群對發酵魚糜品質形成的影響規律,為定向分離篩選適用于羅非魚發酵魚糜的功能微生物、靶向改善羅非魚發酵魚糜品質提供一定理論依據。
1 材料與方法
1.1 材料與試劑
羅非魚、食鹽、蔗糖和葡萄糖(食品級) 廣東廣州市海珠區客村華潤萬家超市。
PCA培養基 廣東環凱微生物科技有限公司;FastDNA?Spin Kit for Soil DNA抽提試劑盒 美國MP Biomedicals公司;NEXTFLEX?Rapid DNA-Seq Kit建庫試劑盒 美國Bioo Scientific公司;MiSeq Reagent Kit v3測序試劑盒 美國Illumina公司;Biowest Agarose瓊脂糖西班牙Biowest公司;FastPfu Polymerase聚合酶 中國TransGen公司。
1.2 儀器與設備
IKA-T25組織勻漿機 德國IKA公司;CT3質構儀美國Brookfield公司;3K30臺式高速冷凍離心機 德國Sigma公司;SZ-22絞肉機 湖南省長沙固利食品機械有限公司;CR-410全自動色差儀 日本柯尼卡美能達光學影像公司;QuantusTMFluorometer微型熒光劑 美國Promega公司;Illumina MiSeq測序儀 美國Illumina公司;ABI GeneAmp?9700型聚合酶鏈式反應(polymerase chain reaction,PCR)儀 美國ABI公司。
1.3 方法
1.3.1 羅非魚發酵魚糜制備
在市場購買鮮活羅非魚(500~750 g/條)后,立刻運往實驗室,去頭、去鱗、去內臟,取背部魚肉并用冷水沖洗,然后用絞肉機絞碎成魚糜。在低溫環境下(4 ℃),將魚糜與0.5%葡萄糖、2%食鹽、0.5%蔗糖等輔料斬拌均勻。使用手動灌腸機灌入直徑30 mm的塑料腸衣中,每根長約15 cm,然后置于30 ℃恒溫恒濕培養箱中發酵30 h,分別在第0、18、24、30小時取樣分析(標記為C0、C18、C24、C30)。
1.3.2 16 S rRNA高通量測序
將3.0 g發酵香腸充分攪碎后,置于27 mL生理鹽水中充分混勻,取含菌的生理鹽水在12 000 r/min、4 ℃離心10 min,獲得菌體沉淀。16S rRNA高通量測序參照文獻[8]方法。采用FastDNA?Spin Kit for Soil試劑盒說明書進行微生物群落總DNA抽提,利用NanoDrop2000檢測DNA純度和濃度。采用上游引物338F(5’-ACTCCTACGGGAGGCAGCAG-3’)和下游引物806R(5’-GGACTACHVGGGTWTCTAAT-3’)對16S rRNA基因V3-V4可變區進行PCR擴增,擴增程序如下:95 ℃預變性3 min;95 ℃變性30 s、55 ℃退火30 s、72 ℃延伸45 s,27 個循環;然后72 ℃穩定延伸10 min,最后在10 ℃進行保存。PCR產物經2%瓊脂糖凝膠電泳鑒定后,使用AxyPrep DNA Gel Extraction Kit純化,用QuantusTMFluorometer進行檢測定量,使用NEXTFLEX?Rapid DNA-Seq Kit進行建庫,利用Illumina MiSeq PE300進行雙末端測序。
1.3.3 質構分析
將發酵香腸切成高25 mm且切面平整的圓柱體,采用質構儀測定發酵魚糜的硬度、彈性、黏性、內聚力、咀嚼性等質構特性。選用4 mm TA44平底圓柱形探頭,TA-RT-KIT夾具,測試速率1 mm/s,距離目標值15 mm,每個樣品進行軸向壓縮2 次,2 次下壓間隔時間1 s,觸發點負載5 g。每次實驗做5 次平行,取平均值。
1.3.4 凝膠強度分析
將發酵香腸切成高25 mm且切面平整的圓柱體,采用質構儀分析魚糜凝膠的凝膠強度。選用壓縮模式測定香腸凝膠的破斷強度(g)和凹陷深度(cm),選用直徑5 mm球形探頭壓縮樣品,下壓位移設置為15 mm,觸發點負載5 g,測試速率1 mm/s。每次實驗做5 個平行,取平均值。凝膠強度按式(1)計算:

1.3.5 色差分析
將發酵香腸切成25 mm且切面平整的圓柱體,采用全自動色彩色差儀在457 nm波長處用標準白板對儀器進行校對,然后測定魚糜凝膠的亮度(L*)值、紅/綠(a*)值、黃/藍(b*)值,按式(2)計算白度值(W):
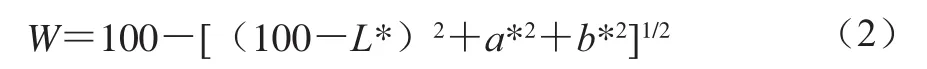
1.3.6 pH值測定
將5.0 g發酵香腸置于45 mL超純水中勻漿,10 000 r/min離心10 min取上清液,用酸度計測定pH值。
1.3.7 菌落總數測定
將3.0 g發酵香腸充分攪碎后,置于27 mL生理鹽水中充分混勻,梯度稀釋后,涂布于PCA培養基上,將平板倒置于37 ℃培養箱內培養24 h后計數。
1.4 數據分析
1.4.1 微生物菌群結構分析
采用Fastp(v0.19.6)軟件對16S rRNA高通量測序原始序列進行質控,利用Flash(v1.2.11)軟件將雙末端測序得到的成對reads進行組裝拼接。使用UPARSE(v7.0.1090)軟件在97%相似度條件下進行操作分類單元(operational taxonomic units,OTU)聚類并剔除嵌合體,利用RDP classifer(v2.11)軟件比對Silva 16S rRNA數據庫(v138)進行物種分類注釋。分別采用Mothur(v1.30.2)和QIIME(v1.9.1)軟件計算樣品的α多樣性值和β多樣性值。
1.4.2 統計學分析
采用單因素方差分析中的Tukey-Kramer檢驗方法,“*”表示不同微生物菌群在不同發酵時間下菌群差異顯著(P<0.05),利用Pearson、PCA等統計學方法分析微生物菌群與發酵魚糜品質變化的相關性。
2 結果與分析
2.1 16S rRNA高通量測序數據統計
利用Illumina MiSeq測序技術研究羅非魚發酵魚糜微生物群落結構和多樣性變化,如表1所示。8 個樣品共得到368 664 條序列,平均每個樣品46 083 條序列,平均長度約為428 bp。序列經過優化后,在97%相似度下將其聚類為用于物種分類的OTU。8 個樣品共獲得246 種OTU,其中發酵24 h的OTU種類最多,平均值達到117 種。
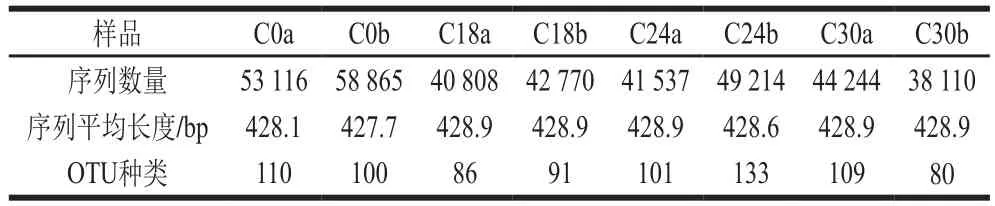
表1 羅非魚魚糜自然發酵過程中微生物16S rRNA高通量測序數據統計Table 1 Statistics of bacterial 16S rRNA gene sequencing data for tilapia surimi during natural fermentation
2.2 羅非魚魚糜發酵過程中微生物多樣性變化
2.2.1α多樣性分析
α多樣性是對單個樣品中物種多樣性進行分析,Sobs指數、Chao指數和ACE指數反映樣品中群落的豐富度,不考慮群落中每個物種的豐度情況,而Shannon指數和Simpson指數則同時考慮群落中物種豐富度和物種均勻度的影響。覆蓋率反映各樣本文庫的覆蓋程度,即樣本中序列被測出的概率。如表2所示,Sobs指數、Chao指數和ACE指數結果顯示,發酵到第24小時,樣品中的微生物豐富度最高;Shannon指數和Simpson指數結果顯示,未發酵時物種的均勻度最高,自然發酵能降低物種的均勻度,發酵后期(24~30 h)物種的均勻度最小;所有發酵時間樣品的序列被捕獲概率均大于0.999,表明測序結果的完整度較高。

表2 不同發酵時間羅非魚魚糜微生物α多樣性分析Table 2 α-Diversity analysis of microbial community in tilapia surimi at different fermentation stages
2.2.2 獨有和特有的OTU組成分析
如圖1所示,4 個發酵時間點共有的微生物種類為54 種;發酵后期即發酵24 h和30 h共有微生物種類最多,達到105 種;發酵24 h時,獨有的微生物種類最多,達到47 種。
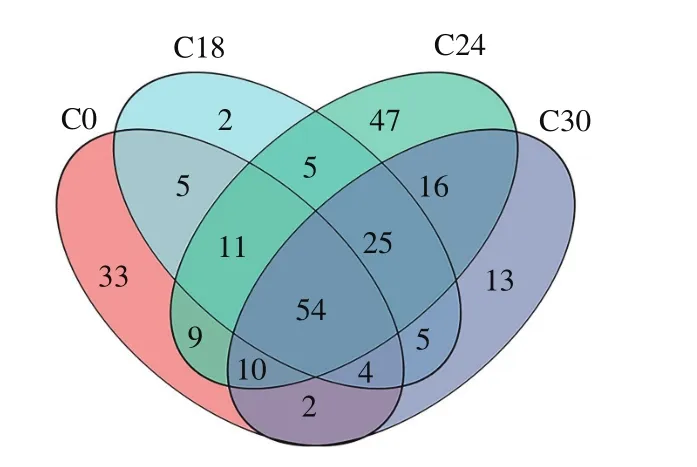
圖1 不同發酵時間羅非魚魚糜微生物群落Venn圖Fig.1 Venn diagram analysis of microbial community in tilapia surimi at different fermentation stages
2.2.3β多樣性分析
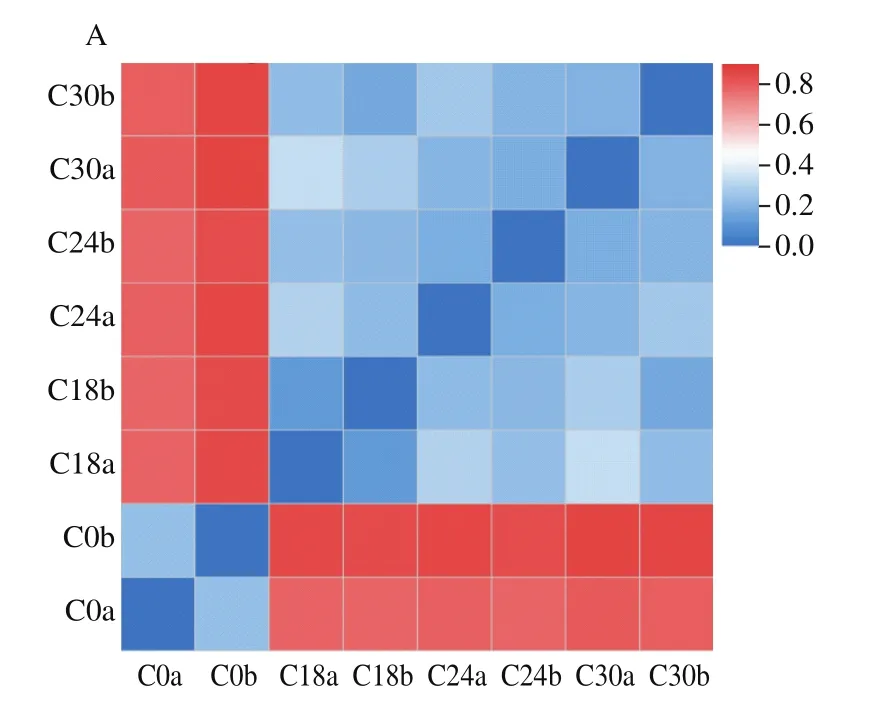
在OTU分類水平上采用Bray-Curtis、unweighted UniFrac和weighted UniFrac距離算法比較不同發酵時間羅非魚魚糜β多樣性,結果如圖2所示。Bray-Curtis的計算只考慮樣品中物種的存在情況,而不考慮序列間的進化距離。結果顯示,平行樣品間微生物菌群差異較小,發酵18、24 h和30 h樣品的微生物菌群差異較小,而與未發酵魚糜中微生物菌群的差異較大(圖2A)。UniFrac是利用系統進化的信息比較不同樣品間物種群落差異,其中unweighted UniFrac不考慮物種豐度,而weighted UniFrac考慮物種豐度。在不考慮物種豐度(圖2B)和考慮物種豐度(圖2C)的情況下,其分析結果均與Bray-Curtis結果相似,平行樣品間微生物菌群差異最小,羅非魚魚糜自然發酵后(18~30 h)與未發酵魚糜的菌群差異較大。
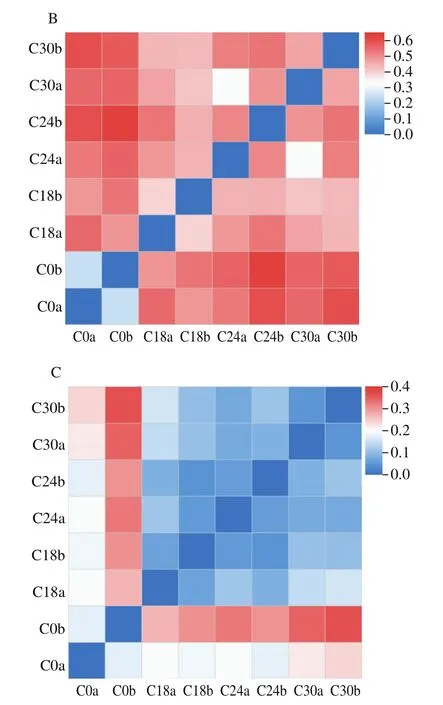
圖2 不同發酵時間羅非魚魚糜微生物菌群β多樣性熱圖Fig.2 Heatmap for β-diversity of microbial community in tilapia surimi at different fermentation stages
2.3 羅非魚魚糜發酵過程中微生物群落結構變化
羅非魚魚糜在自然發酵條件下微生物菌群變化較為明顯(圖3)。在門水平上(圖3A),Firmicutes和Proteobacteria為整個發酵過程主要微生物菌門,隨著發酵時間的延長,Firmicutes中的微生物相對含量逐漸增加,而Proteobacteria相對含量逐漸下降。這與傳統豬肉干制香腸[13]和自然發酵魚露[14]發酵過程門水平上微生物菌群變化結果相似。自然發酵30 h后,Firmicutes成為最主要微生物菌群,相對含量接近90%。

圖3 不同發酵時間羅非魚魚糜微生物菌群在門(A)和屬(B)水平的相對豐度Fig.3 Relative abundance of microbial community in tilapia surimi at different fermentation stages at phylum (A) and genus (B) levels
在屬水平上(圖3B),Lactococcus、Macrococcus、Enterobacter、Citrobacter、Enterococcus等微生物菌屬在整個發酵過程中的變化具有顯著差異(P<0.05)。未發酵前羅非魚魚糜原料中Macrococcus(29.0%)、Acinetobacter(22.2%)、Lactococcus(12.6%)、Floricoccus(11.5%)、Streptococcus(6.9%)為主要微生物菌群。Lactococcus在自然發酵18 h內的相對豐度顯著增長到64.9%,并在隨后的發酵過程中(18~30 h)相對豐度保持穩定,因而是羅非魚發酵魚糜中主要的微生物菌屬。Lactococcus在魚茶[15]、豆腐乳[16]等其他發酵食品中也是主要的發酵菌屬。值得注意的是,Pediococcus在羅非魚魚糜原料中的相對豐度較低(0.02%),隨著發酵時間的延長而增長迅速,發酵30 h的相對含量僅次于Lactococcus,達到13.2%。此外,Enterococcus和Lactobacillus等微生物菌屬的相對豐度也隨著發酵時間的延長呈增長趨勢。由此可見,Lactococcus、Pediococcus、Enterococcus、Lactobacillus等微生物菌屬均可適應羅非魚魚糜發酵環境,這些微生物的代謝作用可能對發酵魚糜品質的形成有很大的影響。
隨著發酵時間的延長,Macrococcus、Acinetobacter、Streptococcus、Floricoccus、Rothia、Enhydrobacter、Psychrobacter、Pseudomonas、Chryseobacterium等微生物菌屬的相對含量呈降低趨勢,同時,Enterobacter、Citrobacter、Photobacterium、Aeromonas、Plesiomonas、Vagococcus、Vibrio在發酵18 h時呈顯著增長趨勢,但發酵后期(24~30 h)含量又逐漸下降。由此可見,這些微生物菌屬可能不適應羅非魚魚糜的弱酸性厭氧發酵環境。Yin等[17]相關研究報道表明添加乳酸菌能顯著抑制發酵鯖魚糜中Pseudomonas、Staphylococcus和Enterobacteriaceae的生長,并在發酵12 h后即成為優勢菌種。因此,發酵后期羅非魚發酵魚糜中有害微生物豐度的顯著降低可能與Lactococcus、Pediococcus、Enterococcus、Lactobacillus等乳酸菌的大量繁殖有關。
2.4 羅非魚魚糜發酵過程中品質特征變化
如圖4所示,凝膠強度是衡量魚糜凝膠制品品質最基本的指標之一。一般來說,具有令人愉悅的彈性口感的凝膠強度在300 g·cm以上[18],而使用加熱法生產的羅非魚魚糜的凝膠強度只有200 g·cm左右[4]。本研究發現,自然發酵能夠顯著提高羅非魚魚糜的凝膠強度,發酵18 h羅非魚魚糜的凝膠強度超過450 g·cm,發酵30 h超過670 g·cm。而且,利用發酵方法得到的羅非魚魚糜的凝膠強度也優于加熱法生產的混合海水魚(馬鮫魚、海鱸魚、金鯧魚等)的羅非魚魚糜[19]。白度也是評價魚糜凝膠制品的標準之一,它直接影響消費者對商品的可接受程度,對于魚糜制品而言,白度越高產品越受消費者喜愛[20]。結果發現,自然發酵能顯著提高魚糜的白度,且發酵時間(18~30 h)對魚糜白度的影響無顯著性差異。發酵后羅非魚魚糜的白度值明顯優于加熱法生產的羅非魚魚糜,且優于海鱸魚、黃花魚、金鯧魚、紅三魚、馬鮫魚等海水魚魚糜[4]。羅非魚魚糜發酵過程中菌落總數呈逐漸上升趨勢,可能與Lactococcus、Pediococcus、Enterococcus、Lactobacillus等乳酸菌的大量繁殖有關。同時,羅非魚魚糜的pH值在18 h內迅速下降,發酵后期(24~30 h)則降速減緩。菌落總數和pH值變化結果與自然發酵鰱魚魚糜的結果一致[21]。pH值是影響魚糜凝膠形成的重要因素,研究表明,由于pH值的變化可以改變氨基酸殘基側鏈的電荷分布,蛋白質分子在等電點處所帶的靜電荷為0,蛋白質分子間就失去了靜電斥力作用,使蛋白分子相互迅速聚集,使產品產生較佳的硬度與彈性[22]。本研究中,羅非魚魚糜發酵過程中pH值的下降對其凝膠強度的改善起著重要作用。
質構特性是影響發酵香腸品質的重要因素,優良發酵香腸的組織結構應該是緊密且切片富有彈力,其中,硬度、彈性和黏性能比較好反映魚糜制品的感官適口性,是表征香腸質地特征的重要力學參數[23]。如圖4所示,羅非魚魚糜發酵前的黏性較高,而隨著發酵的進行,魚糜由溶膠狀態變為膠狀組織,使其黏性逐漸下降。硬度是指第1次壓縮樣品時的壓力峰值,可衡量其熟化程度,通常是由魚肉蛋白的變性和凝膠化以及水分的損失引起。羅非魚發酵魚糜的硬度在發酵過程中始終保持增長趨勢,并在第30小時達到最大。彈性反映的是試樣在第1次壓縮形變后能夠再復原的程度;內聚性反映形成食品形態所需內部結合力的大小[24];咀嚼性反映了魚糜制品從咀嚼的狀態到可吞咽狀態所需要的能量,咀嚼性越高“咬感”就越好[25]。羅非魚發酵魚糜的彈性、內聚性和咀嚼性在發酵過程中的變化趨勢相同,表現為在發酵前18 h明顯下降并在24 h達到最大值。發酵24 h時,羅非魚發酵魚糜具有更佳的硬度與彈性,此特性有助于發酵魚肉香腸被切成薄片。
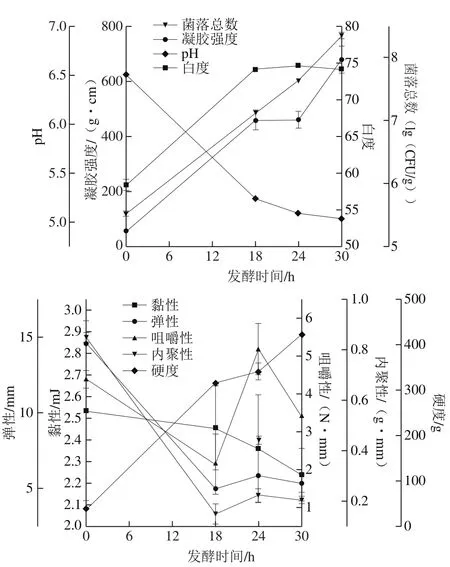
圖4 不同發酵時間羅非魚魚糜的品質特征變化Fig.4 Changes in quality characteristic of tilapia surimi at different fermentation stages
2.5 微生物群落結構對羅非魚發酵魚糜品質特性變化的影響規律
為研究微生物群落結構對羅非魚發酵魚糜品質特性變化的影響規律,篩選相對豐度前10的菌屬(Lactococcus、Macrococcus、Acinetobacter、Enterobacter、Pediococcus、Citrobacter、Floricoccus、Streptococcus、Enterococcus、Rothia),結合發酵魚糜品質指標(凝膠強度、白度、菌落總數、pH值、黏性、彈性、咀嚼性、內聚性、硬度),利用Pearson和PCA等統計學分析方法研究微生物群落結構與魚糜品質的相關性。
如圖5A所示,凝膠強度、白度、硬度等關鍵魚糜品質指標之間具有顯著正相關(P<0.05),同時與菌落總數、Pediococcus、Enterobacter、Citrobacter、Enterococcus、Lactococcus表現出正相關,特別是與Lactococcus的正相關最為顯著(P<0.05),相關系數分別達到0.907、0.980、0.957;與內聚性、彈性和pH值以及Acinetobacter、Rothia、Floricoccus、Streptococcus、Macrococcus等菌屬具有顯著負相關(P<0.05),而與咀嚼性和黏性均無顯著相關。PCA作為一種多變量的相關性分析方法,已被應用于分析中國米酒[26]和韓國Saeu-jeot[27]等發酵食品中主要的菌群與化學物質之間的關系。本研究中,利用PCA方法比較微生物菌群與魚糜品質的相關性,如圖5B所示。結果顯示,PCl的方差貢獻率為77.7%,PC2的方差貢獻率為9.6%,這2 個PC可以反映全部信息的87.3%,提取較為完全,說明這2 個PC能夠替代原始19 個成分反映樣品信息。結果顯示,就不同發酵階段而言,羅非魚魚糜發酵后(18、24、30 h)的樣本與魚糜原料(0 h)的差異性較大。PCA相關性分析結果與Pearson聚類結果類似,19 個成分明顯分為兩大類,一類為凝膠強度、白度、菌落總數、硬度、Lactococcus、Enterobacter、Pediococcus、Citrobacter、Enterococcus,另一類為pH值、黏性、彈性、咀嚼性、內聚性、Macrococcus、Acinetobacter、Floricoccus、Streptococcus、Rothia,每個類別內的成分之間呈正相關,2 個類別間的成分呈負相關,其中發酵過程中Lactococcus相對豐度的增加與凝膠強度、白度、硬度的提高以及魚糜pH值的下降相關性最為顯著(P<0.05)。Pearson相關性分析和PCA結果表明,羅非魚發酵魚糜中Lactococcus、Pediococcus、Enterobacter、Citrobacter、Enterococcus等產酸菌的代謝作用導致發酵魚糜的pH值逐漸下降,從而促進了凝膠強度、白度、硬度等關鍵魚糜品質指標的改善。

圖5 利用Pearson(A)和PCA(B)方法分析微生物群落結構與羅非魚發酵魚糜品質的相關性Fig.5 Correlation analysis of microbial community with quality indexes in fermented tilapia surimi using Pearson correlation analysis (A) and PCA (B)
Lactobacillus、Pediococcus等乳酸菌屬經常被用作商業發酵劑用于淡水魚魚糜發酵,有助于發酵魚糜形成良好的品質[21,28-29]。而本研究中相對豐度最高的Lactococcus在淡水魚發酵魚糜的應用較少。盡管Enterococcus并未列入歐盟安全合理推定和美國公認安全認證的安全性菌屬名單,但有研究報道發現,Enterococcus是奶酪和香腸發酵過程中常見的微生物菌屬,對其品質和風味形成起著重要的作用[28,30]。本研究表明,Lactococcus、Pediococcus、Enterococcus等乳酸菌屬,特別是Lactococcus和Pediococcus與羅非魚發酵魚糜品質形成密切相關。
3 結 論
采用16S rRNA高通量測序技術研究羅非魚魚糜自然發酵過程中微生物群落結構以及多樣性變化。羅非魚魚糜發酵過程中微生物多樣性較為豐富,并在發酵第24小時的OTU總數和獨有的種類最多,發酵18、24和30 h樣品的微生物菌群差異較小,而與發酵初期(0 h)菌群差異較大。Firmicutes和Proteobacteria為整個發酵過程主要微生物菌門,隨著發酵時間的延長,Firmicutes中的微生物相對含量逐漸增加,而Proteobacteria相對含量逐漸下降。Lactococcus、Pediococcus、Enterococcus、Lactobacillus等微生物菌屬均可適應羅非魚魚糜發酵環境,隨著發酵時間的延長而迅速增長。Lactococcus是羅非魚發酵魚糜中主要的微生物菌屬,在自然發酵18 h內的相對豐度從12.6%顯著增長到64.9%,并在隨后的發酵過程中保持穩定,其次是Pediococcus,發酵第30小時的相對含量達到13.2%。隨著發酵時間的延長,凝膠強度、白度、硬度、菌落總數等品質指標逐漸增加,pH值、黏性、彈性、咀嚼性、內聚性等品質指標與發酵前相比有所下降。Pearson相關性分析和PCA發現,羅非魚發酵魚糜中Lactococcus、Pediococcus、Enterococcus、Enterobacter、Citrobacter等產酸菌屬與凝膠強度、白度、硬度等關鍵魚糜品質指標呈正相關而與pH值呈負相關,這些菌的代謝作用(特別是產酸特性)對于發酵魚糜品質的形成發揮著重要作用。基于本實驗結果,后續可以從Lactococcus、Pediococcus等乳酸菌中定向分離篩選菌株,開發適用于羅非魚魚糜的發酵劑,用于靶向改善羅非魚發酵魚糜品質,本研究結果也為其他淡水魚魚糜發酵菌株的定向篩選提供了重要技術參考。

