基于蛋白質結構域特異性的關鍵蛋白質識別算法*
楊増光
(南京理工大學 南京 210094)
1 引言
眾所周知,蛋白質(Protein)在細胞的組成和生物體的生命活動中扮演著極其重要的作用。但不同類型的蛋白質對生物體的重要程度不盡相同,其中那些缺失后會導致生物體病變甚至死亡的蛋白質被稱為關鍵蛋白質(essential proteins),其余的則被稱為非關鍵蛋白質(non-essential proteins)[1~3]。
研究表明,關鍵蛋白質的識別對于我們了解細胞的生長調控過程,研究生物進化的相關機制,以及根據關鍵蛋白質進行藥物設計、藥物標靶鑒定和疾病治療等方面具有著不可忽視的現實意義[4]。
在生物學領域中,識別關鍵蛋白質通常是采用生物醫學實驗的方式進行的,這類方法雖然準確,但是成本高、效率低,無法適用于日益增長的蛋白質數據。隨著高通量技術的發展,越來越多的蛋白質相互作用數據被獲取,這讓我們能夠從網絡水平上識別關鍵蛋白質。
目前,越來越多的研究人員將圖論、復雜網絡等相關知識應用到蛋白質網絡中,并提出多種有效的方法來識別關鍵蛋白質,其中常用的有8種具有代表性的算法:DC[5]、BC[6]、CC[7]、SC[8]、EC[9]、IC[10]、LAC[11]、NC[12]。這些算法雖然能夠有效地識別出關鍵蛋白質,但是由于這類算法容易受到網絡中假陰性和假陽性數據的影響且忽略了蛋白質網絡蘊含的生物信息,因而它們的識別精度不高。
本文,我們提出一種基于蛋白質結構域特異性的關鍵蛋白質識別算法Do-ECC,通過融合蛋白質網絡的拓撲信息和生物信息,能夠有效提高關鍵蛋白質的識別準確度。
2 邊聚集系數
為充分利用蛋白質網絡的拓撲信息,首先需要尋找一個有效的拓撲特征。研究表明,關鍵蛋白質更可能和關鍵蛋白質相連,并且成簇出現,而非關鍵蛋白質則表現稀疏,即關鍵蛋白質在網絡中所處的位置相比于非關鍵蛋白質擁有更高的連通度和模塊化程度[11~12]。基于此,越來越多的研究人員開始使用邊聚集系數作為描述蛋白質網絡的拓撲特征來開展自己的研究,實驗結果也表明,這一特征確實能夠更全面、更準確地描述蛋白質網絡的拓撲信息。
對網絡中的任一條邊,邊聚集系數被定義為該邊在網絡中實際參與構成的三角形個數與該邊最多可能參與構成的三角形個數之比。如對于邊E(u,v),其邊聚集系數可表示為
其中zu,v表示網絡中該邊實際參與構成的三角形的個數,ku和kv分別表示節點u和v的度,則表示該邊最多可能參與構成的三角形的個數。不難看出,邊聚集系數的取值介于0~1之間。對于任一條邊,其邊聚集系數越大,表明其參與網絡模塊結構的比重越多,在網絡中所處位置的聚集程度也越高。
3 蛋白質結構域特異性
大多數蛋白質通常是由一個或者多個功能區域組成,這些區域一般被稱為蛋白質結構域(Protein Domain),是蛋白質結構和功能的基本單位。而在自然界中,復雜的蛋白質分子則是由這些結構域通過不同的組合和重排形成的。研究表明,那些在生物體中出現頻率較少的結構域對于生物體具有更加關鍵的作用;另一方面,包含較多結構域的蛋白質分子,通常執行更多的生物功能,對正常的生命活動更加重要,也更有可能是關鍵蛋白質[13]。
3.1 TF-IDF算法
在信息檢索、文本分類等相關領域,TF-IDF(Term Frequency-Inverse Document Frequency),即“詞頻-逆文本頻率”,是一種常用的加權技術,用以評估一個單詞對于文本和語料庫的區分能力與重要程度。
其中TF(Term Frequency),即詞頻,是指一個單詞在一個文本中出現的頻率,通常表示為這個單詞在文本中出現的次數和該文本包含的單詞的總數之比,即:
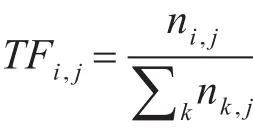
其中,ni,j表示單詞i文本 j中出現的次數,k表示文本 j包含的單詞類型的數目,則表示該文本包含的單詞的總數。
IDF(Inverse Document Frequency),即逆文本頻率的概念,對于一個單詞,它是指整個語料庫中包含該單詞的文本的數量,通常表示為先計算語料庫的文本總數和包含該單詞的文本數之比,然后取對數,即:
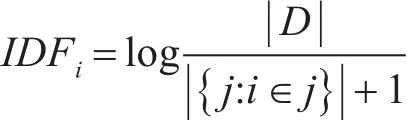
而TF-IDF就是通過結合兩者,用來評估一個單詞對于文本和語料庫的區分能力與重要程度,通常表示為
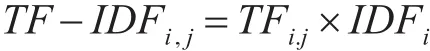
根據上述定義,可以發現,一個單詞的重要性和區分能力隨著它在文本中出現的次數成正比增加,但同時會隨著它在整個語料庫中出現的頻率成反比下降。
3.2 結構域特異性
借鑒TF-IDF算法的思想,我們對蛋白質結構域進行重新審視。如果將每種類型的結構域都當作一個單詞,那么每條蛋白質就相當于一個文本文件,而整個生物體包含的所有蛋白質就組成了一個語料庫。如圖1所示,如果將PF00270、PF00271等幾種結構域視作一種單詞,則蛋白質YER172C、YBL084C、YDL126C的“文本”組成可以表示如圖1所示。
根據IDF的定義,本文提出了IPF(Inverse Protein Frequency)的概念,來描述蛋白質結構域的特異性,即由生物體包含的蛋白質總數除以包含該結構域的蛋白質數目,再將得到的商數取對數,如下所示:
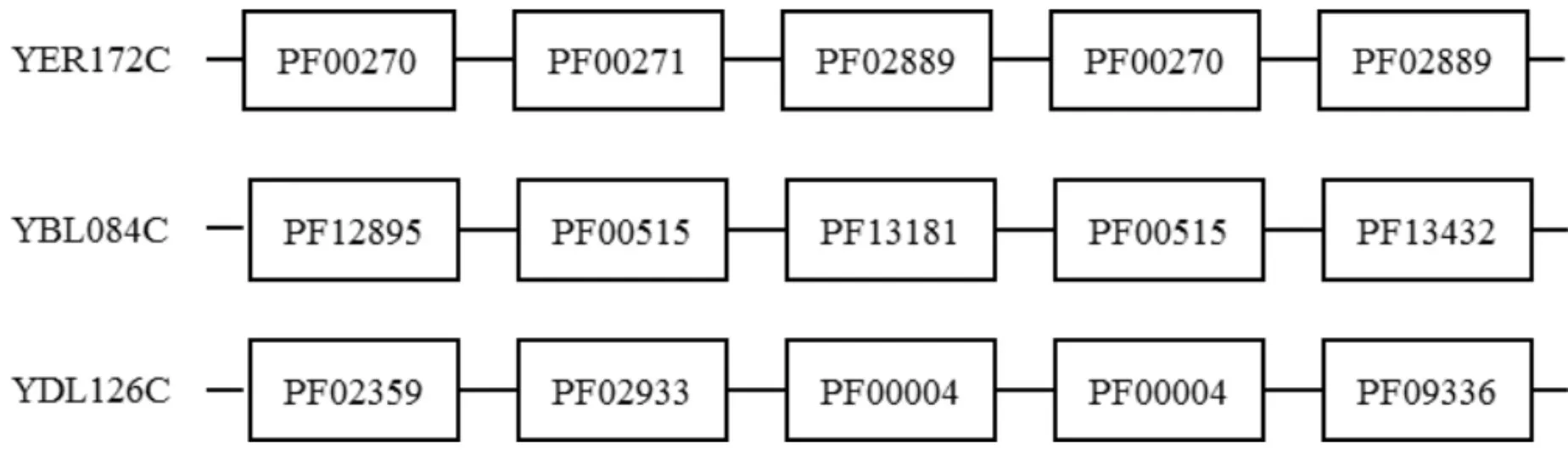
圖1 蛋白質的結構域組成示意圖
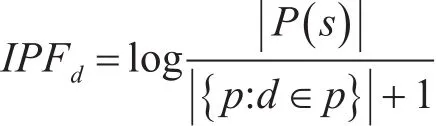
同理,根據TF的定義,本文提出DF(Domain Frequency)的概念,指一個結構域在一個蛋白質分子中出現的頻率,表示為這個結構域在特定蛋白質分子中出現的次數和該蛋白質包含的結構域的總數之比,即:
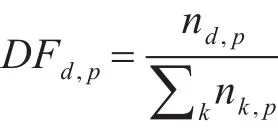
其中,nd,p表示蛋白質結構域d在蛋白質分子 p中出現的頻次,k表示蛋白質分子 p包含的結構域種類數,則表示蛋白質分子 p包含的結構域的總數。
3.3 蛋白質的結構域特異性得分
參照TF-IDF的定義,本文給出DF-IPF的概念。對于結構域d,其特異性為IPFd,在蛋白質 p中出現的頻率為DFd,p,則它對應的DF-IPF值可以表示如下:
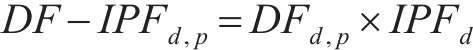
它可以用于描述一個結構域d對蛋白質 p的重要程度,也可以用于度量蛋白質 p基于結構域d獲得的特異性得分。而一個蛋白質可能包含多種類型的結構域,則其總的結構域特異性得分可以表示為
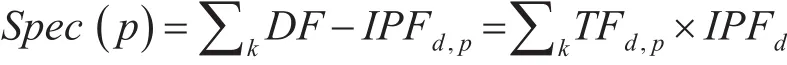
其中,k表示蛋白質 p包含的結構域的種類。
4 Do-ECC算法
如上所述,邊聚集系數能夠描述蛋白質網絡的拓撲信息,蛋白質的結構域特異性得分則反映了蛋白質網絡蘊含的生物信息。本文,我們通過融合這兩種特征,提出一種新的關鍵蛋白質識別算法Do-ECC。
為方便介紹,首先對蛋白質網絡進行建模,將其表示成一個無向圖G(V ,E ),如對于存在相互作用的兩個蛋白質分子,可以將這兩個蛋白質分別表示為節點u和v,而將它們間的相互作用表示邊E(u ,v )。
根據上述定義,對于相互作用E(u ,v) ,其邊聚集系數可以表示為ECC(u ,v)。為能夠和結構域信息進行融合,需對ECC(u ,v)進行歸一化處理,表示為
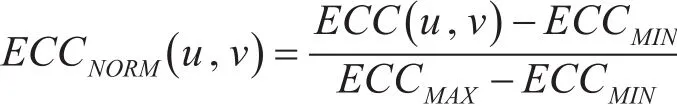
其中,ECCMAX和ECCMIN分別表示所有相互作用的邊聚集系數的最大值和最小值。
對于蛋白質節點u,它的結構域特異性得分可以表示為Spec()u,同樣需要進行歸一化處理,表示為
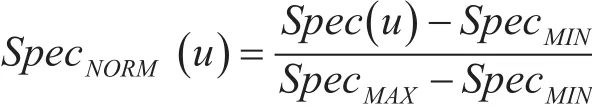
其中,SpecMAX和SpecMIN分別表示所有蛋白質分子的結構域特異性得分的最大值和最小值。同理,對于蛋白質節點v,其歸一化處理后的結構域特異性得分可以表示為SpecNORM()v。
研究表明,蛋白質的關鍵性和蛋白質分子間的相互作用存在密切關系,因此我們可以通過相互作用的兩個蛋白質的結構域特異性計算出這條相互作用的特異性。如對相互作用E(u ,v) ,其結構域特異性得分取決于它對應的兩個蛋白質分子u和v,可以表示為
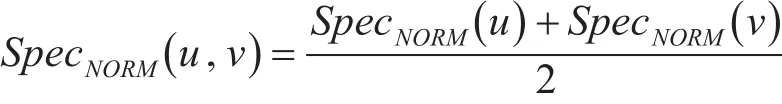
Do-ECC算法就是通過結合兩者來評估蛋白質的關鍵性,如對蛋白質節點u,其關鍵性得分可以表示為
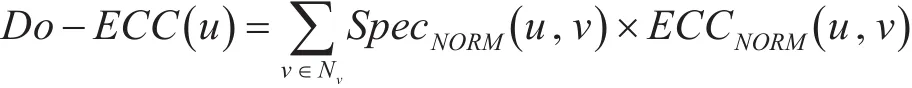
其中Nv是節點u的鄰居節點的集合,v是節點u的鄰居節點且v∈Nv。蛋白質節點的關鍵性得分越高,越可能是關鍵蛋白質。
5 實驗
5.1 實驗數據
1)蛋白質相互作用數據
鑒于酵母的蛋白質相互作用數據的相對完備性,本實驗選擇酵母作為研究對象。所用的蛋白質相互作用數據是從DIP數據庫[14]下載獲得,采用的數據集版本是2017年2月5日更新的釀酒酵母的全部蛋白質相互作用數據集。原始數據集中包含22977條蛋白質相互作用,去除自連接和重復的相互作用后,共提取出22620條相互作用,包含5126個蛋白質分子。
2)蛋白質結構域數據
本實驗所用到的蛋白質結構域數據是從PFAM數據庫[15]中下載獲得的,采用的數據集版本是于2017年3月份更新的Pfam 31.0。因為在PFAM數據庫中,有兩種不同質量水平的結構域序列數據:Pfam-A系列和Pfam-B系列。其中,Pfam-A系列的數據質量水平較高,而Pfam-B系列的數據未經注釋過且質量水平也較低,因此,本實驗僅僅提取酵母的Pfam-A系列的結構域序列數據。在實驗中,我們通過在PFAM數據庫中下載獲取到swisspfam.gz文件,經過預處理后,提取出具有已知的結構域信息的蛋白質共4174個,包含了2829種結構域,而剩余的952個蛋白質則認為沒有已知的結構域信息。
3)已知的關鍵蛋白質和非關鍵蛋白質
通過實驗得到的候選關鍵蛋白質需要和目前已知的關鍵蛋白質數據進行比對,進而分析實驗方法的有效性和準確率。本實驗所選用的已知關鍵蛋白質數據是通過整合數據庫 SGD[16]、DEG[17]和SGDP[18]中的酵母的關鍵蛋白質信息數據得來。最后整合得到的釀酒酵母的關鍵蛋白質1299個,非關鍵蛋白質4982個。將從DIP數據庫中獲取的酵母的5126個蛋白質分子與已知關鍵蛋白質和非關鍵蛋白質數據對比后,我們發現可以將5126個蛋白質分子分為3類:關鍵蛋白質、非關鍵蛋白質和關鍵性未知的蛋白質,其中含有關鍵蛋白質1159個,非關鍵蛋白質3612個,關鍵性未知的蛋白質355個。在實驗過程中,我們將關鍵性未知的蛋白質歸為非關鍵蛋白質一類。
5.2 評價指標
通常來講可以將關鍵蛋白質的識別問題當作非監督的分類問題,然后采用統計學中常用的“排序-篩選”的方法對不同的關鍵蛋白質識別算法的實驗結果進行比較和分析[19]。針對本實驗,“排序-篩選”方法的具體過程如圖2所示。
除此之外,為更加有效地對各個算法的實驗結果進行評估,還可以使用6種常用的測量指標,包括敏感度(Sensitivity,SN)、特異性(Specificity,SP)、F-測度(F-measure)、正確率(Accuracy,ACC)、陽性預測值(Positive Predictive Value,PPV)和陰性預測值(Negative Predictive Value,NPV)。在詳細分析這幾種指標之前,首先需要了解表1中介紹的幾個概念。
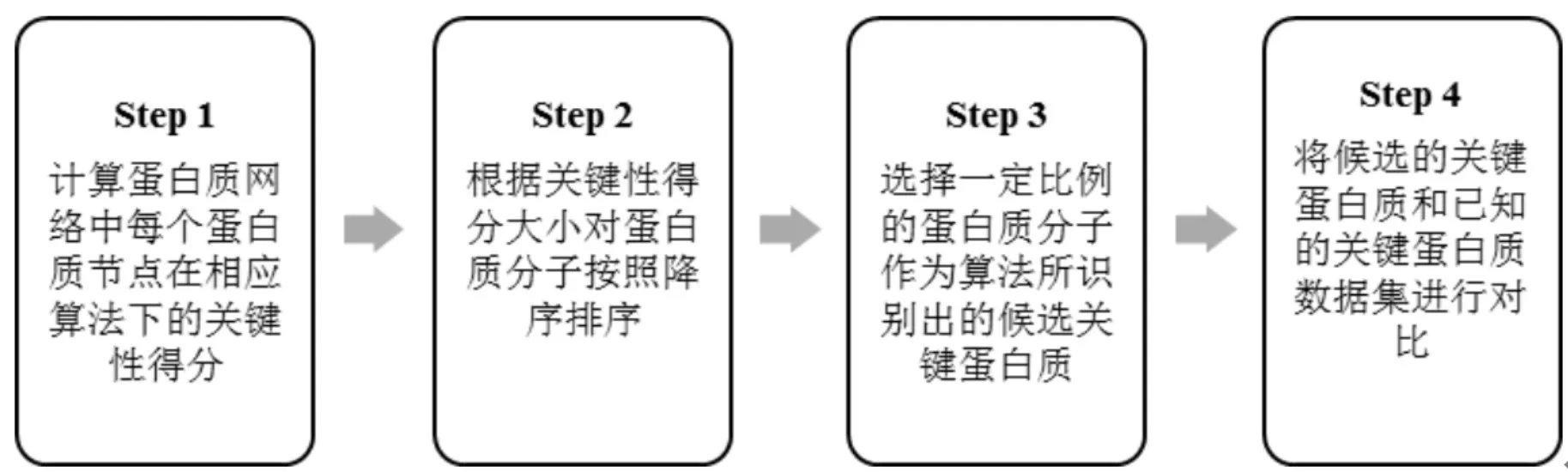
圖2 排序-篩選的流程
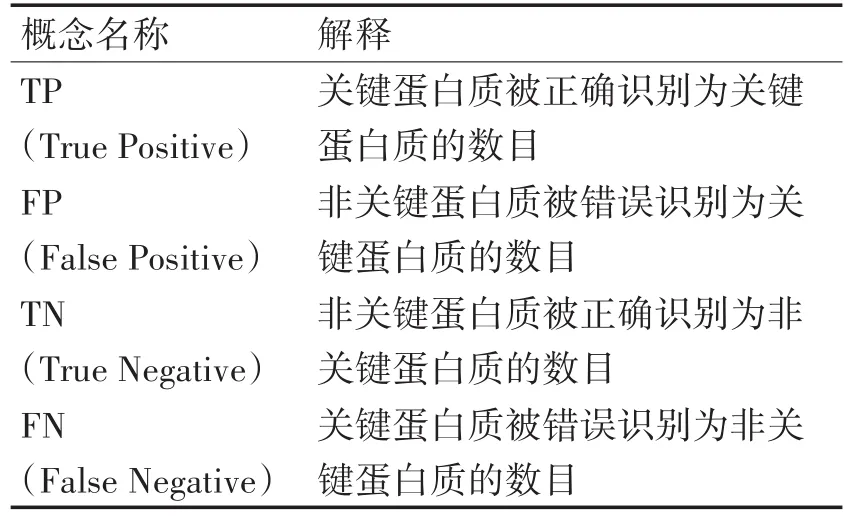
表1 相關概念簡介
基于表1中介紹的四個基本概念,這6種常用的檢測指標定義如下:

5.3 實驗結果與分析
按照“排序-篩選”的方法,我們首先計算出5126個蛋白質節點在上述各個算法下的測度參數并根據測度值按降序排序,然后分別挑選前1%、5%、10%、15%、20%以及25%的部分作為候選的關鍵蛋白質,最后將其和已知的鍵蛋白質數據進行對比,得出各個算法識別出的正確的關鍵蛋白質數目,如表2所示。
由表2展示的實驗結果,可以發現,Do-ECC算法識別出的正確的關鍵蛋白質數目在各個范圍內均顯著多于其他8種算法。
為更加細致地比較各個算法識別關鍵蛋白質的效果,進一步使用SN、SP、F、ACC、PPV和NPV對它們的實驗結果進行評估比較,如表3所示。
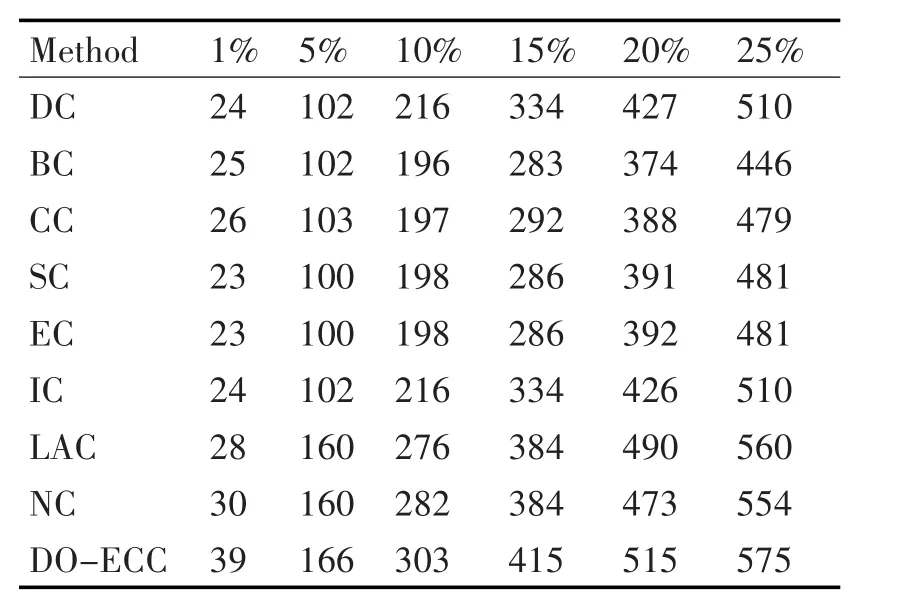
表2 九種算法識別出的正確的關鍵蛋白質數目
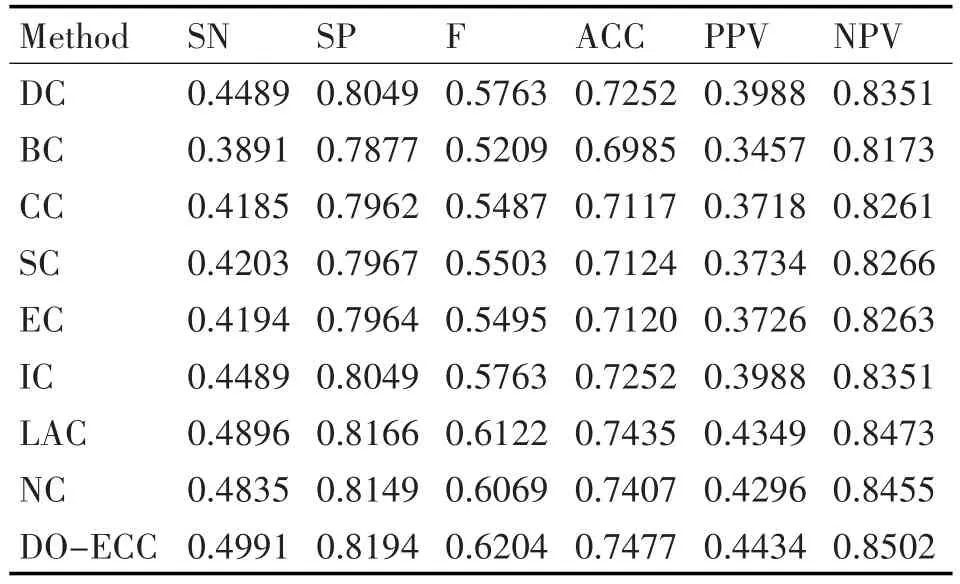
表3 九種算法在6種常用檢驗指標下的實驗結果
由表3不難看出,Do-ECC算法在SN、SP等6種指標下的得分均高于其他8種基于蛋白質網絡拓撲特征的算法。
6 結語
本文使用邊聚集系數刻畫蛋白質網絡的拓撲特征,并借鑒TF-IDF算法的思想,提出蛋白質結構域特異性的概念,然后融合蛋白質網絡的拓撲信息和生物信息,提出一種基于蛋白質結構域特異性的關鍵蛋白質識別算法Do-ECC,最后通過實驗驗證了所提蛋白質結構域特異性和Do-ECC的有效性。

