牛NAD(+)異檸檬酸脫氫酶生物信息學分析
張志國,王 東
(葉爾羌綠洲生態與生物資源研究高校重點實驗室(喀什大學 生命與地理科學學院),新疆 喀什 844000)
異檸檬酸脫氫酶(Isocitrate dehydrogenases, IDH)在細胞內主要催化D-異檸檬酸(D-isocitrate)氧化脫羧成α-酮戊二酸(α-oxoglutarate)和CO2的反應。該酶是三羧酸循環中的關鍵酶,對其活性的調節將直接影響該酶生物學功能的發揮[1]。該酶具有NAD(+)與NADP(+)兩種輔助因子,根據輔助因子的不同,IDH可分為NAD(+)依賴型的IDH(NAD-IDH,EC∶1.1.1.41)和NADP(+)依賴型的IDH(NADP-IDH,EC∶1.1.1.42)[1]。IDHs廣泛分布于原核生物以及真核生物中[2-6]。在哺乳動物組織中存在3種IDHs同工酶,分別為胞質NADP(+)依賴型IDH(IDH1)、線粒體NADP(+)依賴型IDH(IDH2)以及線粒體NAD(+)依賴型IDH(IDH3),其中IDH3被認為在三羧酸循環異檸檬酸脫羧反應中起主要作用,IDH3活性受許多變構調節劑的調節[3]。
目前的研究表明,在各類腫瘤患者中IDH的突變率較高。IDH1與IDH2的突變會抑制宿主免疫應答并且誘導α-酮戊二酸(α-KG)轉化為D型2-羥基戊二酸(D2HG),造成D2HG大量積累進而誘發膠質瘤的發生[4]。IDH3β作為E3泛素連接酶后期促進復合體(APC/C)的新型底物對癌細胞的大量增值具有較為重要的作用[7]。IDH3α在缺氧誘導因子1(HIF-1)介導的癌細胞代謝以及血管生成中發揮作用[24]。該研究還表明,IDH3α的異常表達會促進血管瘤的生長[24]。因此對IDH家族進行深入研究將為癌癥等疾病的治療、分子病理分析和預后提供良好的視角[8, 30]。IDH是目前國際上公認的研究蛋白質結構與功能關系、酶的催化與調節機制以及蛋白質分子進化機制的最好模型之一[9]。目前有關牛IDH的研究報道較少。文獻報道表明,牛心的煙酰胺腺嘌呤二核苷酸依賴性異檸檬酸脫氫酶(NAD-IDH)是由subunit-3/4(α)、subunit1(β)和subunit2(γ)三種亞基構成的異四聚體, 其中各亞基占比分別為2∶1∶1[10]。后來的研究進一步證實哺乳動物中1/β和2/γ亞基是調節性的,而包含一半四聚體的3, 4 /α亞基是催化性的[11-12]。在前人研究的基礎上,本研究采用生物信息學方法分析了牛線粒體NAD-依賴型IDH的理化性質、親疏水性、跨膜區、信號肽、亞細胞定位、二級結構預測、三級結構預測、表面電荷分布以及輔助因子結合位點,從而為進一步研究IDH家族尤其是哺乳動物NAD(+)IDH提供了重要理論依據。
1 材料與方法
1.1 材料
從Uniprot數據庫中查詢并獲得已注冊的牛NAD-IDH的三種亞基IDH3A_BOVIN(P41563)、IDH3B_BOVIN(O77784)以及IDH3G_BOVIN(Q58CP0)的蛋白序列。
1.2 方法
利用生物信息學分析工具(見表1)分析了牛NAD-IDH的理化性質、親疏水性、跨膜區、信號肽、亞細胞定位、二級結構預測、三級結構預測,表面電荷分布以及輔助因子結合位點。

表1 生物信息學分析工具Table 1 Bioinformatic analysis tools
2 結果與分析
2.1 牛NAD(+)IDH理化性質分析
牛NAD(+)IDH三種亞基分析結果表明,該蛋白殘基總數為1 509個,原子總數為23 207個,分子總質量為165 kDa,總分子式為C7259H11670N2048O2144S86。其中占比最高的殘基為Ala(9.7%),占比最低的殘基為Trp(0.6%)。三種亞基中IDH3G_BOVIN和IDH3B_BOVIN兩個亞基呈堿性,IDH3A_BOVIN一個亞基呈酸性且正負電荷殘基僅相差一個,據此可以推斷牛NAD(+)IDH為堿性蛋白質。不穩定系數分析結果表明,三種亞基均不穩定,據此推斷牛NAD(+)IDH蛋白為不穩定蛋白質。親水性分析結果表明,三種亞基均呈親水性,據此推斷牛NAD(+)IDH為親水性蛋白質(見表2)。
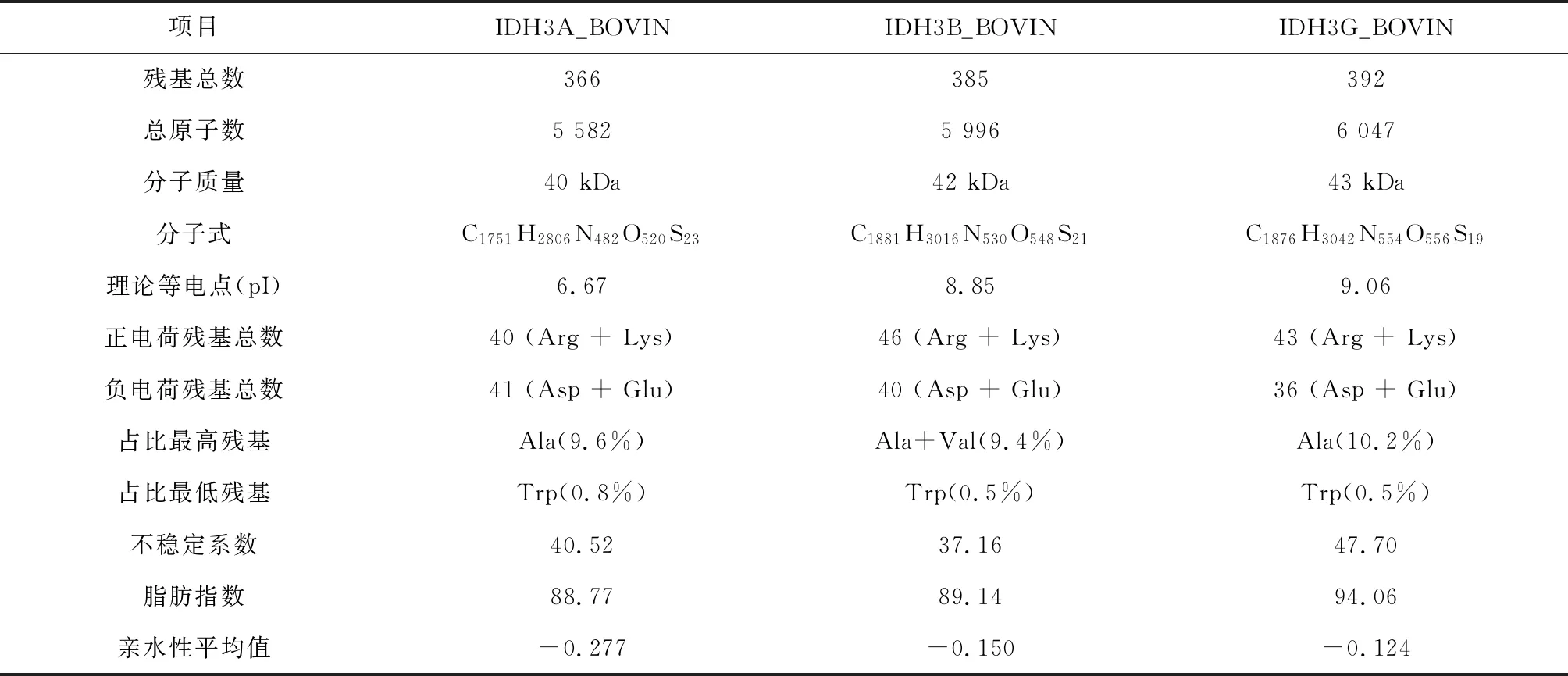
表2 牛NAD(+)IDH理化性質分析Table 2 Analysis of physical and chemical properties of NAD(+)IDH in bovin
2.2 牛NAD(+)IDH親水性/疏水性分析
K-D預測算法分析的牛NAD(+)IDH親水性/疏水性結果表明,IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三個亞基均為親水性,據此推斷牛NAD(+)IDH為親水性蛋白質,這與理化性質分析結果一致。同時分析結果表明,牛NAD(+)IDH各亞基均無明顯疏水區段,表明該蛋白很可能沒有跨膜區,為細胞游離蛋白(見圖1,表3)。
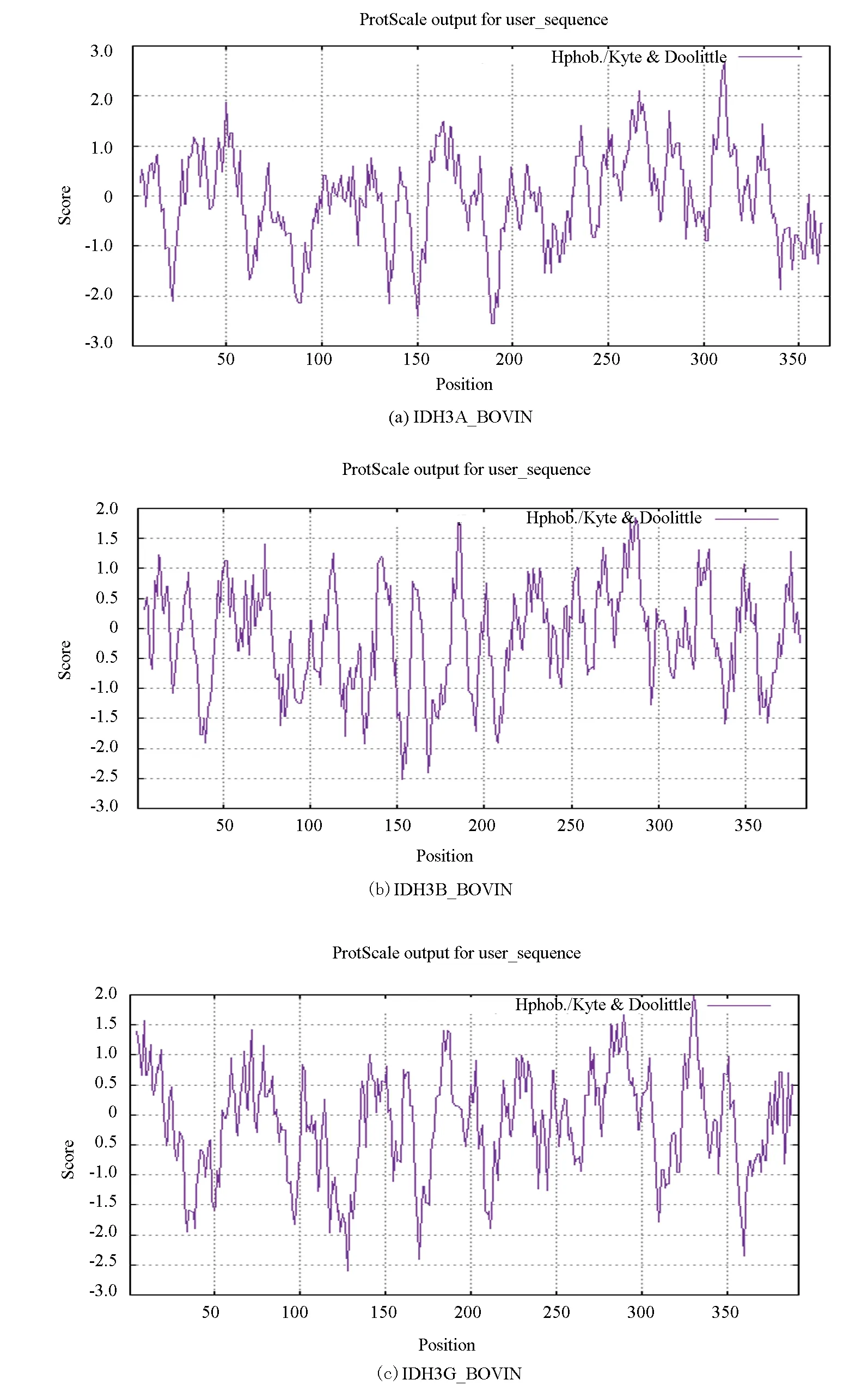
圖1 牛NAD(+)IDH的親水性/疏水性分析 (Kety and Doolittle 算法)Fig.1 Hydrophlicity/hydrophobicity analysis of NAD(+)IDH in bovin (Kety and Doolittle algorithm)
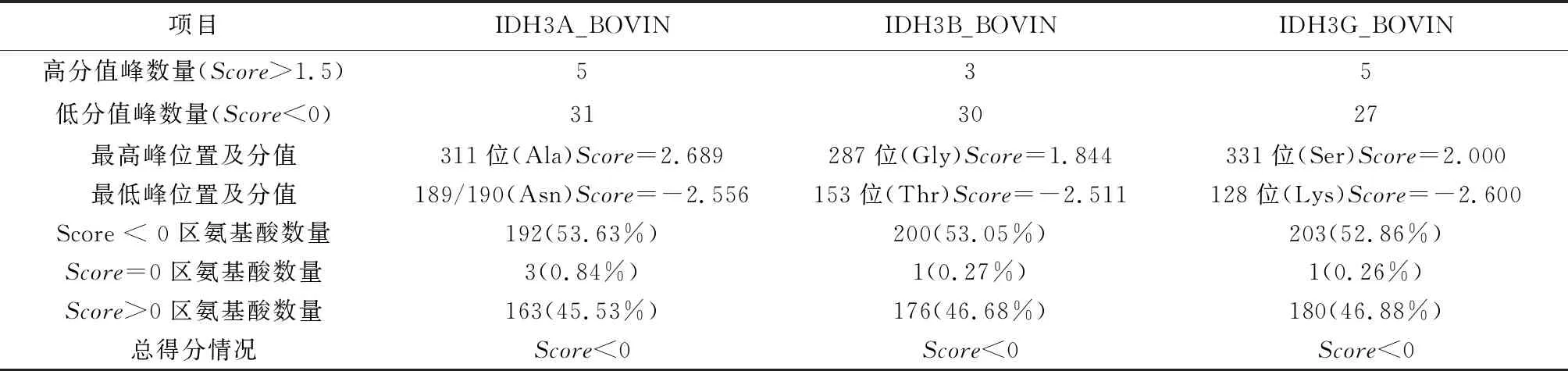
表3 牛NAD(+)IDH的親水性/疏水性分析Table 3 Hydrophlicity/hydrophobicity analysis of NAD(+)IDH in bovin
2.3 牛NAD(+)IDH跨膜區預測
運用隱馬爾可夫模型分別對三個亞基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN進行跨膜區預測分析。結果表明,該蛋白的三個亞基均無跨膜區,據此推斷牛NAD(+)IDH不是跨膜蛋白,在三羧酸循環中以游離狀態發揮作用,這與該蛋白親水性/疏水性分析結果一致(見圖2)。
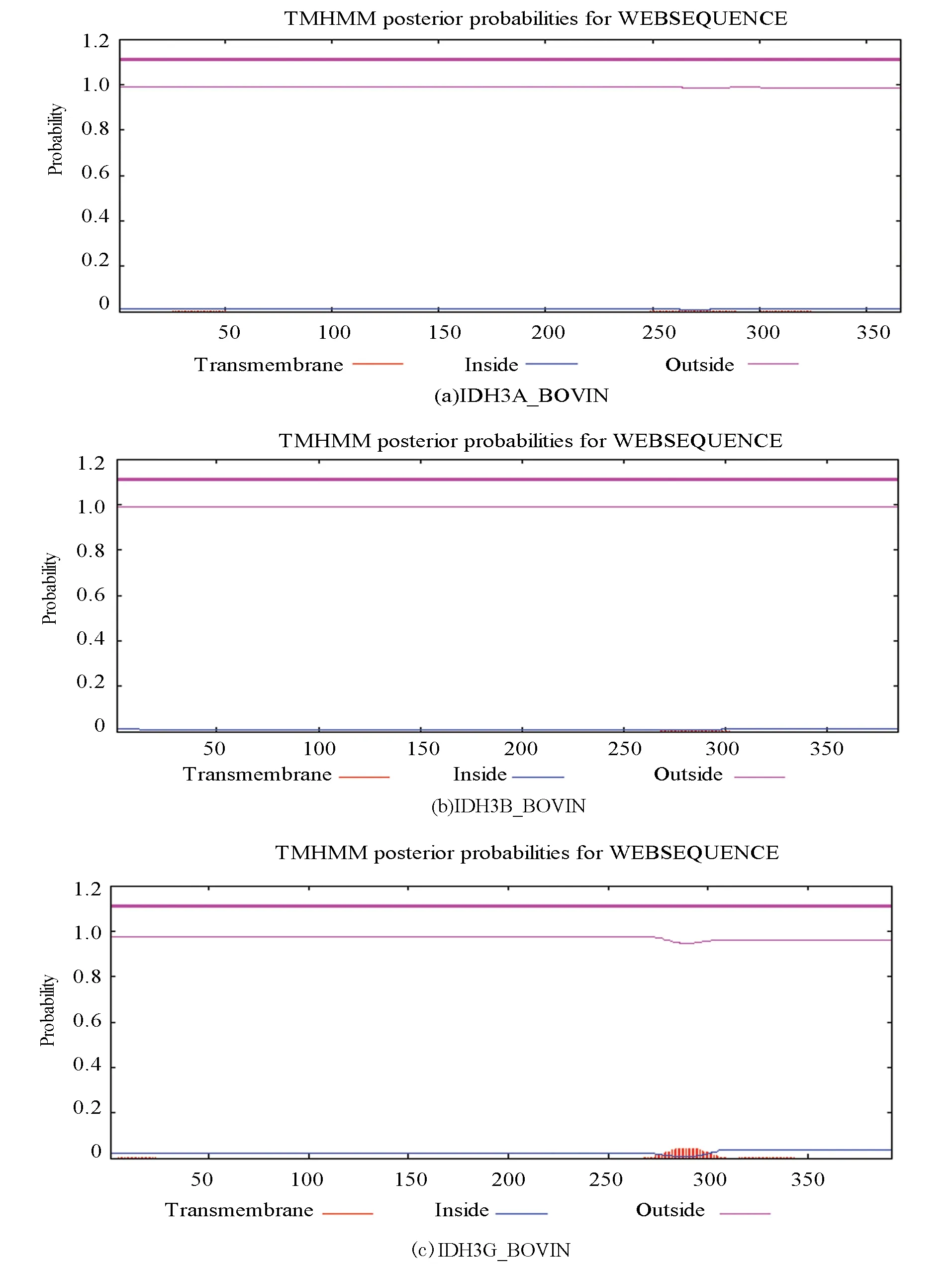
圖2 牛NAD(+)IDH跨膜區預測 (隱馬爾可夫模型)Fig.2 Transmembrane structure prediction of NAD(+)IDH in bovin (Hidden Markov Models)
2.4 牛NAD(+)IDH信號肽預測
基于兩種算法(神經網絡算法和隱馬爾可夫模型)分別對牛NAD(+)IDH三個亞基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN進行信號肽預測分析。神經網絡算法分析結果中C-score表示信號肽酶切位點值;S-score表示信號肽值;Y-score表示綜合得出的剪切位點分值。meanS-score作為判斷蛋白質是否具有信號肽的標準,當meanS-score>0時可以認為是分泌蛋白,具有信號肽,meanS-score<0時則認為是非分泌蛋白,沒有信號肽。結合隱馬爾可夫模型的預測結果,可以推斷出IDH3A_BOVIN和IDH3G_BOVIN兩個亞基沒有信號肽,IDH3B_BOVIN亞基具有信號肽。根據文獻報道,信號肽一般長度為5-30個氨基酸[15],因此排除本研究中隱馬爾可夫模型對IDH3B_BOVIN亞基的預測結果(見圖3,表4)。綜上所述,可以推斷牛NAD(+)IDH存在信號肽,并且該信號肽位于β亞基N端第1至第16位氨基酸,具體序列為MAALSRVRWLTRALVA。
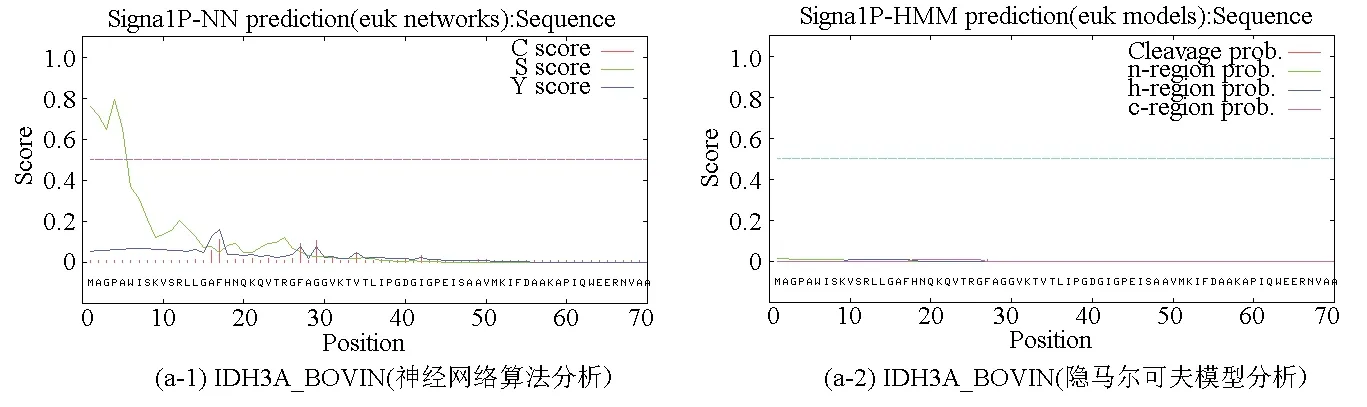
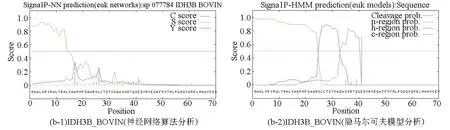
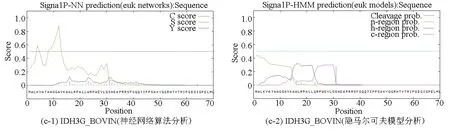
圖3 牛NAD(+)IDH信號肽預測Fig.3 Signal peptide prediction of NAD(+)IDH in bovin

表4 牛NAD(+)IDH信號肽預測Table 4 Signal peptide prediction of NAD(+)IDH in bovin
2.5 牛NAD(+)IDH亞細胞定位預測
基于兩種工具的亞細胞定位預測結果表明,IDH3A_BOVIN、IDH3B_BOVIN與IDH3G_BOVIN三個亞基均定位在線粒體中。與上述信號肽預測結果相吻合,據此推斷牛DNA(+)IDH定位在線粒體中(見表5)。

表5 牛NAD(+)IDH亞細胞定位預測Table 5 Prediction of subcellular localization of NAD(+)IDH in bovin
2.6 牛NAD(+)IDH二級結構預測
蛋白質二級結構預測是聯系一級結構與三級空間結構的橋梁和紐帶[16-17]。本研究分別運用目前廣泛使用的Jpred和SOPMA兩種二級結構在線預測工具對牛NAD(+)IDH的三個亞基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN進行二級結構預測。
SOPMA預測結果表明,IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三個亞基的α螺旋占比分別為49.18%、47.01%和49.23%,β折疊占比分別為10.16%、11.43%和10.71%。Jpred和SOPMA預測結果比對表明,兩種預測工具所預測的結果基本吻合,其中可靠性最強的α螺旋區在表6中以深色底紋顯示,可靠性最強的β折疊區以淺色底紋顯示(見表6,圖4)。
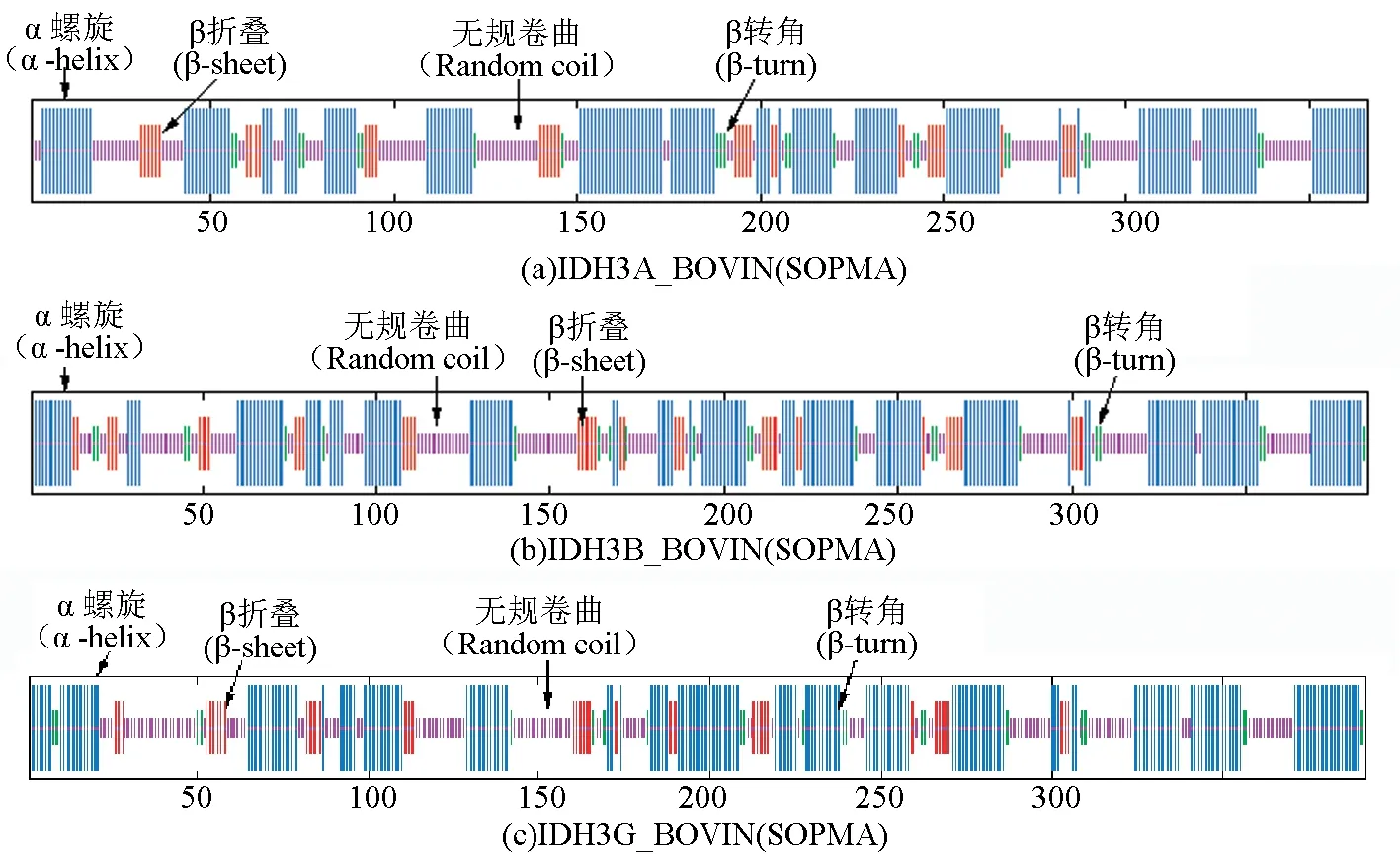
圖4 牛NAD(+)IDH二級結構預測 (SOPMA)Fig.4 Secondary structure prediction of NAD(+)IDH in bovin (SOPMA)
2.7 牛NAD(+)IDH三級結構預測與模型質量評估
空間結構預測對蛋白質結構與功能研究具有較為重要的作用[20]。
2.7.1 同源建模法預測三級結構
采用SWISS-MODEL在線預測工具同源建模法分別預測了牛NAD(+)IDH三個亞基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN的三級結構。其中IDH3B_BOVIN亞基預測結果得到了以PDB ID 2d1c為模板的預測模型,相似度為34.03%,但該預測模型的預測結果表明IDH3B_BOVIN亞基為雙肽鏈蛋白質(見圖5(b)),這一預測結果與文獻報道的經實驗驗證的亞基大小不符合[10],由此推斷同源建模法無法預測IDH3B_BOVIN亞基的三級結構。相比較而言,亞基IDH3A_BOVIN與IDH3G_BOVIN分別得到了以PDB ID 5grh和PDB ID 5grf為模板的高質量預測模型,相似度分別為97.34%和98.31%,全局模型質量評估得分分別為0.90和0.86,均明顯高于其他預測模型。另外,QMEAN得分分別為-0.24和-0.55,比其他預測模型更接近0。進一步分析了預測模型與同源蛋白質三級結構的相似性散點分布圖。結果表明,IDH3A_BOVIN與IDH3G_BOVIN模型(見圖6(a)和(b)中以紅色五角星標記)Z得分均<0,顯示該預測模型結構穩定,是最佳的理想結構(見圖6)。據此可以推斷以PDB ID 5grh為模板的預測結果可以作為IDH3A_BOVIIN亞基的最可能的三級結構(見圖5(a)),以PDB ID 5grf為模板的預測結果可以作為IDH3G_BOVIIN亞基的最可能的三級結構(見圖5(d))。
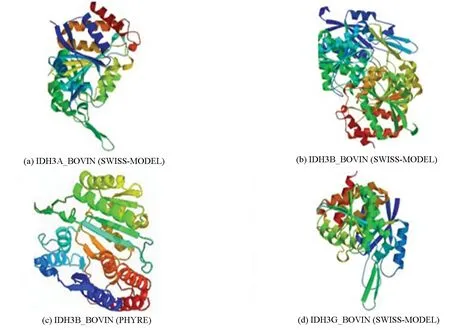
圖5 牛NAD(+)IDH的三級結構Fig.5 Tertiairy structure of NAD(+)IDH predicted model
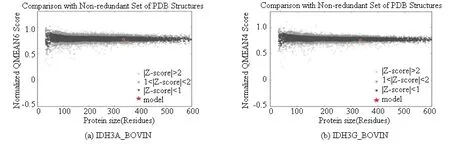
圖6 三級結構相似性散點分布圖Fig.6 Similarity scattergram of the predicted model and the homologous protein
2.7.2 折疊識別法預測三級結構
同源建模法預測結果表明,采用同源建模法無法精確預測IDH3B_BOVIN亞基的三級結構,因此,本研究采用折疊識別法進一步對IDH3B_BOVIN亞基的三級結構進行預測。預測結果顯示有46個模型的準確度達到了90%以上,其中一個模板的折疊子c5grhB_是人異檸檬酸脫氫酶α與γ異二聚體中的γ亞基,該折疊子與IDH3B_BOVIN亞基序列匹配性最好,準確度達到了100%,相似性達到了54%,據此推斷以該折疊子X射線晶體衍射結構折疊識別出來的三級結構可以作為IDH3B_BOVIIN亞基最可能的三級結構(見圖5(c))。
2.7.3 模型質量評估
為了進一步驗證預測模型的可靠性,將預測得到的模型PDB文件分別上傳至SAVES v5.0平臺上的Verify3D和PROCHECK進行在線評估。拉曼圖中從白色到紅色,顏色越深的區域表明Cα的二面角越合理。拉曼圖分析結果表明IDH3A_BOVIN和IDH3G_BOVIN兩個亞基的預測模型中90%以上的殘基都位于紅色最佳允許區域,IDH3B_BOVIN亞基中87.9%的氨基酸殘基(269個)都位于紅色最佳允許區域,10.1%的氨基酸殘基(31個)位于正黃色額外允許區域(見圖7)。表明模型中所有的氨基酸殘基均形成了一種合理的二面角,構成的蛋白質結構穩定。3D-1D得分結果表明,IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三個亞基中3D-1D值大于0.2的殘基均占80%以上,且低質量部分不明顯(見圖8),這表明模型質量合格。綜上所述,可以推斷出上述IDH3A_BOVIN、IDH3B_BOVIN與IDH3G_BOVIN三個亞基的三級結構預測結果穩定可靠。

表6 牛NAD(+)IDH二級結構預測 (Jpred/SOPMA)Table 6 Secondary structure prediction of NAD(+)IDH in bovin (Jpred/SOPMA)

注:AA:氨基酸序列;H:α螺旋;E:β折疊;-:無規卷曲。
2.8 三級結構預測與二級結構預測吻合度分析
將牛NAD(+)IDH三個亞基IDH3A_BOVIN、IDH3B_BOVIN與IDH3G_BOVIN三級結構預測模型進行DSSP二級結構指認,并將指認結果與Jpred和SOPMA兩種二級結構在線預測工具所預測的二級結構信息進行吻合度分析。結果表明,亞基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三級結構模型中指認的二級結構與Jpred和SOPMA所預測二級結構吻合度分別為92%、88%與95%,這表明亞基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三級結構模型中指認的二級結構分布與Jpred和SOPMA所預測的三個亞基二級結構結果是一致的,同時也進一步表明Jpred和SOPMA所預測的二級結構信息是可信的。

圖7 拉曼圖Fig.7 Ramachandran plot of the predicted model
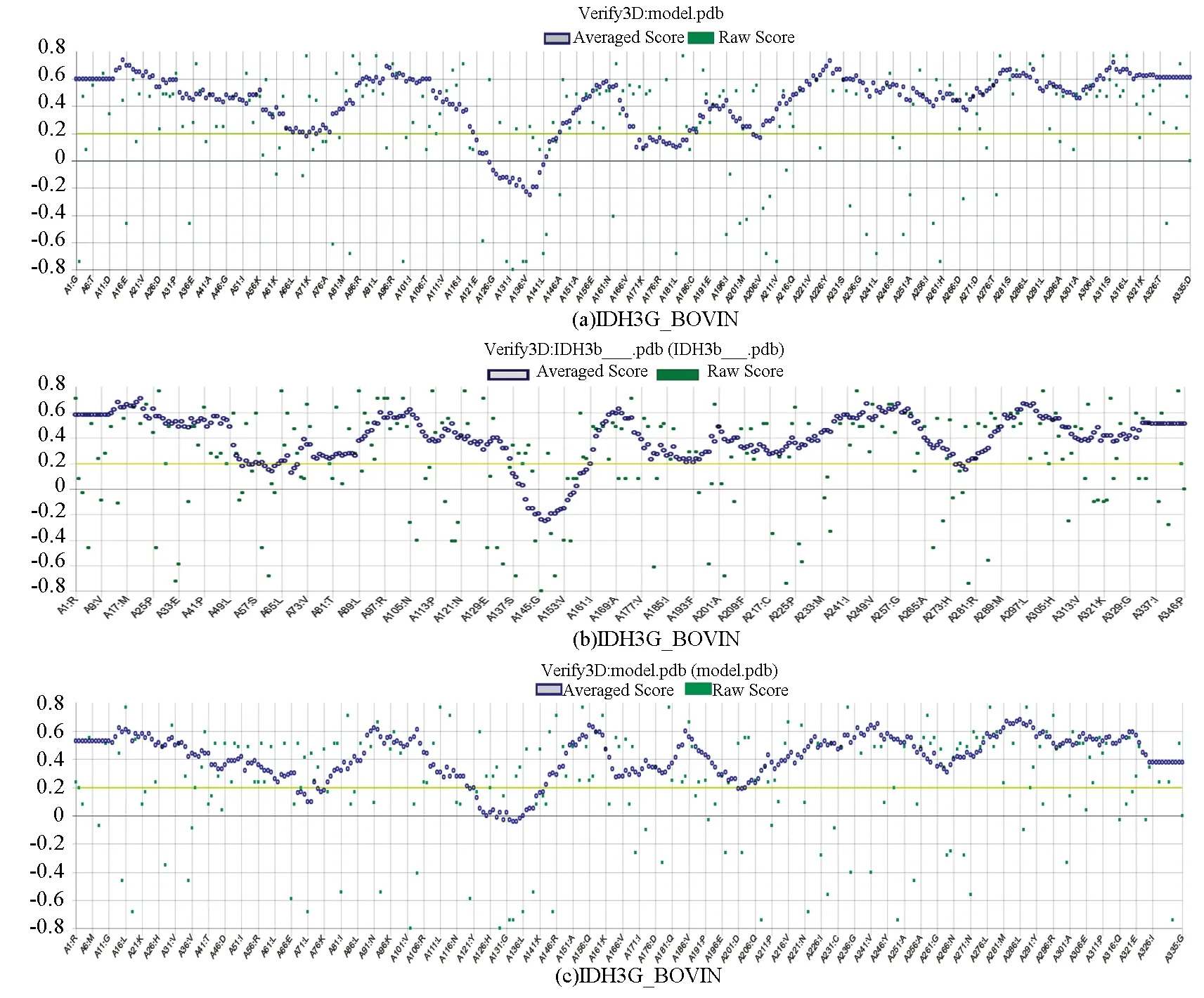
圖8 3D-1D得分散點圖Fig.8 3D-1D score scattergram of the model
2.9 牛NAD(+)IDH蛋白質結構表面電荷分布及輔酶結合位點分析
研究表明,高等動物中NAD(+)IDH不能單獨發揮作用,需要各類輔助因子協同作用促進酶與異檸檬酸的結合并形成α-酮戊二酸,亞基IDH3B_BOVIN和IDH3G_BOVIN具有ADP結合位點,兩者很可能與NAD(+)IDH蛋白活性調節有關。此外,IDH3A_BOVIN亞基為催化亞基,擁有一部分異檸檬酸結合位點[9,12,23]。將獲得的IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三個亞基三級結構數據利用VMD軟件進行分析,通過該軟件中的APBS插件分別計算三個亞基的表面電荷分布。在分析的過程中,用藍色表示所分析蛋白質帶正電荷的區域,紅色表示帶負電荷的區域,灰色表示不帶電荷的區域(見圖9)。分析結果表明,三個亞基三級結構正面均帶有大量正電荷(見圖9(a),(c)和(e)),在β折疊和α螺旋形成的凹槽處尤為明顯,圖中以箭頭所示,推斷該處結構最有可能與異檸檬酸(帶負電)或ADP(帶負電)結合。與正面相比,三個亞基三級結構背面電荷分布均不明顯,且正負電荷分布散亂,推斷這些區域不太可能與異檸檬酸或ADP結合(見圖9(b),(d)和(f))。
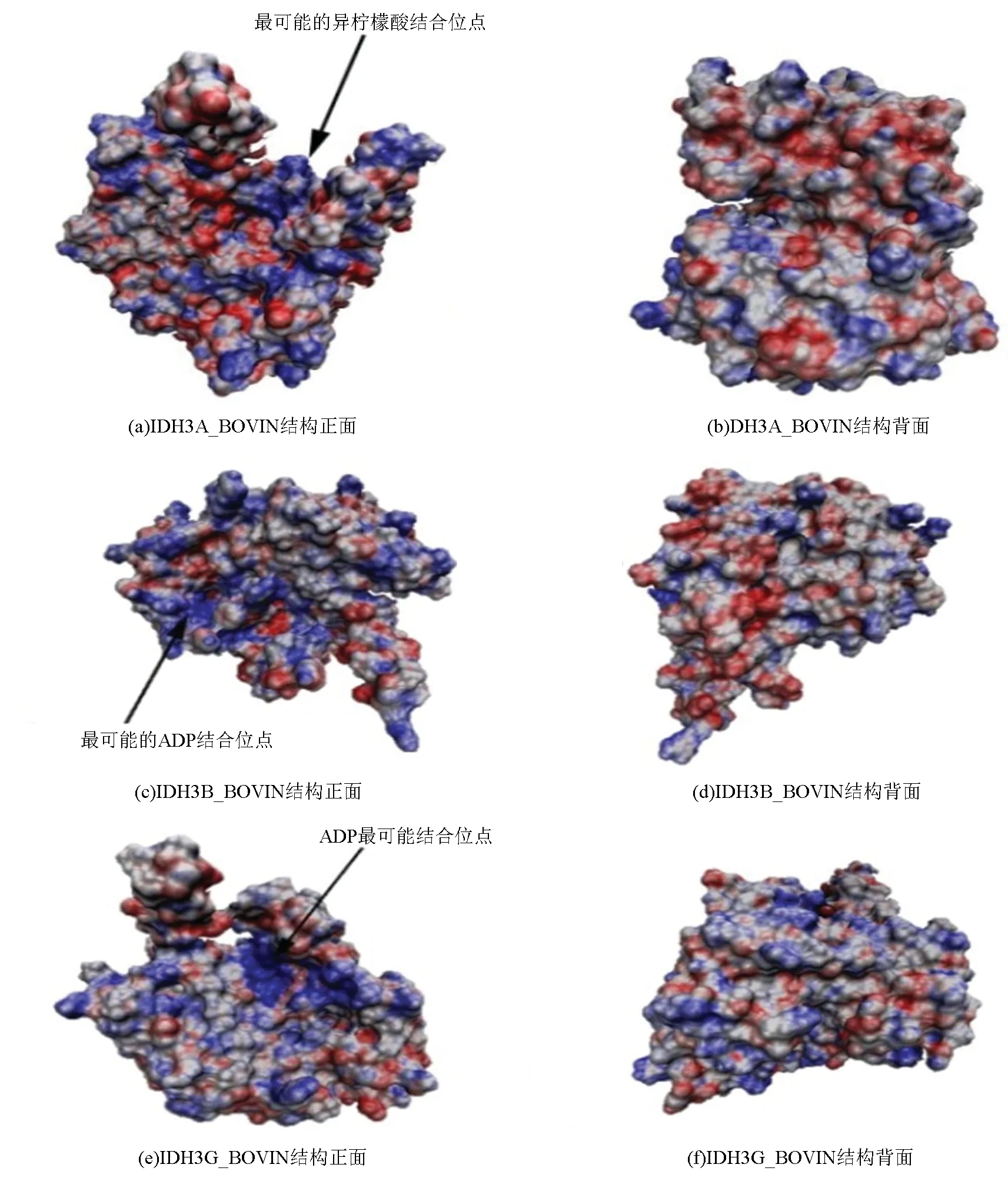
圖9 牛NAD(+)IDH表面電荷分布圖Fig.9 Surface charge distribution of NAD(+)IDH in bovin
3 討 論
異檸檬酸脫氫酶(IDH)催化生物體內的氧化脫羧反應,具有重要的生理作用。該酶受到多種輔助因子的調節,在三羧酸循環反應中發揮著重要作用。自2009年首次在彌漫性膠質瘤中發現IDH突變以來,IDH突變在診斷、預后和膠質瘤預測方面發揮越來越重要的作用[25]。2016年,世界衛生組織(WHO)首次將分子參數用于腦腫瘤診斷,將IDHl與IDH2突變分子病理檢測作為膠質瘤新的分型診斷標準[27]。本研究利用多個生物信息學工具從多個方面分析了牛NAD(+)IDH的理化性質及空間結構。研究發現牛NAD(+)IDH無跨膜結構,具有信號肽且位于β亞基N端第1-16位的氨基酸上,屬于親水性蛋白質,整體呈堿性,亞細胞定位在線粒體中。異檸檬酸脫氫酶是三羧酸循環反應中的關鍵酶之一,在三羧酸循環中發揮著重要作用。三羧酸循環在線粒體中進行[28],據此推斷牛NAD(+)IDH的肽鏈在核糖體上合成之后經進一步折疊組裝,α、β和γ亞基以2∶1∶1的比例相結合,并在β亞基N端1-16位氨基酸殘基信號肽的介導下進入線粒體參與三羧酸循環反應。這一推斷與Chen等(1990)對哺乳動物NAD(+)IDH的研究結果相吻合[13]。
二級結構預測顯示IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三個亞基的二級結構主要為α螺旋,并且α螺旋與β折疊排列緊湊有序,可以形成一定的結構域。此外還發現β亞基與γ亞基的二級結構中α螺旋和β折疊的位置相似,這種相似性與功能的關系有待進一步研究。三級結構預測與二級結構預測吻合度分析結果表明亞基IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三級結構模型中指認的二級結構分布與Jpred和SOPMA所預測的三個亞基二級結構結果相一致,同時也表明Jpred和SOPMA所預測的二級結構信息是可信的。文獻報道表明,Rushbrook等人在牛心線粒體中分離出NAD(+)IDH[10],在本研究中牛NAD(+)IDH亞細胞定位在線粒體中,本研究有關牛NAD(+)IDH亞細胞定位的結果與Rushbrook等人的實驗研究結果相一致。除此之外,本研究中有關牛NAD(+)IDH的二級結構預測、三級結構預測、親/疏水性、信號肽預測以及跨膜區預測等目前尚未發現實驗研究報道,對于本研究中有關牛NAD(+)IDH蛋白的預測結果有待進一步的實驗研究。
哺乳動物中存在的NAD(+)IDH蛋白的各個亞基在進化過程中表現出較高的同源性,然而有關NAD(+)IDH蛋白各個亞基殘基的改變對亞基構象有怎樣的影響以及這種影響的生物學意義目前尚無文獻報道。本研究有關牛NAD(+)IDH蛋白IDH3A_BOVIN、IDH3B_BOVIN和IDH3G_BOVIN三個亞基的生物信息學分析將有助于從生化和分子角度闡明哺乳動物尤其是人的IDH如何影響腫瘤的形成。
此外,有助于闡明DH3β與APC/C的分子對接機制、IDH3α的活性結構域的靶向調節以及腫瘤生長機制等[7, 8, 24, 30]。

