一種假單胞菌Pseudomonas sp.導致的側耳屬細菌病害*
柴紅梅,張小雷,趙永昌,蘇開美,陳衛民
(云南省農業科學院生物技術與種質資源研究所,云南 昆明 650205)
假單胞菌屬細菌是主要的食用菌病菌,能引起平菇 (Pleurotus ostreatus)、雙孢菇 (Agaricus bisporus)、香菇 (Lentinula edodes)、金針菇 (Flammulina velutipes)等的褐斑病[1-4],其繁殖速度快、傳播范圍較廣,容易給食用菌栽培場造成極大的損失。因此,及時鑒別危害病菌的種類和特征、較早預防,可以有效減輕病菌危害。
在食用菌栽培生產中,目前已知的最為普遍的細菌性病原菌為托拉斯假單胞桿菌(Pseudomonas tolaasiiPaine),它能通過產生一種脂多糖類的細胞外毒素tolaasins,有效地破壞食用菌的細胞膜結構,致使細胞破裂[5-6],受感染的子實體表面發粘腐爛并發生褐變。托拉斯假單胞桿菌能侵染多種栽培食用菌,其傳播介體為空氣、土壤、帶病子實體、昆蟲等,能夠在較短時間內感染子實體、產生病斑并快速蔓延。除了托拉斯假單胞桿菌,熒光假單胞菌(Pseudomonas fluorescens)[7]、銅綠假單胞菌 (Pseudomonas aeruginosa)[8]、洋蔥假單胞菌 (Pseudomonas cepaica)[9]、施氏假單胞菌 (Pseudomonas stutzeri)[10]及綠針假單胞菌(Pseudomonas chlororaphis)[11]等也都會對不同的食用菌造成比較大的危害。從平菇和雙孢菇中分離到了托拉斯假單胞菌、熒光假單胞菌和Pseudomonas gingeri[12-13]。另外,在河南的杏鮑菇子實體中發現細菌性病害,表現為子實體畸形,菌肉呈水漬狀,并逐步腐爛,但沒有鑒定出該病原菌的種類[14]。
盡管已有多種方法被用來進行假單胞病原菌的殺滅,包括噴灑二氧化氯、次氯酸鈣及鏈霉素等,但病變發生后很難徹底控制病原菌,因此提前檢測菇房環境并進行阻斷,更有利于后期的生產開展。統計病原菌種類,掌握其生物學特征,特別是DNA序列,可以有效的進行類別鑒定。本研究從云南陸良平菇及杏鮑菇種植場分離獲得了2株假單胞桿菌菌株,分離純化后,擴增其16S rDNA片段,進行序列分析和分類地位分析,為假單胞病原菌在分子水平的研究提供了數據支持。
1 材料與方法
1.1 試驗材料
從云南陸良種植場采集感染病菌的平菇(P.ostreatus)和杏鮑菇(P.eryngii)子實體,分別挑取一個染病部位的菌斑置于無菌水中,吸取菌液涂布到酵母膏胨葡萄糖培養基(蛋白胨0.2%、酵母粉0.2%、葡萄糖2%、瓊脂粉1.5%,pH自然) 固體平板,培養獲得單菌落,保存備用。
1.2 PCR擴增及序列測定
用擴增細菌16S rDNA的通用引物對16SF(5’-AACTGAAGAGTTTGATCCTGGCTC-3’) 和 16SR(5’-TACG GTTACCTTGTTACGACTT-3’) 擴增菌株的16S rDNA片段。平菇和杏鮑菇樣品所分離的2株病原菌落中,都分別隨機挑選3個菌落,進行菌落PCR。反應體系為:2×Power Taq PCR Master Mix 25 μL(天根生化科技有限公司),10 μmol·L-1引物各2 μL,DNA模板1 μL,無菌去離子水補足50 μL。擴增在BIO-RAD公司的C1000TM Thermal Cycler PCR擴增儀上進行,程序如下:95℃預變性10 min;95℃變性 30 s,50℃退火 30 s,72℃延伸 1 min 30 s,35個循環;72℃延伸10 min。PCR產物用1.5%的瓊脂糖凝膠電泳檢測,對符合預測大小的片段膠回收后進行測序。本試驗所用引物的合成及PCR產物序列測定均在昆明碩擎生物技術有限公司完成。
1.3 序列特征及聚類分析
采用DNAStar軟件包SeqMan程序對所得16S rDNA序列進行拼接,并提交GenBank進行Blast搜索,從而獲得近緣種的相似序列。采用ClustalX程序比對拼接序列,去除兩端長短不一和模糊的部分,使序列長度基本一致。采用Mega 4軟件中的鄰接法(Neighbor JoiningAnalysis,NJ)構建系統發育樹。在分析過程中,將Alignment序列中出現的空缺視為信息丟失。采用自舉法進行分支支持率分析,設1000次重復。
2 結果與分析
2.1 細菌侵染子實體特征
在云南陸良種植場出現了大面積的平菇和杏鮑菇病害,從圖1可以看出,平菇顯現出褐腐病特征,受感染子實體表面發粘,出現淡乳白色菌落,然后子實體發軟萎縮,并且發生褐變;杏鮑菇子實體發病時表面先出現水漬狀的斑點,從里到外腐爛并滲出淡褐色液體,之后整個子實體完全消解。另外,該病原感染初期還能造成原基的發育畸形。

圖1 病原菌株Pseudomonas sp.造成的平菇(A) 和杏鮑菇(B) 病害Fig.1 Diseases of Pleurotus ostreatus(A)and Pleurotus eryngii(B)caused by Pseudomonas sp.
2.2 病原菌的分離及分子鑒定
所采集的病原菌經稀釋、平板涂布后挑取單菌落。從平菇和杏鮑菇所獲病原菌菌落中,各隨機選擇3個菌落進行后續的PCR擴增。

圖2 病原菌株Pseudomonas sp.的16S rDNA核酸序列Fig.2 16S rDNA sequences of Pseudomonas sp.
使用特異性引物16S F/R,每個菌株擴增獲得1個特異性片段,膠回收后進行測序。片段含有800多個堿基,去除兩端模糊序列后,序列長度為734 bp(圖2)。Clustal X比對發現,從平菇及杏鮑菇分離到的病原菌菌株的16S rDNA序列完全一致,表明它們為同一個種。序列提交GeneBank blastn分析顯示,與假單胞屬各近似物種的相似度從99.26%到99.56%。根據16S rDNA分析,可確定所獲病原菌為假單胞菌屬,但不能確定具體種。
2.3 病原菌株的系統分析
所分離病原菌Pseudomonassp.與同屬近緣種的16S rDNA序列作聚類分析(圖3),結果顯示其與P.gessardii、P.fluorescens、P.reactans、P.azotoformans等物種能更好的聚在一起,而與常見的P.tolaasii親緣關系較遠。
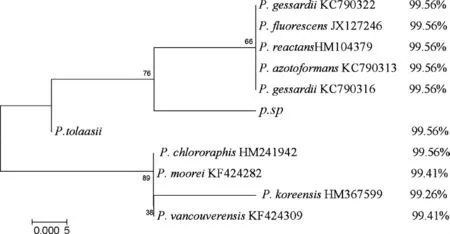
圖3 Pseudomonas 16S rDNA系統聚類圖Fig.3 Phylogeny of Pseudomonas based on 16S rDNA
3 結論
假單胞菌屬多種菌為食用菌的主要細菌性病原菌,這些病原菌具有傳播范圍廣、發病速度快等特點,給栽培場帶來極大的危害。快速鑒定病原菌種類并提前預防可以有效減輕危害帶來的損失,而利用核酸序列進行分析是一個快捷鑒定病原菌的途徑。16S rDNA核酸片段測定已經被廣泛用于細菌物種鑒定,因此本研究使用該方法來對病原菌株進行鑒定。此外,由于細菌通過形態難以進行區分,還需要后續的生理生化試驗進行深入研究。
本研究中從平菇和杏鮑菇感染子實體中分離了假單胞病原菌株Pseudomonassp.,其16S rDNA序列與其他假單胞菌屬真菌相似性最高為99.56%,而聚類結果表明,它與P.gessardii、P.fluorescens、P.reactans、P.azotoformans等物種的親緣性大于P.tolaasii。目前并不能確定這個菌株為假單胞屬的哪一個種,其具體的分類地位仍需后續的生理生化試驗進行證實。
根據序列分析結果,本研究中所獲得的菌株異于常見的托拉斯假單胞菌,而且該菌能侵染平菇及杏鮑菇,說明其寄主具有廣泛性。以前的研究極少有杏鮑菇病原菌相關報道,在此次的研究中,發現并鑒定了假單胞菌相關種侵染杏鮑菇并造成病害的相關特征,為杏鮑菇的種植及病害防治提供了有用的信息。至于其它地區關于杏鮑菇病害特征的描述,由于缺乏核酸信息等方面的證據,不能判斷是否為假單胞菌屬相關物種所造成[14],需要進一步予以證實。這是從杏鮑菇分離到假單胞菌為數不多的相關研究之一,給相關病原菌的鑒定及防治提供了有效支撐,同時也為食用菌細菌性病害研究提供了重要的信息。
此外,根據該病原菌與同屬其它種的16S rDNA序列相似度比對來看,假單胞菌作為食用菌的主要病害,其各個種極為相似,因而需要結合生理生化特征對該屬物種進行準確鑒定,并明確各個種的寄主和造成病害的特征,以便為病害的鑒定及防治提供指導,減輕病原菌給食用菌種植帶來的危害。

