植物OSCA1家族的起源與進化
柴利芳
摘要?OSCA(reduced hyperosmolality-induced [Ca2+]i increase channel)是高滲性門控非選擇性的鈣滲透陽離子通道,它在感受外源和內源的滲透變化以及調節植物生長發育中起著關鍵作用。目前關于OSCA1家族的研究大多集中在功能方面,而關于系統發育分析的研究較少,尤其是在植物界沒有采集大量的樣本并進行深入研究。通過對植物界目前已完成全基因組測序的82個物種OSCA1家族的系統發育分析來探究該家族的起源與進化。研究表明,OSCA1在被子植物中可以分為四大分支(OSCA1.2、OSCA1.5、OSCA17和OSCA1.8),且該家族在整個高等植物中為一個單系;在OSCA1家族的8個成員中均發生了基因重復事件;OSCA1家族在低等到高等植物中的數量分布大致呈現遞增趨勢。以上結果不僅可以完善植物界中OSCA1家族的進化歷史,而且能夠為進一步研究其功能提供充足的理論依據。
關鍵詞?OSCA;通道蛋白;基因重復;系統發育
中圖分類號?Q943文獻標識碼?A
文章編號?0517-6611(2019)20-0001-06
doi:10.3969/j.issn.0517-6611.2019.20.001
開放科學(資源服務)標識碼(OSID):
Origin and Evolution of OSCA1 Family in Plants
CHAI Li?fang
(College of Life Science of Shanxi Normal University,Linfen,Shanxi 041000)
Abstract?OSCA (reduced hyperosmolality?induced [Ca2+]i increase channel) are hyperosmolarity?gated non?selective cation channels that permeates calcium ions. OSCA family plays a key role in sensing exogenous and endogenous osmotic changes and regulating plant growth and development. Most of the current researches on the OSCA1 family focused on functions, but few on phylogenetic analysis.There were no further studies using a large number of samples in plants.In this study, we investigated the origin and evolution of OSCA1 family in 82 species in plants that had completed genome sequencing. The results were as follows:OSCA1s in angiosperms could be divided into four branches (OSCA1.2, OSCA1.5, OSCA1.7 and OSCA1.8), and that OSCA1s was monophyletic in the whole higher plants. There were gene duplication events occurred in all 8 members of OSCA1 family.The number of OSCA1 family in lower to higher plants showed an increasing trend in general.The above results could be not only helpful for us to know the phylogeny of OSCA1 family in plants, but also helpful for further study of its functions.
Key words?OSCA;Channel protein;Gene duplication;Phylogeny
滲透變化的應答對植物生長、發育和繁殖至關重要[1]。而OSCA1是一種Ca2+滲透性陽離子通道蛋白,在高滲應激誘導中起重要作用[2]。真核細胞通過Ca2+傳導通道能夠產生胞質Ca2+信號。Ca2+是動植物適應反應和發育過程中的一種多功能信使。鈣(Ca2+)濃度的空間和時間變化實現了所有真核生物重要的信號調節功能[3-4]。植物和動物在Ca2+通道和Ca2+結合蛋白的互補性上表現出顯著差異。
OSCA1.1是由美國杜克大學裴真明教授研究小組在研究擬南芥(Arabidopsis thaliana)突變體的過程中發現的,他們發現質膜蛋白OSCA1.1是一種介導Ca2+增加的離子通道。OSCA1.1形成非選擇性陽離子通道,其滲透質膜上的Ca2+被高滲激活[5]。在滲透壓脅迫下,OSCA1.1對保衛細胞和根細胞的滲透性造成影響,從而影響Ca2+信號傳導,減少水分蒸騰和調節根系生長。研究發現,擬南芥OSCA1家族共由8個成員組成,分別為OSCA1.1、OSCA1.2、OSCA1.3、OSCA1.4、OSCA1.5、OSCA1.6、OSCA1.7、OSCA1.8[6]。
2014年,Hou等[7]在擬南芥鑒定過程中獲得了膜整合蛋白(CSC?At4g22120,也被稱為OSCA1.2),它是調控Ca2+滲透性的陽離子通道,并與OSCA1.1保持高度的同源性。該蛋白可以在高滲脅迫下增加細胞內Ca2+濃度,對K+和Na+具有一定的滲透性。
OSCA1是一種非常重要的蛋白,它編碼植
物細胞膜中的蛋白質,感知水的可用性變化。基于該蛋白家族,筆者對物種間的系統發育關系進行更進一步的研究,構建系統進化樹,并分析OSCA1家族在植物界中的分布與進化,并增加從水生植物到陸生植物、從關鍵低等植物到高等植物的采樣,以期為后續OSCA1家族的功能驗證提供參考和依據。
1?材料與方法
1.1?序列搜集與比對
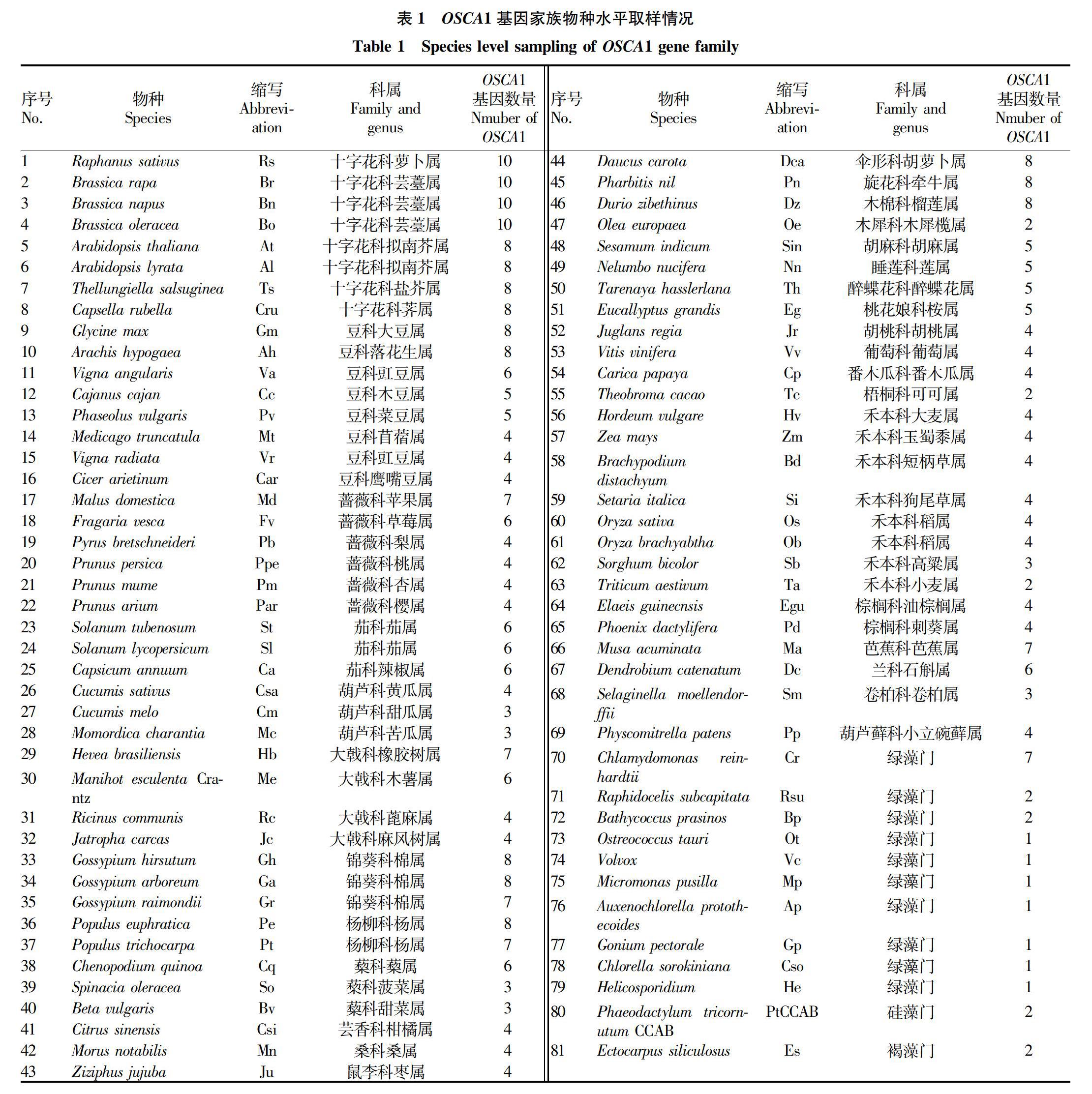
通過一系列的進化分析探索了植物中OSCA1家族的進化歷史。首先,通過雙向BLAST的方法在GeneBank(http://www.ncbi.nlm.nih.gov)數據庫中搜索OSCA1蛋白。為了計算OSCA1在植物中不同進化階段的拷貝數目及變化情況,根據已完成的基因組測序物種的分布情況,選擇其中的82個物種,它們涵蓋了藻類植物、苔蘚植物、蕨類植物、被子植物及其大部分內部支系的四大分支(表1),剔除過短的序列和歧義位點過多的序列,經過整理和分析,保留了高質量的且具有類群代表性和拷貝數代表性的序列。
在進行多序列比對時,先使用CLUSTALW生成原始矩陣,再用Bioedit對最初的比對結果進行手工調整。在獲得相對可靠的矩陣后,再產生一棵樹,并重復上面的操作,進行反復調整,直至序列比對比較可靠、合理。
1.2?系統發育分析
考慮到數據量較大,在系統發育分析中,主要使用MEGA7.0軟件,用鄰接法(neighbor?joining,NJ)和最大簡約法(maximum parsimony,MP)構建系統發育樹。其中NJ樹3個參數的設置:①test of phylogeny(建樹檢驗方法),是用來檢驗建樹質量的。默認的檢驗方法是步長檢驗(bootstrp method),步長測試需要設定檢查次數,通常為100的倍數,該研究的默認設置為1 000。②substitution model,它是選擇遺傳距離時使用的計算模型。理論上應嘗試各種模型,并根據測試結果選擇最合適的模型計算。實際上,可嘗試選擇更簡單的距離模型,比如p-distance,該研究中,氨基酸矩陣使用p-distance模型。③缺口/缺失數據處理,大多數樹構建方法需要刪除多序列比對中具有多個空位的列。但是根據遺傳距離測量的不同方法,刪除原則也不同。如果遺傳距離是通過序列之間不同殘基的數量來衡量的,則需要選擇完全缺失。該研究中,選擇部分刪除(partial deletion),刪除程度設定為50%。MP樹最早源于形態性狀方面的研究,現在已擴展到分子序列的進化分析中。最大簡約法對所有可能的拓撲結構進行計算,能夠計算出所需替代數最小的拓撲結構,并將其作為最優樹,適用于親緣關系較近的序列。該研究使用TBR模型,支持率以自展(bootstrap)法獲得,重復取樣1 000次。BS值只顯示50%以上的支持率。
2?結果與分析
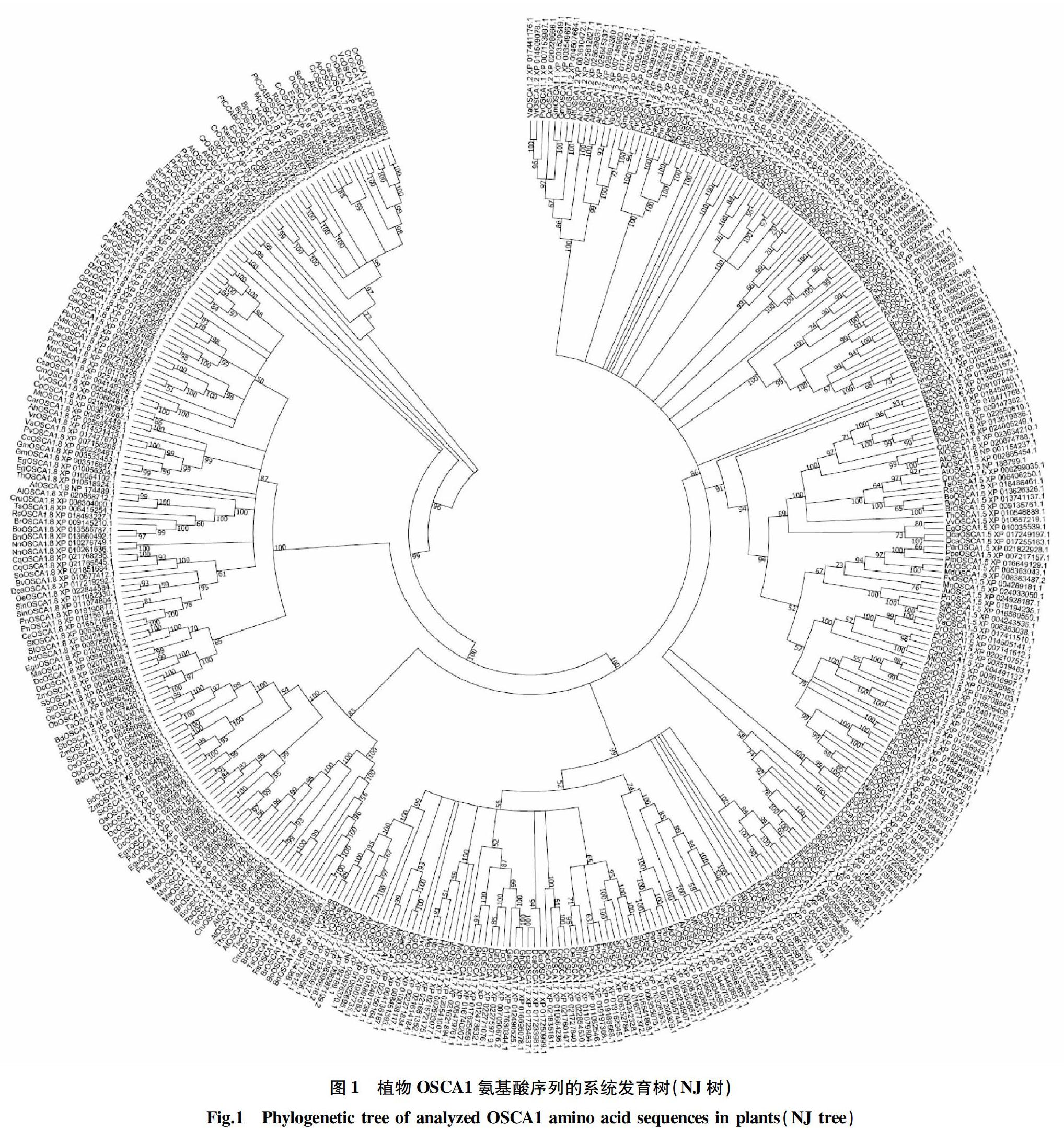
2.1?OSCA1的系統發育分析
用NJ法對植物OSCA1進行系統發育分析,結果如圖1所示。因親源關系較遠,其中基部類群的綠藻門單獨聚為一支,且與高等植物無支持率,在其物種內部發生了一次基因重復事件。通過對在綠藻門中分布的OSCA1的分析,可以看出,OSCA1是一類非常重要的蛋白,它會發生多次重復事件,并且將保留每次迭代產生的新基因。苔蘚植物小立碗蘚(Physcomitrella patens)和蕨類植物江南卷柏(Selaginella moellendorffii)也都分別發生了基因重復事件。基因樹與物種樹大致相符。
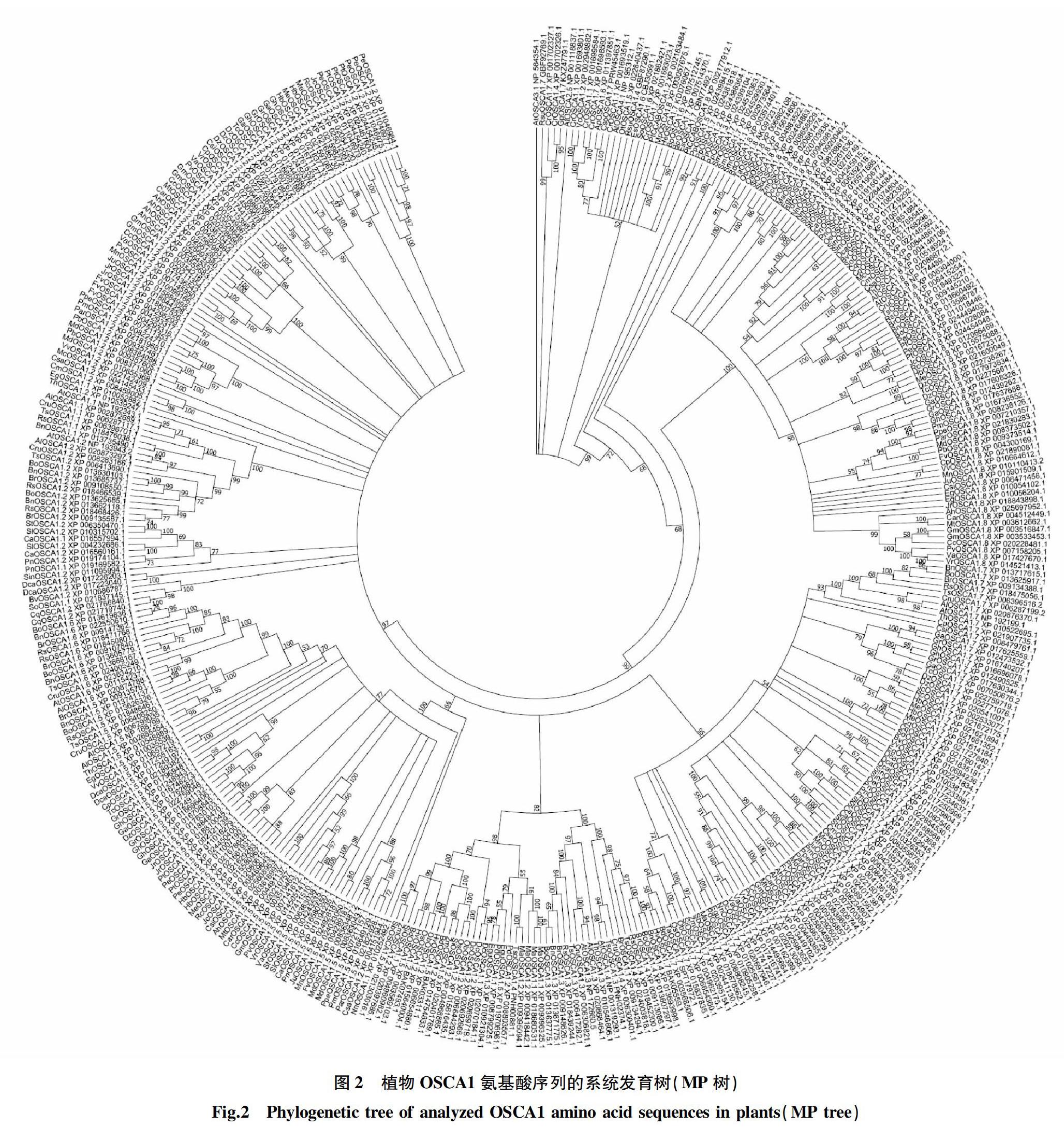
用MP法對植物OSCA1進行系統發育分析,結果如圖2所示。可以看出,OSCA1在被子植物中廣泛分布,分別有基因重復事件的發生,且在整個植物界只有一個大分支,這與NJ樹的結果基本保持一致。
為更明顯直觀地分析植物OSCA1的系統發育關系,分別對OSCA1.1~OSCA1.6、OSCA1.7和OSCA1.8這三大分支進行進一步分析。
2.2?OSCA1.1~OSCA1.6的系統發育分析
植物OSCA1.1、OSCA1.2、OSCA1.3、OSCA1.4、OSCA1.5和OSCA1.6的系統發育分析見圖3。OSCA1.2和OSCA1.5在被子植物中廣泛分布,數量不一,而OSCA1.3、OSCA1.4和OSCA1.6僅在真雙子葉植物的十字花科植物中分布。在單子葉植物的禾本科和棕櫚科起源之前分別發生了一次基因重復事件。
OSCA1.2在錦葵科棉屬、楊柳科楊屬以及十字花科蘿卜與蕓薹屬起源之前分別發生了基因重復事件,在薔薇科植物中,蘋果(Malus domestica)和梨(Pyrus bretschneideri)發生了一次基因重復事件。OSCA1.5在錦葵科棉屬和楊柳科楊屬起源之前分別發生了重復事件。OSCA1.6僅在真雙子葉植物的十字花科中有分布,其中,在蕓薹屬植物白菜(Brassia rapa)、歐洲油菜(Brassica napus)、甘藍(Brassica oleracea)和蘿卜(Raphanus sativus)起源之前發生了基因重復事件。
在十字花科起源之前發生了基因重復事件,其中OS?CA1.2重復產生OSCA1.?OSCA1.5重復產生OSCA1.6,OSCA1.3和OSCA1.4也來自于同一個祖先。OSCA1.1、OSCA1.2、OSCA1.3和OSCA1.4這4個成員中,OSCA1.1與OSCA1.2、OSCA1.3與OSCA1.4可能來自于一次重復事件,它們可能都來源于OSCA1.2。在菊分支和薔薇分支分開之前發生了一次基因重復事件,出現了OSCA1.2和OSCA1.5這兩大分支,在十字花科起源之前由2個成員演變為6個,即OSCA1.2演變為OSCA1.1、OSCA1.2、OSCA1.3和OSCA1.4,OSCA1.5演變為OSCA1.5和OSCA1.6。
2.3?OSCA1.7的系統發育分析
植物OSCA1.7的系統發育分析見圖4。OSCA1.7在被子植物中廣泛分布。其中,在真雙子葉植物茄科、大戟科木薯屬、錦葵科棉屬起源之前分別發生了基因重復事件。藜科植物藜麥(Chenopodium quinoa)、傘形科植物胡蘿卜(Daucus carota)、木棉科植物榴蓮(Durio zibethinus)和豆科植物花生(Arachis hypogaea)中分別發生了一次基因重復事件。
2.4?OSCA1.8的系統發育分析
植物OSCA1.8的系統發育分析見圖5。OSCA1.8也在被子植物中廣泛分布。其中,在真雙子葉植物楊柳科楊屬起源之前發生了基因重復事件。在睡蓮科植物蓮(Nelumbo nucifera)和旋花科植物牽牛花(Pharbitis nil)中分別發生了一次基因重復事件。
3?討論
OSCA1編碼植物葉片和根細胞膜中的蛋白質,作為離子通道,在干旱期能夠允許Ca2+進入細胞。OSCA1家族在功能上與擬南芥的滲透脅迫反應相關,是植物滲透感受機制的重要成分。基因重復事件在進化過程中起重要作用,為植物提供了豐富的遺傳原料,以應對環境脅迫機制的演變,從而促進植物進化。通過對82個物種的OSCA1家族進行系統發育分析,可以得出在該家族的8個成員中均有基因重復事件的發生。
OSCA1.1與OSCA1.2、OSCA1.3與OSCA1.4、OSCA1.5與OSCA1.6兩兩之間分別聚為一支。OSCA1.1、OSCA1.3、OSCA1.4、OSCA1.6僅在十字花科中分布,在十字花科起源之前發生了多個基因重復事件,其中,OSCA1.2重復產生OSCA1.1、OSCA1.5重復產生OSCA1.6,因此可以看作是十字花科起源之前的一次重復事件造成的OSCA1.1與OSCA1.2、OSCA1.3與OSCA1.4、OSCA1.5與OSCA1.6的基因重復。禾本科植物單獨聚為一支,且位于基因樹的基部,這可能與禾本科植物基因的快速進化有關。OSCA1.7與OSCA1.8分別單獨聚為一支。
從小立碗蘚開始,整個高等植物中只有一支OSCA?且支持率都高達100%。通過OSCA1各成員在植物中的分布情況以及相關基因重復事件的發生可以得出,OSCA1在被子植物中可能最多有四大分支(即OSCA1.2、OSCA1.5、OSCA1.7和OSCA1.8)。
古多倍化也稱為全基因組加倍,是物種進化的重要機制之一,對物種進化有推動作用[8-9]。全基因組加倍也可能對基因組的進化有推進作用[10-11]。此前有研究表明,真雙子葉植物的祖先在115百萬~130百萬年前經歷了一次全基因組三倍化事件[12],此后十字花科植物經歷了2次十字花科作物共有的加倍(brassicaceae common duplication,BCD),分別稱為BCD-β、BCD-α,隨后蘿卜與蕓薹屬作物共同經歷了一次蕓薹屬全基因組三倍化[13]。該研究中的基因重復事件可能與這些全基因組加倍事件相關。
該研究通過對82個物種的OSCA1家族進行取樣,結果表明,OSCA1家族在藻類植物中已有分布,且其在低等到高等植物中的數量分布大致呈現遞增趨勢。
參考文獻
[1] GU X Y,WANG P,LIU Z, et al.Genome?wide identification and expression analysisof the OSCA gene family in Pyrus bretschneideri[J].Canadian journal of plant science,2018,98(4):918-929.
[2] 李建偉,楊珺凱,賈博為,等.大豆基因組中OSCA基因家族的進化和表達分析[J].中國油料作物學報,2017,39(5):589-599.
[3] BERRIDGE M J,LIPP P,BOOTMAN M D.The versatility and universality of calcium signalling[J].Nature reviews molecular cell biology,2000,1(1):11-21.
[4] KUDLA J,BATISTICˇ O,HASHIMOTO K.Calcium signals:The lead currency of plant information processing[J].The plant cell,2010,22(3):541-563.
[5] BATISTICˇ O,KUDLA J.Analysis of calcium signaling pathways in plants[J].Biochimica et biophysica act,2012,1820(8):1283-1293.
[6] YUAN F,YANG H M,XUE Y,et al.OSCA1 mediates osmotic?stress?evoked Ca2+ increases vital for osmosensing in Arabidopsis[J].Nature,2014,514(7522):367-371.
[7] HOU C C,TIAN W,KLEIST T,et al.DUF221 proteins are a family of osmosensitive calciumpermeable cation channels conserved across eukaryotes[J].Cell research,2014,24:632-635.
[8] 季磊,楊艷梅.十字花科開花相關基因SBP進化的基因組信息學分析[J/OL].分子植物育種,2018-11-06[2019-04-05].http://kns.cnki.net/kcmc/detail/46.1068.S.20181102.1644.008.html.
[9] JIAO Y N,LEEBENS?MACK J,AYYAMPALAYAM S,et al.A genome triplication associated with early diversification of the core eudicots[J].Genome biology,2012,13(1):1-14.
[10] JIAO Y N,WICKETT N J,AYYAMPALAYAM S,et al.Ancestral polyploidy in seed plants and angiosperms[J].Nature,201?473(7345):97-100.
[11] SOLTIS D E,VISGER C J,MARCHANT D B,et al.Polyploidy:Pitfalls and paths to a paradigm[J].Amrican journal of botany,2016,103(7):1146-1166.
[12] DENOEUD F,CARRETERO?PAULET L,DEREEPER A,et al.The coffee genome provides insight into the convergent evolution of caffeine biosynthesis[J].Science,2014,345(6201):1181-1184.
[13] MURAT F,LOUIS A,MAUMUSF,et al.Understanding Brassicaceae evolution through ancestral genome reconstruction[J].Genome biology,2016,16:1-17.

