基于同層多尺度核CNN的單細胞圖像分類
郝占龍,羅曉曙,趙書林
1.廣西師范大學 電子工程學院,廣西 桂林 541004
2.廣西師范大學 化學與藥學學院,廣西 桂林 541004
1 引言
CNN已經在機器學習領域中得到了廣泛的應用[1-3]。CNN模型作為一種有效的圖像分類工具,已經應用在人臉識別、字符識別、自然圖像分類等領域[4-6]。CNN模型對圖像進行圖像分類,避免了一些傳統的分類方法中對圖像特征提取困難,特征提取復雜度高,通用性差等問題[7-11]。經典的CNN模型都需要較多的數據進行參數的訓練才能具有一定的分類能力,而HEp-2數據集中僅有訓練集為721張、測試集為738張總計1 459張單細胞圖像,無法直接進行CNN模型的訓練,并且經典的CNN模型普遍采用每層相同尺度卷積核的結構;為了使模型在不同尺度上對圖像進行特征提取,針對以上問題,本文采用數據提升方法對訓練集單圖像進行擴充,并設計同層多尺度核CNN模型,仿真測試表明本文數據提升方法有效訓練了網絡參數,并且本文同層多尺度核CNN模型提高了單細胞圖像的識別率。
2 針對小數據集單細胞圖像數據提升
盡管CNN使用局部感受野,權值共享的方式減少了大量的參數,但是相對于本文使用的細胞圖像數據集,網絡模型的參數數量依然很多,訓練這樣的網絡仍然具有很大的挑戰。
在本文使用的兩個細胞圖像數據庫中,hep2的單細胞圖像標準訓練集為721張,測試集為738張;深度學習是一種自動學習特征的機器學習方法,需要足夠的訓練樣本才能夠更加有效地調整網絡參數,顯然如此小規模的圖像顯然無法直接有效訓練卷積神經網絡中的眾多參數。對于每一張醫學圖像都來自承受著疾病痛苦的病人,應該想辦法盡量充分利用這些圖片中的更多信息。為了盡量利用有限的訓練數據,可以通過使用一些方法對數據進行提升,這樣,模型將看不到兩張完全相同的圖片,也有利于抑制過擬合,使得模型更有效地調整參數,提高泛化能力。
數據提升的方法主要有:裁剪、旋轉[12]、亮度、對比度變換[13]、規范化等。
通過三階插值變換對圖像的尺寸進行規范化,然后通過裁剪、旋轉、亮度、對比度變換、規范化等方式對數據集進行提升;通過數據提升,使得可用訓練集單細胞圖像樣本數量得到了擴充,每張尺寸為72×72的單細胞圖像進行64×64裁剪即可得到64張不同的圖像,也即使原始單細胞圖像數據集擴大64倍;若每張圖像只進行90°步長的旋轉,即可得到4張不同的圖像,單細胞數據集再次擴大4倍,通過這兩種方法,原來的HEp-2訓練集中721張單細胞圖像即可擴大到184 576張,擴大256倍,再通過對比度、亮度變化所得到的圖像數量將更加龐大。通過這些方法擴大了數據集,有利于卷積神經網絡中參數的訓練和模型的泛化;為下一步卷積神經網絡的設計和訓練提供了基礎。具體數據提升公式可表示為:

其中 y為輸出圖像,I為輸入圖像,Fa、Fb分別為亮度、對比度變換系數,crop為裁剪矩陣,rot為旋轉矩陣。
如圖1所示為一張單細胞圖像某次數據提升效果圖,第一張為原圖,數據提升1~3參數如表1所示,其中crop為裁剪矩陣起點坐標,rot為旋轉矩陣所旋轉的角度。
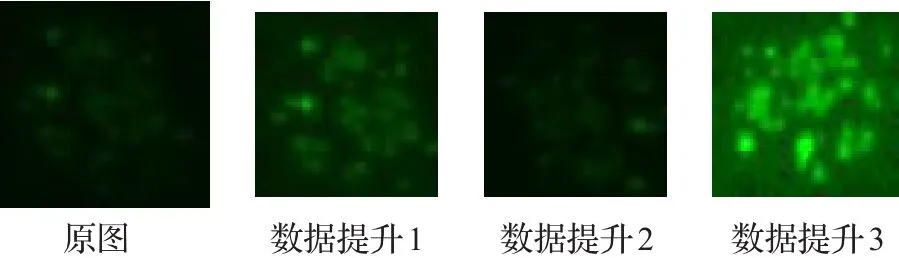
圖1 單細胞圖像數據提升效果圖
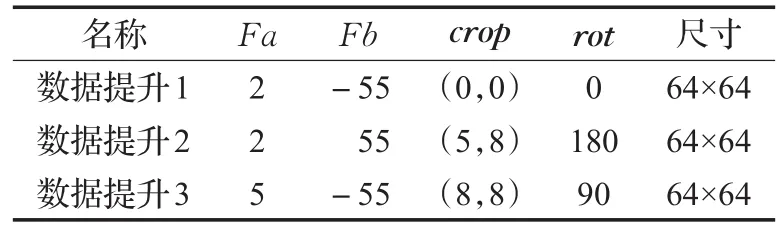
表1 數據提升參數表
3 基于同層多尺度核卷積神經網絡模型
LeNet-5[14]等經典模型使用固定的尺度對細胞圖像進行觀察,也即每層卷積核尺度是單一固定的,這意味著感受野是固定的;而人類視覺過程中,隨著人觀察事物的關注點的不同,感受野的大小并不是單一的,所以在單層卷積時使用多個不同尺度的卷積核同時卷積做為下一層的輸入,這樣有利于網絡在不同的尺度上對圖像的特征信息進行更充分的提取。
考慮到人類視覺過程隨著興趣點的不同感受野會不同的特點,參考LeNet-5模型結構,將第一個卷積層改為一個5×5和一個7×7兩個不同尺度的卷積核ω1、ω2,使得卷積神經網絡可以通過不同的尺度處理輸入的圖像。則第一個卷積層的輸出Out為:

其中I為輸入圖像,?代表same方式卷積。如圖2所示為same方式卷積和valid方式卷積示意圖。
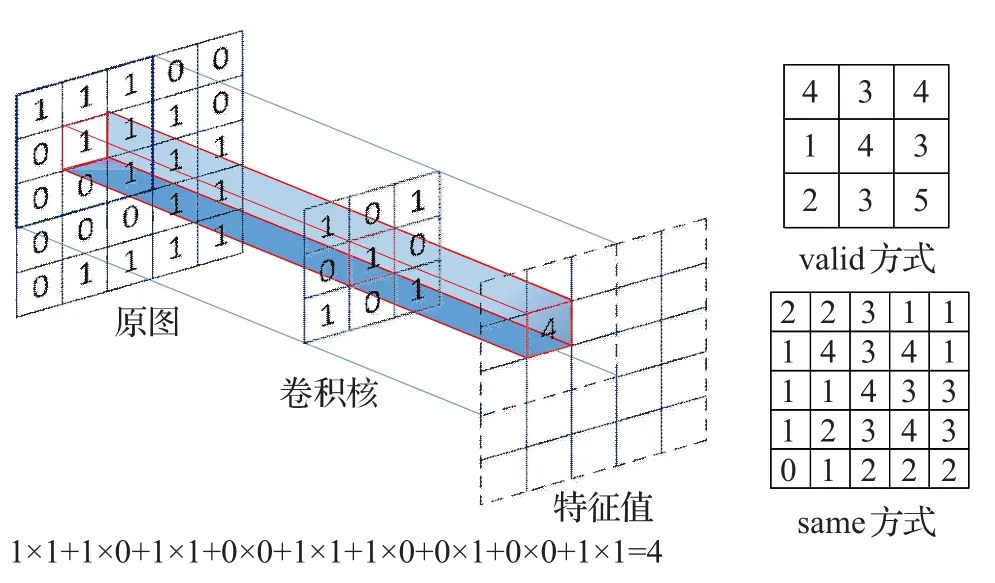
圖2 兩種卷積運算示意圖
如圖3所示為本文所述同層多尺度核卷積神經網絡模型。其中C代表卷積(Convolutional),P代表池化(Pooling),例如C1.1@64×64×8代表第一層第一個卷積核卷積得到的特征圖,尺寸為64×64,一共有8張;模型使用ReLUs激活函數,每次最大池化后進行局部歸一化再輸入到下一層。用表示經過ReLUs的神經元在(x,y)處應用核函數i的響應,則局部響應歸一化可表示為[15]:
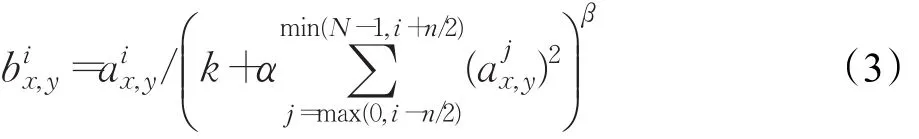
其中N是該層核函數的總數,n是參與競爭的鄰域數量,超參數 k,n,α和 β 可設置為[15]k=2,n=5,α=10-4,β=0.75。
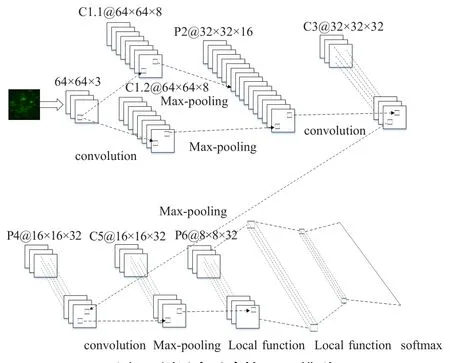
圖3 同層多尺度核CNN模型
4 仿真實驗
為了研究部分參數對CNN模型的影響,調整部分參數,和本文模型不同的是,CNN1去掉了卷積層Conv3,CNN2去掉了全連接函數Localfunction2,CNN3去掉Conv1.2卷積核函數,所有模型均采用最大池化——maxpooling,具體參數如表2所示。
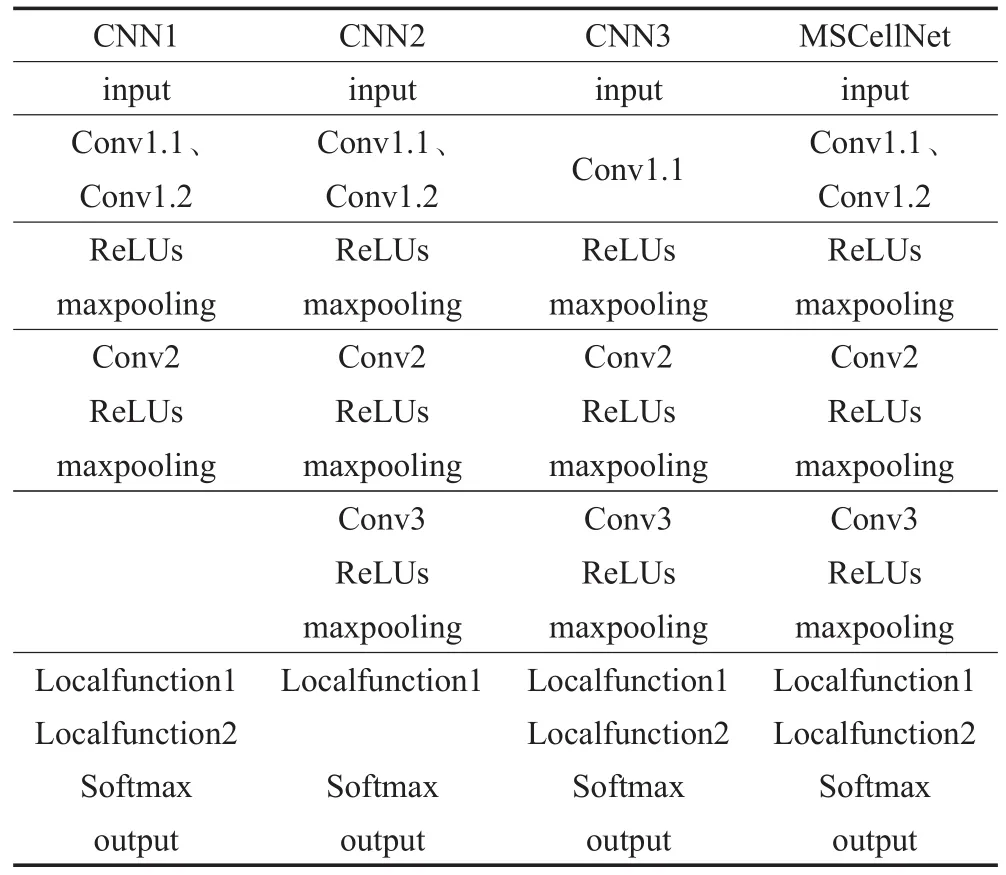
表2 幾種CNN模型參數列表
圖4為幾種不同模型下測試集準確率曲線,從圖中可以看出,本文模型較其他參考模型測試集預測準確率都更高,說明本文所述模型中任何一部分結構都是缺一不可的,最下面一條曲線為使用本文模型時的無數據提升模型識別率,說明數據提升后能夠更加有效地訓練網絡參數;表3為4種模型的HEp-2單細胞識別率,和Faraki[7],Nosaka[8]方法不同的是本文模型使用數據提升后單細胞圖像進行訓練,使得模型對于殘缺、對比度亮度變化、旋轉具有一定的適應性,本文模型識別率為72.1%比單尺度模型CNN4提高2.7%。表4為本文模型和近年其他文獻對HEp-2單細胞圖像六分類識別率的對比,本文所述模型對HEp-2單細胞六分類識別率72.1%分別比ICPR2012競賽最佳方法68.7%提高了3.4%,比ICPR2012競賽中的CNN模型分類識別率提高了12.3%;對比其他方法也都有一定程度的識別率提升。
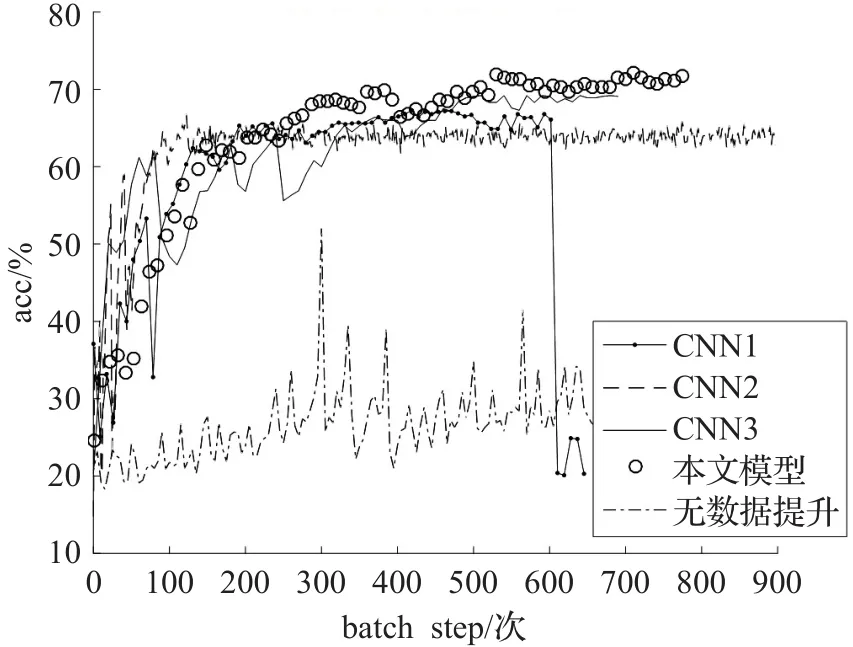
圖4 不同模型HEp-2測試集準確率變化曲線

表3 4種模型細胞分類識別率 %
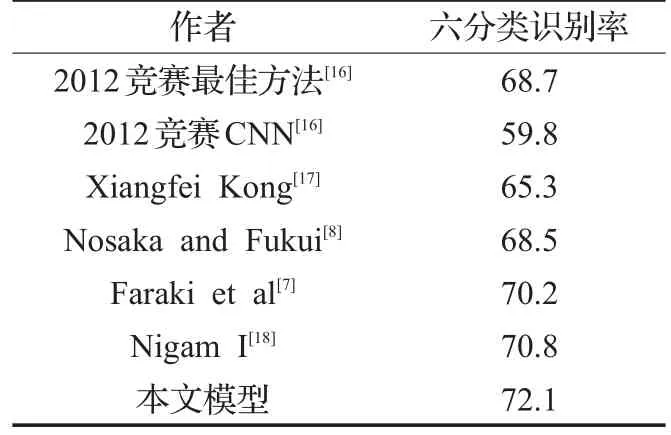
表4 本文模型和近年其他文獻對HEp-2細胞圖像六分類識別率 %
5 結束語
本文所述的同層多尺度核CNN模型避免了Faraki[7],Nosaka[8]細胞圖像分類方法特征提取困難、計算復雜、只適用特定的細胞類型等缺點,具有較好的魯棒性和抗擾性,對于圖像殘缺、對比度亮度不同以及旋轉等都仍然能夠很好地完成HEp-2單細胞圖像的六分類,具有一定的應用價值。

