黃淮麥區主要推廣小麥品種(系)DNA指紋圖譜構建及遺傳多樣性分析
楊艷紅,尹華燕,高雨,趙金曉,馬信,王宏偉,李安飛,孔令讓,李憲彬
?
黃淮麥區主要推廣小麥品種(系)DNA指紋圖譜構建及遺傳多樣性分析
楊艷紅,尹華燕,高雨,趙金曉,馬信,王宏偉,李安飛,孔令讓,李憲彬*
山東農業大學農學院, 山東 泰安 271018
小麥作為世界上重要的糧食作物,其豐富的遺傳變異對小麥的遺傳改良工作至關重要。本研究利用均勻分布在小麥A、B、D基因組上的84對SSR引物對119份黃淮麥區推廣小麥品種進行了DNA指紋數據庫的構建,并進行遺傳多樣性分析。結果表明,84對SSR引物共檢測到170個等位變異,每對引物檢測出1~5個等位變異,其中有13對引物可以將某一小麥與其他品種區分開。根據SSR基因型聚類結果可將119份小麥品種 (系)分為5大類群,基本能夠分辨各個推廣品種間的親緣關系,為小麥親本選擇及雜交組配提供了一定的理論依據。
小麥; SSR引物; 指紋圖譜; 遺傳相似性
小麥是世界上主要的糧食作物之一,在我國小麥約占糧食總產的27%,是我國第二大糧食作物。近年來,由于長期品種間的雜交選育,現有育成品種間同質性較高,種質資源多樣性愈來愈狹窄,小麥遺傳改良難以實現突破性進展[1-3]。前人已利用多種方法對小麥遺傳多樣性進行評價分析,其中分子標記由于不受環境影響且遍布整個基因組,被普遍認為是研究遺傳差異的理想工具。簡單重復序列SSR(Simple Sequence Repeat),也稱作微衛星,是由1~6個堿基組成的基本序列串聯重復組成的短片段,具有簡便、快速、穩定性好和等位基因多樣性高等優點,現已成功應用于小麥的遺傳差異和遺傳演化研究等諸多方面[4]。DNA指紋圖譜是指建立在DNA標記基礎上的某一品種所具有的區別于其他品種的特異DNA片段,利用SSR標記進行遺傳多樣性分析、DNA指紋圖譜構建、功能基因標記和比較作圖等方面具有公認的優越性和廣闊的應用前景[5]。本研究擬采用SSR分子標記技術,通過均勻分布在小麥A、B、D基因組上的84對SSR引物對黃淮地區119份推廣小麥品種的遺傳多樣性進行研究分析,建立DNA指紋圖譜數據庫,從而掌握小麥品種資源的遺傳基礎和遺傳多樣性狀況,對于有效促進小麥育種工作具有重要意義。
1 材料與方法
1.1 試驗材料
利用均勻分布在小麥A、B、D基因組上的84對SSR標記(表2)對119份近年來黃淮地區育成的小麥新品種(系)和其他一些地方品種(表1)進行指紋構建和遺傳多樣性分析。引物由上海生工生物工程有限公司(Shanghai Sangon Co.Ltd.上海)合成。試驗材料種植于山東農業大學試驗基地。

表1 小麥品種(系)名稱、編號及系譜信息
1.2 實驗方法
1.2.1 DNA提取于小麥分蘗期取新鮮健康的葉片,按照CTAB法提取基因組DNA[6]。
1.2.2 PCR擴增選取均勻分布于小麥A、B、D基因組上的84對SSR引物對上述119份推廣小麥品種進行PCR分析。PCR反應體系為10 μL,含5 μL 2*Taq Plus Master Mix,20 ng-1μL前后引物各1 μL,20 ng-1μL基因組DNA1 μL,2 μL ddH2O[7]。PCR擴增程序如下:95 ℃預變性5 min,94 ℃變性30 s,55~65 ℃復性(因不同引物而異)40 s,72 ℃延伸40 s,35個循環;72 ℃延伸10 min;4 ℃保存。
1.2.3 電泳檢測擴增產物PCR結束后在擴增產物中加入適量Loading Buffer,混勻后在8%聚丙烯酰胺非變性凝膠上電泳,上樣量為1.5 μL 1xTBE,120 V,電泳3~3.5 h(根據片段大小適當延長或縮短電泳時間)。電泳結束后將膠塊銀染顯影,用凝膠成像系統照相,對帶型清晰的膠塊進行讀帶。
1.3 數據處理與分析
根據PCR擴增結果,將同一引物擴增出的大小相同的目的帶視為1個等位變異[8,9],并進行記錄。用同一引物對119份材料進行擴增檢測時,相同的帶型用同一數字進行賦值,無帶賦值為“0”,數據采用Powermarker軟件進行聚類,繪制聚類圖。
2 結果與分析
2.1 SSR標記在119個小麥品種之間多態性分析
84對引物在119個小麥品種中共檢測出170個等位變異,每對引物檢測出1~5個等位變異,平均為2.02個。其中,有13對引物(gwm124、gwm617、cfa2019、wmc173、wmc752、barc96、barc126、gwm294、barc35、gwm174、cfd22、gwm397、wmc474)可以將某一品種與其他品種( 系 )區分開,如引物gwm124在“山農29”中檢測到的等位變異與其他材料不同(圖1),因此可以將“山農29”與其他品種(系)區分開來;同理,引物gwm617可以將“92145E8”與其他品種(系)區分開來;引物cfa2019可以將“良星66”與其他品種(系)區分開;引物wmc173可以將“揚麥158”與其他品種(系)區分開;wmc752可以將“山農15381”與其他品種(系)分開。此外,利用同一個引物還可將2~3個品種從119份品種(系)中區分開,如引物wmc623可以將“封麥6號”“鄭麥379”、“周麥27”與其他品種(系)區分開;引物wmc617可以將“揚麥158”、“石麥15”、“煙19”與其他品種(系)區分開來;引物cfd9可以將“徐麥2178”、“邯優3475’與其他材料區分開來;引物gwm480可以將“泰山818”和“煙5158”與其他品種(系)區分開來,引物gwm164可以將“太麥198”和“山農紫麥1號’與其他品種(系)區分開來;cfd48可以將“邯6172”“豫農949”與其他品種(系)區分開。其他引物單獨使用時不能鑒別出119份小麥品種(系)中的任何一個或幾個材料。
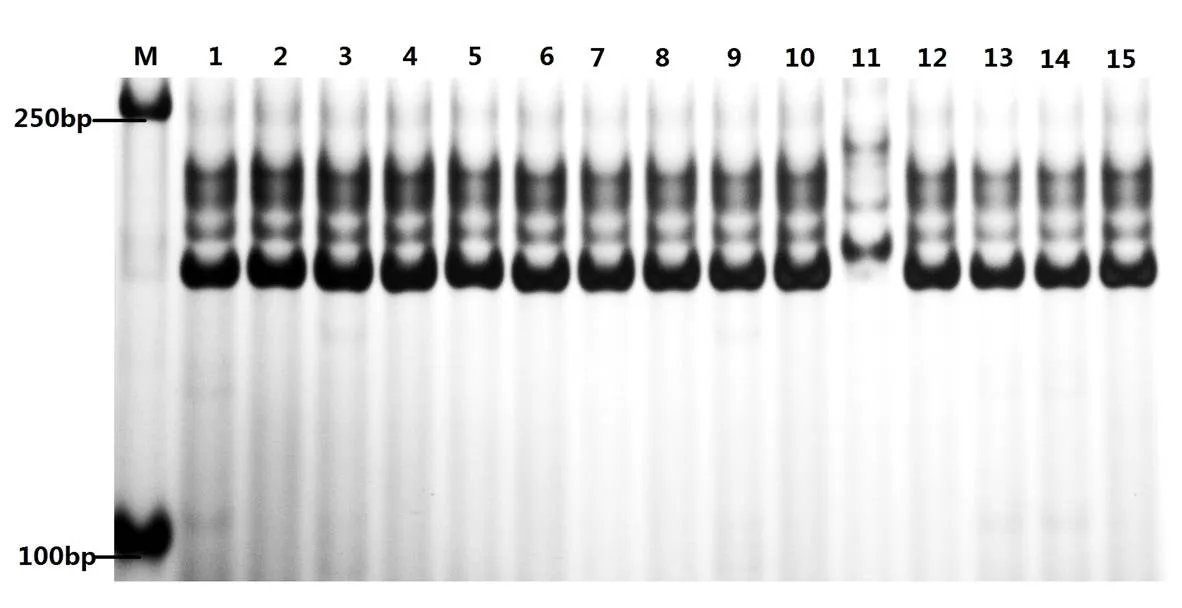
圖 1 標記Xgwm124在部分小麥品種中的擴增結果
M:D2000; 1:濰麥33;2:寧麥13;3:鎮麥168;4:揚麥158;5:太麥198;6:山農紫麥1號;7:山農20;8:山農23;9:山農24;10:山農25;11:山農29;12:山農30;13:山農31;14:山農32;15:山農15381
M:D2000; 1:Weimai 33;2:Ningmai 13;3:Zhenmai 168;4:Yangmai 158;5:Taimai 198;6:Shannongzimai1hao;7:Shannong20;8:Shannong 23;9:Shannong 24;10:Shannong 25;11:Shannong 29;12:Shannong 30;13:Shannong 31;14:Shannong 32;15:Shannong 15381
2.2 119份小麥種質材料的DNA指紋數據庫的初步構建
由于本試驗中單個引物僅能區分出少數品種,因此,為了準確鑒別119份小麥品種(系),選擇其中10對帶型清晰且穩定的引物,將每一對引物擴增的帶型進行統計并編號,得到了119份小麥品種(系)的DNA指紋數據庫(附表1)。84對SSR引物共鑒定出170個等位變異,所檢測到的不同等位變異及所處染色體位置等基本信息詳見表2。

表 2 84對 SSR 引物在119份小麥品種中檢測到的等位基因
2.3 119份小麥種質材料的遺傳多樣性分析
利用84對SSR引物所檢測出的170個等位變異,對其進行聚類(圖2)后可以將119份品種(系)基本劃分為5個類群。
第一類群包括“鄭麥1325”、“山農664”、“石03-Y119”三個品種;第二類群包括“新鄉9852”、“小偃54號”、“鄭麥306”3個品種;第三類群包括“德勝508”、“金麥73”、“臨麥8號”等18個品種;第四類群包括“周麥26”、“豫麥416”“山農3509”、“矮抗58”等42個品種;第五類群包括“中育211”、“濟麥23”、“許科129”、“鄭麥379”等53個小麥品種。將上述SSR標記聚類結果就所選材料的系譜關系(表1)進行比較,聚類結果在一定程度上反映了品種之間的親緣關系,如“周麥27”和“許科316”均為“周麥16”作為母本選擇的品種,因此劃分到同一類群,但因為其父本分別為“百農64”和“矮抗58”,所以具體分類時,聚類到兩個小類群里;“山農23”和“山農24”均是從創建的Ta1(Ms2)小麥輪選群體中選擇出的小麥品種,二者在同屬一個大類的同時也單獨聚為一小類;同樣,“新麥35”、“新麥36”均由“周麥22”作為母本選育而成,聚類時聚到同一類群;“孟麥023”、“淮麥39(MS)”、“冠麥1號”均由周麥13作為母本選育出來,因此聚為同一類群里。但也有系譜親緣關系較近的品種與SSR聚類不一致的現象,如“菏麥0643”就是以“矮抗58”為母本、“濟麥19”為父本雜交選育的,但二者SSR基因型存在一定的差異,因此劃分為不同的兩個類群。出現這種現象一方面可能因為本研究選用分子標記較少,代表性不夠;另一方面可能與當下小麥育種資源交流頻繁,導致品種的地域性差異程度降低[10]有關。

圖 2 119 份材料的SSR聚類分析
3 討論
由于長期小麥品種間雜交選育,小麥品種遺傳多樣性的降低已成為世界范圍內的共識[11-16]。在我國1586份小麥初選核心種質中,以20世紀50年代的品種具有最高的遺傳多樣性,到90年代降至最低[17],因此提高小麥種質材料的遺傳多樣性對促進遺傳改良工作至關重要。分子標記被普遍認為是研究遺傳差異的最理想工具,新發展起來的微衛星SSR分子標記技術具有簡便、快速、穩定性好和等位基因多樣性高等優點,已成功應用于小麥遺傳差異和遺傳演化研究等方面[18]。王立新[19]等采用15個SSR標記和20個AFLP-SCAR標記,分析了來自我國不同麥區的455個小麥品種,證明用分子標記建立小麥DNA指紋可以更加全面地反映品種的遺傳多樣性。高睦槍等[20]用53對 SSR引物對全國1999~2000年北方冬小麥及黃淮冬麥區觀察圃中選出的48個新品種(系)進行了遺傳差異分析,共檢測到367個等位變異。本研究通過84對SSR標記對119份黃淮地區推廣小麥品種(系)進行遺傳多樣性分析,共檢測到170個等位變異,遺傳變異較小,經聚類分析后發現,119份材料主要聚為5個類群,這種結果一方面可能因為我們選用的分子標記較少且部分標記在種質材料中多態性較低,另一方面可能也與選用材料大部分為主栽品種有關系。同時本研究結果也為育種工作者選擇親本組配雜交組合提供了一定的理論依據。
[1] 李興普.不同矮稈基因小麥農藝性狀的遺傳差異[J].華北農學報,1995,10(1):1-5
[2] 相吉山,楊欣明,李秀全,等.小麥骨干親本南大2419對衍生品種(系)HMW-GS的貢獻分析[J].植物遺傳資源學 報,2013,14(6):1053-1058
[3] 劉玉平,李亞軍,陳希勇,等.河北省審定優質麥主要性狀分析[J].河北農業科學,2006,10(2):50-53
[4] Lelley T, Stachel M, Grausgruber H,. Analysis of relationship between Aegilops tauschil and the D genome of wheat utilizing microsatellites[J]. Genome, 2000,43:661-668
[5] 張學勇,李大勇.小麥及其近親基因組中的DNA重復序列研究進展[J].中國農業科學,2000,33(5):14-24
[6] 樓巧君,陳亮,羅利軍.三種水稻基因組DNA快速提取方法的比較[J].分子植物育種,2005,3(5):749-752
[7] 梁宏偉,王長忠,李忠,等.聚丙烯酰胺凝膠快速、高效印染方法的建立[J].技術與方法,2000,30(10):1379-1382
[8] 李根英, Susanne D, Marilyn LW,等.小麥指紋圖譜數據庫的建立及SSR分子標記試劑盒的研發[J].作物學 報,2006,36(12):1771-1778
[9] Nei M, Li WH. Mathematical mode for studying genetic varia-tion in terms of restriction endonucleases[J]. Proceedings of the National Academy of Sciences of the United States of Ameri-ca, 1979,76:5269-5273
[10] 程保山,徐海風,顧正中,等.淮安地區主栽小麥品種指紋圖譜的構建及遺傳多樣性分析[J].浙江農業學 報,2011,23(1):20-24
[11] Fu YB, Peterson GW, Richards KW,. Allelic reduction and genetic shift in the Canadian hard red spring wheat germplasm re-leased from 1845 to 2004[J]. Theor Appl Genet, 2005,110:1505-1516
[12] Huang XQ, B?rner A, R?der MS,. Assessing genetic diversity of wheat(L.) germplasm using microsatellitemarkers[J]. Theor Appl Genet, 2002,105:699-707
[13] Orabi J, Jahoor A, Backes G. Changes in allelic frequency over time in European bread wheat(L. ) varieties re-vealed using DArT and SSR markers[J]. Euphytica, 2014,197(3):447-462
[14] Roussel V, Koenig J, Beckert M,. Molecular diversity in Frenchbread wheat accessions related to temporal trends and breeding pro-grammes[J]. Theor Appl Genet, 2004,108:920-930
[15] 謝煒,郭青云,郭小敏,等.同名地方品種小紅芒和小紅芒麥形態學和HMW-GS組成的演變分析[J].植物遺傳資源 學報,2011,12(3):381-388
[16] 李遠,趙檀,王睿輝,等.河北省小麥品種基于SSR 標記的遺傳多樣性研究[J].河北農業大學學報,2012,4(35):1-5
[17] Smale M, Reynolds MP, Warburton M,. Dimensions of diver-sity in modern spring bread wheat in developing countries from 1965[J]. Crop Sci, 2002,42:1766-1779
[18] 郝晨陽.五十年來我國小麥育成品種的遺傳多樣性演變及西北春麥區核心種質的構建[D].蘭州:甘肅農業大學,2004
[19] 王立新,李云伏,常利芳,等.建立小麥品種DNA指紋的方法研究[J].作物學報,2007,33(10):1738-1740
[20] 高睦槍,劉冬成,郭小麗,等.我國部分冬小麥新品種(系)SSR標記遺傳差異的研究[J].農業生物技術學 報,2001,9(1):49-54
附表1 10對SSR引物構建的 119個小麥品種(系)的SSR指紋圖譜
Attached list 1 Data of the SSR fingerprint bands with 10 pairs of SSR primer among 119 wheat cultivars

編號No.名稱Varieties引物名稱及條帶編號 Primers and band numbergwm294wmc317wmc622gwm369gwm617cfa2019wmc623cfd48wmc752cfd7 1中麥1751114112111 2許科1292114112112 3冠麥1號1114131111 4中育11234114211111 5周麥305114211111 6封麥6號3114233111 7洛麥291113231111 8鄭麥3791114113112 9鄭麥08561124111111 10鄭麥76981111111121 11山農146281533111212 12山農35091312241212 13濟麥221424141212 14山農221424112212 15山農26581121111222 16鄭麥13421114111321 17鄭麥3691121142221 18良星661111122121 19山農281121141112 20鄭麥18601111232111 21徐農0291112211211 22徐農21781412132111 23存麥181112132111 24國麥1271114111111 25柱麥3051314212121 26孟麥0231314132111 27中原181111131111 28濮興1114132112 29偃展11101341112122 30中麥661112112121 31良星5181124112222 32盈滿2081124142212 33淮麥39(MS)1414111211 34河北4158*4453112131 35菏麥20*1521142132 36菏麥0643*1124142212 37中育12111411212111 38中育14281314131111 39新麥351114112121 40新麥361111112121 41百農4161112232111 42周麥361111212111 43豫麥1581114131111 44鄭品麥241112131111 45濟麥231124142111 46濟麥441114242111 4792145E83121432111 48豫農2111111131111 49魯原5021114312102 50Classic2144132101 51德盛5084531112101 52濟麥191141312101 53濟寧13號2111231101 54金麥6號4111112101 55金麥731132131101 56科信8號1112112101 57萊農03011421212202 58聊17091151312101 59聊98313414311102 60聊麥16號1143111101 61臨麥7號1424341102 62臨麥8號1423332102 63魯麥211124212101 64魯麥222211211101 65山農6641112212102 66石03-Y1194541111111 67素農24133422131111 68泰山8181121111131 69濰麥8號1141342132 70汶農141124142112 71汶農151124131112 72西農5號1552132112 73西農9791111111131 74小偃54號1454131111 75小偃6號3114211132 76新鄉98523113112112 77徐州40431121131112 78煙1801121211111 79煙193421131111 80煙51581111111111 81煙農1811411311111 82矮抗581112132111 83邯61721124131311 84鄭麥3063121131111 85周麥221111111131 8602H171141131131 87鄭麥8831111212131 88鄭麥13251214231312 89國麥3011414211211 90豫農9492111142131 91良星991421131212 92安農11241114211131 93衡觀352131111131 94邯優34753114111132 95滄麥193122112132 96中麥1551124242112 97登海50481104131131 98豫農8021102232131 99宿12642101231111 100許科3164201211131 101豫麥4161104111111 102周麥261304132111 103周麥27(粉)1102113111 104石麥151102311131 105濰麥333104112231 106寧麥131403112212 107鎮麥1681401112312 108揚麥1581101111112 109太麥1983102112111 110山農紫麥1號1103112111 111山農201414111111 112山農231414111112 113山農241114112112 114山農251114112111 115山農291413141112 116山農303114111112 117山農313414111111 118山農321414141111 119山農153813412111131
Establishment of DNA Fingerprinting and Genetic Diversity Analysis of Main Wheat Cultivars in Huanghuai Region
YANG Yang-hong, YIN Hua-yan, GAO Yu, ZHAO JIN-xiao, MA Xin, WANG Hong-wei, LI An-fei, KONG Ling-rang, LI Xian-bin*
271018,
Wheat is an important food crop in the world, its rich genetic variation is crucial to wheat improvement. Eighty-four SSR primers distributed on A, B, Dgenome were used to build DNA fingerprinting database and genetic diversity analysis in 119 samples of main wheat cultivars in Huanghuai region. The study showed that 170 variations were detected by using the 84 pairs of SSR primers, and each pair of primers exhibited 1-5 alleles in 119 wheat cultivars in Huanghuai region. Out of the 84 pairs of SSR primers, 13 pairs of primers can detect one of wheat cultivars different from the other varieties respectively. According to SSR gene clustering dendrogram, 119 wheat varieties could be divided into 5 groups to some extent, which could provide some theoretical information for wheat improvement in the future.
Wheat; SSR primers; fingerprinting; genetic diversity
S512.1
A
1000-2324(2018)03-0371-08
2017-12-1
2017-12-18
山東省自主創新重大關鍵技術(2014GJJS0201-3-1);山東省農業良種工程
楊艷紅(1993-),女,碩士研究生,主要從事小麥種質資源創新工作. E-mail:yyh19930106@163.com
Author for correspondence. E-mail:lqlwlxb@163.com

