生物土壤結皮土生對齒蘚轉錄組SSR位點分析
陳莉 趙浩然 黃琳淞 玉蘇甫江
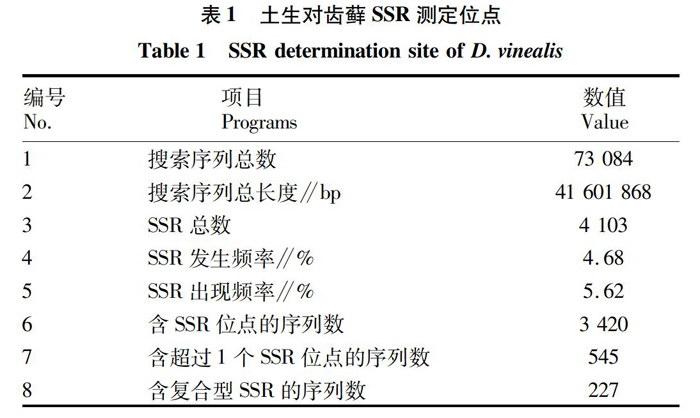
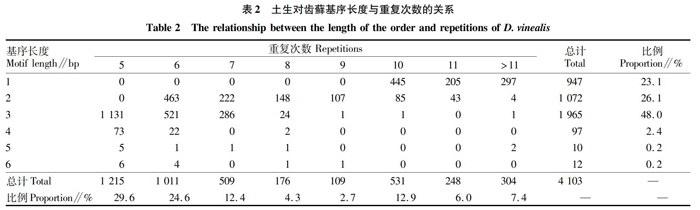
摘要[目的]分析生物土壤結皮土生對齒蘚轉錄組SSR位點。[方法]以生物土壤結皮土生對齒蘚為材料,分析土生對齒蘚轉錄組數據,開發土生對齒蘚SSR分子標記技術。[結果]通過MISA軟件對土生對齒蘚高通量測序獲得的73084條unigenes進行SSR篩選。從搜索序列中發現4103個符合條件的SSRs,分布在3420條unigenes上,出現頻率為5.62%。三核苷酸和二核苷酸為主要重復類型,分別占SSR總數的47.9%和26.1%,AGC/CTG(31.9%)和AG/CT(75.5%)分別是其主要重復基元。69.0%的基序長度為12~20bp。[結論]土生對齒蘚轉錄組SSR位點出現頻率高、類型豐富、多態性潛能大,為土生對齒蘚分子生物學機制研究等方面提供理論依據。
關鍵詞 土生對齒蘚;轉錄組;SSR
中圖分類號 S188 文獻標識碼A 文章編號0517-6611(2018)11-13078-03
土生對齒蘚(Didymodonvinealis)屬于土壤結皮蘚類植物,廣泛存在于干旱、半干旱地區,對于維持生物土壤結皮的穩定,抵御風蝕、水蝕起著關鍵作用。人們對生物土壤結皮中苔蘚的干旱適應性形態特征和繁殖特性已有相關研究。
簡單重復序列(simplesequencerepeat,SSR)又稱微衛星DNA(microsatdliteDNA),是由1~6個核苷酸串聯重復單元組成,長度為10~100bp。SSR具有分布廣泛、多態性強、數量豐富和共顯性遺傳易于分析等特點,可作為較為理想的遺傳標記。SSR標記已被廣泛應用于水稻、大麥、小麥、玉米等植物的遺傳圖譜構建、遺傳多態性分析、品種鑒定、分子標記輔助育種等研究。
隨著新一代高通量測序技術的成熟,轉錄組學得到快速發展,除小立碗蘚之外,對蘚類植物轉錄組學的測定鮮有報道。筆者通過高通量測序方法,從分子水平對土生對齒蘚的SSR進行多態性研究,為進一步探索土生對齒蘚分子生物學機制提供理論依據。……