利用SSR標記分析蠶豆品種(品系)與優異種質的遺傳多樣性
張紅巖 郭興蓮 楊 濤 劉 榮 黃宇寧 季一山 王 棟宗緒曉*
(1中國農業科學院作物科學研究所,北京100081;2青海大學新農村發展研究院,青海西寧 810016;3青海省海東市互助縣農業技術推廣中心,青海海東 810500)
蠶豆(Vicia fabaL.)為豆科蝶形花亞科巢菜屬的唯一栽培種,為越年生(秋播)或一年生(春播)草本植物(鄭卓杰,1997)。據FAO統計,2005~2014年全世界干蠶豆平均生產面積為243.25萬hm2,總產量429.80萬t,其中中國生產面積為96.99萬hm2,總產量164.877萬t,分別占世界的35.76%和38.36%(FAO,2017)。蠶豆在我國已有2 000余年的栽培歷史,是一種重要的糧食、蔬菜和飼料、綠肥兼用作物(馬鈺,2012),是豆類蔬菜中重要的食用豆之一。
種質資源的多樣性是育種工作的基礎。早期,人們主要采用形態學的方法對蠶豆的遺傳多樣性進行研究(Robertson & El-Sherbeeny,1991;Polignano et al.,1999),該方法簡便、經濟,但鑒定周期較長、易受環境影響(王偉 等,2009)。隨著分子生物技術的快速發展,RAPD(Link et al.,1995)、AFLP(Zeid et al.,2004)、ISSR(Fernandez et al.,2002;Terzopoulos & Bebeli,2008)等分子標記技術被應用于蠶豆種質資源的遺傳多樣性研究之中。Link等(1995)利用RAPD標記對3個蠶豆自交系群體進行研究,結果表明,地中海種和歐洲小粒種被分為截然不同的2組,歐洲大粒種介于兩者之間。Zong等(2009)利用10對AFLP引物對204份國內冬性蠶豆資源和39份國外冬性資源進行遺傳多樣性分析,聚類分析和主成分分析結果表明,中國蠶豆資源與國外蠶豆資源界限明顯。王海飛等(2011)利用11對ISSR標記對國內外蠶豆資源的遺傳多樣性進行分析,揭示了中國春播型蠶豆資源遺傳多樣性較為豐富,美洲蠶豆種質資源遺傳基礎相對狹窄。Gong等(2011)利用11對EST-SSR標記對29份國內外蠶豆品種進行遺傳多樣性分析,發現參試中國蠶豆品種遺傳背景較為狹窄。目前,我國蠶豆種質資源的研究水平較為落后,其龐大的基因組嚴重限制了從分子水平研究蠶豆資源遺傳多樣性的步伐。
基于全自動遺傳分析儀的SSR熒光標記具有省時、高通量、高準確度等特點,目前已應用于農作物的遺傳多樣性分析。雍洪軍等(2009)基于該項技術對我國7個省的90份糯玉米進行遺傳多樣性分析,初步將供試玉米品種劃分為3個類群。王瑞云等(2017)利用15對特異性SSR熒光標記掃描我國11個省的132份糜子資源,聚類分析可將參試糜子資源劃分成4類。
蠶豆染色體組2n=12,基因組片段大小約為13 000 kb,基因組龐大、基因組學研究滯后、開發的SSR標記十分有限。本試驗利用中國農業科學院作物科學研究所冷季食用豆類課題組自主開發的99對SSR熒光標記引物,對102份國內育成蠶豆品種(品系)及優異種質資源的遺傳多樣性進行系統研究,旨在揭示中國代表性蠶豆品種(品系)和種質資源間的遺傳多樣性差異,為我國蠶豆育種產業的快速推進、科學合理選配親本及挖掘和利用優異資源提供理論依據。
1 材料與方法
1.1 試驗材料
供試蠶豆材料共計102份,其中青海育成品種(品系)27個、云南育成品種19個、江蘇育成品種6個、優異種質資源50份。優異種質資源主要是指綜合農藝性狀優良、具有抗病蟲性及抗逆性等特殊性狀的蠶豆資源。參試材料均取自國家種質資源中期庫,由中國農業科學院作物科學研究所提供,2016年11月種植于中國農業科學院作物科學研究所網室內。參試材料具體信息見表1。
1.2 SSR熒光標記
利用中國農業科學院作物科學研究所冷季食用豆類課題組構建的蠶豆SSR遺傳連鎖圖譜,經8%非變性凝膠電泳多次篩選,獲得在連鎖群上均勻分布、多態性較高、PCR擴增較穩定的99對SSR標記,標記的5′端分別用FAM(藍色)、HEX(綠色)熒光染料標記。SSR引物合成及PCR擴增產物熒光檢測均委托北京梓熙生物科技有限公司。2×TaqPCR Master Mix購自北京康潤誠業生物科技有限責任公司。DNA Marker購自北京莊盟國際生物基因科技有限公司。
1.3 DNA提取
每份參試材料選擇生長良好的4個單株,取幼嫩葉片混合后,采用改良的CTAB法(Wang et al.,2015)提取DNA,提取緩沖液中加入一定量的水溶性PVP和β-羥基乙醇(Gentile et al.,2017;Sokol et al.,2017),可以有效防止酚類物質的氧化作用。采用1.2%的瓊脂糖凝膠電泳檢測DNA質量,采用NanoDrop 2000軟件(Yang et al.,2015)檢測DNA濃度,根據檢測結果將其稀釋到工作液濃度(20~30 ng·μL-1)后于-20 ℃冰箱保存備用。
1.4 SSR-PCR擴增及產物熒光檢測
采用10 μL的擴增反應體系,其中2×TaqPCR Master Mix 5 μL,2 μmol·L-1的上、下游引物各 1 μL,DNA(30 ng·μL-1)模板 1.5 μL,ddH2O 2.5 μL。PCR反應程序:95 ℃預變性5 min;95 ℃變性35 s,55~60 ℃退火40 s,72 ℃延伸45 s,共32個循環;72 ℃延伸5 min,10 ℃保溫7 min。
將FAM(藍色)和HEX(綠色)熒光標記的PCR產物用超純水稀釋25倍,分別取等體積的上述2種稀釋液混合,吸取1 μL混合液、0.1 μL LIZ500分子量內標和8.9 μL去離子甲酰胺分別加入到DNA分析儀專用深孔板孔中;然后在PCR儀上95 ℃變性5 min,取出后立即置于碎冰上,冷卻15 min左右;瞬時離心10 s,在ABI3700 DNA遺傳分析儀上進行檢測。
1.5 數據處理
采用DNA Collection和Genemapper軟件進行原始數據收集和分析,建立相應數值數據庫。采用PopGenversion 1.32軟件(孫源文 等,2012)計算群體的等位變異數(observed number of alleles,Na)、有效等位變異數(effective number of alleles,Ne)、Shannon信息指數(I)。采用PowerMarker 3.25軟件計算標記位點的多態性信息量(polymorphism information content,PIC), 以 鄰 接 法(neighborjoining)計算參試材料間Nei’s遺傳距離,進行聚類分析并用FigTree軟件(Rambaut,2009)做樹狀聚類圖。標記系數(marker index,MI)按Senior等(1998)的公式MI=Allele×PIC計算,Allele為該引物的等位基因數。采用Structure 2.2軟件(Evanno et al.,2005)進行群體遺傳結構分析,Burnin Period和after Burnin各為10 000,K值為1~9,每個K值各運行15次。利用GenALEX 6軟件(Peakall &Smouse,2006)計算材料間的遺傳距離并進行主成分(PCA)分析。
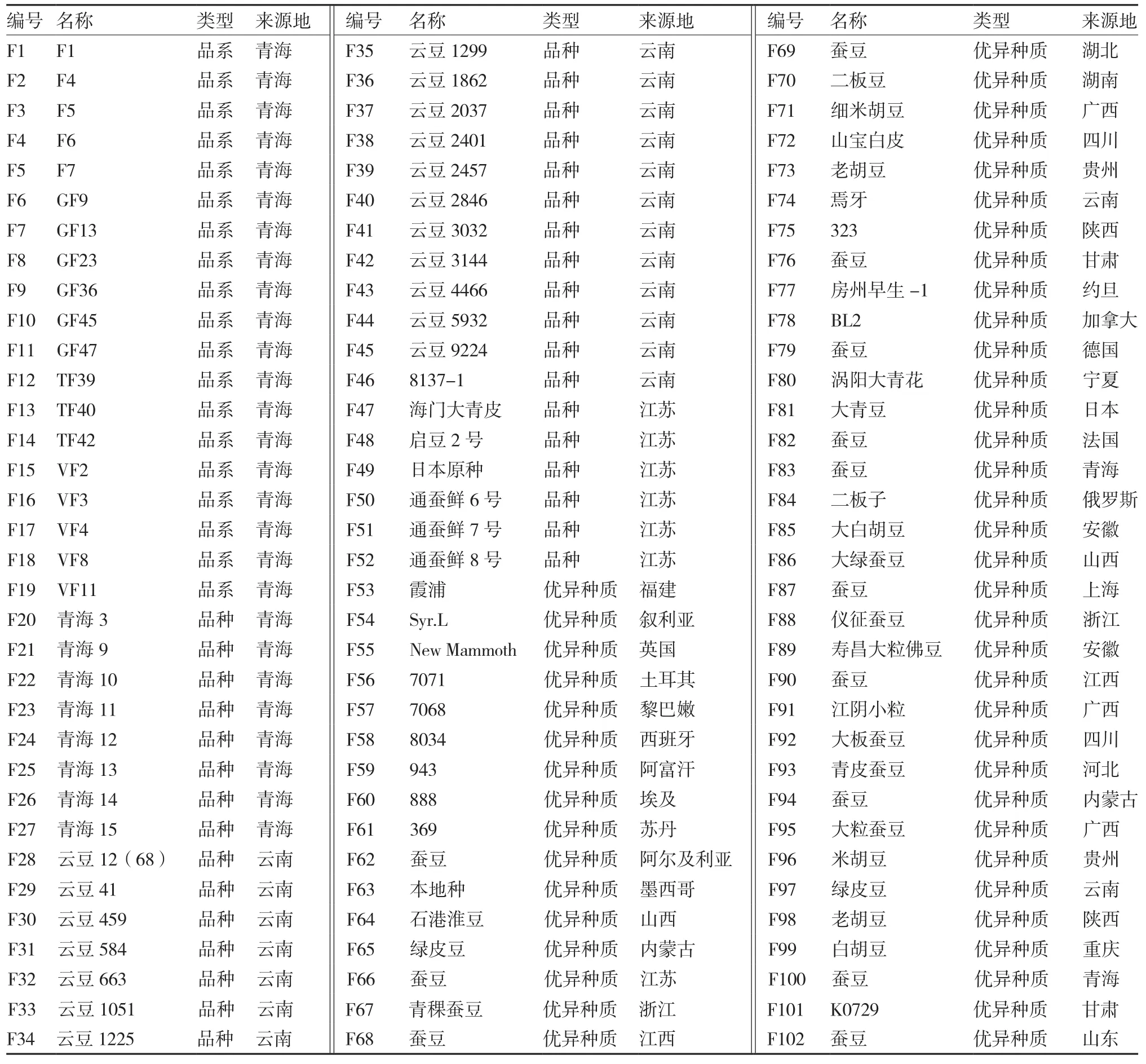
表1 參試蠶豆類型及來源地
2 結果與分析
2.1 遺傳多樣性分析
102 份國內蠶豆育成品種(品系)及優異種質在99個SSR位點(表2)共檢測出937個等位變異,每對引物平均檢測出4~19個等位變異,平均為9.46個。99個位點的多態性信息量PIC值變化范圍為0.38~0.88,平均為0.63;標記系數MI變化范圍為1.74~15.40,平均為6.19;有效等位變異變化范圍為1.79~9.22,平均為3.41;Shannon信息指數變化范圍為0.79~2.44,平均為1.45。其中,標記SSR12192的等位變異最多(19個),PIC值為0.78,MI值為14.77,有效等位變異為4.98,Shannon信息指數為2.01;SSR13513、SSR10910與EST2045的等位變異均為4個,它們的PIC值、MI值、有效等位變異與Shannon信息指數也相應較低。
表明等位基因數、多態性信息量PIC、MI值、有效等位基因數與Shannon信息指數整體呈現出一致的變化趨勢。
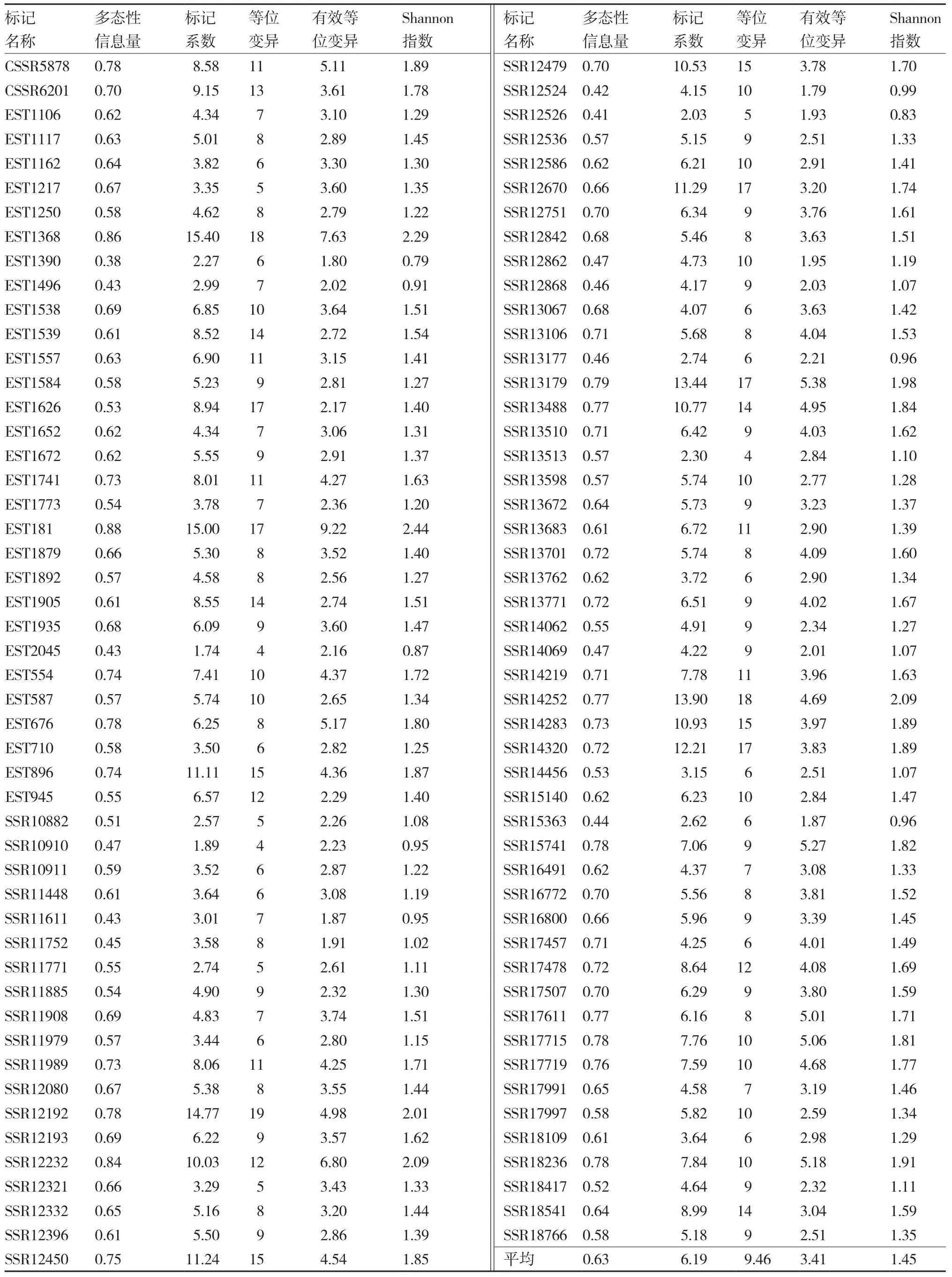
表2 99對SSR標記在102份蠶豆材料中的遺傳多樣性參數
2.2 不同種質間的遺傳關系
2.2.1 聚類分析 基于NJ聚類法,102份參試材料在遺傳距離為0.324時被分成兩大類(圖1)。第一大類全部為青海蠶豆品種(品系),表現為春播生態類型。第二大類為云南、江蘇育成品種和蠶豆優異種質資源,整體表現為秋播生態類型。來源于青海的品種(品系)與其他供試材料界限較為明顯,可能由于育成品種的骨干親本多以當地優異農家種為主,或者在獨特的地理環境下長期自然選擇的結果。來源于青海的兩份具有抗逆特性的蠶豆資源F83和F100與青海育成品種(品系)并沒有聚類在一起,可能是由于引種所致,這兩份資源是否為外來資源還需進一步驗證。國內外蠶豆優異種質資源與云南和江蘇蠶豆品種聚類在一起,這為我國秋播型蠶豆育種研究、科學選配親本及挖掘和利用優異資源提供了依據。
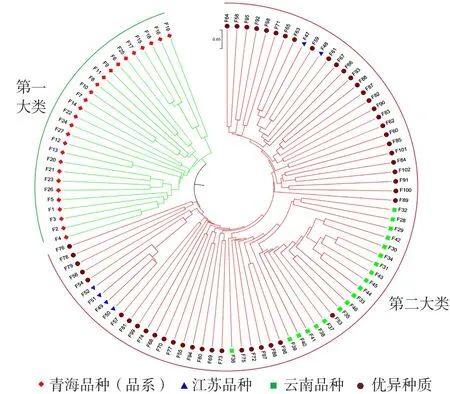
圖1 102份蠶豆材料的NJ聚類圖
由表3可以看出,優異蠶豆種質資源和不同省份蠶豆品種的等位變異不同,其中優異蠶豆資源組群最高,其次為青海組群和云南組群,江蘇組群最低。有效等位變異最高為優異蠶豆資源,其次為青海組群和云南組群,江蘇組群最低。Shannon信息指數變化趨勢與有效等位變異相同,優異蠶豆資源>青海組群>云南組群>江蘇組群。綜合各組群間的遺傳多樣性參數可知,優異蠶豆資源遺傳多樣性較高,其次為青海組群和云南組群,江蘇組群遺傳多樣性相對最低。
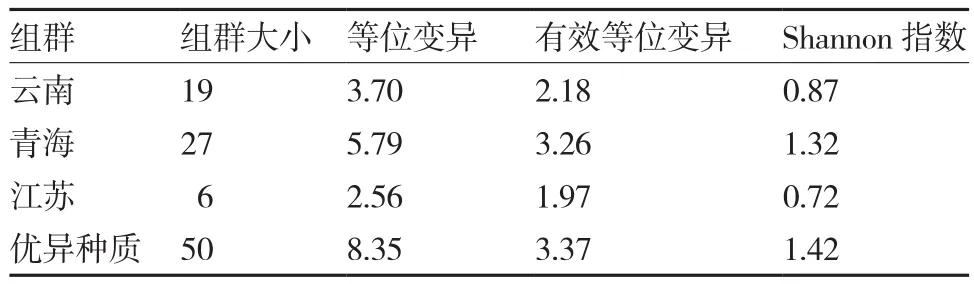
表3 優異蠶豆種質與不同省份蠶豆品種(品系)間遺傳多樣性參數
由表4可以看出,優異種質資源與云南品種和青海品種(品系)之間的親緣關系相對較近,與江蘇品種的親緣關系相對較遠,各蠶豆組群之間的親緣關系從某種程度上反映了優異種質資源在蠶豆主要產區的適應性。青海品種(品系)與江蘇品種、云南品種親緣關系相對較近,云南品種與江蘇品種之間親緣關系相對較遠。綜合來看,不同地理來源的蠶豆材料具有一定的地域生態性。
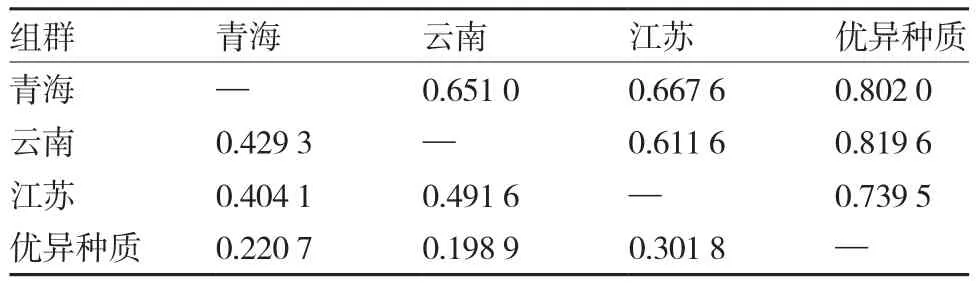
表4 優異蠶豆種質與不同省份蠶豆品種(品系)組群的遺傳相似系數(右上)與遺傳距離(左下)
2.2.2 群體結構分析 采用Structure 2.2軟件對102份參試材料進行群體結構分析,因后驗概率值〔lnP(D)〕隨亞群數的增大而增大,故采用基于ΔK的最大似然估計確定適宜亞群數。結果表明,102份參試材料在K=3時ΔK出現峰值,即參試材料從群體結構上可被分為3個亞群(圖2)。進一步研究Structure軟件分析得到的亞群和地理生態型間的關系發現,亞群Ⅰ主要包括青海全部品種(品系)和小部分優異種質資源;亞群Ⅱ主要包括云南大部分品種以及小部分優異種質資源;亞群Ⅲ主要包括江蘇全部品種、大部分優異種質資源以及小部分云南品種。可見,亞群分類結果與參試材料地理來源及生態類型存在一定程度的相關性。
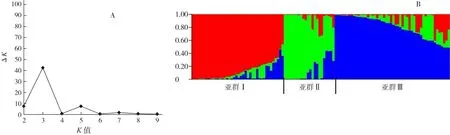
圖2 102份蠶豆材料的群體遺傳結構圖
2.2.3 主成分分析 主成分分析結果與Structure群體遺傳結構分析結果基本一致。102份蠶豆材料呈現出交叉分布的現象,在一定程度上說明其遺傳基礎較為廣泛(圖3)。第一大類主要以云南育成品種為主,中間混雜個別優異種質資源;第二大類全部為青海育成品種(品系);第三大類主要為大部分優異種質資源以及全部江蘇育成品種。
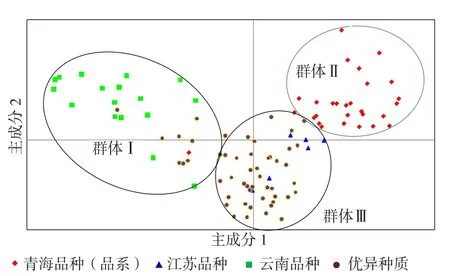
圖3 102份參試蠶豆材料的PCA分析
3 結論與討論
3.1 SSR熒光標記技術
SSR熒光標記檢測技術利用不同熒光基團染料(例如HEX、ROX、TET、FAM、Texas、Red)標記引物,通過全自動DNA分析測序儀精確分析PCR擴增產物的片段大小。傳統的銀染法經濟,但靈敏度較低、數據的有效整合性較差,同一膠板或不同膠板上樣品譜帶間相對位置難以有效識別,嚴重制約了數據的準確性。相對而言,SSR熒光標記檢測技術具有高通量、高靈敏度、高準確性、數據一致性高等優點,可實現不同板數樣品間數據的有效整合。目前,該項技術已廣泛應用于玉米(張全芳 等,2017)、枸杞(尹躍 等,2017)、水稻(王玲等,2015)、甘藍型油菜(周夢妍 等,2015)、高粱(王瑞 等,2015)等作物。郝晨陽等(2005)利用24對SSR標記擴增小麥基因組,通過銀染法檢測到235個等位變異,每對引物平均檢測到3~20個等位變異,平均為9.8個;基于SSR熒光標記技術檢測到312個等位變異,每對引物平均檢測到4~24個等位變異,平均為13.0個。通過分析兩種SSR分型方法,相對于銀染技術,熒光標記檢測技術可以檢測到更多的等位變異,檢測效果更為理想,靈敏度更高,更適于當前作物遺傳多樣性和指紋圖譜構建的分析與研究。
3.2 蠶豆遺傳多樣性研究進展
早期人們主要通過蠶豆的主要農藝性狀(生育期、種皮顏色、株高、籽粒大小、分枝數、有效莢數等)開展遺傳多樣性研究。康智明等(2015)對6份國內秋播型蠶豆品種和6份國外主栽品種進行農藝性狀和品質的遺傳多樣性分析,結果表明品種的親緣關系與地理來源關系不大。徐東旭等(2010)通過分析國內外不同地理來源的637份蠶豆種質資源的18個農藝性狀,推斷出參試資源由三大基因庫組成,國內春、冬性蠶豆種質資源間以及國內外資源間具有較大的遺傳變異。隨著分子標記技術的日益成熟,不同類型的分子標記被應用到蠶豆基礎性研究之中。本試驗利用99對熒光SSR標記分析102份中國代表性蠶豆品種(品系)和優異種質,聚類分析顯示春播型和秋播型蠶豆界限較為明顯,這與前人研究結果一致。優異蠶豆資源的遺傳多樣性明顯高于育成品種(品系),優異種質資源與云南品種和青海品種(品系)之間的親緣關系相對較近,從某種程度說明優異種質整體上可以適應兩地生態環境。在今后蠶豆育種過程中,建議育種家們擴大種質資源的篩選,充分挖掘和利用優異種質資源,提高育種效率。
3.3 種質資源創新是蠶豆育種的關鍵
優異的種質資源是培育優良作物品種的遺傳物質基礎,調查、收集和保存蠶豆種質資源顯得尤為重要。蠶豆作為世界上第六大類食用豆類作物,我國擁有豐富的蠶豆種質資源,同時也是全球蠶豆栽培面積和總產量最高的國家。當前我國蠶豆育種主要目標以產量為主,部分骨干親本的過度使用造成了蠶豆品種間的遺傳基礎狹窄。本試驗通過對102份蠶豆品種(品系)和優異種質資源進行遺傳多樣性分析,云南、青海和江蘇育成品種界限較為明顯,表明蠶豆具有生態適應性狹窄和極強的地域生態特點,這一結論與龐雯等(2002)的研究結果一致。加強地方蠶豆品種整理與鑒定、引種、系統選育以及雜交育種是當前蠶豆育種的主要方式。尚啟兵等(2003)通過引種和系統選育成功選育出蠶豆新品系Divine,該品系具有單寧含量低、高蛋白、多抗性以及適應性廣等特點,目前已經成為多個蠶豆新品種的育種親本。呂梅媛等(2016)通過雜交育種方式成功選育出適應性廣、產量較高、耐寒的蠶豆新品種云豆690。充分挖掘和利用蠶豆優異種質資源,合理選配雜交組合是蠶豆獲得高產穩產的主要途徑。
郝晨陽,王蘭芬,賈繼增,董玉琛,張學勇.2005.SSR熒光標記和銀染技術的比較分析.作物學報,31(2):144-149.
康智明,鄭開斌,徐曉俞,李愛萍.2015.不同蠶豆品種農藝及品質性狀的遺傳多樣性分析.福建農業學報,30(3):249-252.
呂梅媛,王麗萍,楊峰,代程,于海天,何玉華.2016.廣適、高蛋白優質蠶豆新品種“云豆690”選育.中國科技成果,17(18):17-19.
馬鈺.2012.蠶豆SSR標記的開發及遺傳連鎖圖譜的構建﹝碩士論文﹞.北京:中國農業科學院.
龐雯,楊示英,宗緒曉,蔡慶生,韋廣天.2002.廣西原產和外引蠶豆種質資源鑒定評價.植物遺傳資源學報,3(4):39-43.
尚啟兵,王志剛,李云霞,溫啟錄,張耀輝,宗緒曉.2003.蠶豆新品系Divine的篩選和利用.內蒙古農業科技,(4):21-22.
孫源文,陳鈺輝,劉富中,張映,連勇.2012.基于SSR分子標記的栽培種茄子遺傳多樣性分析.中國蔬菜,(22):17-23.
王海飛,關建平,孫雪蓮,馬鈺,宗緒曉.2011.世界蠶豆種質資源遺傳多樣性和相似性的ISSR分析.中國農業科學,44(5):1056-1062.
王玲,左示敏,張亞芳,陳宗祥,黃世文,潘學彪.2015.中國南方八省(自治區)水稻紋枯病菌群體遺傳結構的SSR分析.中國農業科學,48(13):2538-2548.
王瑞,張福耀,程慶軍,田承華,凌亮.2015.利用SSR熒光標記構建20個高粱品種指紋圖譜.作物學報,41(4):658-665.
王瑞云,季煦,陸平,劉敏軒,許月,王綸,王海崗,喬治軍.2017.利用熒光SSR分析中國糜子遺傳多樣性.作物學報,43(4):530-548.
王偉,楊文鵬,張文龍.2009.貴州48個玉米雜交種及其親本SSR指紋圖譜的構建與分析.貴州農業科學,37(11):1-8.
徐東旭,姜翠棉,宗緒曉.2010.蠶豆種質資源形態標記遺傳多樣性分析.植物遺傳資源學報,11(4):399-406.
尹躍,安巍,趙建華,李彥龍,樊云芳,曹有龍.2017.枸杞品種SSR熒光指紋圖譜構建及遺傳關系分析.西北林學院學報,32(1):137-141.
雍洪軍,張世煌,張德貴,李明順,李新海,郝轉芳,劉曉鑫,白麗,謝傳曉.2009.利用SSR熒光標記分析90個糯玉米地方品種的遺傳多樣性.玉米科學,17(1):6-12.
張全芳,梁水美,李燕,劉艷艷,范陽陽,郭慶法,魯守平,步迅.2017.基于熒光SSR標記的玉米自交系遺傳結構解析.植物遺傳資源學報,18(1):19-31.
鄭卓杰.1997.中國食用豆類學.北京:中國農業出版社:93-140.
周夢妍,吳金鋒,許鯤,李鋒,陳碧云,伍曉明.2015.甘藍型油菜核心種質和新品種(系)的SSR等位變異分析.分子植物育種,(6):1248-1258.
Evanno G,Regnaut S,Goudet J.2005.Detecting the number of clusters of individuals using the software structure:a simulation study.Molecular Ecology,14(8):2611-2620.
FAO.2017.FAO Statistical Database.http://www.fao.org.
Fernandez M,Figueiras A,Benito C.2002.The use of ISSR and RAPD markers for detecting DNA polymorphism,genotype identification and genetic diversity among barley cultivars with known origin.Theoretical and Applied Genetics,104(5):845-851.
Gentile F,Arcaro A,Pizzimenti S,Daga M,Cetrangolo G P,Dianzani C,Lepore A,Graf M,Ames P,Barrera G.2017.DNA damage by lipid peroxidation productions:implications in cancer,inflammation and autoimmunity.AIMS Genetics,4(2):103-137.
Gong Y M,Xu S C,Mao W H,Li Z Y,Hu Q Z,Zhang G W,Ding J.2011.Genetic diversity analysis of faba bean(Vicia fabaL.)based on EST-SSR markers.Agricultural Sciences in China,10(6):838-844.
Link W,Dixkens C,Singh M,Schwall M,Melchinger A E.1995.Genetic diversity in European and Mediterranean faba bean germplasm revealed by RAPD markers.Theoretical and Applied Genetics,90(1):27-32.
Peakall R,Smouse P E.2006.Genalex 6:genetic analysis in Excel.Population genetic software for teaching and research.Molecular Ecology Notes,6(1):288-295.
Polignano G B,Alba E,Uggenti P,Scippa G.1999.Geographical patterns of variation in Bari faba bean germplasm collection.Genetic Resources and Crop Evolution,46(2):183-192.
Rambaut A.2009.FigTree.v1.3.1.Available:http://tree.bio.ed.ac.uk/software.
Robertson L D,El-Sherbeeny M.1991.Distribution of discretely scored descriptors in a pure line faba bean(Vicia fabaL.)germplasm collection.Euphytica,57(1):83-92.
Senior M L,Murphy J P,Goodman M M,Stuber C W.1998.Utility of SSR for de-termining genetic similarities and relationships in maize using an agarose gel system.Crop Science,(38):1088-1098.
Sokol E,Nijenhuis M,Sjollema K A,Jonkman M F,Pas H H,Giepmans B N G.2017.Particle bombardment of ex vivo skin to deliver DNA and express proteins.Methods in Molecular Biology,1559:107-118.
Terzopoulos P J,Bebeli P J.2008.Genetic diversity analysis of Mediterranean faba bean(Vicia fabaL.)with ISSR markers.Field Crops Research,108(1):39-44.
Wang F,Yang T,Burlyaeva M,Li L,Jiang J,Fang L,Redden R,Zong X.2015.Genetic diversity of grasspea and its relative species revealed by SSR markers.PLoS One,10(3):34-38.
Yang T,Fang L,Zhang X Y,Hu J G,Bao S Y,Hao J J,Li L,He Y H,Jiang J Y,Wang F,Tian S F,Zong X X.2015.Highthroughput development of SSR markers from pea(Pisum sativumL.)based on next generation sequencing of a purified Chinese commercial variety.PLoS One,10:103-107.
Zeid M,Sch?n C C,Link W.2004.Hybrid performance and AFLP-based genetic similarity in faba bean.Euphytica,139(3):207-216.
Zong X,Liu X,Guan J,Wang S,Liu Q,Paull J G,Redden R.2009.Molecular variation among Chinese and global winter faba bean germplasm.Theoretical and Applied Genetics,118(5):971-978.

