一種基于基因拓撲重要性的通路識別方法
方宏源, 昝鄉鎮,沈良忠,劉文斌*
(1.溫州大學 物理與電子信息工程學院,浙江 溫州 325035;2.溫州商學院 信息工程學院,浙江 溫州 325035)
基于微陣列的高通量技術產生了大量的基因表達數據,如何從這些海量基因表達數據中獲得洞察性的認識,進而理解生命現象的機制仍然是擺在世界各國科學家面前的一個嚴峻的挑戰。生物通路是一組完成特定功能的基因之間的相互作用關系,主要有信號傳導通路和代謝通路。在信號傳導通路中,節點代表基因(或基因產物),邊代表從一個基因轉導到另一個基因的信號。在代謝通路中,節點代表生化化合物,邊代表通過酶編碼的化合物之間的生物化學反應,酶是為基因編碼的。常用的通路數據庫有KEGG[1]和Reactome[2]數據庫,它們提供了基因之間相互作用的可視化形式。在過去十多年中,研究者開發了很多基于通路的基因表達差異分析方法,來識別各種癌癥或疾病相關的通路。
2005年,PNAS上發表了兩篇重要的通路分析方法的論文,一個是Tian等[3]提出的基于功能的顯著通路分析方法,這種方法綜合考慮了一個基因集合中基因表達與集合外基因表達差異的顯著性(行置換),以及該基因集基因表達與表型相關性的的顯著性(列置換)。另一個是Subramanian等[4]提出著名的基因集富集分析方法GSEA方法,其主要思想是根據通路中基因表達情況與給定表型之間的相關性對所有基因進行排序,然后確定給定通路P的Kolmogorov-Smirnov統計量在排序列表中靠近極端處程度的得分。該方法中,Kolmogorov-Smirnov統計量的顯著性根據樣本的列置換確定。2006年,Zahn等[5]使用Van der Waerden統計量代替Kolmogorov-Smirnov統計量并用自舉抽樣代替置換檢驗方法該方法考慮了通路中兩個基因表達水平的相關性以及與其他因素的相關性。同年,EFRON等[6]用最大-均值統計量替代Kolmogorov-Smirnov統計量來計算通路分數,然后通過行置換方法對該分數進行標準化,最后利用列置換來檢驗通路分值的顯著性,這就是著名的GSA方法。
從系統生物學的角度,基因之間的相互作用及其動力學的變化是導致各種疾病及癌癥發生的主要原因[7-12]。因此,癌癥相關通路的識別應盡可能考慮到通路中包含基因的各種信息,如基因的上下游位置、調控基因的數量、基因之間的作用關系等等因素。2009年,Tarca等[13]考慮了通路中基因的上下游位置關系提出了著名的信號通路影響分析(SPIA)方法。同年,Thomas等[14]提出了一種考慮通路中基因拓撲結構的方法,主要思想是位于上游和下游的基因比上下游中間位置具有更高權重,并且在打分上使得緊密連接的基因比不緊密連接的基因具有更高的分數。在通路中,有些基因頻繁出現在很多通路中,這些基因可以看作是非特異性基因,其變化對特定通路的影響相對較小;反過來,另外一些基因僅在特定通路出現,即其特異性很高,這些基因的變化對該通路的影響往往很大。2012年,Tarca 等[15]在GSA方法的基礎上加入了基因特異性的影響,提出重疊基因降權的通路分析方法(PADOG)。最近,Liu等[14]提出了稱為基因相互作用富集和網絡分析(GIENA)的方法,以表示協同、競爭、冗余,表達水平的依賴性的失調的基因相互作用。
由KEGG中的Ras信號傳導通路,可看出其中的Ras基因調節該通路中的許多下游基因。由于Ras基因參與控制細胞分裂和細胞死亡的許多信號傳導通路,已有研究表明該基因的過表達和突變與許多癌癥相關,如胰腺、結腸、肺(30%)、甲狀腺、膀胱、卵巢、乳腺、皮膚、肝臟、腎臟和一些白血病等。顯然在通路中,調控大量基因的基因應該比僅調控少量基因的基因更為重要,它們的差異對通路的功能應該具有更大的影響。考慮這一現象,本文將基因平均出度的大小定義為基因的重要性,并和PADOG方法中的基因的特異性結合起來,提出了一種基于重要性和特異性的通路識別方法PAGIS。在結腸癌、肺癌和胰腺癌3個數據集上的結果表明,改進后的方法能夠提高癌癥相關通路的識別精度。
1 材料與方法
1.1 數據集
本文主要分析了3個癌癥數據集。
1)結腸癌數據集GSE4107,該數據集包括12個結腸癌樣本與10個正常樣本(Affymetrix HG-U133 Plus 2.0微陣列平臺)。
2)肺癌數據集GSE27262,該數據集包括25個肺癌樣本和25個正常樣本(Affymetrix Human Genome U133 Plus 2.0微陣列平臺)。
3)胰腺癌數據集GSE16515,包括36個胰腺癌樣本和16個正常樣本。
1.2 頻度和平均出度的分布
如圖1所示是KEGG數據庫中204個信號通路的基因的頻度和出度分布圖,其中圖1(a)是基因的平均頻度分布,可以看出大多數基因僅出現在一兩條通路中,只有少數基因出現在多條通路中。圖1(b)是基因的平均出度分布,可以看出僅有少數基因調控大量下游基因,而大多數基因的平均出度在0~5之間。圖1(c)是基因的頻度和平均出度的散點圖,可以看出僅有部分平均出度大且頻度低的基因。本文把平均出度在前100名的基因在DAVID數據庫中進行GO功能注釋,結果發現顯著富集在一些癌癥相關通路中,如pathways in cancer, adipocytokine signaling pathway, neurotrophin signaling pathway, thyroid cancer, ErbB signaling pathway, PPAR signaling pathway,和renal cell carcinoma。這說明這些平均出度大的基因與癌癥的發生發展具有密切的關系,提高它們在癌癥相關通路中的權重具有生物學意義。
基因在通路中出現的頻度實際上反映了一個基因的特異性,頻繁出現在很多通路中的基因屬于一些“公共基因”,它們對通路的影響相對較小;僅在一條或幾條通路中出現的基因其特異性高,它們的差異表達對通路的影響基因就大。在PADOG方法中,文獻[15]定義基因的特異性權重為

式中:max(f)、min(f)分別為204條KEGG通路中最大頻度和最小頻度;wf(g)反映基因在通路中特異程度,該值越大則基因在通路中特異程度越高,反之則特異程度越低,wf(g)取值在1~2之間。
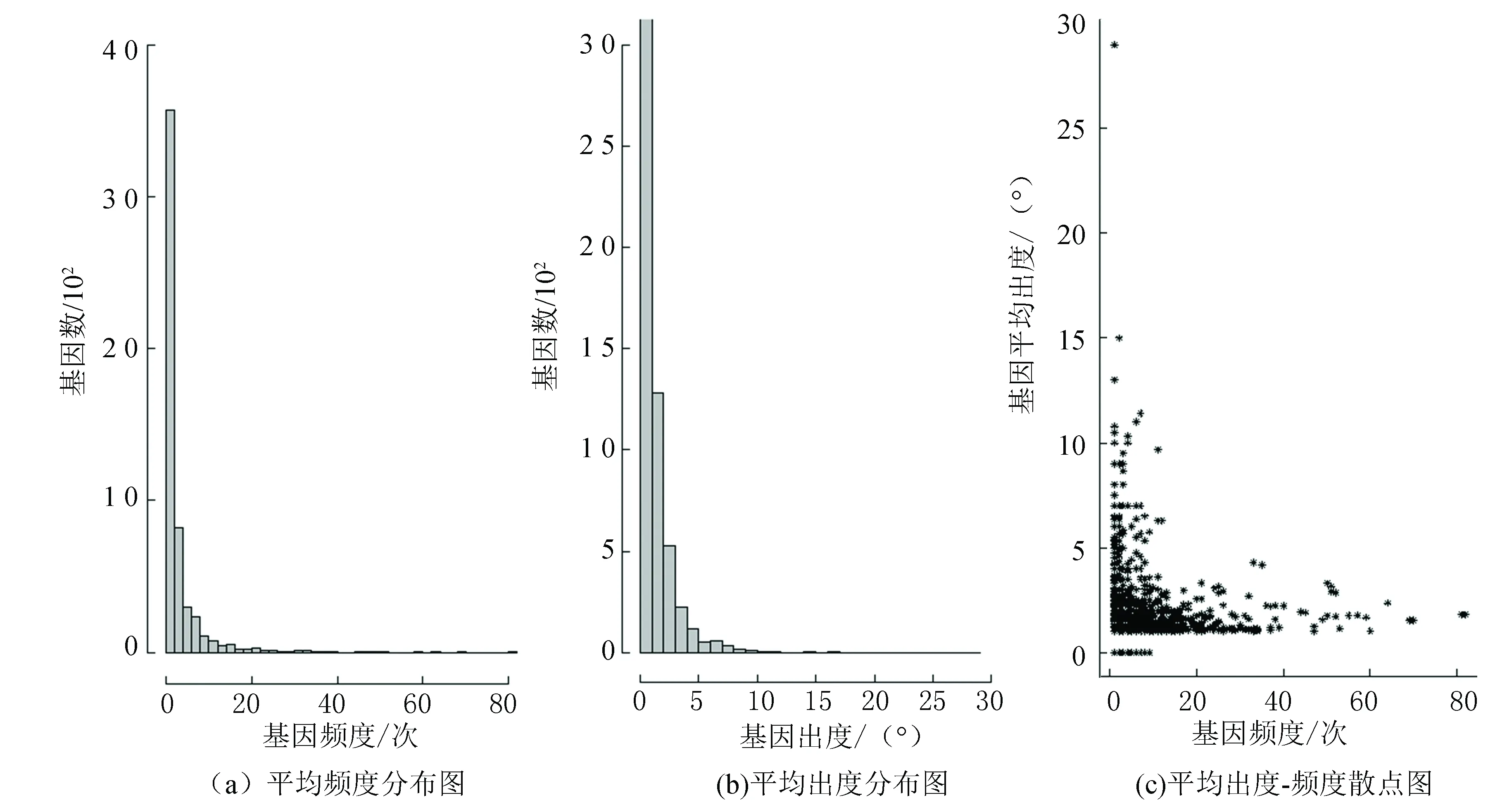
圖1 204條KEGG通路基因平均出度-頻度分布圖Fig.1 Distribution of the average gene out-degrees and frequencies across the 204 KEGG signaling pathways
由于基因出度表示的是一個基因調控的下游基因的數量,因此,出度越大的基因,對通路的影響就越大。為此,本文定義基因重要性的權重為
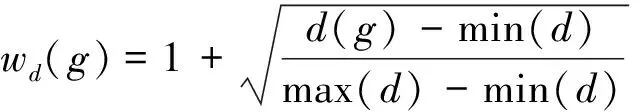
式中:max(d)、min(d)分別為204條KEGG通路中基因最大平均出度和最小平均出度;wd(g)反映基因在通路中的重要性,該值越大則基因在通路中重要程度越高,值越小則基因在通路中重要程度越低,取值也在1~2之間。
1.3 癌癥相關通路分析方法
本文簡要介紹GSEA方法、GSA方法、PADOG方法,進而引出本文的改進方法。假定所有基因總數為N, 給定一個通路S,通路中基因數為M,GSEA的主要過程如下。
Step1按照每個基因g與表型間相關性r(或t統計量)對N個基因排序wd(g)L=[g1,...,gj,...gN]。
Step2用帶權值的Kolmogorov-Smirnov統計量計算通路的富集分數ES0(S)為
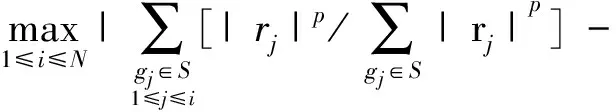
式中p為用來校正ES的權值,p一般取1。
Step3隨機置換樣本標簽Nite次,并重新計算通路S的分數ESite(S)。
Step4計算該通路富集分數ES0(S)的顯著性p-value。
在GSA方法中,文獻[6]使用“最大均值”統計量代替Kolmogorov-Smirnov統計量來計算通路分數ES。公式如下:
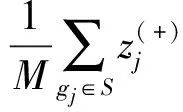
1.4 基于重疊基因降權通路分析方法(PADOG)
使用通路中所有基因的加權絕對矯正t分數和的均值來計算通路S分數ES0(S),公式如下:
式中:Τ(gj)為基因gj在兩類樣本中矯正t分數;wf(gj)為基因gj的權重。
利用行隨機化和置換排列方法計算通路顯著性p-value。公式如下:
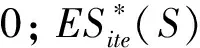
1.5 基于基因重要性和特異性的通路分析方法(PAGIS)
為將基因的平均出度引入到PADOG方法框架中,本文合并權重wf(g)和wd(g)成w(g),公式如下:
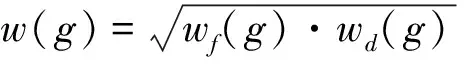
式中:wf(g)為基因頻度的權重;wd(g)為基因平均出度的權重;w(g)為合并權重且值取1~2;w(g)反映基因在通路中的重要性和特異性的程度,基因在通路中重要程度和特異程度越高則該值越大,相反基因的重要程度或特異程度越低則該值越小。本文將w(g)作為PADOG計算通路分數的新權重并提出PAGIS方法。
2 結果與分析
本文比較PADOG和PAGIS方法在3個癌癥數據集上的結果,PADOG的R語言包由文獻[15]開發。由于不同方法p值計算有所不同,僅僅比較p值不夠合理。本文基于通路的p值升序排列并比較排名,通路排名越靠前則該通路傾向被認為與癌癥顯著相關。表1~3列出PADOG和PAGIS方法在前30名中與癌癥相關的通路排名。在3個癌癥數據集中,PADOG和PAGIS共識別出21、23、15條癌癥相關通路。

表1 PAGIS和PADOG方法在結腸癌數據集中前30名癌癥相關通路和排名Table 1 The rank of top 30 cancer-related pathway in colorectal cancer
圖2(a)~(c)分別是PADOG和PAGIS方法在結腸癌、肺癌和胰腺癌數據集中癌癥相關通路的排名折線圖。該圖中橫軸對應表1~3中Pathway No字段,縱軸對應表1~3中PADOG和PAGIS方法中的通路排名。由圖3可看出,相比PADOG方法PAGIS能夠顯著提高某些癌癥相關通路的排名。如圖2(a)所示,通路Metabolic pathways, Pathways in cancer和Ubiquitin mediated proteolysis在PADOG方法中排名是82、79和114,而PAGIS是1、11和22;在肺癌數據集(圖2(b))中ECM-receptor interaction和Metabolic pathways在PADOG方法中排名分別是53、195,而PAGIS是20、29;在胰腺癌數據集中(圖2(c))中通路ECM-receptor interaction,Cell cycle和Regulation of actin cytoskeleton,在PADOG方法中排名分別是25、35和31,而PAGIS是5、15和16。
表1~3列出PADOG和PAGIS方法在3個癌癥數據集中識別出癌癥相關通路的平均排名,PADOG方法識別出癌癥相關通路的平均排名分別為30.14、29.43和15.87,而PAGIS分別為17.62、16.91和14.13,排名值越小越靠近排名列表的頂端位置,意味著總體與癌癥相關程度越高;排名值越大越靠近排名列表的底端位置,意味著總體相關程度越低。顯然在3個癌癥數據集中PAGIS方法識別出的癌癥相關通路平均排名位置比PADOG方法更靠近頂端位置。

表2 PAGIS和PADOG方法在肺癌數據集中前30名癌癥相關通路和排名Table 2 The rank of top 30 cancer-related pathway in lung cancer

表3 PAGIS和PADOG方法在胰腺癌數據集中前30名癌癥相關通路和排名Table 3 The rank of top 30 cancer-related pathway in pancreatic cancer
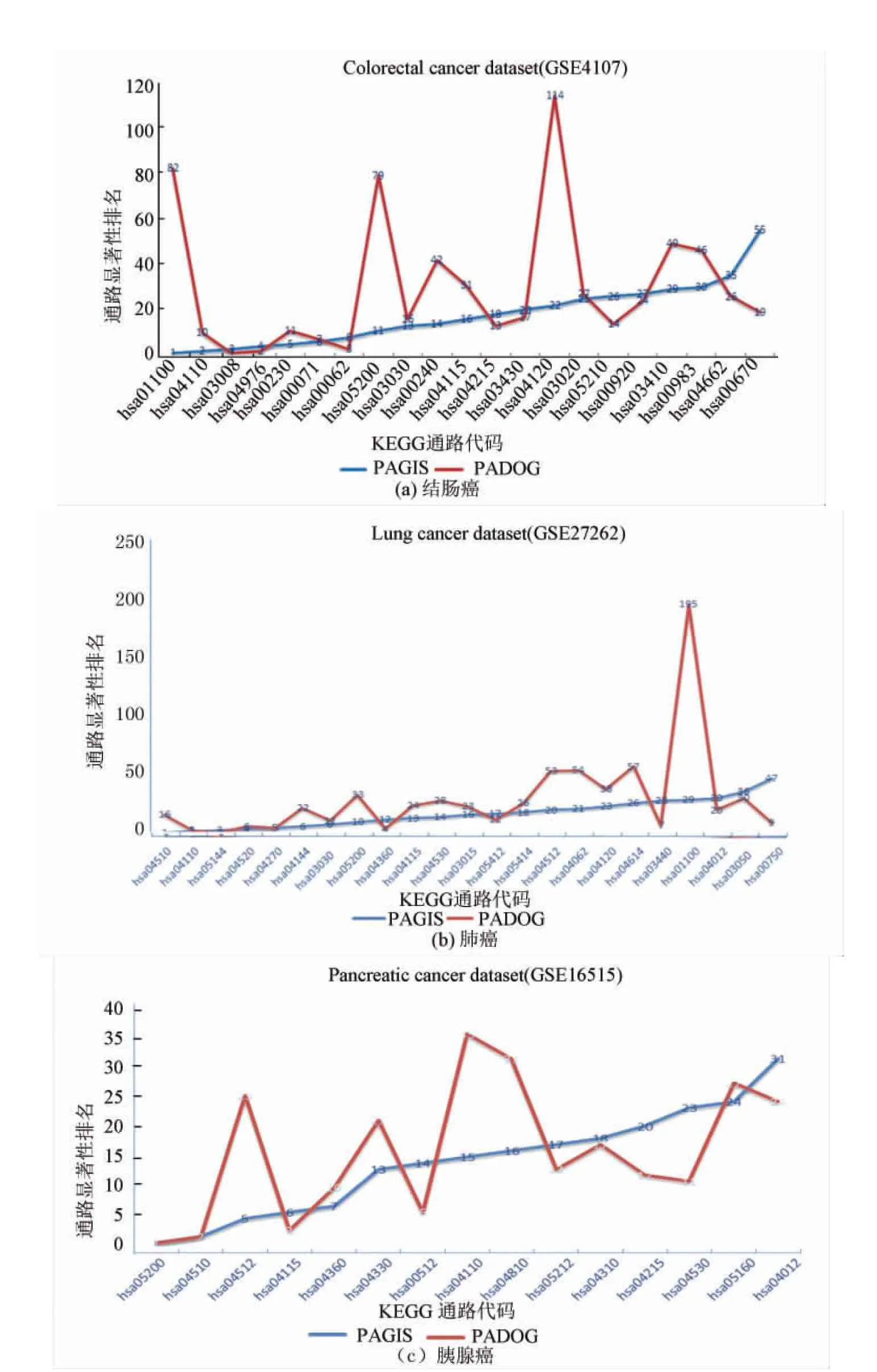
圖2 PADOG和PAGIS方法在3個數據集中癌癥相關通路排名折線圖
Fig.2 Ranks of the cancer related pathways by PAGIS and PADOG in the three cancer datasets
另一方面如圖2中虛線所示,在結腸癌數據集中,排名在30名后的通路PADOG方法有7條,而PAGIS僅有2條;在肺癌數據集中PADOG方法有6條,PAGIS僅有2條;在胰腺癌數據集中PADOG方法有2條而PAGIS有1條。顯然PAGIS方法能識別出更多的癌癥相關通路。為進一步比較PADOG和PAGIS方法在3個癌癥數據集中的性能,本文分別列出PADOG和PAGIS方法在前10、20和30名中識別出的癌癥相關通路的數目,見表4。表中在結腸癌數據集中前10名與癌癥相關的通路PAGIS方法識別出7條,PADOG識別出5條,前20名中PAGIS方法識別出13條而PADOG識別出11條,前30名中PAGIS方法識別出19條而PADOG識別出14條。其他兩個數據集的結果和結腸癌數據集類似,這說明在各段排名中PAGIS方法能穩定的識別出比PADOG更多的癌癥相關通路,PAGIS具有比PADOG更好的性能優勢。
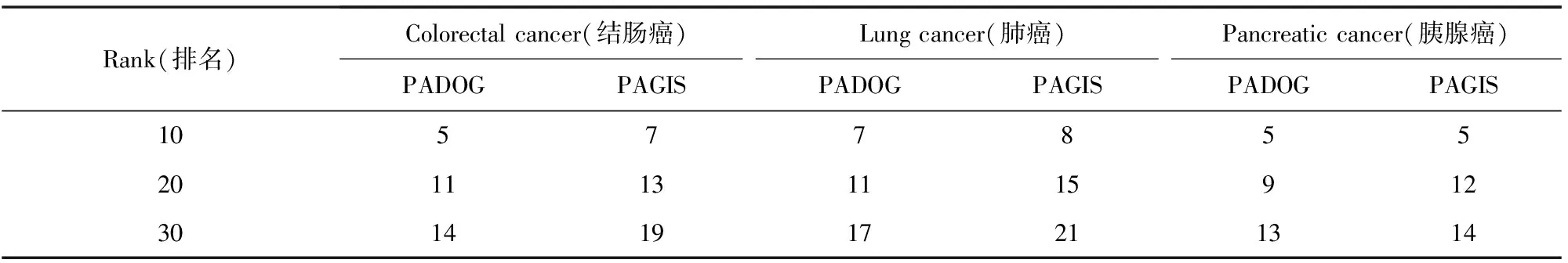
表4 PADOG和PAGIS方法在前10、20、30名中識別癌癥相關通路數目Table 4 Numbers of cancer-related pathway in top 10, 20, 30 identified by PADOG and PAGIS
3 結 論
1)本文統計了KEGG數據庫中204條信號通路中基因的頻度和出度,并計算出每個基因的平均出度。
2)在基因特異性加權的通路分析方法(PADOG)基礎上引入基因的平均出度,并用平均出度表示基因在通路中的重要程度。
3)合并基因特異性和重要性的權值,提出一種基于基因拓撲重要性的通路識別方法(PAGIS),并將該方法應用在結腸癌、肺癌和胰腺癌數據集中。
4)總體上PAGIS方法比PADOG方法識別出更多的癌癥相關通路,能穩定提高癌癥相關通路的識別率。
References)
[1]KANEHISA M, FURUMICHI M, TANABE M, et al. KEGG: new perspectives on genomes, pathways, diseases and drugs[J]. Nucleic Acids Research, 2017, 45(D1): D353-D361. DOI: 10.1093/nar/gkw1092.
[2] FABREGAT A, SIDIROPOULOS K, VITERI G, et al. Reactome pathway analysis: a high-performance in-memory approach[J]. BMC Bioinformatics, 2017, 18(1): 142. DOI: 10.1186/s12859-017-1559-2.
[3]TIAN Lu, GREENBERG S A, KONG S W,et al. Discovering statistically significant pathways in expression profiling studies[J].Proceedings of the National Academy of Sciences of the United States of America,2005,102(38), 13544-13549.DOI:10.1073/Pnas.0506577102.
[4]SUBRAMANIAN A, TAMAYO P, MOOTHA V K, et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles[J]. Proceedings of the National Academy of Sciences, 2005, 102(43):15545-15550. DOI:10.1073/pnas.0506580102.
[5]ZAHN J M,SONU R VOGEL H,et al. transcriptional profiling of aging in human muscle reveals a common aging signature[J]. PLoS Genetics, 2016,2(7):e115.DOI:10.1371/journal.pgen.0020115.
[6]EFRON B B, TIBSHIRANI R. On testing the significance of sets of genes[J].The Annals of Applied Statistics, 2007, 1(1): 107-129. DOI: 10.1214/07-AOAS101.
[7]KHATRI P, SIROTA M, BUTTE A J. Ten years of pathway analysis: current approaches and outstanding challenges-supplementary notes[J]. Plos Computational Biology, 2012, 8(2):e1002375. DOI: 10.1371/journal.pcbi.1002375.
[8]TURNBULL C, SEAL S, RENWICK A, et al. Gene-gene interactions in breast cancer susceptibility[J]. Human Molecular Genetics, 2012, 21(4):958-962. DOI: 10.1093/hmg/ddr525.
[9]JEONG H H, LEEM S, WEE K, et al. Integrative network analysis for survival-associated gene-gene interactions across multiple genomic profiles in ovarian cancer[J]. Journal of Ovarian Research, 2015, 8(1):42.DOI: 10.1186/s13048-015-0171-1.
[10]ZHANG Jigang, LI Jian, DENG Hongwen. Identifying gene interaction enrichment for gene expression data[J]. Plos One, 2009, 4(11):e8064. DOI: https://doi.org/10.1371/journal.pone.0008064.
[12]DUTTA B, WALLQVIST A, REIFMAN J. PathNet: a tool for pathway analysis using topological information[J]. Source Code for Biology and Medicine, 2012, 7(1):10. DOI: 10.1186/1751-0473-7-10.
[13]TARCA A L,DRAGHICI S,KHATRI P,et al. A novel signaling pathway impact analysis[J]. Bioinformatics, 2009,25(1): 75-82.DOI:10.1093/bioinformatics/BTN577.
[14]THOMAS R, GOHLKE J M, STOPPER G F, et al. Choosing the right path: enhancement of biologically relevant sets of genes or proteins using pathway structure[J]. Genome Biology, 2009, 10(4):R44. DOI: 10.1186/gb-2009-10-4-r44.
[15]TARCA A L, DRAGHICI S, BHATTI G, et al. Down-weighting overlapping genes improves gene set analysis[J]. BMC Bioinformatics, 2012, 13(1):136. DOI: 10.1186/1471-2105-13-136.
[16]LIU Yu, KOYUTüRK M, BARNHOLTZ-SLOAN J S, et al. Gene interaction enrichment and network analysis to identify dysregulated pathways and their interactions in complex diseases[J]. BMC Systems Biology, 2012, 6(1):65. DOI: 10.1186/1752-0509-6-65.

