基于ITS序列位點特異性PCR鑒別桃仁與苦杏仁
高琳惠 尹 艷 李 佳 馬玥萌 李文斌 李博文 張夏楠
(1 首都醫科大學中醫藥學院,北京,100069; 2 貴州大學,貴陽,550025)
基于ITS序列位點特異性PCR鑒別桃仁與苦杏仁
高琳惠1尹 艷2李 佳1馬玥萌1李文斌1李博文1張夏楠1
(1 首都醫科大學中醫藥學院,北京,100069; 2 貴州大學,貴陽,550025)
目的:對桃仁和苦杏仁進行分子鑒定研究,建立基于ITS序列位點特異性的PCR鑒定方法。方法:收集桃仁、苦杏仁樣品,提取基因組總DNA,用ITS通用引物進行測序,通過BioEdit軟件進行序列比對,根據特異位點設計鑒別引物進行特異性PCR反應,并對PCR反應條件進行了優化。結果:基于ITS序列設計的特異性鑒別引物G4-7可以用于桃仁和苦杏仁的鑒別研究,在位點特異性PCR反應條件考察中,25 μL反應體系DNA模板用量適宜范圍為0.05~1 ng,退火溫度在59 ℃時特異性最佳,實驗建立的方法對不同DNA聚合酶和PCR儀均具有普適性。結論:該研究設計的位點特異性引物在一定條件下的PCR反應中,桃仁能夠擴增出333 bp清晰條帶,而苦杏仁沒有該條帶,從而實現了桃仁與苦杏仁的快速、準確鑒別。
桃仁;苦杏仁;ITS;位點特異性PCR;分子鑒定
桃仁為薔薇科植物桃Prunuspersica(L.)Batsch或山桃P.davidiana(Carr.)Franch.的干燥成熟種子,性平,味苦、甘,歸心、肝、大腸經,具有活血祛瘀,潤腸通便,止咳平喘之功效,用于經閉痛經,癥瘕痞塊,肺癰腸癰,跌撲損傷,腸燥便秘,咳嗽氣喘[1]。苦杏仁為薔薇科同屬植物山杏P.armeniacaL.var.ansu Maxim.、西伯利亞杏P.sibiricaL.、東北杏P.mandshurica(Maxim.)Koehne或杏P.armeniacaL.的干燥成熟種子,性微溫、味苦,歸肺、大腸經,具有降氣止咳平喘,潤腸通便之功效,用于咳嗽氣喘,胸滿痰多,腸燥便秘[1]。桃仁與苦杏仁在外觀上非常相似,經脫皮、炮制加工后更難區分,然而兩者藥理作用顯著不同,桃仁屬于活血化瘀藥,杏仁屬于止咳平喘藥。且兩者主要化學成分均為苦杏仁苷,應用傳統鑒別方法容易出現誤差。此外,苦杏仁中苦杏仁苷含量較高,該化合物被苦杏仁酶代謝分解后會產生有毒的氫氰酸,過量則會引起中毒,桃仁和苦杏仁的混用容易引起潛在的用藥風險[2]。因此,建立一種快速、簡易、準確鑒定桃仁和苦杏仁的分子鑒定體系,對于控制桃仁的藥材質量以及用藥安全都具有十分重要的意義。本實驗基于ITS序列位點特異性PCR技術對桃仁和苦杏仁藥材樣品進行鑒別,設計特異性鑒別引物并對特異性PCR反應條件進行了摸索和優化,實驗確定的鑒別方法穩定、快速,為桃仁和苦杏仁的準確鑒別提供依據。
1材料與方法
1.1 樣品 桃仁與苦杏仁樣品來源有:購自河北安國中藥材市場和安徽亳州藥材市場的藥材,由首都醫科大學中醫藥學院李佳副教授鑒定(編號:1,2,10,11,12,19);采集自山西、河北、北京的桃仁、山桃仁、苦杏仁樣品由北京中醫藥大學劉春生教授鑒定(編號:8,9,18,24,25,26);全國中藥資源普查中心提供的桃仁與苦杏仁樣品(編號:3,4,5,6,7,13,14,15,16,17,20,21,22,23),樣品信息見表1。桃仁標準藥材(山桃,121560-201302,編號T)、苦杏仁標準藥材(杏,121554-201204,編號X)購于中國食品藥品檢定研究院。NCBI下載序列見表2。
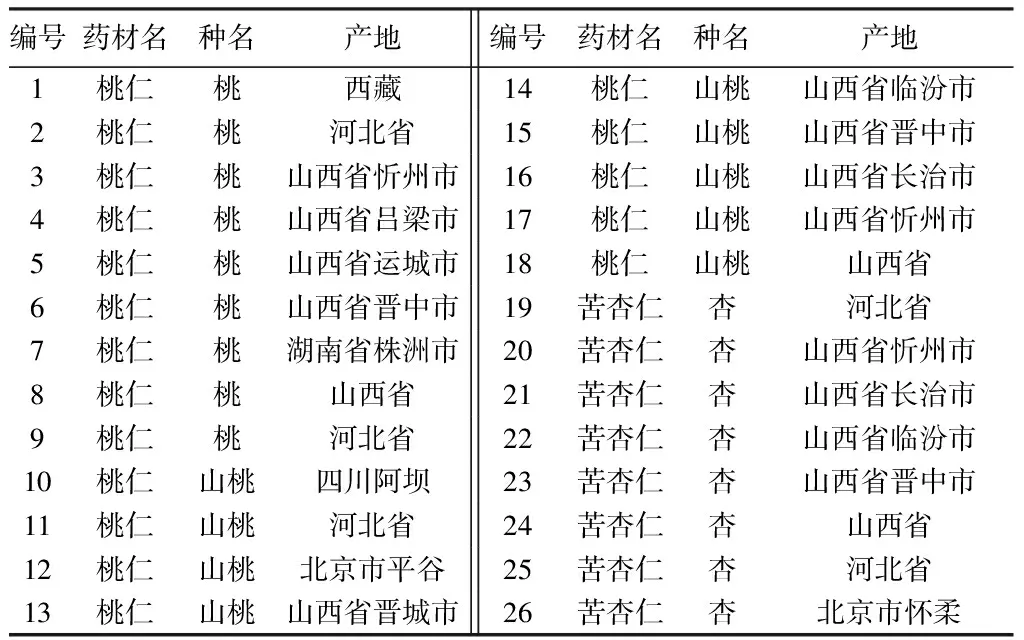
表1 本實驗所用樣品材料
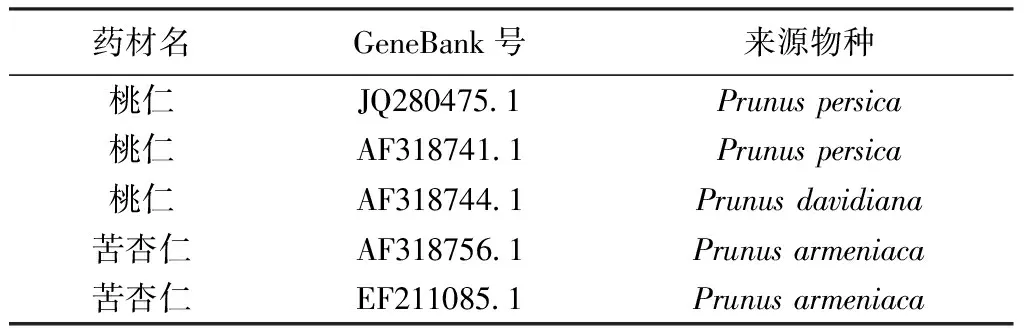
表2 NCBI下載序列信息
1.2 儀器 PCR儀(Applied Biosystems Veriti;Eppendorf AG 22331);微量核酸定量儀(NanoDrop 2000C);高級熒光化學發光系統(FUSION-SL)。
1.3 試劑 植物基因組DNA提取試劑盒(離心柱型DP305-2,TIANGEN);2×EasyTaq PCR SuperMix(AS111,TRANS);2×Taq PCR StarMix(A112,GenStar);Ex Taq DNA聚合酶(RR53AM,TakaRa);Phusion超保真PCR Master Mix(M0531,BioLab);2K DNA Marker(E110,TransGen)。
1.4 方法
1.4.1 基因組DNA的提取、通用引物PCR擴增及測序 取樣品約100 mg,經粉碎后,用植物基因組DNA提取試劑盒提取各藥材總DNA,最后用50 μL無菌水洗脫。樣品用通用引物ITS[3](序列見表1)以高保真酶擴增樣品DNA并進行雙向測序。PCR擴增體系為Phusion超保真PCR Master Mix 12.5 μL,正反向引物各0.75 μL,DNA 2 μL,無菌水9 μL,總體系25 μL。PCR擴增程序見表3。PCR產物由北京睿博興科生物技術有限公司測序部測序,各樣品均采用正反雙向測序,以保證結果的準確性。

表3 引物及PCR反應條件
1.4.2 序列分析 將測序結果與網上桃仁與苦杏仁序列用CLUSTAL X軟件進行序列比對,使用MEGA6用鄰接(NJ)法構建系統聚類樹,Bootstrap(1000次重復)檢驗各分支的支持率。
1.4.3 特異性鑒別引物的設計 將所測得的序列與GenBank中的桃仁與苦杏仁ITS序列用BioEdit軟件進行比對分析,找出具有穩定差異的特異性片段,篩選獲得變異位點,通過該位點運用Premier Primer 5.0軟件針對桃仁的特異性片段設計多對鑒別引物,并由睿博興科生物技術有限公司合成。
1.4.4 PCR引物特異性篩選 用所設計引物進行PCR反應,PCR所用25 μL反應體系為:2×EasyTaq PCR Super Mix 12.5 μL,正反向引物各0.5 μL,DNA稀釋20倍后取1 μL,無菌水補齊。PCR產物采取瓊脂糖凝膠電泳方法檢測。以結果出現整齊、清晰條帶為陽性,其余為陰性,由此判斷引物特異性。
1.4.5 特異性PCR反應條件優化 為獲得最優的位點特異性PCR反應條件,對其反應條件進行優化,分別考察了DNA模板濃度(0.005 ng/μL、0.01 ng/μL、0.02 ng/μL、0.05 ng/μL、0.1 ng/μL、0.2 ng/μL、0.25 ng/μL、0.5 ng/μL、1 ng/μL、2 ng/μL)、退火溫度(55 ℃、57 ℃、59 ℃、61 ℃、63 ℃)、聚合酶種類(Ex Taq DNA聚合酶、2×Taq PCR StarMix、2×EasyTaq PCR SuperMix)、不同PCR儀(Applied Biosystems Veriti;Eppendorf AG 22331)對PCR反應的影響。反應體系為:DNA聚合酶12.5 μL,正反向引物各0.5 μL,DNA 1 μL,無菌水補足25 μL。
2結果與分析
2.1 通用引物PCR擴增電泳結果 ITS通用引物擴增樣品的成功率為100%,即全部具有明亮清晰條帶。擴增結果如圖1。
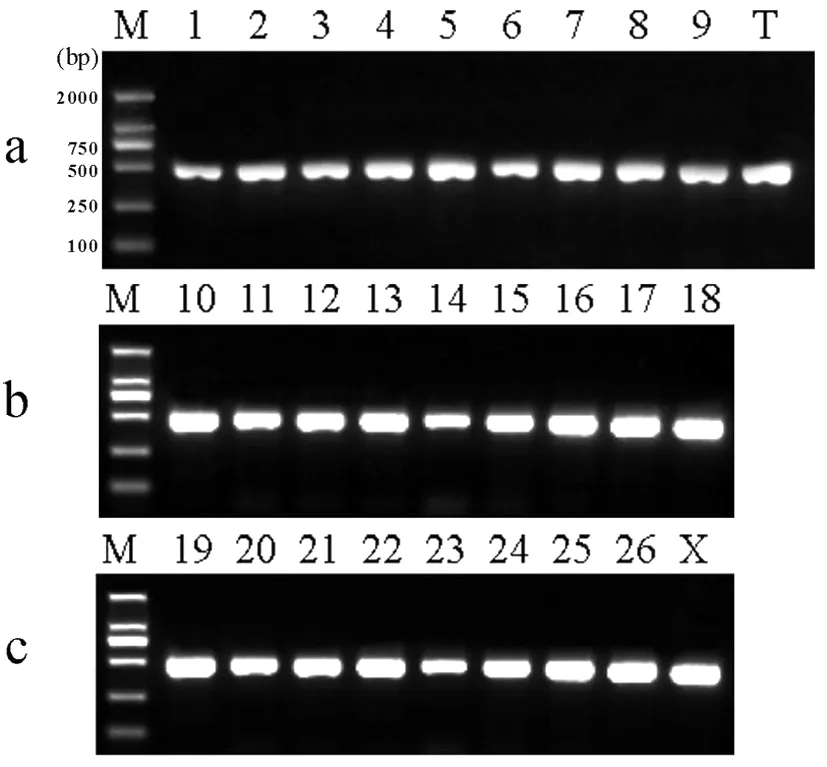
圖1 通用引物PCR結果
注:a:桃仁1-9以及桃仁標準藥材T;b:山桃10-18;c:杏仁19-26以及苦杏仁標準藥材X;M:DNA分子標記
2.2 基于ITS序列桃仁與苦杏仁樣品的系統進化樹構建 采用Mega 6分析軟件進行桃仁與苦杏仁系統發育樹的構建,采用鄰接法(NJ)構建系統發育樹。見圖2,采用Kimura2-parameter距離,系統樹各分支的置信度用bootstrap method檢驗,共進行1 000次循環。由圖2可知,桃仁樣品與桃仁標準藥材聚為一支,苦杏仁與苦杏仁標準藥材聚為一支,表明ITS序列用于桃仁與苦杏仁的分子鑒定具有一定的理論依據。
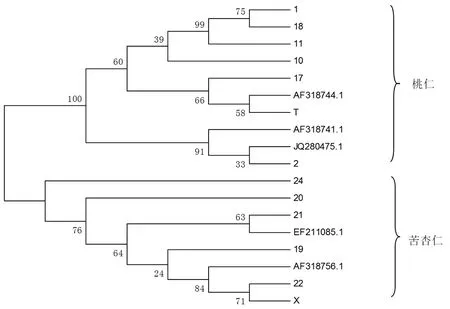
圖2 基于ITS序列構建的桃仁與杏仁藥材的鄰接(NJ)樹
注:桃仁:1,2,10,11,17,18,T,AF318744.1,AF318741.1,JQ280475.1;苦杏仁:19,20,21,22,24,X,AF318756.1,EF211085.1
2.3 特異性引物PCR擴增 用篩選的特異PCR引物G4-7(序列見表3)對提取的桃仁和苦杏仁DNA模板進行擴增。如圖3所示,桃仁標準藥材與桃仁樣品特異性擴增結果呈陽性,條帶整齊、清晰明亮,大小333 bp,而苦杏仁標準藥材與苦杏仁樣品特異性擴增結果均呈陰性,證明該引物具有良好的特異性。

圖3 特異引物PCR結果
注:a:桃1-9以及桃仁標準藥材T;b:山桃10-18;c:杏19-26以及苦杏仁標準藥材X M:DNA分子標記
2.4 PCR擴增反應條件優化
2.4.1 DNA模板量的考查 對25 μL PCR反應體系中的模板DNA用量進行了考察,結果如圖4,DNA模板濃度在2 ng/μL時苦杏仁出現假陽性條帶,而在0.02 ng/μL時桃仁不能出現清晰、明亮條帶,且其引物二聚體明顯增多(見圖4),因此確定本方法DNA模板濃度適宜范圍應為0.05~1 ng/μL。實驗采用0.05 ng/μL DNA模板濃度繼續進行其他條件的考察。

圖4 DNA模板濃度優化結果
注:1-13:桃仁,19-22:苦杏仁;M:DNA分子標記
2.4.2 退火溫度的考查 設置退火溫度分別為55 ℃、57 ℃、59 ℃、61 ℃、63 ℃進行考察,結果見圖5。從結果可見,桃仁均出現整齊、清晰條帶,而苦杏仁均無條帶,表明退火溫度對其無明顯影響,采用59 ℃作為此方法的退火溫度。
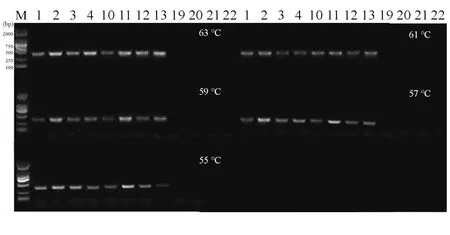
圖5 退火溫度考查結果
注:1-13:桃仁,19-22:苦杏仁;M:DNA分子標記
2.4.3 DNA聚合酶的考察 對Ex Taq DNA聚合酶,2×Taq PCR StarMix,2×EasyTaq PCR SuperMix 3種聚合酶進行考察。結果如圖6。采用不同聚合酶的PCR反應,偽品均無條帶,而正品均有333 bp特異性擴增條帶,說明特異性引物可用多種酶進行實驗,無單一局限性。
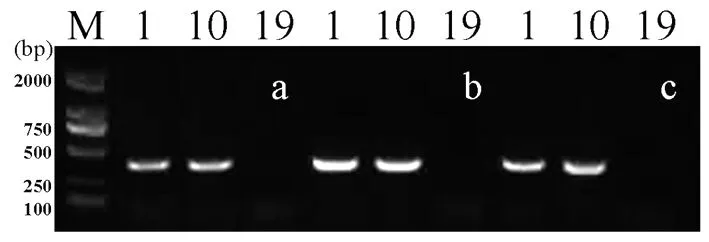
圖6 不同酶考查結果
注:1,10:桃仁,19:苦杏仁。a:Ex Taq DNA聚合酶,b:2×Taq PCR StarMix,c:2×EasyTaq PCR SuperMix;M:DNA分子標記
2.4.4 PCR儀對實驗結果重復性的影響 選用2種不同型號的PCR儀(Eppendorf AG 22331、Applied Biosystems Veriti)進行考察,結果桃仁均出現整齊、明亮清晰的條帶,苦杏仁均無條帶出現(圖7),表明此方法穩定性好,重復性高,PCR儀對其結果無明顯影響。
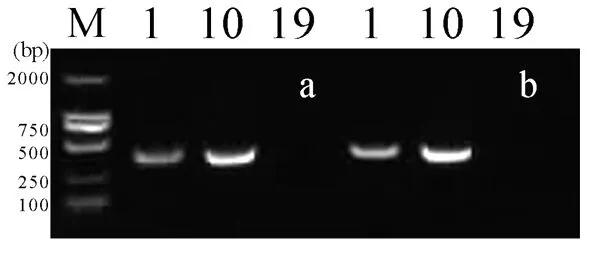
圖7 不同PCR儀考查結果
注:1,10:桃仁,19:苦杏仁。a:Eppendorf AG 22331 PCR儀;b:Applied Biosystems Veriti PCR儀;M:DNA分子標記
3討論
由于桃仁品種繁雜,形態變異大,且與苦杏仁形態相似,尤其是山桃仁在形態上與杏仁更加難以區分,故傳統鑒別在實際應用中很難將兩者準確區別。位點特異性引物PCR具有操作簡單、成本低、重復性好等優點,使用特異性引物可以極大降低PCR的假陽性率,保證鑒定結果的準確性[4],特別是近年來快速PCR技術迅速發展,使得中藥材分子鑒定在現場快速鑒定上的應用成為了可能[5-6]。如李振華等[7]通過位點特異PCR的方法證明了荒漠肉蓯蓉能擴增出331 bp的條帶,而混偽品不能,從而實現了荒漠肉蓯蓉與其混偽品的快速、準確鑒別。陳康等[8]在位點特異性引物PCR方法的基礎上改良實驗條件,應用快速PCR方法通過顯示熒光區分真偽蛇類藥材,所建立的蛇類藥材快速PCR真偽檢測方法可在30~45 min完成。趙丹等[9]對太子參藥材及其混偽品的rDNA-ITS片段進行比對分析,應用特異性引物PCR方法將太子參藥材及其混偽品進行了很好的區分。
DNA條形碼技術在物種鑒別分類上起到了越來越重要的作用[10-12]。植物常用鑒別序列是ITS序列,包括ITSl、ITS2,分別位于18S~5.8S rDNA、5.8S~26S rDNA之間,長度分別為187~298 bp和187~252 bp,而18S、5.8S、26S rDNA的序列又非常保守,所以其通用引物可適用物種范圍廣泛[13]。Chen等[3]比較了藥用植物中7個候選的DNA條形碼(psbA-trnH、rbcL、matK、rpoC1,ycf5,ITS2以及ITS),并且測試了ITS2鑒別753個屬4 800個物種的6600個樣品的成功率,結果為其鑒別成功率在種水平上為92.7%,最終認為ITS2可做為更廣泛的藥用植物鑒別DNA條形碼。林韻涵等對藥材桃仁及其近緣種進行分子鑒定,結果表明ITS2序列作為DNA條形碼可以有效地鑒定藥材桃仁及其近緣種。因此本實驗最初選用桃仁及苦杏仁藥材ITS2序列進行測序,但由于其親緣關系較近,序列相似度較高,且多處均為單個堿基的不同,因此難以設計鑒別度較好的特異引物。中國植物條形碼工作組[14]對來自42目75科141屬1 757個物種的6 286個種子類植物樣本的rbcL,matK,psbA-trnH,ITS序列進行研究,結果表明ITS有最高的鑒別能力,建議ITS/ITS2應成為種子植物的核心條形碼。因此本實驗即對桃仁及苦杏仁藥材ITS序列進行測序,2種藥材ITS序列中Poly結構較多,突變缺失情況較嚴重,并不是所有藥材均能測序成功,但根據所測序結果以及與網上序列進行比對分析,設計特異性鑒別引物,結果表明其引物特異性好,鑒別能力高,可作為桃仁及苦杏仁藥材鑒別的特異引物。
本實驗通過特異引物篩選和條件優化,最終確定25 μL PCR反應體系時,引物G4-7在DNA模板加樣量0.05~1 ng范圍內,退火溫度55~63 ℃時可將桃仁與苦杏仁進行有效的鑒別。該方法靈敏度高,穩定性好,可用于桃仁與苦杏仁的分子鑒別以及桃仁藥材分子鑒定的推廣與應用,且為桃仁藥材的快速鑒別提供了參考依據。
[1]國家藥典委員會.中華人民共和國藥典(一部)[M].北京:中國醫藥科技出版社,2015:201-202,277-278.
[2]杜虹韋,張愛華,趙欣蕾.苦杏仁毒性及其解毒方法研究進展[J].黑龍江中醫藥,2013,43(4):58-59.
[3]Chen S,Yao H,Han J,et al.Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J].PLoS One,2010,5(1):e8613.
[4]黃璐琦.中藥分子鑒定操作指南[M].上海:上海科學技術出版社,2014.
[5]蔣超,侯靜怡,黃璐琦,等.快速PCR方法在金銀花真偽鑒別中的應用[J].中國中藥雜志,2014,39(19):3668-3672.
[6]蔣超.中藥現場、快速鑒別方法的建立—以金銀花為例[D].北京:中國中醫科學院,2013.
[7]李振華,龍平,鄒德志,等.基于ITS序列位點特異性PCR的肉蓯蓉與其混偽品的鑒別研究[J].中國中藥雜志,2014,39(19):3684-3688.
[8]陳康,蔣超,袁媛,等.快速PCR方法在蛇類藥材真偽鑒別中的應用[J].中國中藥雜志,2014,39(19):3673-3677.
[9]趙丹,周濤,江維克,等.太子參藥材的快速分子鑒定[J].中國中藥雜志,2014,39(19):3689-3694.
[10]韋健紅,吳文如,喻良文,等.DNA條形碼技術在生物分類中的應用[J].廣東藥學院學報,2010,26(4):430-433.
[11]裴男才,陳步峰.生物DNA條形碼:十年發展歷程、研究尺度和功能[J].生物多樣性,2013,21(5):616-627.
[12]韓建萍,宋經元,姚輝,等.中藥材DNA條形碼鑒定的基因序列比較[J].中國中藥雜志,2012,37(8):1056-1061.
[13]程龍,葛斐林,劉澤邦,等.中藥資源DNA條形碼鑒定方法研究進展[C].北京:第四屆中國中藥商品學術大會暨中藥鑒定學科教學改革與教材建設研討會論文集,2015:398-404.
[14]Li DZ,Gao LM,Li HT,et al.Comparative analysis of a large dataset indicates that internal transcribed spacer(ITS)should be incorporated into the core barcode for seed plants[J].Proc Natl Acad Sci U S A,2011,108(49):19641-19646.
(2016-11-30收稿 責任編輯:楊覺雄)
IdentificationofPersicaeSemenandArmeniacaeSemenAmarumbyPCRAmplificationofSpecificAllelesBasedonITSSequences
Gao Linhui1,Yin Yan2,Li Jia1,Ma Yuemeng1,Li Wenbin1,Li Bowen1,Zhang Xia′nan1
(1SchoolofTraditionalChineseMedcine,CapitalMedicalUniversity,Beijing100069,China; 2SchoolofPharmaceuticalSciences,GuizhouUniversity,Guiyang550025,China)
To identify Persicae semen amarum using DNA molecular identification,and establish classical PCR identification mesthod.The internal transcribed spacer (ITS) of nuclear ribosomal DNA of Persicae Semen and Armeniacae semen amarum were amplified and bidirectionally sequenced.ITS sequences were analysed by BioEdit.Specific identification primers were designed according to the ITS sequences of specific alleles,and PCR reaction system was optimized.Persicae Semen and Armeniacae Semen Amarum can be identificated by the specfic peimers G4-7 based on ITS sequences.In the condition′s concentration is between 0.05-1 ng for a 25 μL PCR reaction,the best annealing temperature is 59 ℃.This method is well used for different DNA polymerases and different PCR instruments.The result showed that the 333 bp identification band can be amplified in Persicae semen,while it can not be amplified in Armeniacae semen amarum.It is confirmed that the PCR amplification system of specific alleles can be used to identify Persicae semen and Armeniacae semen amarum fastly and accurately.
Persicae Semen; Armeniacae Semen Amarum; ITS; PCR amplification of specific alleles; Molecular identification
R284;R917
:Adoi:10.3969/j.issn.1673-7202.2017.09.050
國家自然科學基金(81303166);國家中醫藥管理局中醫藥行業專項(201407003)
高琳惠(1993.07—),女,在讀研究生,研究方向:分子生藥學,E-mail:gaolinhui1993@qq.com
張夏楠(1982.04—),女,博士,講師,研究方向:分子生藥學,E-mail:xnzhang@ccmu.edu.cn

