革蘭陰性桿菌血流感染的病原菌分布、耐藥性及碳青霉烯酶基因的檢測與分析
鄒 穎, 徐曉剛, 郭慶蘭, 李光輝
·論著·
革蘭陰性桿菌血流感染的病原菌分布、耐藥性及碳青霉烯酶基因的檢測與分析
鄒 穎1,2, 徐曉剛1, 郭慶蘭1, 李光輝1
目的 調查復旦大學附屬華山醫院革蘭陰性桿菌血流感染的病原菌分布情況、對常用抗菌藥物的耐藥性以及碳青霉烯酶基因的流行現狀。方法 收集2012年3月1日—2013年2月28日該院血流感染分離獲得的132株非重復革蘭陰性桿菌菌株,按統一方案采用紙片擴散法進行抗菌藥物敏感性試驗,共篩選出33株碳青霉烯類不敏感菌株。采用PCR方法檢測KPC、NDM、VIM、IMP、GIM、SPM、OXA23、OXA24、OXA51、OXA58型碳青霉烯酶基因。結果 革蘭陰性桿菌血流感染前5位的病原菌依次為克雷伯菌屬(28.0%,37/132)、大腸埃希菌(26.5%,35/132)、不動桿菌屬(13.6%,18/132)、銅綠假單胞菌(9.8%,13/132)和腸桿菌屬(7.6%,10/132)。33株碳青霉烯類不敏感革蘭陰性桿菌中有75.8%(25/33)的菌株檢測出碳青霉烯酶基因,KPC、OXA23、OXA51、NDM型碳青霉烯酶基因檢出率分別為42.4%(14/33)、30.3%(10/33)、30.3%(10/33)、3.0%(1/33),未檢測到其他類型碳青霉烯酶基因。13株對碳青霉烯類藥物不敏感的肺炎克雷伯菌100%攜帶KPC型耐藥基因,10株對碳青霉烯類藥物不敏感的鮑曼不動桿菌100%攜帶OXA23、OXA51型基因。結論 革蘭陰性桿菌血流感染中肺炎克雷伯菌、鮑曼不動桿菌對碳青霉烯類抗生素耐藥形勢嚴峻。攜帶KPC、OXA23型碳青霉烯酶基因是導致肺炎克雷伯菌和鮑曼不動桿菌對碳青霉烯類抗生素耐藥的主要機制之一。
血流感染; 碳青霉烯酶; 金屬β內酰胺酶; 碳青霉烯酶基因
1 材料與方法
1.1 革蘭陰性桿菌血流感染診斷標準
血培養分離出革蘭陰性桿菌,并伴有下列癥狀或體征中的一項:①發熱>38℃或低體溫<36℃,可伴有寒顫;②有入侵門戶或遷徙病灶;③有全身感染中毒癥狀而無明確感染灶;④收縮壓低于90 mmHg,或較原收縮壓下降超過40 mmHg。回顧性調查血標本分離出革蘭陰性桿菌患者的病史資料,所有患者均符合血流感染診斷標準。
1.2 菌株分離、培養、鑒定及藥敏試驗
菌株來自復旦大學附屬華山醫院2012年3月1日—2013年2月28日送檢的血液標本,按醫院常規方法進行細菌分離、培養、鑒定。剔除同一患者同一次發病重復分離的菌株,共獲得132株革蘭陰性桿菌。全部菌株經法國生物梅里埃公司產品VITEK 2-Compact或API 20系列鑒定。標準菌株為大腸埃希菌ATCC 25922和銅綠假單胞菌ATCC 27853。采用紙片擴散法測定菌株對常用抗菌藥物的敏感性,結果按美國臨床和實驗室標準化協會(CLSI) 2012年標準判讀。頭孢哌酮-舒巴坦判斷標準參照文獻[2]。藥敏試驗結果對亞胺培南、美羅培南、多立培南任一碳青霉烯類為中介或耐藥即判定為碳青霉烯類不敏感菌株。
1.3 碳青霉烯酶基因的檢測與分析
1.3.1 DNA提取 挑取1~2個菌落細菌加入500 μL TE(pH 8.0)溶液中混勻,100 ℃煮沸10 min后放入4 ℃冰箱,冷卻后5 000 r/min離心1 min,收集上清液作為細菌DNA模板。
1.3.2 耐藥基因擴增 KPC、NDM、IMP、VIM、 GIM、SPM型碳青霉烯酶基因采用PCR方法擴增(PCR儀為美國ABI公司產品,ABI 9600型)。反應總體積50 μL,其中10×buffer 5 μL,dNTP 4 μL,Taq酶0.25 μL(TaKaRa公司),上下游引物各1 μL(20 μmol/L),模板DNA 4 μL,ddH2O補足至50 μL。KPC、NDM、IMP、VIM引物應用DNAstar 7.1軟件自行設計。PCR反應條件為:94 ℃預變性5 min,94 ℃變性30 s,54 ℃退火30 s,72 ℃延伸30 s,共35個循環,最后72 ℃延伸6 min。GIM、SPM引物序列及PCR反應條件參照文獻[3-4]。OXA23、OXA24、OXA51、OXA58型碳青霉烯酶基因采用多重PCR方法進行擴增,引物序列及PCR反應條件參照文獻[5],反應總體積50 μL,其中10×buffer 5 μL,dNTP 4 μL,Taq 酶0.25 μL,每條引物各0.5 μL(20 μmol/L),模板DNA 3 μL,ddH2O補足至50 μL。各型碳青霉烯酶基因引物序列見表1,由生工生物(上海)有限公司合成。
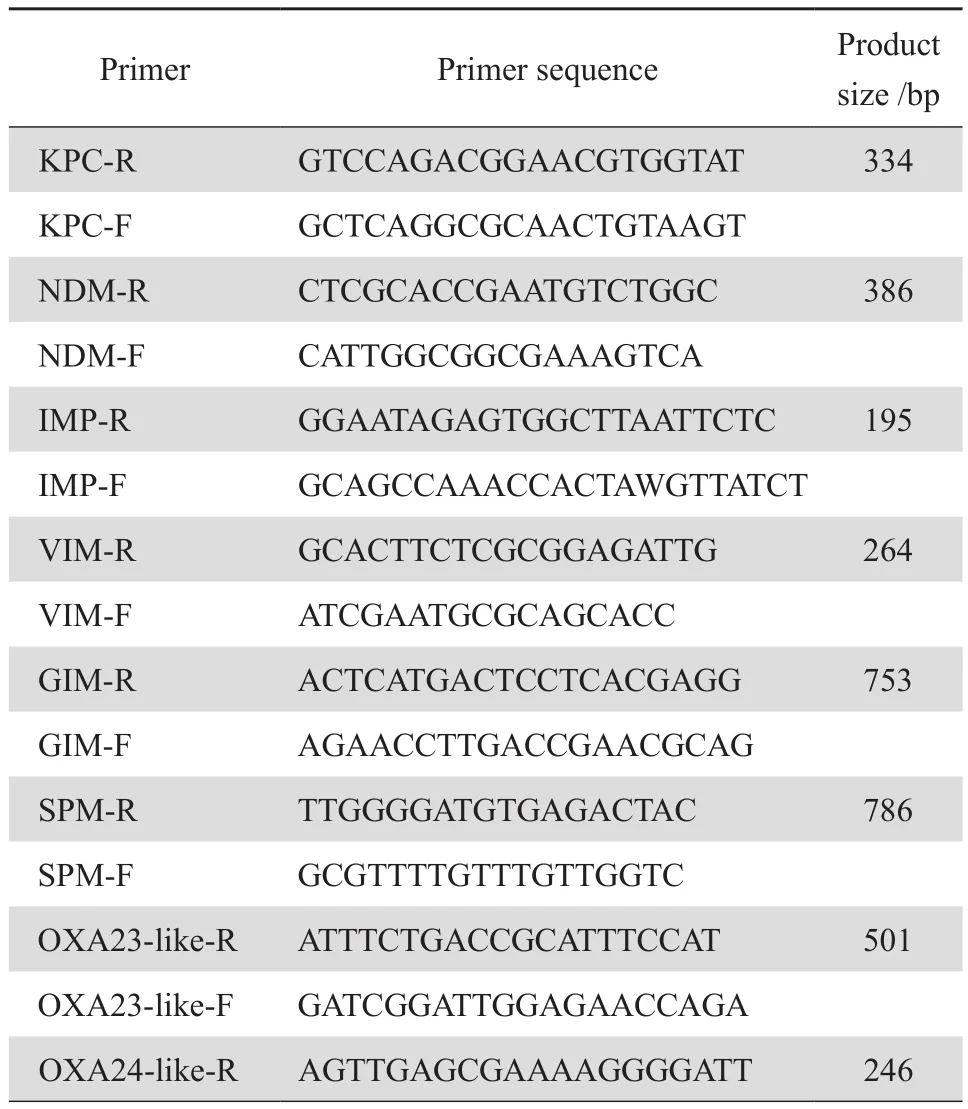
表1 碳青霉烯酶基因引物序列及片段大小Table 1 Primer sequences and fragment length of carbapenem resistance genes
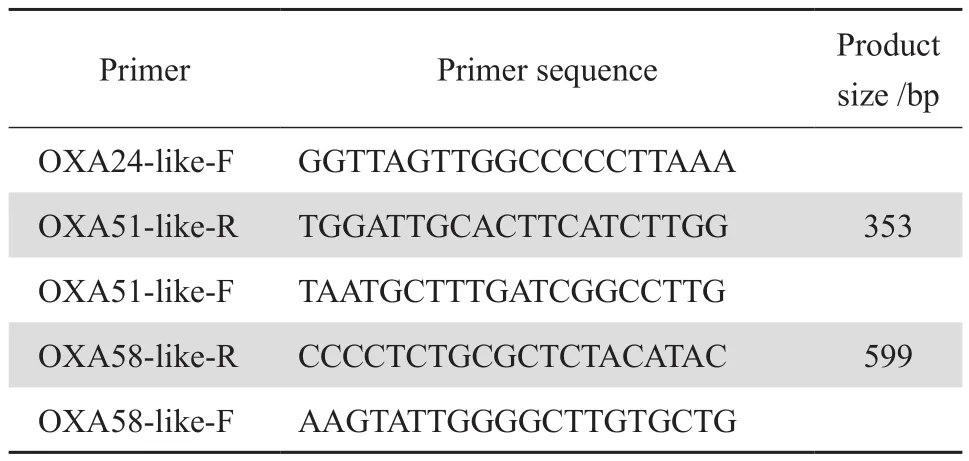
表1(續)Table 1(continued)
1.3.3 PCR產物分析 取5 μL PCR擴增產物與6×上樣緩沖液1 μL混勻,經1.2%瓊脂糖凝膠以120V電泳35 min,溴乙錠染色20 min,流水漂洗,最后在凝膠成像分析系統(美國Bio-Rad公司產品)上觀察結果。采用DNA Marker DL2000為分子量標準,出現目的條帶者判定為陽性。取陽性結果PCR產物送生工生物(上海)有限公司測序,測序結果與GenBank中的核苷酸序列進行同源性比對。
2 結果
2.1 病原菌分布
2012年3月—2013月2月復旦大學附屬華山醫院共發生132例革蘭陰性桿菌血流感染,血培養分離獲得革蘭陰性桿菌132株。病原菌分布前5位依次為克雷伯菌屬(28.0%,37/132,其中肺炎克雷伯菌36株,占97.3%)、大腸埃希菌(26.5%,35/132)、不動桿菌屬(13.6%,18/132,其中鮑曼不動桿菌16株)、銅綠假單胞菌(9.8%,13/132)、腸桿菌屬(7.6%,10/132,其中陰溝腸桿菌6株),見表2。
2.2 藥敏試驗結果
前5位病原菌對常用抗菌藥物的敏感率見表3。132株革蘭陰性桿菌中腸桿菌科細菌、銅綠假單胞菌、不動桿菌屬對碳青霉烯類的敏感率分別為81.3%(74/91)、92.3%(12/13)、38.9%(7/18)。克雷伯菌屬對碳青霉烯類的敏感率(62.2%)顯著低于大腸埃希菌(97.1%~100%)和腸桿菌屬細菌(80.0%~100%);對第三、第四代頭孢菌素的敏感率(46.0%~51.4%)高于大腸埃希菌(22.9%~42.9%);對頭孢哌酮-舒巴坦和哌拉西林-他唑巴坦的敏感率均在50%以上。大腸埃希菌對哌拉西林-他唑巴坦的敏感率達74.3%;克雷伯菌屬對氨基糖苷類的敏感率為51.4%~62.2%,而大腸埃希菌為45.7%~80.0%;克雷伯菌屬對環丙沙星的敏感率雖顯著高于大腸埃希菌,但均不足半數(48.6%比28.6%)。腸桿菌屬細菌對頭孢吡肟、碳青霉烯類、氨基糖苷類和喹諾酮類均高度敏感,敏感率為80.0%~100%。不動桿菌屬對碳青霉烯類的敏感率(38.9%)顯著低于銅綠假單胞菌(92.3%)。不動桿菌屬對所有受試藥物的敏感率均未超過50%。銅綠假單胞菌對常用受試藥物的敏感率為84.6%~92.3%,但對頭孢哌酮和氨曲南僅分別為53.8%和69.2%。
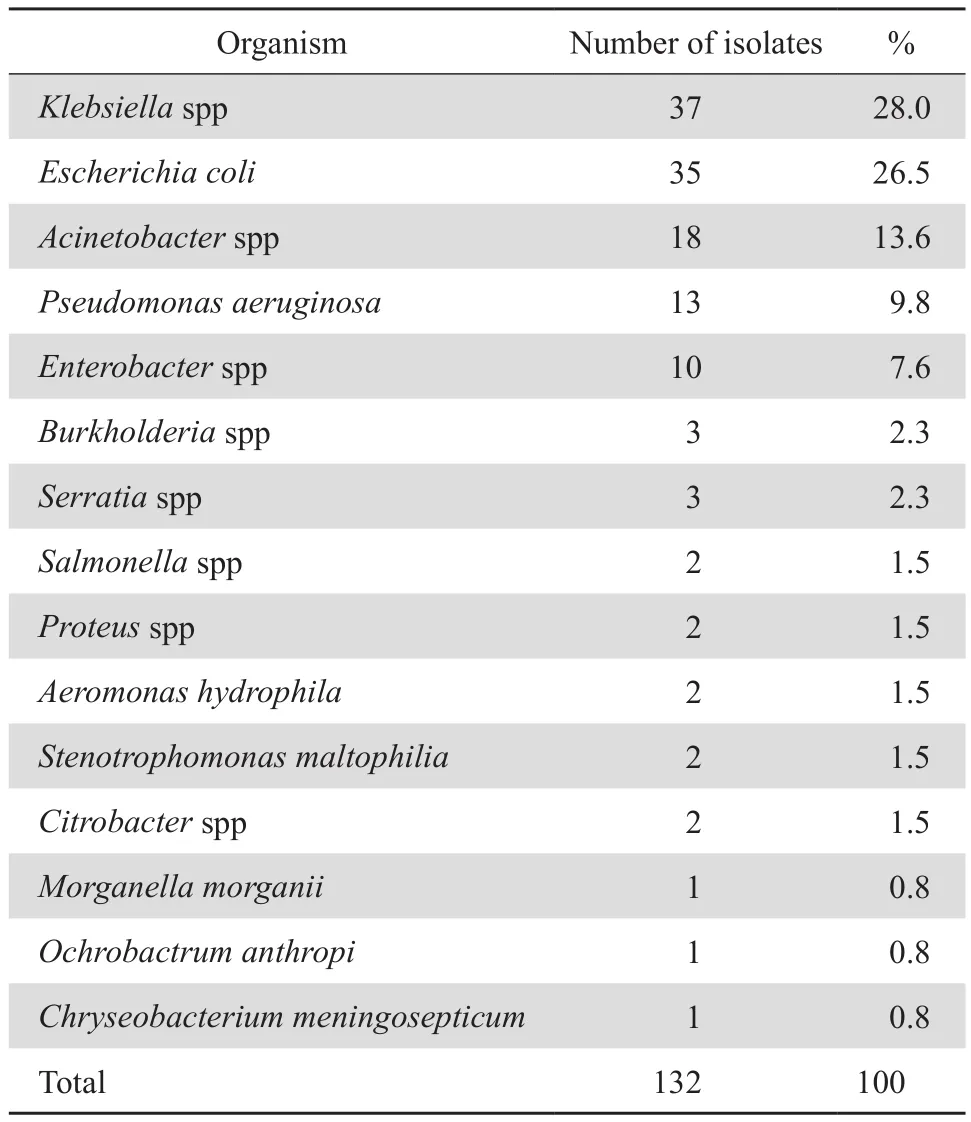
表2 血培養分離132株革蘭陰性桿菌的分布Table 2 Distribution of 132 strains of gram negative bacteria isolated from blood samples
33株對碳青霉烯類不敏感菌株中克雷伯菌屬占42.4%(14/33,其中肺炎克雷伯菌13株)、不動桿菌屬占33.3%(11/33,其中鮑曼不動桿菌10株)、腸桿菌屬占6.1%(2/33),其余為大腸埃希菌、銅綠假單胞菌、嗜水氣單胞菌、嗜麥芽窄食單胞菌、摩根摩根菌、洋蔥伯克霍爾德菌各1株。碳青霉烯類不敏感克雷伯菌屬對甲氧芐啶-磺胺甲唑的敏感率為57.1%,對環丙沙星和阿米卡星的敏感率為7.1%,對其他受試藥物均耐藥。碳青霉烯類不敏感不動桿菌屬對阿米卡星、慶大霉素、環丙沙星、頭孢哌酮-舒巴坦、甲氧芐啶-磺胺甲唑的敏感率為9.1%~18.2%,對其他受試藥物均耐藥,見表4。
2.3 碳青霉烯類不敏感菌株科室分布
33株碳青霉烯類不敏感菌株主要分布在綜合ICU(10株)、神經外科ICU(7株)、神經外科急救病房(4株),其余分布在感染科(2株)、皮膚科(2株)、血液科(2株)、神經內科(2株)、神經外科(2株)、普外科(1株)和急診內科(1株)。
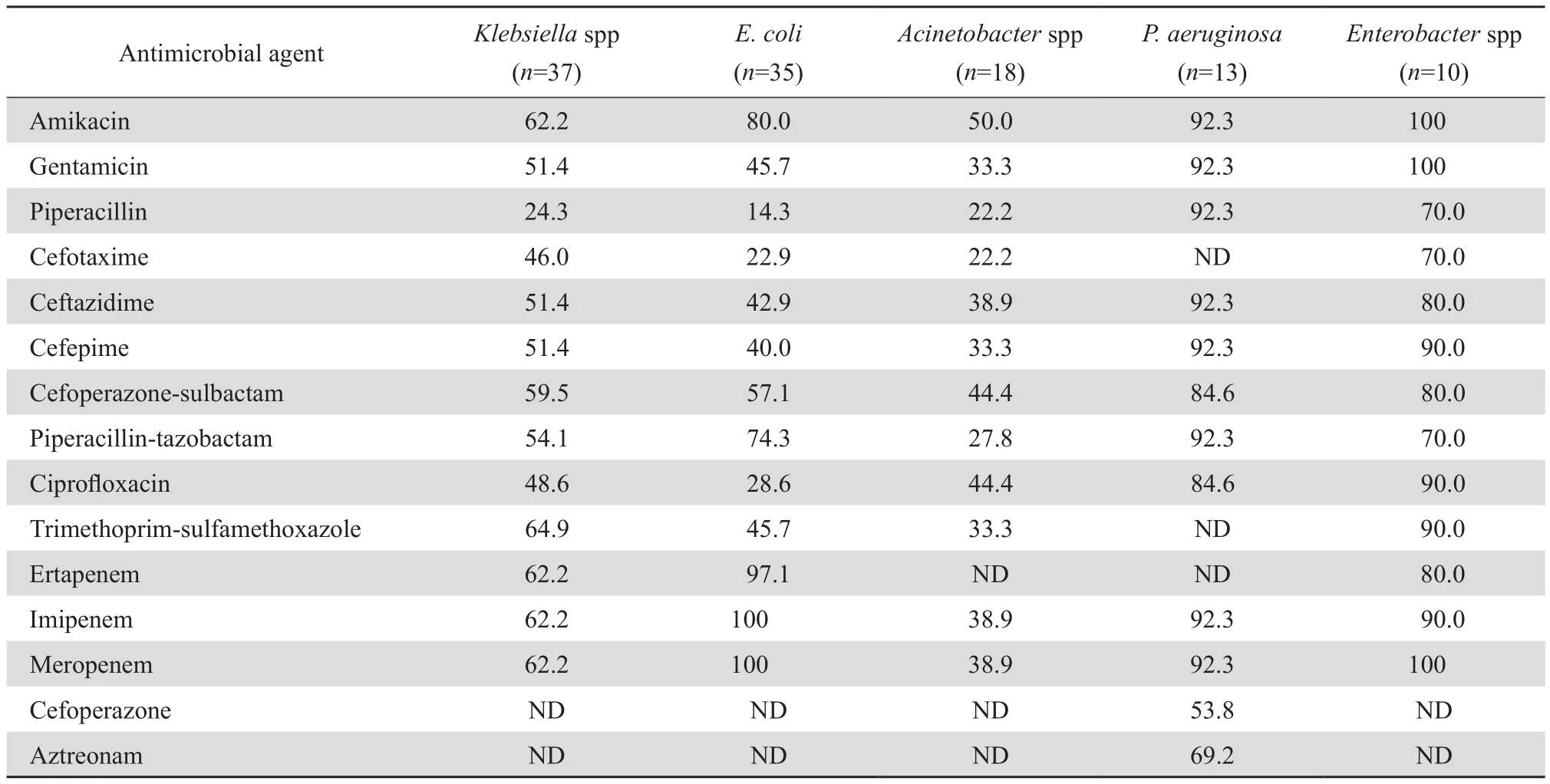
表3 血標本分離前5位革蘭陰性桿菌對常用抗菌藥物的敏感率Table 3 Susceptibility of the top five gram negative bacterial species to selected antimicrobial agents (%)
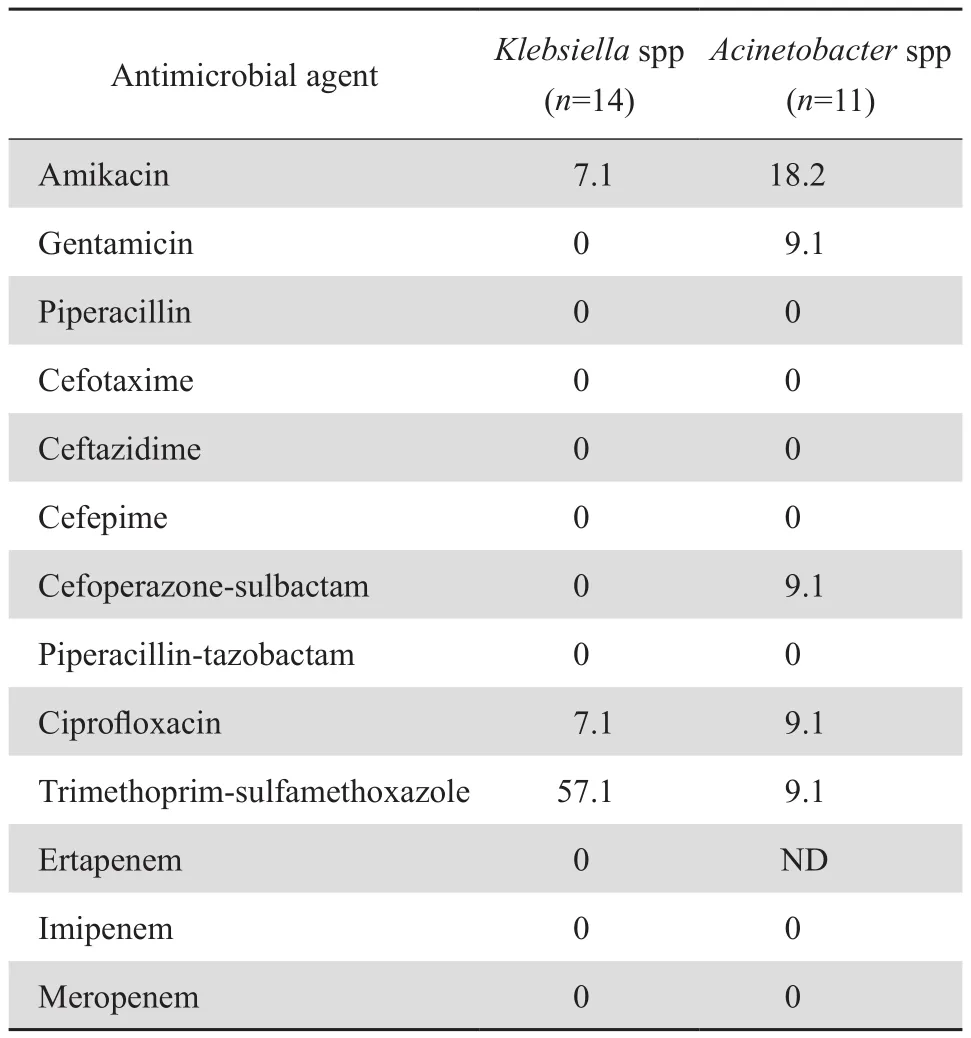
表4 25株碳青霉烯類不敏感克雷伯菌屬和不動桿菌屬菌株對常用抗菌藥物的敏感率Table 4 Susceptibility of 25 carbapenem-nonsusceptible strains to selected antimicrobial agents(%)
2.4 碳青霉烯酶基因檢測
33株對碳青霉烯類不敏感革蘭陰性桿菌中有75.8%(25/33)菌株檢測出碳青霉烯酶基因。KPC、OXA23、OXA51、NDM型碳青霉烯酶基因檢出率分別為42.4%(14/33)、30.3%(10/33)、30.3%(10/33)、3.0%(1/33),未檢測到其他類型碳青霉烯酶基因。13株對碳青霉烯類不敏感的肺炎克雷伯菌100%攜帶KPC型基因,10株對碳青霉烯類不敏感的鮑曼不動桿菌100%攜帶OXA23、OXA51型基因。不同菌株碳青霉烯酶基因攜帶情況見表5。KPC、NDM、OXA23、OXA51型基因擴增產物電泳圖見圖1~3。
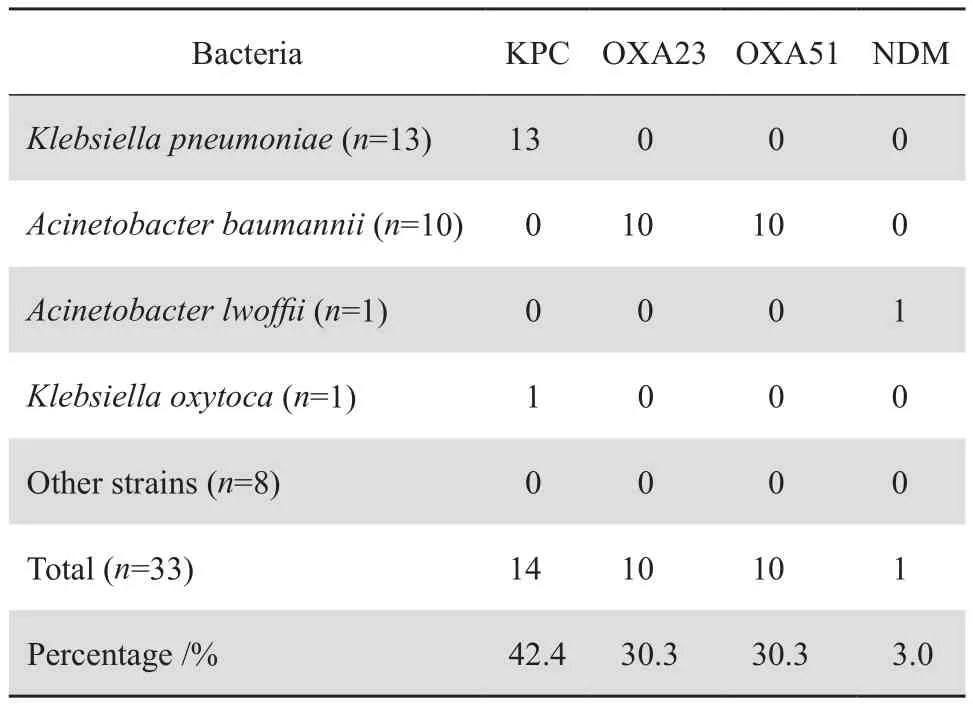
表5 33株對碳青霉烯類不敏感菌株碳青霉烯酶基因攜帶情況Table 5 Detection of carbapenem resistance genes from 33 carbapenem-nonsusceptible strains

圖1 KPC型碳青霉烯酶擴增產物電泳圖(目的片段:334 bp)Figure 1 Electrophoretic pattern of KPC type carbapenemase gene amplified by PCR (target fragment size: 334 bp)

圖2 NDM型碳青霉烯酶擴增產物電泳圖(目的片段:386 bp)Figure 2 Electrophoretic pattern of NDM type carbapenemase gene amplified by PCR (target fragment size: 386 bp)
3 討論
血流感染是常見的嚴重衛生保健相關感染[6],其中由革蘭陰性桿菌引起的感染占有相當重要的地位。依據2012年CHINET監測數據,革蘭陰性桿菌在血標本分離菌中占44.0%[7]。雖然凝固酶陰性葡萄球菌是血液最常分離到的細菌,但相當一部分可能為污染,而血培養分離到腸桿菌科細菌、不發酵糖革蘭陰性桿菌往往具有診斷意義。

圖3 OXA23、OXA51型碳青霉烯酶擴增產物電泳圖(OXA23、OXA51目的片段大小分別為:501 bp、353 bp)Figure 3 Electrophoretic pattern of OXA type carbapenemase gene amplified by multiplex PCR (target fragment size of OXA23 and OXA51 genes: 501 bp and 353 bp)
本研究中革蘭陰性桿菌血流感染占前5位的病原菌依次為克雷伯菌屬、大腸埃希菌、不動桿菌屬、銅綠假單胞菌和腸桿菌屬細菌,與歷年來CHINET血標本監測數據中前5位的病原菌基本一致,唯排位順序略有不同,克雷伯菌屬取代大腸埃希菌居第1位,兩者所占比例非常接近。從耐藥率方面來看,克雷伯菌屬和大腸埃希菌對第三、第四代頭孢菌素敏感率偏低,尤其是大腸埃希菌敏感率均低于50%,提示產ESBL菌株可能占有相當比例。大腸埃希菌對碳青霉烯類仍保持高度敏感,所有35株大腸埃希菌中僅1株對厄他培南藥敏試驗結果為中介,對美羅培南和亞胺培南仍為敏感。因此,臨床對于懷疑由大腸埃希菌導致的血流感染可在知道藥敏結果之前經驗性選擇碳青霉烯類。相比之下,克雷伯菌屬對碳青霉烯類的耐藥性明顯高于大腸埃希菌。所有37株克雷伯菌屬中有36株為肺炎克雷伯菌,超過1/3肺炎克雷伯菌(36.1%,13/36)對臨床常用3種碳青霉烯類呈現高度耐藥,且這13株耐碳青霉烯類菌株除了對甲氧芐啶-磺胺甲唑敏感率略高外,對其余9種常用的抗菌藥物敏感率均不足10%,一旦感染臨床幾乎無藥可選,耐藥形勢極為嚴峻。
另一個在病原菌組成中占相當比例且耐藥情況極為嚴重的革蘭陰性桿菌為不動桿菌屬。18株不動桿菌屬菌株對全部12種受試藥物的敏感率均未超過50%,61.1%(11/18)菌株對碳青霉烯類不敏感,且這11株菌株對其余受試藥物的敏感率也相當低,敏感率最高的阿米卡星也僅為18.2%,成為多重耐藥革蘭陰性桿菌的主要菌種之一。
從臨床科室分布情況來看,對碳青霉烯類不敏感菌株有集中分布的趨勢。綜合ICU、神經外科ICU、神經外科急救病房3個科室共分離出21株耐藥菌株,占總數的63.6%(21/33),因此不能排除存在多重耐藥菌株醫院內播散可能,可通過菌株同源性檢測進一步驗證。
碳青霉烯類治療產ESBL革蘭陰性桿菌引起的感染往往能取得令人滿意的療效,因此被認為是臨床上針對多重耐藥革蘭陰性桿菌最有效的抗菌藥物之一。然而,隨著碳青霉烯類的廣泛應用以及存在一些不規范使用乃至濫用的情況,近年來國內外細菌耐藥性監測數據提示耐碳青霉烯類的革蘭陰性桿菌比例不斷上升。CHINET監測數據表明肺炎克雷伯菌對亞胺培南和美羅培南的耐藥率分別從2005年的0.9%、1.3%升至2013年的10.5%、13.5%;同期鮑曼不動桿菌對碳青霉烯類抗生素的耐藥率也從30%~40%升至60%左右[8-9]。英國抗菌藥物耐藥監測數據顯示2010年產碳青霉烯酶細菌的數量較2003年增加了近300倍[10]。本研究中血液標本分離肺炎克雷伯菌對亞胺培南、美羅培南的耐藥率均為36.1%(13/36),而鮑曼不動桿菌這一數據高達62.5%(10/16),以肺炎克雷伯菌、鮑曼不動桿菌為代表的革蘭陰性桿菌對碳青霉烯類耐藥性逐年上升已是不爭的事實。
導致對碳青霉烯類耐藥的重要原因之一是耐藥菌株產生β內酰胺酶,它能水解碳青霉烯類藥物,因而被稱為碳青霉烯酶。KPC型、OXA型以及新近發現的以NDM型為代表的金屬酶是其中最重要的分型[11-13]。本研究中33株對碳青霉烯類不敏感的革蘭陰性桿菌中有75.8%(25/33)菌株檢測出碳青霉烯酶基因,KPC、OXA23、OXA51、NDM型碳青霉烯酶基因檢出率分別為42.4%(14/33)、30.3%(10/33)、30.3%(10/33)、3.0%(1/33)。KPC型、OXA23、OXA51型碳青霉烯酶基因集中分布于特定菌種,且在這些耐碳青霉烯類菌株中占據相當比例。本研究中除1株產酸克雷伯菌產KPC型碳青霉烯酶外,其他13株攜帶KPC型、10株攜帶OXA23、OXA51型碳青霉烯酶基因菌株均分布于肺炎克雷伯菌和鮑曼不動桿菌。13株碳青霉烯類不敏感的肺炎克雷伯菌均攜帶KPC型基因,10株碳青霉烯類不敏感的鮑曼不動桿菌全部攜帶OXA23、OXA51型基因。OXA51型基因普遍存在于鮑曼不動桿菌染色體中,對碳青霉烯類水解能力較弱,通常情況下并不表達活性[14]。近年研究表明,OXA23型基因與鮑曼不動桿菌對碳青霉烯類的耐藥性密切相關。本研究提示攜帶KPC及OXA23型碳青霉烯酶基因是導致肺炎克雷伯菌和鮑曼不動桿菌對碳青霉烯類耐藥的主要機制之一。
近年來臨床分離到耐碳青霉烯類的銅綠假單胞菌逐年增多,其中產金屬β內酰胺酶菌株成為耐藥機制研究中的熱點[15-16]。IMP、VIM、GIM、SPM型金屬酶是耐碳青霉烯類銅綠假單胞菌中最常分離到的亞型[17]。本研究中共分離出13株銅綠假單胞菌,僅1株菌株藥敏試驗顯示對碳青霉烯類不敏感,均未檢測到上述4個亞型的金屬酶。2009年在印度新德里1株肺炎克雷伯菌中分離到一種新的金屬β內酰胺酶,它能水解除氨曲南以外所有β內酰胺類,包括碳青霉烯類,被命名為新德里I型金屬β內酰胺酶(New Delhi Metalloβ-Lactamase, NDM-1)[18]。此后世界各地不斷有產NDM-1酶菌株感染的病例報道,涉及菌種不僅有肺炎克雷伯菌,亦有大腸埃希菌、陰溝腸桿菌、弗勞地枸櫞酸桿菌等其他腸桿菌科細菌,同時在鮑曼不動桿菌、銅綠假單胞菌等不發酵糖革蘭陰性桿菌中檢出更多[19],它的出現再次給全世界敲響了細菌耐藥的警鐘。NDM型碳青霉烯酶基因可以通過質粒轉移到其他細菌DNA,本研究中發現1株產NDM型基因碳青霉烯酶的洛菲不動桿菌,提示NDM型基因碳青霉烯酶在不同菌種之間存在進一步的擴散趨勢。
本研究中耐碳青霉烯類革蘭陰性桿菌對臨床常用的其他抗菌藥物也有相當高的耐藥率,由這些耐藥菌導致的血流感染往往是致命的。雖然替加環素、多黏菌素等對耐藥菌株有較好的抗菌活性,臨床也可嘗試聯合用藥來提高療效[20],但是替加環素血藥濃度偏低,不宜用于血流感染患者,多黏菌素有顯著的腎毒性、神經毒性,聯合用藥會產生不良反應及費用增加等一系列問題,實際應用受到不少限制,療效也未必令人滿意。因此加強醫院內病原菌菌種譜和耐藥性監測,進行耐藥基因篩查及耐藥菌株同源性分析,為醫院感染的防控提供流行病學依據十分必要。醫務人員應重視本地區耐碳青霉烯類革蘭陰性桿菌的分布情況,合理選擇抗菌藥物,加強醫院感染防控從而延緩細菌耐藥性的發展。
[1]RODRIGUEZ-CREIXEMS M, ALCALA L, MUNOZ P,et al. Bloodstream infections: evolution and trends in the microbiology workload, incidence, and etiology, 1985-2006.[J]. Medicine(Baltimore),2008,87(4):234-249.
[2]BARRY AL, JONES RN. Criteria for disk susceptibility tests and quality control guidelines for the cefoperazone-sulbactam combination[J]. J Clin Microbiol,1988,26(1):13-17.
[3]DONG F, XU XW, SONG WQ, et al. Characterization of multidrug-resistant and metallo-beta-lactamase-producing Pseudomonas aeruginosa isolates from a paediatric clinic in China[J]. Chin Med J,2008,121(17):1611-1616.
[4]SHIBATA N, DOI Y, YAMANE K, et al. PCR typing of genetic determinants for metallo-β-lactamases and integrases carried by gram-negative bacteria isolated in Japan, with focus on the class 3 integron[J]. J Clin Microbiol,2004,41(12):5407-5413.
[5]WOODFORD N, ELLINGTON MJ, COELHO JM. Multiplex PCR for genes encoding prevalent OXA carbapenemases in Acinetobacter spp.[J]. Antimicrob Agents,2006,27(4):351-353.
[6]DIEKEMA DJ, BEEKMANN SE, CHAPIN KC, et al. Epidemiology and outcome of nosocomial and community-onset bloodstream infection[J]. Clin Microbiol, 2003,41(8):3655-3660.
[7]李光輝, 朱德妹, 汪復,等. 2012年中國CHINET血培養臨床分離菌的分布及耐藥性[J]. 中國感染與化療雜志, 2014,14(6):474-481.
[8]汪復. 2005年中國CHINET細菌耐藥性監測結果[J]. 中國感染與化療雜志, 2006,6(5):289-295.
[9]胡付品, 朱德妹, 汪復,等. 2013年中國CHINET細菌耐藥監測[J]. 中國感染與化療雜志,2014,14(5):369-378.
[10]Health Protection Agency. Guidance on carbapenem resistance producers[EB/OL]. 2011-01-28[2015-01-28]. http://www. hpa.org.uk/web//HPAweb&Page&HPAwebAutoListName/ Page/1294740725255.
[11]POIREL L, PITOUT JD, NORDMANN P, et al. Carbapenemases:molecular diversity and clinical consequences[J]. Future Microbiol,2007,2(2):501-512.
[12]YIGIT H, QUEENAN AM, ANDERSON GJ, et al. Novel carbapenem-hydrolyzing beta-lactamase, KPC-1, from a carbapenem-resistant strain of Klebsiella pneumoniae[J]. Antimicrob Agents Chemother,2001,45(4):1151-1161.
[13]WALSH TR. Emerging carbapenemases: a global perspective[J]. Int J Antimicrob Agents,2010,11,36 Suppl 3:s8-s14.
[14]WALTHER-RASMUSSEN J, H?IBY N. OXA-type carbapenemases[J]. J Antimicrob Chemother,2006,57(3):373-383.
[15]LEE MF, PENG CF, HSU HJ, et al. Molecular characterization of the metallo-beta-lactamase genes in imipenem-resistant Gram-negative bacteria from a university hospital in southern Taiwan[J]. Int J Antimicrob Agents,2008,32(6):475-480.
[16]ROSSOLINI GM, LUZZARO F, MIGLIAVACCA R, et al. First countrywide survey of acquired metallo-beta-lactamases in gram-negative pathogens in Italy[J]. Antimicrob Agents Chemother,2008,52(11):4023-4029.
[17]WALSH TR, TOLEMAN MA, POIREI L, et al. Metallo-betalactamases: the quiet before the storm? [J]. Clin Microbiol Rev,2005,18(2):306-325.
[18]KUMARASAMY KK, TOLEMAN MA, WALSH TR,et al. Emergence of a new antibiotic resistance mechanism in India, Pakistan, and the UK: a molecular, biological, and epidemiological study[J]. Lancet Infect Dis,2010,10(9):597-602.
[19]BHASKAR E. Clinical correlates of New Delhi metallo-beta lactamase isolates—a survey of published literature[J]. Indian J Med Res,2012,136(6):1054-1059.
[20]LIVERMORE DM. Has the era of untreatable infection arrived? [J]. J Antimicrob Chemother,2009,64 Suppl 1:i29-i36.
Distribution, antibiotic resistance and production of carbapenemases in gramnegative bacteria isolated from blood samples
ZOU Ying, XU Xiaogang, GUO Qinglan, LI Guanghui. (Institute of Antibiotics, Huashan Hospital, Fudan University, Key Laboratory of Clinical Pharmacology of Antibiotics, Ministry of Health, Shanghai 200040,China)
Objective To investigate the distribution, antibiotic resistance and prevalence of carbapenemases genes in gramnegative bacteria isolated from blood samples in Huashan Hospital. Methods Antimicrobial susceptibility testing was carried out according to a unified protocol using Kirby-Bauer method for 132 strains of gram-negative bacteria isolated from March 2012 to February 2013. KPC, NDM, VIM, IMP, GIM, SPM, OXA23, OXA24, OXA51 and OXA58 type carbapenemases genes were identified in 33 carbapenem-nonsusceptible strains by using PCR method. Results The top five bacterial species were Klebsiella spp. (28.0%, 37/132), E. coli (26.5%, 35/132), Acinetobacter spp. (13.6%, 18/132), P. aeruginosa (9.8%, 13/132), and Enterobacter spp. (7.6%, 10/132). Carbapenem resistance genes were identified in 75.8% (25/33) of the carbapenemnonsusceptible strains. The prevalence of KPC, OXA23,OXA51, NDM type carbapenem resistance genes was 42.4% (14/33), 30.3% (10/33), 30.3% (10/33), and 3.0% (1/33),respectively. KPC type carbapenem resistance gene wasidentified in all 13 strains of carbapenem-non-susceptible K. pneumoniae while OXA23 and OXA51 type carbapenem resistance genes were identified in all the 10 strains of carbapenem-nonsusceptible A. baumannii. Conclusions Resistance to carbapenem is increasing in K. pneumoniae and A. baumannii strains isolated from blood samples. KPC and OXA23 type carbapenem resistance genes may be one of the main mechanisms contributing to carbapenem resistance in K. pneumoniae and A. baumannii.
bloodstream infection; carbapenemase; metallo-beta-lactamase; carbapenemase gene血流感染是臨床常見的重癥感染性疾病,具有較高的發病率及病死率,對患者生命威脅極大。近年來,血流感染發病率呈持續增高趨勢[1],與此同時細菌耐藥性也日益嚴重。定期監測細菌耐藥性,開展流行病學調查有助于臨床醫師掌握本地區病原菌的分布及耐藥情況,有助于初始治療選用恰當的抗菌藥物,對于醫院感染的防控也有積極意義。
R378.2
A
1009-7708(2016)02-0214-07
10.16718/j.1009-7708.2016.02.016
2015-06-15
2015-08-16
上海市科學技術委員會醫學引導類科技支撐項目(10411961500)。
1. 復旦大學附屬華山醫院抗生素研究所,衛生部抗生素臨床藥理重點實驗室,上海 200040;
2. 上海市公共衛生臨床中心重癥肝炎科。
鄒穎(1980—),女,碩士研究生,主治醫師,主要從事感染性疾病的診斷與治療。
李光輝,E-mail: liguanghui@fudan.edu.cn。

