阿爾茨海默氏病新易感基因的研究
黃旭程,王瑩,黃智慧
(溫州醫科大學,浙江 溫州,325035,1.檢驗醫學院;2.基礎醫學院)
·綜 述·
阿爾茨海默氏病新易感基因的研究
黃旭程1,王瑩1,黃智慧2
(溫州醫科大學,浙江 溫州,325035,1.檢驗醫學院;2.基礎醫學院)
在阿爾茨海默氏病(AD)新易感基因領域研究中,經典的基于連鎖基因和候選基因的關聯研究已經逐漸被外顯子測序、全基因組測序(針對孟德爾型AD)和全基因組關聯研究(針對非孟德爾型AD)等替代。通過新技術尋找到的新易感基因有助于研究潛在的疾病機制。除了大樣本檢測新風險因素外,新一代測序方法可以對很小數量的患者進行檢測。今后的研究重點將更注重轉化醫學的研究、單個患者的測序和個體患者生物材料的收集,這些將成為遺傳子研究的核心單位。當然這一轉變需要遺傳科學家和臨床神經科醫師的緊密合作。本文對AD遺傳學的最新發現和應用進行綜述。
阿爾茨海默氏病;新易感基因;外顯子測序;全基因組測序;全基因組關聯研究
預計2030年全球將有6 600萬老年癡呆癥患者,2050年患者人數將會高達1.15億。阿爾茨海默氏病(Alzheimer’s disease,AD)是老年癡呆癥患者的主要疾病之一,且研究表明AD患者的病情越重,其認知功能和生活自理能力越差[1]。臨床診斷上可將其分為早發型(<65歲)和遲發型(>65歲)兩類。AD的病理學特征是存在淀粉樣Aβ肽纏結斑塊以及神經細胞中高度磷酸化的微管相關蛋白tau(microtubule-associated protein tau,MAPT)。目前已知,無論是早發型還是遲發型AD都有遺傳因素。迄今在早發型AD中已發現三個突變基因,這三個基因的突變奠定了淀粉樣蛋白在AD中的核心作用,淀粉樣蛋白通路因此成為了AD機制中研究最廣泛的通路。然而,除了常染色體顯性遺傳的家系外,AD的遺傳模式并不是簡單的常規遺傳方式,而是由多種遺傳和環境因素綜合造成的,也就是說更適合定義為一種復雜遺傳背景的疾病。另一方面關于遲發型AD,研究預測其具有高達80%的遺傳力[2]。而先前許多年來的研究只證明一個遺傳風險因子:ApoE ε 4等位基因,明確地參與了AD的發生發展。隨著科學技術的進步,大規模的全基因組關聯研究項目開始出現,能同時識別整個基因組數以百萬計的遺傳變異,因此能迅速識別出遲發型AD中十多個風險基因[3-4]。同時這些風險基因的發現會促使人們去關注其他相關通路,如脂質代謝、免疫系統和突觸功能的機制等。
通過AD全基因組關聯研究的Meta分析有助于闡明更多的危險因素[3-4]。這些新一代的測序方法可以應用于某些小型家系原因不明的AD和一些不相關的患者,同時它擴大了探針的數量,有助于檢出潛在的變異風險。臨床應用這些先進的技術方法對患者生物材料的采樣和遺傳診斷篩查具有重大的意義。我們對AD遺傳學研究進行了綜述,著重討論最新的發現及其應用,以及面臨的挑戰。
1 最新發現的遺傳因子
1.1 復雜基因型AD的新風險位點 長期以來AD的遺傳學研究難以進行,而自2009年以來國際間大型合作的開展已經改變了這個現象,使得近幾年來AD的遺傳學研究取得較大進展。通過歐洲和國際性全基因組關聯的合作,已至少找到九個新的易感基因位點。加利福尼亞路德大學(California Lutheran University,CLU)發布了此結果。此外全基因組關聯和復制研究已經發現補體受體1(complement receptor,CR1)、PICALM和Myc基因盒依賴的相互作用蛋白1(Myc box-dependent-interacting protein 1,BIN1)及其附近位點的單核苷酸多態性。這些基因都是在第一波的大型合作性的全基因組關聯研究中檢測到的,不過仍需要繼續開展合作,確定在跨膜四域蛋白A(Membrane-spanning 4-domains subfamily A,MS4A)集群、CD2相關蛋白(CD2-associated protein,CD2AP)、CD33、EphA1和三磷酸腺苷結合盒轉運子(ATP-bingding cassette A7,ABCA7)中的單核苷酸多態性之間的關聯,AD常見的變異及其作用,見表1[3-4]。
AD國際基因組學研究小組將會繼續補充此列表的內容,此項目匯集了來自全球四個最大的全基因組關聯組織的數據,并用一系列方法進行了分析。例如研究影響發病年齡的遺傳因素、探索基因與整個AD生理途徑的相互作用等。通過與帕金森病和額顳葉變性全基因組關聯研究結果的對比,發現致病因子都是全基因組關聯的最常見的風險基因,但淀粉樣前體蛋白(amyloid precursor protein,APP)、早老蛋白1(presenilin 1,PSEN1)和早老蛋白2(presenilin 1,PSEN2)這三個常見變異并不是復雜型AD的危險因素[4-5]。
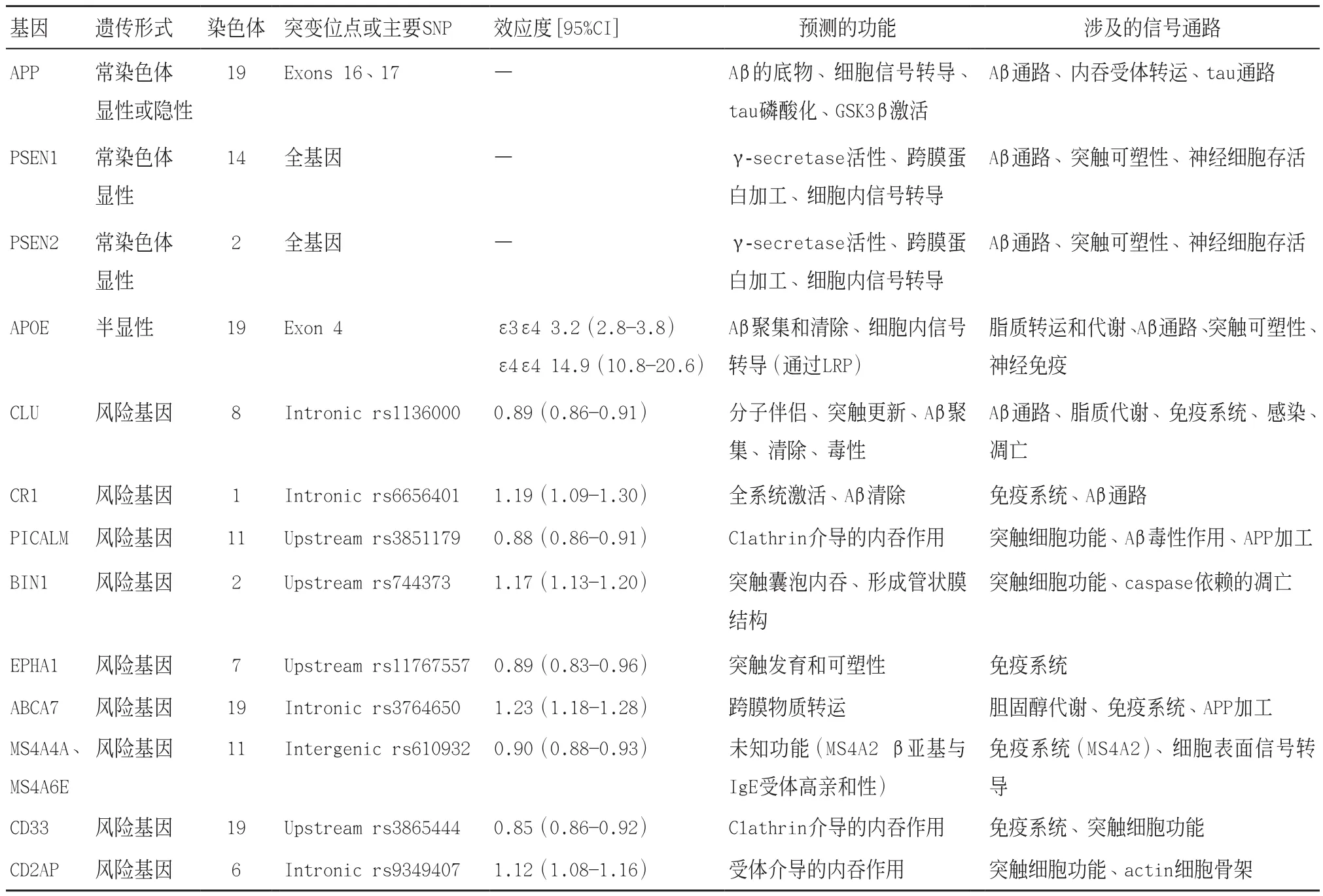
表1 孟德爾形式和非孟德爾形式阿爾茨海默氏癥間的因果關系和風險基因
1.2 早發型AD和孟德爾型AD的風險基因重檢 新一代DNA測序技術的進步給人類基因組測序帶來了前所未有的改革,這使研究者們開始重新審視早發型或家族性AD患者的遺傳子研究,尤其是對于傳統的連鎖分析研究來說規模太小的家庭。對新一代DNA測序技術來說,僅僅對幾個相關患者全外顯子或全基因組的測序比較就足以揭示孟德爾疾病新的致病突變。因此對AD的分子遺傳子來說,其核心應是單個患者而不是家系。到目前為止,已報道了兩個AD患者的全外顯子測序研究。第一個研究意外發現NOTCH3(Homo sapiens notch 3)上的錯義突變與AD有關,這個基因以前一直被認為與常染色體顯性腦動脈病伴皮層下梗死和白質腦病有關[6],因此并沒有作為家系的先證者被篩查出來。同樣,通過對肌萎縮側索硬化癥患者進行全外顯子測序,并在臨床、病理和遺傳學方面與額顳葉變性相互結合研究,揭示了某意大利家庭常染色體顯性遺傳的VCP突變[7]。VCP突變以前曾在包涵體肌病、Paget氏病和額顳葉癡呆癥患者中被檢測到[7]。
我們知道單個基因的突變可以解釋一系列神經退行性疾病的表型,如AD的MAPT Arg406Trp突變、皮克病的PSEN1 Gly183Val突變、AD和帕金森病的顆粒體蛋白(Granulin,GRN)的無義突變和錯義突變、AD的C9orf72重復擴增等,這一發現至今仍具有重要的臨床意義,為遺傳原因不明的AD患者進行所有已知神經退行性基因的診斷篩查提供了根據。由于神經退行性疾病相關基因的數量太大,通過Sanger測序來進行診斷顯然不能實現,而新一代測序技術能夠同時篩選所有參與神經退行性疾病的基因。因此,這種新技術將大大有助于神經退行性疾病的個性化治療,特別是使針對目標分子引起的治療方式變得可行。
第二個在早發型AD患者中進行全外顯子測序的研究表明:在分揀蛋白相關受體-1(sortilinrelated receptor receptor 1,SORL1)上存在錯義突變和無義突變[8],它編碼一種神經元性整合蛋白能結合APP,并將其定位至內涵體回收通路。盡管缺乏SORL1變異體功能性的證據,但編碼SORL1變異體在AD中的高頻出現尤其值得注意,因為這個基因先前被認為是復雜型AD的一個危險因素。這個發現意味著一個基因的致病變異與常規風險變異都可能在疾病中發揮作用。
全外顯子測序是最常被用來研究單基因遺傳孟德爾疾病的方法,主要是因為與全基因組測序相比,外顯子組只占整個基因組1%~2%;不僅成本低,而且大多數序列變化所導致的嚴重表型效應都只位于基因組的編碼部分。然而越來越多的證據表明,非編碼區的變異,如肌萎縮性側索硬化癥的內含子可溶性超氧化物歧化酶(superoxide dismutase1,SOD1)突變,AD的啟動子變異也會增加神經退行性疾病的風險。此外,全基因組測序比全外顯子測序提供了更好的外顯子覆蓋范圍。隨著測序成本的下降和生物信息學技術的推進,全基因組測序將來會廣泛應用。有研究通過對1 795個冰島人全基因組測序數據的研究,在β-分泌酶(β-site APP cleaving enzyme 1,BACE1)酶切位點附近發現一個保護性的錯義突變(ala676thr),與那些沒有這個突變的人相比,AD患者淀粉樣肽形成有所降低,并有更好的認知能力[9]。這一發現支持了一種觀點,即降低BACE1的剪切能夠預防疾病,并可能減少淀粉樣蛋白形成和減弱淀粉樣蛋白毒性作用。
全基因組測序研究仍面臨著巨大挑戰,因為與一個外顯子組(有20 000~50 000單核苷酸多態性)相比,一個基因組約有300萬,因此進行驗證和跟蹤的基因變異數量相當巨大。由于這需要大范圍收集臨床和生物化學表型并進行隨訪,與臨床醫生密切合作顯得尤為重要。目前開展的大型合作,如顯性遺傳性阿爾茨海默網絡(DIAN)聯盟[10]旨在了解罕見的單基因形式的AD,還有歐洲早發型老年癡呆癥協會[11]針對早發型AD患者開展臨床與遺傳轉化研究,這兩個組織的研究工作將在今后發揮重要作用。
2 從間接相關到遺傳病因的確定
2.1 潛在的疾病變異的研究 全基因組關聯研究已經證實了常見的遺傳變異對于復雜AD的作用。風險變異的存在為疾病的病理機制提供了新思路。然而,在這些風險基因位點的關聯變異往往沒有出現明顯的功能性疾病和連鎖不平衡現象(兩個或多個基因位點的等位基因的非隨機聯系)。最相關的單核苷酸多態性位于成簇蛋白(Clusterin,CLU)上,例如某一個內含子的突變起初被認為并不影響CLU的表達或功能,但事實上是CLU最相關的單核苷酸多態性[4]。
仔細研究全基因組關聯位點對于鑒定潛在的風險等位基因很有必要。不僅僅是檢測最顯著的全基因組相關的單核苷酸多態性,而且也應研究在這些位點完整的遺傳變異,包括常見的、罕見的變異和拷貝數變異。至今,大多數遺傳因子的后續研究都是基于第一波全基因組關聯的基因,即CLU、CR1、PICALM和BIN1,只有少數基于第二波全基因組關聯基因。
特別是CLU,很多研究已經證實了其與AD的相關性,是全基因組相關單核苷酸多態性中最顯著的一個[12]。一個常見的多態性連鎖不平衡(顯著單核苷酸多態性)已被認為是可能的功能變異。然而其他的研究并沒有重復出現此相關性,表明這種多態性不能或至少不完全能解釋在CLU上的全基因組關聯性。有研究正在重新對CLU編碼的區域進行不同程度的測序,以確認可能的致病變異。最大的一項研究發現,在AD患者中,CLU的β-鏈外顯子上存在罕見致病變種聚集模式,其次是等位基因頻率<5%、存在非同義替換、9個堿基的插入或缺失,暗示此蛋白亞單位在疾病風險中發揮作用[13]。
與CLU相似,CR1的首次研究也局限于全基因組相關單核苷酸多態性的結果。此單核苷酸多態性位于非編碼序列,沒有明顯的功能相關性。CR1位點的特點是序列高度重復,這使對遺傳危險因素的真實性驗證產生了一定困難。但通過對AD具有易感性的CR1拷貝數的個體變異的檢測找到了AD和功能性拷貝數變異之間的關聯。此功能性的拷貝數決定CR1蛋白的長度,并決定了在補體級聯C3b或C4b和輔因子結合位點的數目[14]。與此拷貝數變異聯合可以解釋早期全基因組關聯研究中所發現的單核苷酸多態性。
而對于四種新發現的AD基因位點,與其關聯的最強信號都在基因上游或下游,這使確定致病變異的難度有所增加,見表1。以PICALM為例,全基因組關聯信號是基因的遠上游,這一發現在后續研究中得以驗證[15],但不一定是編碼PICALM的變異。同樣,在散發性、家族性遲發型AD[15]和全基因組關聯研究的Meta分析[3]中,證實了與BIN1的上游具有關聯性。但強的關聯并不一定意味著這些單核苷酸多態性的因果關系,一個基因的上游非編碼DNA變異也可能對疾病表型有直接的作用,例如在膽固醇的代謝中,分揀蛋白(sortilin 1,sort1)sort1啟動子上游120 kb的一個非編碼變異就能調控該基因的表達。
早先的Sanger測序需耗費大量勞力,且不適用于在基因組非編碼區識別遺傳風險因素。相比之下,新一代測序技術能檢測出參與疾病或對疾病有影響的常見的、罕見的變異以及拷貝數變異,這些檢測已開始應用在復雜性疾病的研究中,難度是要找到合適的算法和足夠大的樣本數據來進行分析。這種結合新一代測序技術、算法和足夠的樣本數據的分析方法,已運用于鑒定與AD相關聯的罕見CLU變異,并證實了這一策略具有卓越的應用價值[13]。此外,新一代測序方法也證實了NCSTN上的罕見變異和遲發型AD之間具有一定的關聯[16],此變異也與家族性早發型AD相關[17]。一項功能性遺傳學研究調查了不同nicastrin單倍體,提出了不同單倍體和NCSTN的單核苷酸多態性在常見AD中的功能差異[18]。一些罕見的變異可能會積累并最終超過疾病的易感性閾值,由于AD的風險基因如CLU[13]可能是罕見的致病變異,這種在等位基因范圍內的轉變需要對所有新的風險基因作進一步的詳細或者針對性的再測序。對于與復雜AD無關的患者,新一代測序研究將在致病基因和風險變異的識別上發揮至關重要的作用。功能學研究將進一步鑒定這些變異的致病作用。
2.2 遺傳變異的生物學效應 那么這些檢測出的遺傳變異是否都具有生物學效應,且其生物學效應都集中在哪些方面呢。事實上,AD的九個新風險基因在功能上具有一定的相關性,大多數都與脂質傳遞假說(CLU和ABCA7)、補體系統、炎癥免疫系統(CLU、CR1、ABCA7、CD33和EphA1)和細胞突觸功能(如細胞內吞作用PICALM、BIN1、CD33和CD2AP)相關[19],見表1。MS4A集群可能作用于信號傳導,具體功能未知。雖然還不能確定其發病機制,但可以據此作出猜想。例如clusterin是一種多功能蛋白質,其疾病相關的變異可能會影響一系列的功能,包括腦內淀粉樣Aβ42的清除、細胞凋亡、膽固醇轉運和炎癥反應[20]。同樣,CR1變異能激活補體反應并減少大腦中淀粉樣斑塊的形成。PICALM基因的遺傳變異可能影響突觸功能,因為PICALM基因能夠調節突觸小泡相關膜蛋白(vesicle-associated membrane protein,VAMP2)轉運,這對突觸小泡的融合及學習記憶是至關重要的[21]。因此AD易感基因并不是隨機發現的,而是基于對AD全基因組關聯數據的信號通路分析得出的,即用生物信息學方法對參與疾病易感性的信號通路進行分析,同時結合全基因組測序中單核苷酸多態性的重要性來共同發現新的信號途徑[19]。目前科學家通過結合生物學預測、功能學分析和患者生物標本研究,來驗證AD的變異模式。研究主要集中在CLU和CR1,見圖1。
第一,調查疾病相關性變異是否影響基因產物的量(在RNA轉錄和蛋白質翻譯水平),及其是否可以作為疾病的早期標志物或潛在的治療靶點。有意思的是發現AD基因在調控區與拷貝數變異區具有很強的關聯性。幾項研究已檢測出CLU蛋白濃度以期判斷CLU的蛋白濃度是否可以用作生物標記物[22],同時檢驗遺傳變異是否與信使RNA和蛋白質濃度相關[22-23]。研究發現AD患者腦血漿和腦脊液中的CLU濃度會明顯增加[24],但蛋白表達和基因水平的變異并不完全一致。下一步需對更大樣本量的AD患者展開研究,開展完整的CLU基因序列并重點關注其異構體,也許能揭示AD風險因子的變異如何真正調控CLU的表達。
第二,深入認識疾病的發病機制,研究已檢測在血液、腦脊液和腦中,遺傳變異對淀粉樣蛋白Aβ 1-42和tau(t-tau和p-tau181)的作用。對CLU、CR1和PICALM的研究指出,PICALM能降低腦脊液中淀粉樣蛋白Aβ 1-42濃度,CR1單核苷酸多態性能增加腦脊液中β淀粉樣蛋白Aβ 1-42濃度[14]。但AD腦脊液生物標志物和全基因組關 聯研究中,并沒有發現與生物標志物濃度相關的新基因位點[25]。因此尚沒有明確的證據顯示基因型與β-淀粉樣蛋白或tau蛋白的表型相關,見表2。這或許是由于研究設計或小尺度效應的問題,長期以來妨礙了AD候選基因的研究,將來真正的風險等位基因的發現可能會得到更強相關性的結果。
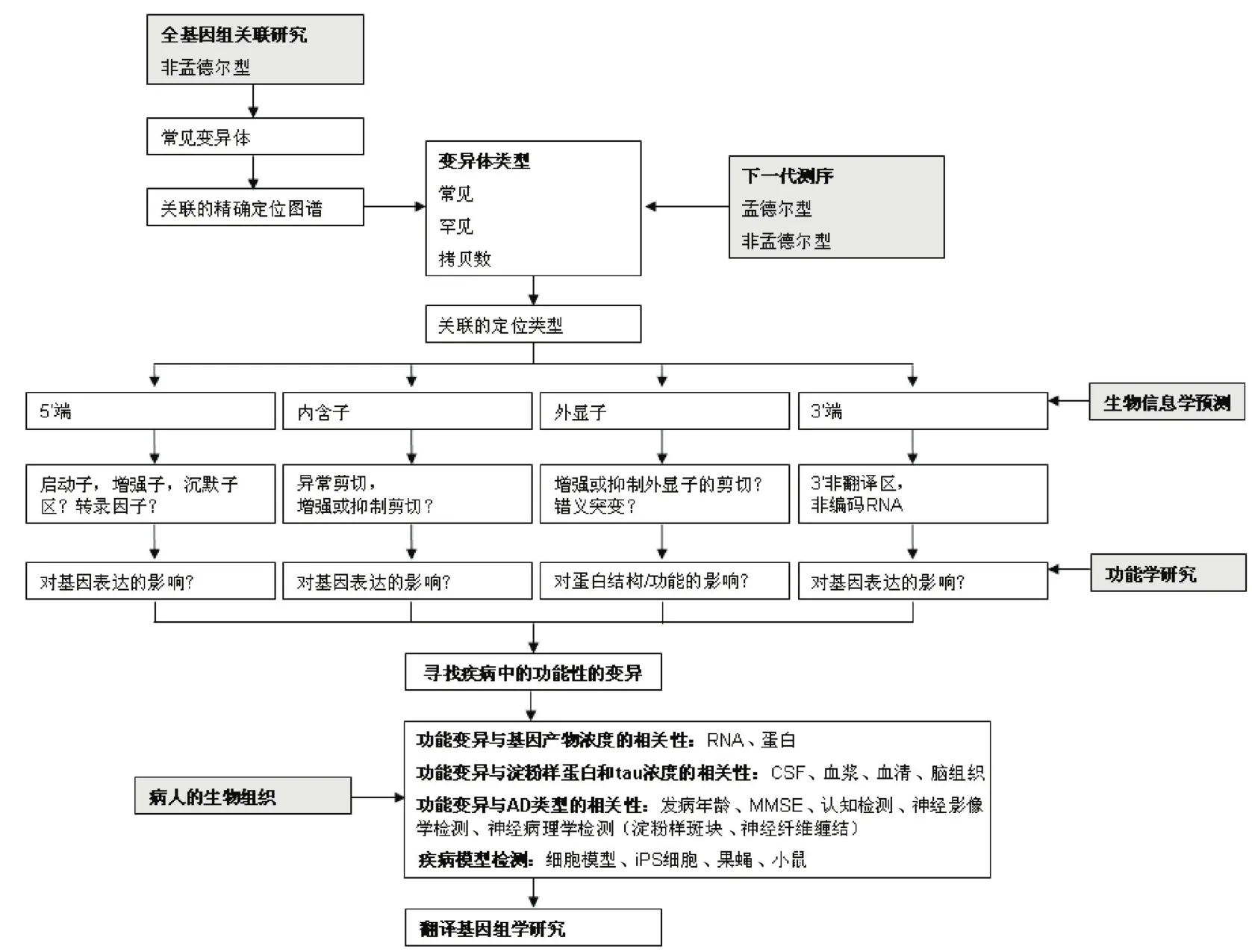
圖1 對AD的遺傳相關性、潛在的功能性變體和生物學功能的鑒定
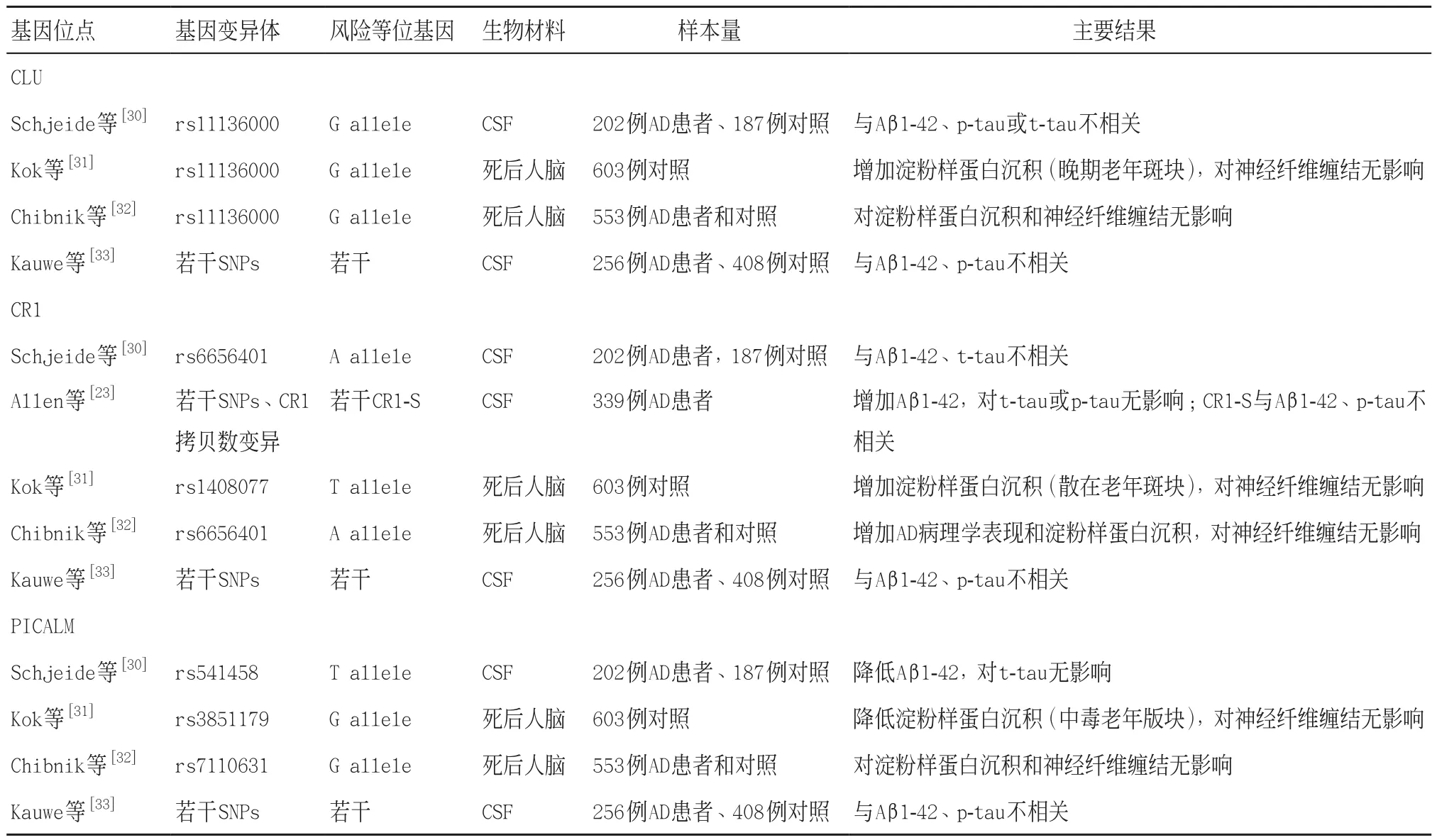
表2 第一波全基因組關聯基因研究發現與阿爾茨海默病相關的生物標志物
第三,通過模式生物研究揭示未知的相關性。例如通過檢測幾種新基因在Aβ和tau通路中的作用,證實了在酵母、線蟲和大鼠皮層神經元中,clathrin介導的內吞基因PICALM、BIN1、CD2AP與淀粉樣β蛋白毒性效應有直接關系[26]。此外,通過模式生物(如果蠅)的功能篩選可以得出人類全基因組關聯研究的結果。通過在體觀察tau轉基因果蠅眼睛的表型,來研究該基因對AD tau的神經毒性[27]。因此,今后應對所有顯著或者可能相關的基因在模式生物系統上進行功能檢測,觀察其對tau和Aβ毒性的作用,這為可能的作用機制提供了依據。
第四,結合相關遺傳表現與內在表型數據分析致病機制。分析相關遺傳表現與內在表型數據(如神經影像學特征、發病年齡、精神狀態和認知能力)之間的關系有助于研究疾病機制。基因位點(CLU、CR1、PICALM和BIN1)對神經影像學相關指標(如內嗅皮質厚度)和AD神經病理學的影響表明:這些性狀至少部分地取決于這些位點的基因組序列,見表3。如果大樣本量能重復此結果,會使這種聯系得到進一步驗證。
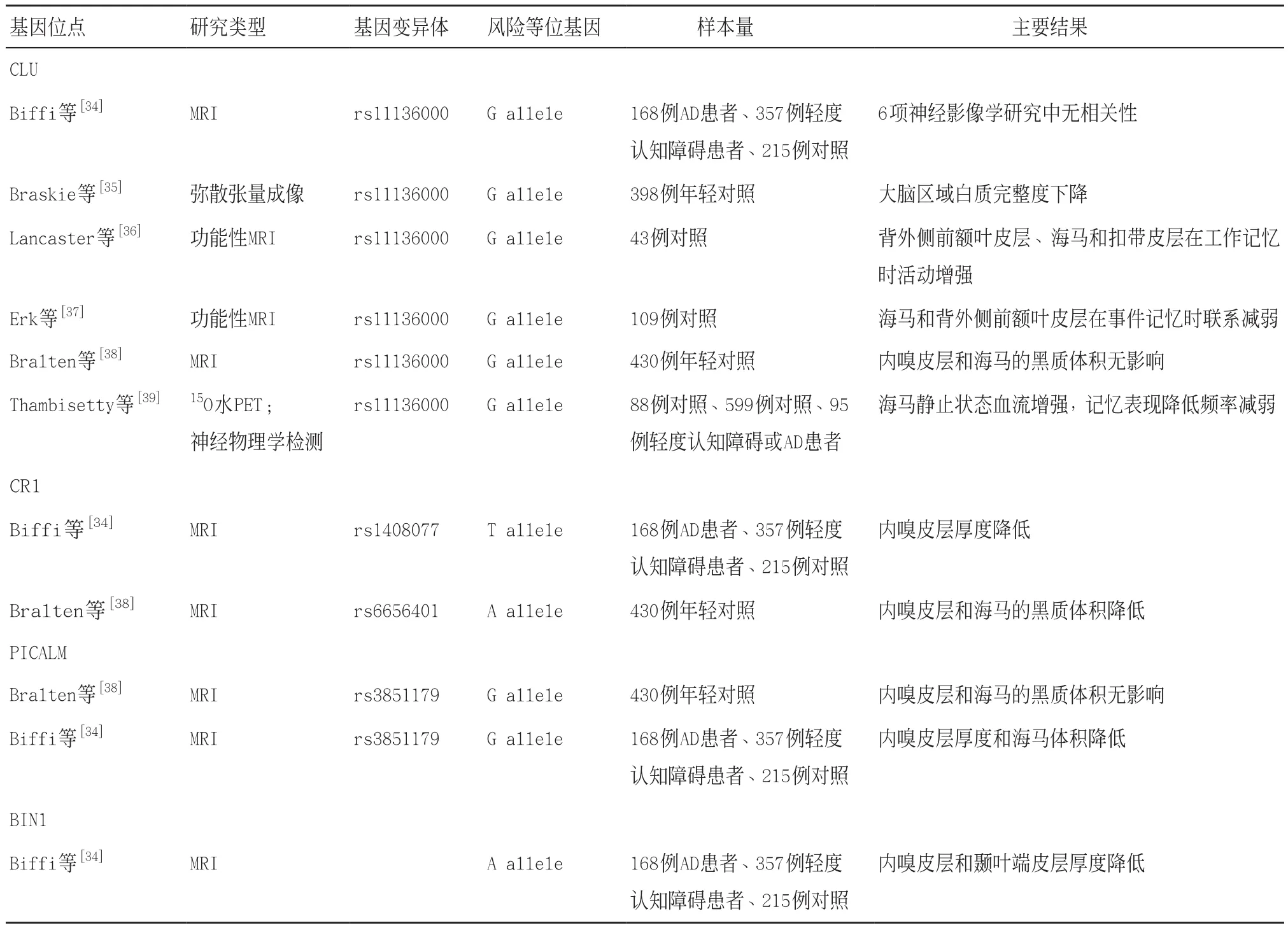
表3 若干第一波的全基因組關聯基因的神經影像學研究
通過全基因組關聯研究尋找遺傳變異對AD神經影像學表型的影響和生物標志物的濃度等的研究已經開展,但新發現的AD全基因組關聯基因幾乎沒有與影響影像學表型的基因重疊,除了除了發現PICALM風險等位基因rs3851179與內嗅皮質厚度的增加有關[28]。一項正在開展的AD神經影像學研究(Neuroimaging Initiative)將會收集和驗證AD疾病中的生物標記物的所有數據,包括血檢和腦脊液檢測,同時結合MRI和PET掃描,將有助于找到遺傳變異和神經影像學表型之間的聯系。采用多國合作的大樣本合并全基因組單核苷酸多態性和結構性MRI數據的方法(經Meta分析聯合神經影像遺傳學研究)有效地確定了遺傳變異與海馬、顱內、全腦體積之間的關聯[29]。此外,國際AD基因組學研究項目(IGAP)的一系列Meta分析(臨床、認知檢測、影像學及生物標志物)將有助于確定影響這些性狀的遺傳變異。一旦在風險基因中檢測到潛在遺傳變異,就能更好地證明其與內表型的相關性。
3 展望
高通量基因組技術的出現已經改變了阿爾茨海默氏病的遺傳變異的研究模式,大隊列的全基因組關聯研究使得人們對復雜的遺傳形式有了深入的認識,至今已經發現9個具有小尺度效應的風險基因。隨著新一代測序方法的應用,包括假說自由的方式(全基因組和全外顯子組測序)和假說驅動的方式(針對全基因組關聯的候選基因再測序)將有助于AD的檢測和開發靶點治療藥物。
[1] 麻小莉, 董樂丹, 傅曄, 等. 中重度阿爾茨海默病患者的神經心理學癥狀研究[J]. 溫州醫學院學報, 2010, 40(6): 536-538.
[2] Gatz M, Reynolds CA, Fratiglioni L, et al. Role of genes and environments for explaining Alzheimer disease[J]. Arch Gen Psychiatry, 2006, 63(2): 168-174.
[3] Hollingworth P, Harold D, Sims R, et al. Common variants at ABCA7, MS4A6A/MS4A4E, EPHA1, CD33 and CD2AP are associated with Alzheimer’s disease[J]. Nat Genet, 2011, 43(5): 429-435.
[4] Lambert JC, Heath S, Even G, et al. Genome-wide association study identifies variants at CLU and CR1 associated with Alzheimer’s disease[J]. Nat Genet, 2009, 41(10): 1094-1099.
[5] Van Deerlin VM, Sleiman PM, Martinez-Lage M, et al. Common variants at 7p21 are associated with frontotemporal lobar degeneration with TDP-43 inclusions[J]. Nat Genet, 2010, 42(3): 234-239.
[6] Guerreiro RJ, Lohmann E, Kinsella E, et al. Exome sequencing reveals an unexpected genetic cause of disease: NOTCH3 mutation in a Turkish family with Alzheimer’s disease[J]. Neurobiol Aging, 2012, 33(5): 1008-1023.
[7] Johnson JO, Mandrioli J, Benatar M, et al. Exome sequencing reveals VCP mutations as a cause of familial ALS[J]. Neuron, 2010, 68(5): 857-864.
[8] Pottier C, Hannequin D, Coutant S, et al. High frequency of potentially pathogenic SORL1 mutations in autosomal dominant early-onset Alzheimer disease[J]. Mol Psychiatry, 2012, 17(9): 875-879.
[9] Jonsson T, Atwal JK, Steinberg S, et al. A mutation in APP protects against Alzheimer’s disease and age-related cognitive decline[J]. Nature, 2012, 488(7409): 96-99.
[10] Bateman RJ, Xiong C, Benzinger TL, et al. Clinical and biomarker changes in dominantly inherited Alzheimer’s disease [J]. N Engl J Med, 2012, 367(9): 795-804.
[11] van der Zee J, Gijselinck I, Dillen L, et al. A pan-European study of the C9orf72 repeat associated with FTLD: geographic prevalence, genomic instability and intermediate repeats[J]. Hum Mutat, 2013, 34(2): 363-373.
[12] Ross OA, Soto-Ortolaza AI, Heckman MG, et al. Association of LRRK2 exonic variants with susceptibility to Parkinson’s disease: a case-control study[J]. Lancet Neurol , 2011, 10(10): 898-908.
[13] Bettens K, Brouwers N, Engelborghs S, et al. Both common variations and rare non-synonymous substitutions and small insertion/deletions in CLU are associated with increased Alzheimer risk[J]. Mol Neurodegen, 2012, 7: 3.
[14] Brouwers N, Van CC, Engelborghs S, et al. Alzheimer risk associated with a copy number variation in the complement receptor 1 increasing C3b/C4b binding sites[J]. Mol Psychiatry, 2011, 17(2): 223-233.
[15] Lambert JC, Zelenika D, Hiltunen M, et al. Evidence of the association of BIN1 and PICALM with the AD risk in contrasting European populations[J]. Neurobiol Aging, 2011, 32 (4): 756-765.
[16] Lupton MK, Proitsi P, Danillidou M, et al. Deep sequencing of the nicastrin gene in pooled DNA, the identification of genetic variants that affect risk of Alzheimer’s disease[J]. PLoS One, 2011, 6(2): e17298.
[17] Dermaut B, Theuns J, Sleegers K, et al. The gene encoding nicastrin, a major gamma-secretase component, modifies risk for familial early-onset Alzheimer disease in a Dutch population-based sample[J]. Am J Hum Genet, 2002, 70(6): 1568-1574.
[18] Hamilton G, Killick R, Lambert JC, et al. Functional and genetic analysis of haplotypic sequence variation at the nicastrin genomic locus[J]. Neurobiol Aging, 2012, 33(8): 1848.
[19] Karolien Bettens, Kristel Sleegers, et al. Genetic insights in Alzheimer’s disease[J]. Lancet Neurol, 2013, 12(1): 92-104.
[20] Nuutinen T, Suuronen T, Kauppinen A, Salminen A. Clusterin: a forgotten player in Alzheimer’s disease[J]. Brain Res Rev, 2009, 61(2): 89-104.
[21] Harel A, Wu F, Mattson MP, Morris CM, Yao PJ. Evidence for CALM in directing VAMP2 traff cking[J]. Traffc, 2008, 9(3): 417-429.
[22] Schurmann B, Wiese B, Bickel H, et al. Association of the Alzheimer’s disease clusterin risk allele with plasma clusterin concentration[J]. J Alzheimers Dis, 2011, 25(3): 421-424.
[23] Allen M, Zou F, Chai HS, et al. Novel late-onset Alzheimer disease loci variants associate with brain gene expression[J]. Neurology, 2012, 79(3): 221-228.
[24] Schrijvers EM, Koudstaal PJ, Hofman A. Breteler MM. Plasma clusterin and the risk of Alzheimer disease[J]. JAMA, 2011, 305(13): 1322-1326.
[25] Kim S, Swaminathan S, Shen L, et al. Genome-wide association study of CSF biomarkers Abeta1-42, t-tau, and ptau181p in the ADNI cohort[J]. Neurology, 2011, 76(1): 69-79.
[26] Treusch S, Hamamichi S, Goodman JL, et al. Functionallinks between Abeta toxicity, endocytic traffcking, and Alzheimer’s disease risk factors in yeast[J]. Science, 2011, 334 (6060): 1241-1245.
[27] Shulman JM, Chipendo P, Chibnik LB, et al. Functional screening of Alzheimer pathology genome-wide association signals in Drosophila[J]. Am J Hum Genet, 2011, 88(2): 232-238.
[28] Furney SJ, Simmons A, Breen G, et al. Genome-wide association with MRI atrophy measures as a quantitative trait locus for Alzheimer’s disease[J]. Mol Psychiatry, 2010, 16 (11): 1130-1138.
[29] Stein JL, Medland SE, Vasquez AA, et al. Identifcation of common variants associated with human hippocampal and intracranial volumes[J]. Nat Genet, 2012, 44(5): 552-561.
[30] Schjeide BM, Schnack C, Lambert JC, et al. The role of clusterin, complement receptor 1, and phosphatidylinositol binding clathrin assembly protein in Alzheimer disease risk and cerebrospinal fuid biomarker levels[J]. Arch Gen Psychiatry, 2011, 68(2): 207-213.
[31] Kok EH, Luoto T, Haikonen S, Goebeler S, Haapasalo H, Karhunen PJ. CLU, CR1 and PICALM genes associate with Alzheimer’s-related senile plaques[J]. Alzheimers Res Ther, 2011, 3(2): 12.
[32] Chibnik LB, Shulman JM, Leurgans SE, et al. CR1 is associated with amyloid plaque burden and age-related cognitive decline[J]. Ann Neurol, 2011, 69(3): 560-569.
[33] Kauwe JS, Cruchaga C, Karch CM, et al. Fine mapping of genetic variants in BIN1, CLU, CR1 and PICALM for association withcerebrospinal fluid biomarkers for Alzheimer’s disease[J]. PLoS One, 2011, 6(2): e15918.
[34] Biff A, Anderson CD, Desikan RS, et al. Genetic variation and neuroimaging measures in Alzheimer disease[J]. Arch Neurol, 2010, 67(6): 677-685.
[35] Braskie MN, Jahanshad N, Stein JL, et al. Common Alzheimer’s disease risk variant within the CLU gene aff ects white matter microstructure in young adults[J]. J Neurosci, 2011, 31(18): 6764-6770.
[36] Lancaster TM, Baird A, Wolf C, et al. Neural hyperactivation in carriers of the Alzheimer’s risk variant on the clusterin gene[J]. Eur Neuropsychopharmacol, 2011, 21(12): 880-884.
[37] Erk S, Meyer-Lindenberg A, Opitz von Boberfeld C, et al. Hippocampal function in healthy carriers of the CLU Alzheimer’s disease risk variant[J]. J Neurosci, 2011, 31(49): 18180-18184.
[38] Bralten J, Franke B, Arias-Vasquez A, et al. CR1 genotype is associated with entorhinal cortex volume in young healthy adults[J]. Neurobiol Aging, 2011, 32(11): 2106.e7-11.
[39] Thambisetty M, Beason-Held LL, An Y, et al. Alzheimer risk variant CLU and brain function during aging[J]. Biol Psychiatry, 2013, 73(5): 399-405.
(本文編輯:吳彬)
R394
CDOI: 10.3969/j.issn.2095-9400.2015.03.018
2014-04-21
溫州醫科大學2013年度校級學生科研立項課題資助(wyx201301035)。
黃旭程(1993-),男,浙江金華人,本科生。
黃智慧,副教授,Email:hzhzju021@163.com。

