前列腺癌TMPRSS2-ETS融合基因的研究進展
陶 晶 綜述 孫忠全 審校復旦大學附屬華東醫院泌尿外科(上海 200040)
·綜 述·
前列腺癌TMPRSS2-ETS融合基因的研究進展
陶 晶 綜述 孫忠全 審校
復旦大學附屬華東醫院泌尿外科(上海 200040)
前列腺癌是男性常見惡性腫瘤,據統計,前列腺癌發病率在世界范圍內位居第二位,其中在歐美國家高居第一位[1]。前列腺癌發病率有明顯的地理和種族差異,歐美國家發病率明顯高于亞洲國家。盡管如此,據統計近年來我國前列腺癌發病率明顯呈現上升趨勢,在北京、上海、廣州三城市均已超過男性膀胱癌的發病率,居男性泌尿生殖系腫瘤發病率第一位[2]。
早期前列腺癌通常沒有癥狀,主要診斷方法包括直腸指檢、超聲、磁共振和血清前列腺特異性抗原(PSA)測定。直腸指檢和經直腸超聲及磁共振診斷前列腺癌敏感度和特異度不高。雖然PSA在前列腺癌篩查、診斷、治療和隨訪中發揮著重要作用,但其缺乏腫瘤特異性,在前列腺的良性疾患中也會升高,在4~10ng/L的PSA灰度區間的敏感度和特異度較差,同時在<4ng/L的正常區間亦存在較高的漏診率[3]。PSA篩查雖然大大提高了早期前列腺癌的發現率,但研究表明大部分前列腺癌是無進展的,PSA篩查造成了對早期低級別前列腺癌的過度診斷穿刺活檢和治療[4]。因此,臨床上需要更加可靠的檢測前列腺癌和判斷預后的生物學因子。自2005年Tomlins等首次報導前列腺癌的跨膜絲氨酸蛋白酶編碼基因TMPRSS2 (transmembrane protease serine 2)與ETS轉錄因子家族成員ERG、ETV1之間發生融合后[5],國內外對融合基因及其蛋白表達在前列腺癌發病機制、檢測、診斷及預后判斷等方面做出了廣泛的研究,揭示了其在前列腺癌的診斷和評估預后方面所具有的潛在價值。
一、ETS家族和MRPSS2基因簡述
ETS家族為一系列擁有與鳥類白血病病毒E26高度保守的同源序列的基因,根據E26(E-twenty six)縮寫將其命名為ETS,已知包括27個家族成員,其中前列腺癌中與TMPRSS2融合的主要包括ERG、ETV1、ETV4、ETV5等[6]。ERG、ETV1和ETV4分別定位于21q22.2、7p21.2及17p21。ETS家族基因的共同特點是含有高度保守的DNA結合域,能夠與特定序列結合調控靶基因的表達。研究表明,ETS蛋白既可作為轉錄激活物,又可作為轉錄抑制物,ETS轉錄因子在正常的細胞增殖、分化、凋亡及腫瘤的血管形成、轉移等生物學行為中扮演重要作用,與尤文肉瘤、白血病、胸腺癌等多種腫瘤相關[7,8]。
TMRPSS2基因為跨膜絲氨酸蛋白酶編碼基因,受雄激素調節,編碼的蛋白屬于Ⅱ類跨膜絲氨酸蛋白[9],對細胞的生長和維持正常形態具有重要作用。該蛋白含有70個氨基酸的N端胞內結構域,30個氨基酸的跨膜結構域,A類低密度脂蛋白受體,富含半胱氨酸的清道夫受體和膜外的C端絲氨酸蛋白酶區域。TMPRSS2基因定位于21q22,研究表明其在前列腺上皮細胞表達明顯高于其他人體組織如結腸、肺、肝臟,且在原發及轉移前列腺癌中高度表達[9]。
二、前列腺癌中TMPRSS2-ETS融合基因的檢測和表達
目前已發現前列腺中存在超過20種融合基因,如3’端的ETS家族其他成員ETV4和ETV5,以及5’端的其他基因伴侶SLC5A3,HERV-K22q11.23,C15orf21和HNRPA2B1等,但其中以TMPRSS2-ERG融合為最常見類型,發生率約為50%,而TMPRSS2-ETV1融合率約為5%~10%,其余各種類則不足1%[6,10,11],因此對TMPRSS2-ERG的相關研究也最多。TMPRSS2與ETS家族融合可誘導特定生化蛋白的合成或對ETS蛋白表達的調節[12],例如TMPRSS2-ERG融合可導致細胞ERG蛋白過度表達,因而可利用特異性的抗體通過免疫組化間接檢測TMPRSS2-ERG融合。表1中列出了部分樣本量較大的關于TMPRSS2-ERG的研究結果,可以發現不同研究中心的檢測結果存在一定差異,而這可能與研究的人種、標本類型、樣本量大小、檢測方法等的不同相關。
表1中數據提示,前列腺融合基因的發生率在不同人種之間存在明顯差異,歐美人種前列腺癌的TMPRSS2-ERG發生率約為50%,且明顯高于亞洲人種。Magi等應用FISH檢測出白種人、非裔美國人和日本人的前列腺癌中TMPRSS2-ERG發生率分別為50.0%、31.3% 和15.9%[25]。鑒于其在亞洲人群發生率較低,因而不具備篩查前列腺癌的作用。目前常用的研究標本包括前列腺癌穿刺和根治標本,部分研究者采用了經尿道電切標本及淋巴結或內臟轉移灶。不同標本的人群分布可能存在差異。穿刺標本多基于PSA篩查,經尿道電切的前列腺標本則多為偶發癌,而根治標本主要屬于早期局限性前列腺癌患者。檢測融合基因主要有三種方法:反轉錄PCR(RT-PCR)、熒光原位雜交(FISH)和免疫組化(IHC)。RT-PCR和FISH能檢測融合基因的融合形式,但操作較困難,結果閱讀困難,耗費也相對較大。IHC檢測則操作簡單,易于推廣,但其不能鑒別融合基因的種類和形式[26,27]。
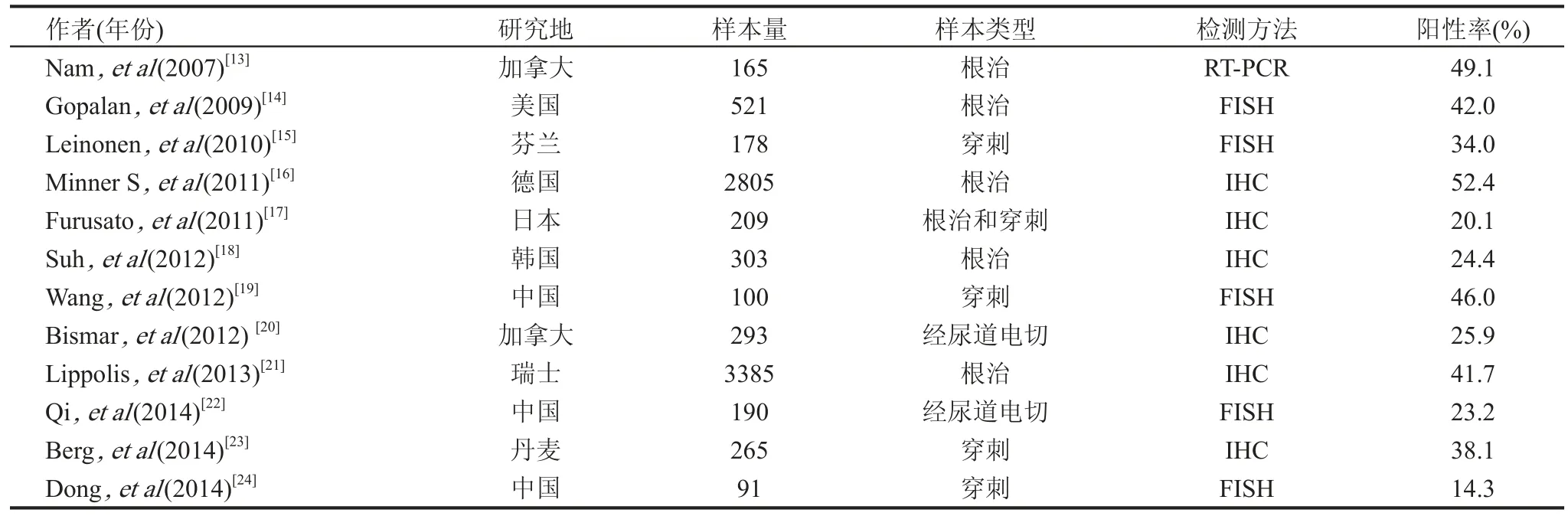
表1 不同研究中心的檢測融合基因TMPRSS2-ERG陽性率的結果
TMPRSS2-ERG融合基因對前列腺癌有較高的特異性。研究表明,ERG基因重排僅發生于前列腺癌和上皮內瘤變(PIN),正常前列腺上皮細胞并不會出現融合基因[28],同時在膀胱癌、腎癌、肺癌等上皮腫瘤中亦未檢測出[29]。各種病理類型的前列腺癌包括小細胞癌都可檢測出融合基因,這對臨床上診斷不明來源的小細胞有重要意義。因此,TMPRSS2-ERG融合基因檢測可作為特異性指標診斷前列腺癌。此外,研究發現在多病灶的前列腺癌標本中,可同時存在融合陽性病灶和陰性病灶,提示不同癌灶可能具有不同的克隆來源[28,30],而轉移灶的融合基因種類一般與原發腫瘤的一致[31]。鑒于外周帶前列腺癌發病率明顯高于移形帶,Falzarano等利用FISH檢測了根治標本中移形帶和外周帶腫瘤的TMPRSS2-ERG發生率,結果分別為12%和34%[32],Wernert等應用PCR檢測20例前列腺癌患者正常外周帶和移形帶的ETS家族成員的表達情況,發現外周帶中ELF-5、ERG、ETV1明顯表達上調[33],這也為研究前列腺發病機制提供了線索。
三、前列腺癌中TMPRSS2-ETS融合的融合機制和形式
TMPRSS2-ETS融合基因的發生機制尚未明確。TMPRSS2和ERG基因位于同一染色體,而TMPRSS2-ERG融合的發生率明顯高于其他家族成員,提示基因組的空間位置相近可能具有重要作用。而TMPRSS2亦可與其他位于不同染色體的ETS家族成員發生融合,這可能是由于細胞在特定的環境下誘導兩者靠近[34]。有研究指出,雄激素受體的激活在基因的融合過程中有重要作用。Lin及Haffner等的研究指出受雄激素受體(AR)誘導的拓撲異構酶Ⅱ(TOP2B)可誘導特定位點的雙鏈DNA斷裂(DNA double-stranded breaks, DSBs),這也是空間靠近的基因組發生融合的關鍵步驟[34,35]。盡管如此,基因融合的過程十分復雜,仍需進一步研究探索。
由于TMPRSS2和ERG基因位于同一染色體,因而TMPRSS2-ERG融合可有缺失(deletion)和易位(translocation)兩種形式:兩個基因組間的區域遺失造成缺失型,或通過復雜的染色體基因重排即易位型(也稱為插入型)(圖1)。各個研究中兩種融合模式的發生率不盡相同,但都提示前列腺癌病灶中存在異質性,在多病灶的標本中可同時存在兩種融合模式。
四、TMPRSS2:ETS融合基因與前列腺癌發病機制的研究
TMPRSS2-ETS家族融合在前列腺癌的發病和進展的作用目前尚未明確。體外細胞學研究顯示ETS基因的過度表達能誘導良性上皮細胞的侵襲性,但并不促發細胞過度增生,同時敲除ERG或ETV1基因的前列腺癌細胞系的侵襲性出現降低[36]。在動物模型體內研究方面,Tomlin和Klezovitch等分別發現TMPRSS2-ETS融合基因能誘導轉基因小鼠前列腺正常上皮細胞出現PIN,即前列腺癌的癌前病變,但不轉化為侵襲性癌[36,37];然而,Chen等發現在同時伴抑癌基因PTEN缺失時,小鼠前列腺可產生侵襲性癌[38]。因此,單獨的TMPRSS2-ETS融合基因似乎不足以誘導前列腺癌,其可能作為前列腺癌的前期重要驅動因素。AR和ERG蛋白的基因組結合域有重疊部分,因而ERG過度表達可干擾和改變雄激素調節基因的表達,Yu等提出ERG過度表達抑制雄激素調節的細胞分化,而通過激活多梳家族蛋白H3K27甲基轉移酶EZH2使細胞去分化[39],與腫瘤形成相關。此外,需要指出的是:不同的ETS蛋白可能通過不同的機制致癌。由于目前對融合基因和前列腺癌發病機制的相關性仍知之甚少,因而需要更多的實驗研究闡明機制機理。
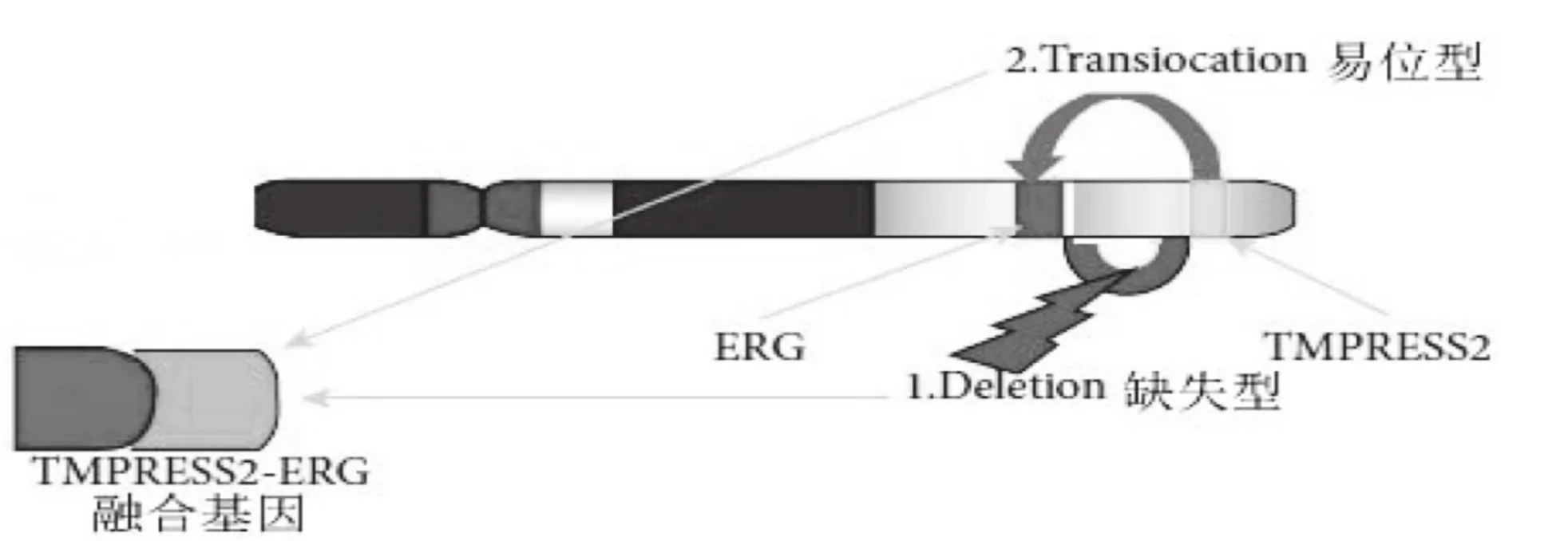
圖1 融合模式注: 1. 缺失型; 2. 易位型
五、TMPRSS2-ETS融合基因與前列腺癌臨床相關性研究及展望
臨床工作者十分重視TMPRSS2-ETS融合基因在前列腺癌臨床診治方面的價值。國內外學者也對TMPRSS2-ETS融合基因與前列腺的臨床指標如發病年齡、PSA、Gleason評分、臨床病理分期等指標的相關性做出了大量的研究,但目前得出的結論存在明顯爭議。以融合基因與Gleason評分的關系研究結果為例,Gopalan等利用FISH檢測521例早期前列腺癌發現TMPRSS2-ERG基因重排與低Gleason評分相關[14],Bismar等研究利用IHC檢測亦發現ERG蛋白高表達與低Gleason評分相關[40],多數研究者提出TMPRSS2-ERG主要分布于Gleason評分6~7分,Darnel等進一步分析在主Gleason分級為3分的前列腺癌更常見[41]。此外,亦有部分研究認為其與Gleason評分無關[22,42],甚至融合基因陽性的前列腺癌疾病進展風險更大。各研究中心的研究方法、樣本選擇和樣本量大小的差異,造成了這些爭議性的報道結果,因此,有關前列腺癌融合基因的臨床價值仍需大量的研究。
前列腺癌是一種明顯的異質性疾病,目前存在主動監測、手術、內分泌治療、放療、化療等多種治療方法,而研究者對于前列腺癌融合基因是否對前列腺癌治療預后具有判斷價值也做出了大量研究,研究結果同樣存在明顯爭議。盡管多數研究結果表明融合基因或ERG蛋白監測對前列腺癌治療預后無判斷價值[14,16,21],但部分研究中心卻有不同發現。一項針對前列腺癌主動監測患者的研究提示ERG蛋白檢測陽性的患者疾病進展風險比陰性患者高[23],亦有研究報道融合基因陽性患者根治術后生化復發風險大[13]。Saramaki等研究150例根治病例發現陽性患者預后優于陰性組[43]。部分研究者利用ERG免疫組化強度判斷預后,Bismar等研究發現ERG蛋白高表達的前列腺癌患者總生存率高[40],但Spencer等研究結果卻與之相反[44]。此外,Attard等[45]提出2條或2條以上染色體上與TMPRSS2融合的ERG5’端的DNA片段缺失而3’端保留(2+Ddel’)的類型提示預后不良,Toubaji等[46]則發現21號染色體擴增對前列腺癌的預后具有判斷價值,而并非是TMPRSS2-ERG融合基因。
前文已述及TMPRSS2-ETS家族融合基因在前列腺癌中較高的發生率以及特異性,因此其在前列腺癌臨床檢測方面有重要意義,可作為臨床前列腺癌特異性指標應用于臨床病理診斷。有研究者提出應用RTPCR檢測尿液中TMPRSS2-ERG融合基因轉錄物,進而無創特異性診斷前列腺癌。Rice等[47]研究顯示檢測尿液中ERG的mRNA對PSA<4ng/mL的前列腺癌患者亦有較好的診斷價值,為PSA處于正常區間而前列腺癌穿刺結果陰性的患者提供了一個很好的參考指標。此外,部分研究者聯合尿液中PCA3檢測及血清PSA提高該方法的敏感度[48]。但對于亞洲人群,其較低的發生率可能限制其尿液篩查前列腺癌的作用。
由于融合基因與前列腺癌的發病分子機制研究尚未明確,有關TMPRSS2-ETS對前列腺癌治療的研究還比較缺乏。Rahim等[49]對YK-4-279進行相關研究,發現YK-4-279能抑制前列腺癌ERG和ETV1的轉錄,進而降低了癌細胞的侵襲性,但是未發生基因融合的癌細胞則對之無反應。Kissick等[50]研究認為TMRPSS2-ERG融合基因可作為研究前列腺癌疫苗的靶點,這些都為前列腺癌的治療提供了新的思路。
六、總結
前列腺癌中TMRPSS2-ETS家族融合基因的發現大大提高了對前列腺癌發病的分子生物學機制及疾病進展的認識,為進一步研究前列腺癌提供了新思路。目前的研究已揭示了TMRPSS2-ETS家族融合基因在前列腺癌中表達的特異性,可作為特異性診斷指標輔助診斷。盡管有關融合基因對前列腺癌的預后判斷價值尚存在爭議,但隨著研究的進一步加深,必然為前列腺癌的臨床治療提供其特有的價值,甚至為前列腺癌提供一種新的治療策略。TMRPSS2-ETS家族融合基因對前列腺癌的臨床診治應具有廣闊的應用前景。
TMPRSS2-ETS; ERG; 前列腺腫瘤;融合基因
1 Center MM, Jemal A, Lortet-Tieulent J, et al. J Eur Urol 2012; 61(6): 1079-1092
2 韓蘇軍, 張思維, 陳萬青, 等. 臨床腫瘤學雜志 2013;18(4): 330-334
3 Tormey WP. J Surgeon 2014;12(6):323-327
4 Daskivich TJ, Chamie K, Kwan L, et al. Cancer 2011;117(10): 2058-2066
5 Tomlins SA, Rhodes DR, Perner S, et al. Science 2005;310(5748): 644-648
6 Rubin MA, Maher CA,Chinnaiyan AM. J Clin Oncol 2011; 29(27): 3659-3668
7 Hollenhorst PC, Mcintosh LP, Graves BJ. Annu Rev Biochem 2011; 80: 437-471
8 Wei GH, Badis G, Berger MF, et al. EMBO J 2010;29(13): 2147-2160
9 Lucas JM, True L, Hawley S, et al. J Pathol 2008; 215(2): 118-125
10 Kumar-Sinha C, Tomlins SA, Chinnaiyan AM. Nat Rev Cancer 2008; 8(7): 497-511
11 Hermans KG, van der Korput HA, van Marion R, et al. Cancer Res 2008; 68(18): 7541-7549
12 Gasi Tandefelt D, Boormans J, Hermans K, et al. Endocr Relat Cancer 2014; 21(3): R143-R152
13 Nam RK, Sugar L, Yang W, et al. Br J Cancer 2007; 97(12): 1690-1695
14 Gopalan A, Leversha MA, Satagopan JM, et al. Cancer Res 2009; 69(4): 1400-1406
15 Leinonen KA, Tolonen TT, Bracken H, et al. Clin Cancer Res 2010; 16(10): 2845-2851
16 Minner S, Enodien M, Sirma H, et al. Clinical Cancer Res 2011; 17(18): 5878-5888
17 Furusato B, van Leenders GJ, Trapman J, et al. Pathol Int 2011; 61(7): 409-414
18 Suh JH, Park JW, Lee C, et al. Korean J Pathol 2012;46(5): 423-428
19 Wang JJ, Liu YX, Wang W, et al. Asian Pac J Cancer Prev 2012; 13(10): 4935-4938
20 Bismar TA, Dolph M, Teng LH, et al. Eur J Cancer 2012;48(4): 538-546
21 Lippolis G, Edsj? A, Stenman UH, et al. Prostate Cancer Prostatic Dis 2013; 16(2): 145-150
22 Qi M, Yang X, Zhang F, et al. PLoS ONE 2014; 9(2): e84959
23 Berg KD, Vainer B, Thomsen FB, et al. Eur Urol 2014;66(5): 851-860
24 Dong J, Xiao L, Sheng L, et al. Asian Pac J Cancer Prev 2014; 15(7): 3099-3103
25 Magi-Galluzzi C, Tsusuki T, Elson P, et al. Prostate 2011;71(5): 489-497
26 Fernández-Serra A, Rubio L, Calatrava A, et al. Biomed Res Int 2013; 2013: 465179
27 Park K, Tomlins SA, Mudaliar KM, et al. Neoplasia 2010;12(7): 590-598
28 Furusato B, Tan SH, Young D, et al. Prostate Cancer Prostatic Dis 2010; 13(3): 228-237
29 Scheble VJ, Braun M, Beroukhim R, et al. Mod Pathol 2010; 23(8): 1061-1067
30 Mertz KD, Horcic M, Hailemariam S, et al. Hum Pathol 2013; 44(12): 2727-2735
31 Guo CC, Wang Y, Xiao L, et al. Hum Pathol 2012; 43(5): 644-649
32 Falzarano SM, Navas M, Simmerman K, et al. Mod Pathol 2010; 23(11): 1499-1506
33 Shaikhibrahim Z, Lindstrot A, Ellinger J, et al. Mol Med Rep 2012; 5(2): 313-316
34 Lin C, Yang L, Tanasa B, et al. Cell 2009; 139(6): 1069-1083
35 Haffner MC, Aryee MJ, Toubaji A, et al. Nat Genet 2010;42(8): 668-675
36 Tomlins SA, Laxman B, Varambally S, et al. Neoplasia 2008; 10(2): 177-188
37 Klezovitch O, Risk M, Coleman I, et al. Proc Natl Acad Sci U S A 2008; 105(6): 2105-2110
38 Chen Y, Chi P, Rockowitz S, et al. Nat Med 2013; 19(8): 1023-1029
39 Yu J, Yu J, Mani RS, et al. Cancer Cell 2010;17(5):443-454
40 Bismar TA, Dolph M, Teng LH, et al. Eur J Cancer 2012;48(4): 538-546
41 Darnel AD, Lafargue CJ, Vollmer RT, et al. Cancer Biol Ther 2009; 8(2): 125-130
42 Teng LH, Wang C, Dolph M, et al. ISRN Urol 2013;2013: 786545
43 Saramaki OR, Harjula AE, Martikainen PM, et al. Clin Cancer Res 2008; 14(11): 3395-3400
44 Spencer ES, Johnston RB, Gordon RR, et al. Prostate 2013; 73(9): 905-912
45 Attard G, Clark J, Ambroisine L, et al. Oncogene 2007;27(3): 253-263
46 Toubaji A, Albadine R, Meeker AK, et al. Mod Pathol 2011; 24(11): 1511-1520
47 Rice KR, Chen Y, Ali A, et al. Clin Cancer Res 2010;16(5): 1572-1576
48 Salami SS, Schmidt F, Laxman B, et al. Urol Oncol 2013;31(5): 566-571
49 Rahim S, Beauchamp EM, Kong Y, et al. PLoS ONE 2011; 6(4): e19343
50 Kissick HT, Sanda MG, Dunn LK, et al. Cancer Immunol Immunother 2013; 62(12): 1831-1840
(2015-05-18收稿)
10.3969/j.issn.1008-0848.2015.08.014
R 737.25

