奶牛α-1,3-巖藻糖基轉移酶基因的克隆表達
姚 晶 , 張陽旸 , 吳正鈞 , 任 婧 *
(1.乳業生物技術國家重點實驗室,光明乳業股份有限公司 技術中心,上海 200436;2.上海海洋大學 食品學院,上海 201306)
α -1,3- 巖 藻 糖 基 轉 移 酶 (alpha-(1,3)-fucosyltransferase,FT)參與Lewis糖鏈合成最后一步的巖藻糖化反應,FT有6類,包括FT-Ⅲ、FT-Ⅳ、FT-Ⅴ、FT-Ⅵ、FT-Ⅶ和FT-Ⅸ,其主要功能是催化GDP-巖藻糖的巖藻糖基轉移至糖鏈中N-乙酰氨基乳糖單位的N-乙酰氨基葡萄糖(GlcNAc)上。Lewis是一組存在于細胞表面糖脂和糖蛋白糖鏈(主要在O-糖鏈,也可在N-糖鏈)上的糖類抗原[1-3]。唾液酸化Lewis(Slex)則是一種細胞粘附分子——選擇素(selectin)的共同糖配體,選擇素主要參與了細胞粘附和遷移,這與細胞的生長發育、組織正常功能的維持、炎癥的發生及腫瘤的形成和轉移等多種生理過程密切相關,但其中的作用機制,如Slex的表達與癌細胞的增殖、轉移、凋亡之間的關系等,并不十分清楚[4-5]。FT是Slex合成過程中必不可少的糖基轉移酶,催化合成反應的最后一步,調節和控制著Slex的表達。由于FT與免疫應答反應、腫瘤的發生發展及轉移等體內生理活動密切相關,國內外學者對于FT的研究多集中在FT的酶活、表達量、參與合成產生的Slex的結構與體內這些生理活動的關系,以及FT基因的調控作用等方面[6-9]。為了研究FT基因的作用,相關學者已經成功克隆了人、鼠、兔等的FT基因,并分別在人表皮癌細胞系A431、小鼠脾細胞、原核表達系統pET30中成功表達;而牛的FT相關基因的克隆表達尚未見報道[10-12]。作者通過對牛的其中一種FT基因開展相關的分子生物學研究,為進一步理解其在體內的生理功能和作用機理,及相關疾病新型藥劑的研發奠定基礎。
1 材料與方法
1.1 菌株和質粒
大腸桿菌Escherichia coli DH5α,為光明乳業股份有限公司技術中心實驗室保存,基因型:supE44ΔlacU169(φ80 lacZΔM15)hsdR recA1 endA1 gurA1 thi-1 relA1。畢赤酵母 GS115,為 Invitrogen公司產品,由復旦大學惠贈。
質粒pMD19-T,為TaKaRa公司產品。畢赤酵母分泌型表達載體pPIC9K,為 Invitrogen公司產品,由上海生物工程有限公司惠贈。
1.2 試劑和儀器
1.2.1 主要試劑 限制性內切酶NotⅠ-HF、AvrⅡ、BalⅡ、Sac I-HF ,NEB 公司產品;T4 DNA 連接酶、溴酚藍指示劑(6x buffer)、X-Gal、硫酸卡納霉素、氨芐青霉素、血液組織細胞基因組提取試劑盒、蛋白loading buffer、標準蛋白Marker,天根公司產品;Taq DNA聚合酶,probest DNA聚合酶,Wide range DNA Marker,DL2000 DNA Marker,TAKARA公司產品;其余常規試劑均為國產分析純。
1.2.2 主要儀器設備 TC-25/H型PCR儀,杭州博日科技有限公司制造;EPS-300A型數顯雙穩電泳儀,上海天能科技有限公司制造;412951型凝膠成像儀,BIO-RAD公司制造。
1.3 FT基因片段的生物信息學分析
1.3.1 基本理化性質預測分析 根據NCBI(www.ncbi.nlm.nih.gov)上公布的FTⅨ的蛋白質序列(獲取 號 :NP_777160.1), 登 錄 http://www.expasy.ch/tools/protparam.html,在線對其相對分子質量、氨基酸組成、等電點及疏水性等基本理化性質進行分析[13]。
1.3.2 蛋白質組學預測分析 利用網站工具軟件PROF 軟件(http://www.predictprotein.org/)和 HNN軟件(http://npsa-pbil.ibcp.fr)預測 FT 的二級結構。利 用 網 站 工 具 軟 件 Scan Prosite (http://www.predictprotein.org/)對FT進行結構域分析。利用網站工具軟件 SWISS-MODEL 軟件(http://swissmodel.expasy.org/)進行建模,預測該酶的三級結構,結果用蛋白質三維圖象軟件java 2.0查看[14-16]。
1.3.3 蛋白質序列的同源性分析與系統進化樹的構建 將氨基酸序列提交至NCBI數據庫中,利用網站工具軟件BLAST軟件(http://blast.ncbi.nlm.nih.gov/Blast.cgi)進行同源性比對分析。進而利用Clustal W軟件進行多序列同源性比對和聚類分析,系統進化樹使用MEGA 5.0軟件的鄰接歸并法(NJ法)構建[17]。 供試物種為:家犬(Canis lupus familiaris,NP_001005380.1),大熊貓(Ailuropoda melanoleuca,XP_002927989.1),黑 猩猩(Pan troglodytes,NP_001 008978.1), 家 兔 (Oryctolagus cuniculus,XP_002714 636.1),家鼠(Mus musculus,NP_034373.1),馬(Equus caballus,XP_001503875.1),褐鼠(Rattus norvegicus,NP_445917.1),鴨嘴獸 (Ornithorhynchus anatinus,XP_001506677.1),短尾猊(Monodelphis domestica,XP_001367612.1),紅原雞(Gallus gallus,NP_00107 2970.1),火雞(Meleagris gallopavo,XP_003204387.1),安樂蜥(Anolis carolinensis,XP_003225370.1)。 還有人(Homo sapiens,CAB41890.1)。
1.4 FT基因片段的擴增
由于奶牛的FT基因不含有內含子,因此可以直接從基因組DNA直接擴增獲得。從荷斯坦奶牛靜脈采血5 mL,分裝置至含有抗凝劑的1.5 mL的離心管中,液氮速凍,后置于-80℃的超低溫冰箱貯存。用血液、組織、細胞基因組DNA抽提試劑盒抽提基因組,并電泳檢測,保存基因組DNA于TE緩沖液中備用,主要操作按照試劑盒說明書。
參考 GenBank(www.ncbi.nlm.nih.gov)數據庫荷斯坦奶牛 α-1,3-巖藻糖基轉移酶Ⅸ(alpha-(1,3)-fucosyltransferase,FTⅨ)編碼區序列,設計引物:
P1:ACTGAGCCTAGGATGACCTCAGCATCCAA AGG;P2:AAGGAAAAAAGCGGCCGCTTAATTCCAA AACCATTTCTCTAAATTACC,進行 PCR,用質量分數0.8%瓊脂糖凝膠電泳檢測PCR產物,并對目的DNA回收純化。
1.5 重組質粒pMD19-FT的構建
將上述PCR產物溶液于質量分數0.8%瓊脂糖凝膠上電泳后,以Marker為參照估算擴增產物的物質的量。在與T載體連接前,首先進行一個加A反應。然后將目的基因片段和載體pMD19-T按摩爾比1∶3~1∶10進行連接反應并轉化。對轉化的重組子進行藍白斑篩選,挑選出目的重組子,并對重組子進行PCR鑒定和雙酶切鑒定。將PCR鑒定與雙酶切鑒定均為陽性的轉化子送至Invitrogen公司進一步測序鑒定。送至Invitrogen公司測序,測序引物為M13(+)、M13(-),對測序結果進行比對分析。
1.6 表達質粒pPIC9K-FT的構建
制備具有雙黏性末端的重組質粒pMD19-FT以及pPIC9K質粒片段,按一定比例加入到含有T4連接酶緩沖液和T4連接酶的體系中,16℃連接過夜,轉化。并按上述方法進行重組子的篩選及鑒定,測序引物為α-factor/3’AOX,并根據測序結果,對目的基因的序列及位置進行驗證。
1.7 重組酵母GS115-FT轉化子制備及誘導表達
抽提鑒定正確的重組質粒pPIC9K-FT,電轉化至P.pastoris GS115感受態細胞,電轉化電壓1.5 kV,電擊時間5 ms。電擊結束后取900 μL山梨醇溶液混勻菌體并移至1.5 mL離心管中,30℃靜置培養1h。取200 μL轉化液涂布至MD平板,30℃培養2~3 d至出現His+轉化子。
待轉化子長至肉眼可見時,將轉化子菌落編號并一一對應地點在含有G418硫酸鹽質量濃度為2 mg/mL的MD和MM平板上,30℃培養2~3 d,篩選在MD、MM平板上都正常生長的轉化子His+Mut+。隨機挑取5個His+Mut+轉化子,用Lysis buffer for microorganism to direct PCR試劑進行菌落PCR,PCR 引物為 α-factor/3’AOX。
取鑒定His+Mut+轉化子接種至100 mL的BMGY培養基中,30℃ 250 r/min,培養至OD600=2.0~6.0。3 000 g離心5 min收集菌體,用BMMY培養基重懸并稀釋至OD600=1.0,分別進行誘導培養24、48、72 h,以 1%(體積分數)甲醇為誘導劑進行誘導表達。蛋白質樣品15 000 r/min離心2 min收集上清液,取1 mL置液氮中速凍并保存于-80℃。添加甲醇至 V(上清)∶V(甲醇)=1∶1,4 ℃靜置 1 h,12 000 r/min 離心 5 min,沉淀復溶于 100 μL ddH2O,質量分數12%SDS-PAGE檢測蛋白質表達情況。
2 結果與分析
2.1 FT基因的生物信息學分析
2.1.1 FT基因基本理化性質 FT cDNA為1 101 bp,有1個ORF(堿基序列9-1088),不含有內含子。通過其序列的在線分析發現,FT的化學式為C1930H2885N489O535S15,原子總數為5 854,相對分子質量為41 978.9,包括359個氨基酸殘基,等電點理論值為7.61,負電荷殘基(Asp+Glu)總數為34,正電荷殘基(Arg+Lys)總數為35,不穩定系數為45.20,故認為該蛋白質為不穩定蛋白質。脂肪系數為80.33,親水性評估為-0.275。依據氨基酸分值越低親水性越強和分值越高疏水性越強的規律,可見該酶為親水性蛋白質。該酶的氨基酸組成如表1所示,其中相對含量比較多的氨基酸為Ser(31個,占8.6%),含量比較少的氨基酸是Tyr(6個,占1.7%)。其在哺乳動物體內的半衰期預測值為30 h,在酵母菌中則>20 h,在大腸桿菌中為>10 h。
2.1.2 FT基因的蛋白質組學分析 通過PROF和HNN軟件對FT進行二級結構的預測,結果如表2所示。從預測的結果可以看出,兩種方法所得出的二級結構中,包含的α螺旋基本一致,而β折疊、自由卷曲則有一定差別。利用ScanPROSITE對FT的功能域分析發現,它有3個N糖基化位點,1個cAMP和cGMP依賴的蛋白激酶磷酸化位點,6個蛋白激酶C磷酸化位點,3個酪蛋白激酶Ⅱ磷酸化位點,1個酪氨酸激酶磷酸化位點以及2個N豆蔻酰基化位點,如表3所示。利用SWISS-MODEL軟件預測的FT的三維結構如圖1所示。
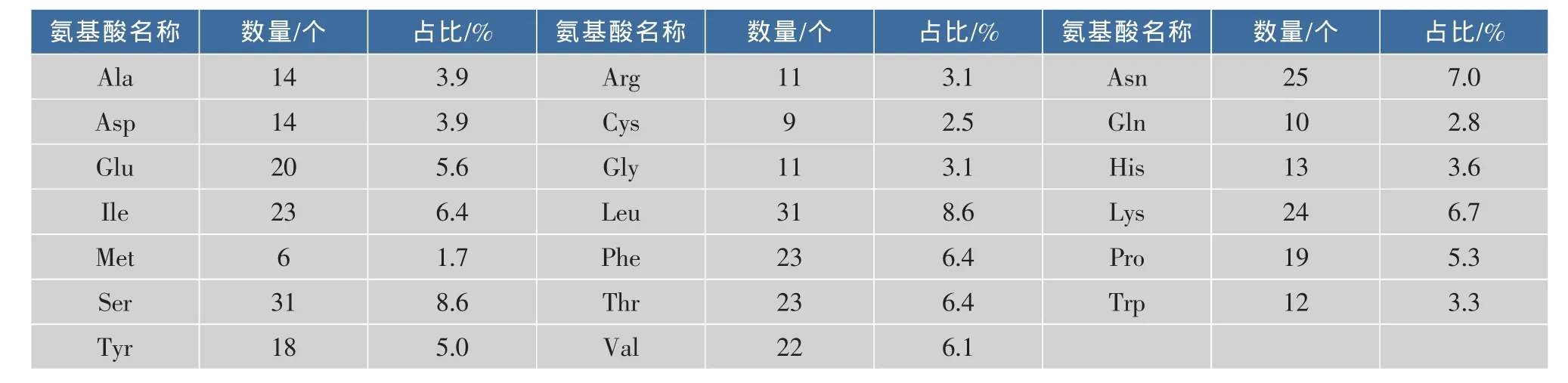
表1 FT的氨基酸組成Table 1 Amino acid composition of alpha-(1,3)-fucosyltransferase Ⅸ
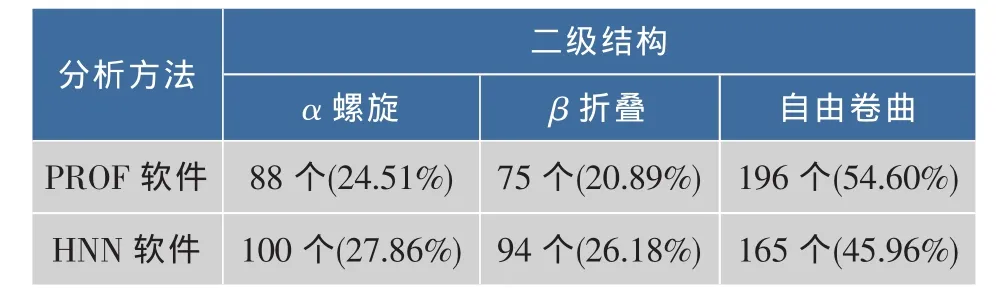
表2 FT二級結構分析Table 2 Predicted secondary structure of alpha-(1,3)-fucosyltransferaseⅨ
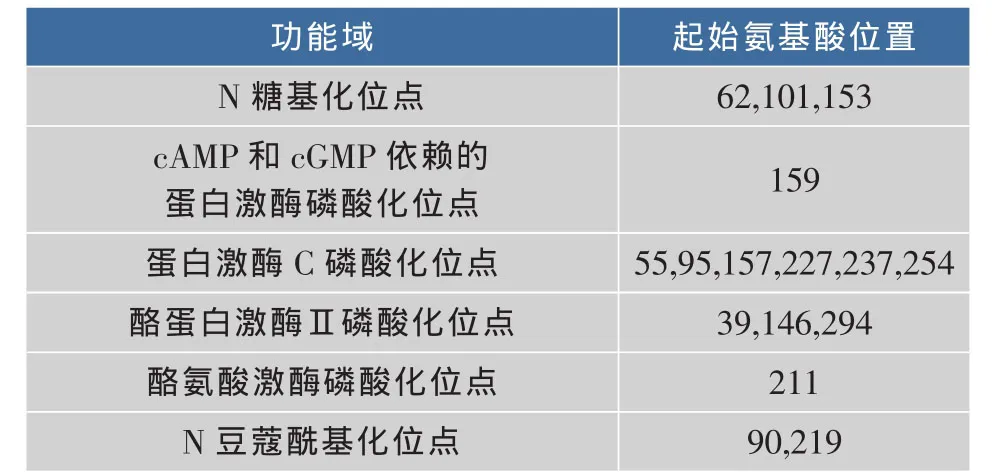
表3 FT蛋白功能域分析Table 3 Predicted protein function of alpha-(1,3) -fucosyltransferaseⅨ
2.1.3 蛋白序列同源性分析與系統進化樹的構建將蛋白質序列在NCBI上進行BLAST,通過多重序列比對,發現和供試物種的同源度都非常高。通過MEGA 5.0建立的系統進化樹如圖2所示,可以看出,供試的14個物種的FT被聚成2大類:紅原雞、火雞和安樂蜥的FT為一類。其中雉科的紅原雞、火雞為一支。褐鼠、家鼠等其他11個哺乳動物的FT聚為另一類。其中,短尾猊的FT自為一支,其他10個的FT聚為一支。在此10個物種的FT中,鴨嘴獸的FT為一分支,其他9種聚為另一分支。這9個物種FT又分為2個分支:褐鼠、家鼠、黑猩猩和家兔,以及人的FT聚為一個分支,其中家兔又自成一分支,褐鼠和家鼠聚為一組,黑猩猩和人聚為一組,兩兩組成另一個分支;大熊貓、家犬、馬和牛的FT聚為另一分支,其中大熊貓自成一支,馬和牛的FT組成的小分支和家犬聚為另外一分支。
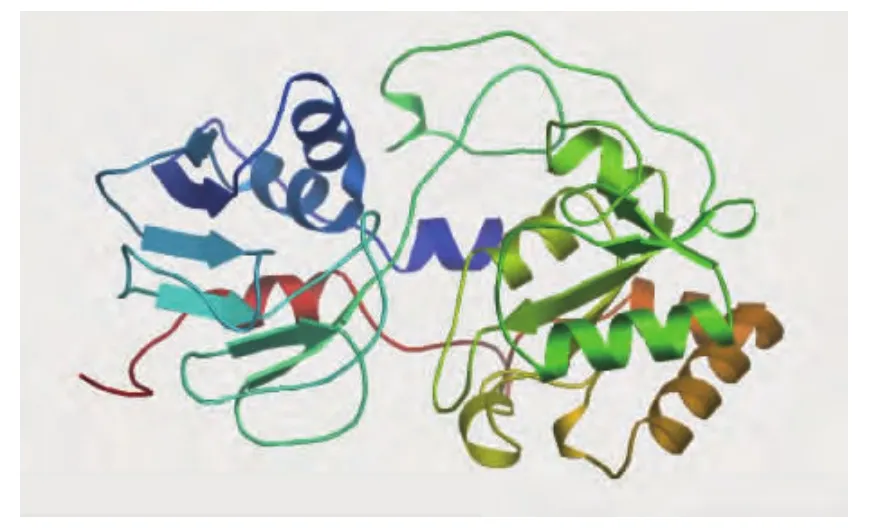
圖1 SWISS-MODEL預測的FT三維結構Fig.1 Predicted 3D structure of alpha-(1,3) -fucosyltransferaseⅨby SWISS-MODEL
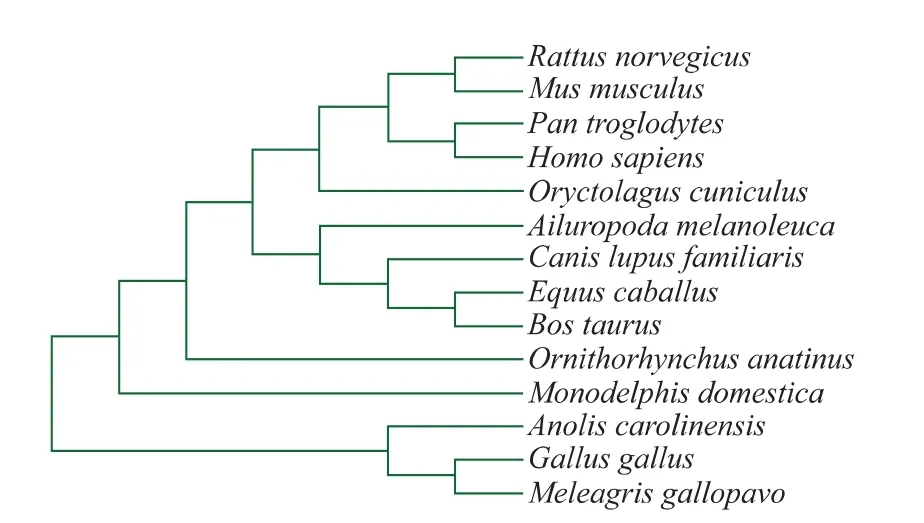
圖2 FT的系統發育樹Fig.2 Phylogenetic tree of alpha-(1,3)-fucosyltransferaseⅨ
2.2 荷斯坦奶牛基因組DNA的提取和目的基因的擴增
為得到奶牛α-1,3-巖藻糖基轉移酶的相關基因,根據GenBank數據庫中報道的巖藻糖基轉移酶Ⅸ基因設計PCR引物,并結合表達載體引入相關酶切位點。用血液、組織、細胞基因組DNA抽提試劑盒按照試劑盒說明書抽提基因組,并進行電泳檢測,如圖3所示,可以看出,基因組DNA條帶清晰、整齊,說明該試劑盒提取的基因組DNA純度較高,品質較好。保存基因組DNA于TE緩沖液中,作為擴增目的基因的模板備用。
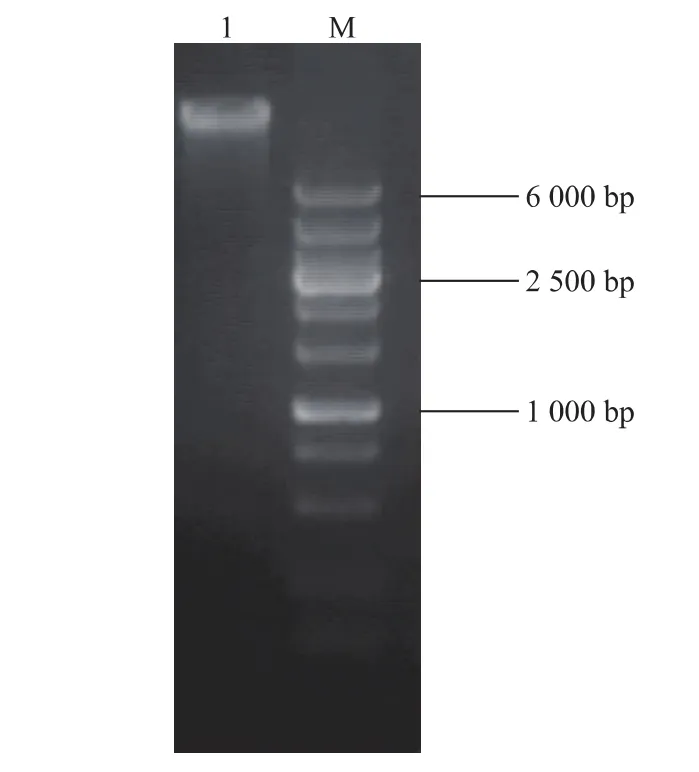
圖3 基因組DNA電泳結果Fig.3 Electrophoresis result of genomic DNA
將上步抽提的基因組DNA作為模板,加入到50 μL PCR體系中進行PCR反應,電泳檢測在約1.1 kb處出現目的條帶,如圖4所示。
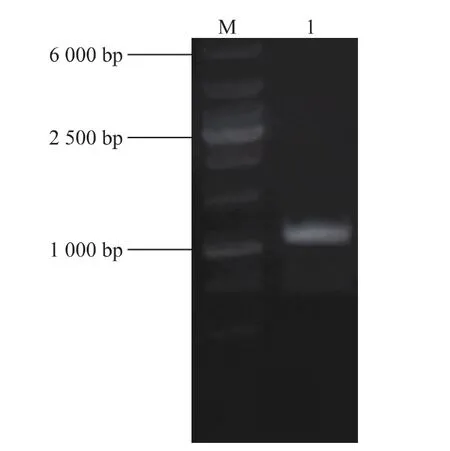
圖4 PCR產物電泳圖Fig.4 Electrophoresis result of PCR product
2.3 重組質粒陽性克隆篩選及鑒定
上步PCR擴增產物經回收試劑盒純化后,進行加A反應。按照載體與插入片段摩爾比約為1∶3~1∶10,進行T載體連接。連接產物轉化至E.coil DH5α感受態細胞,利用藍白斑篩選轉化子。經過夜培養,平板上可明顯觀察到藍白斑。對平板上正常生長的白色菌斑隨機挑取5個,進行PCR鑒定,結果如圖5所示,發現①號、②號菌落為陽性克隆。繼而將②號菌落進行雙酶切鑒定,可以得到較好的酶切結果(圖6)。將②號菌保種后送樣測序分析。將測序結果與NCBI公布的FT基因比對,結果發現PCR產物發生了一個堿基突變。通過多次重復實驗,發現在同一位點均發生相同的堿基突變,由于本實驗中PCR模板為中國荷斯坦乳牛,而genebank中公布的序列來源為美國乳牛,推測這個位點的突變屬于個體差異。至此,構建出重組質粒并命名為pMD19-FT。

圖5 重組質粒的PCR鑒定Fig.5 PCR analysis of the DH5α
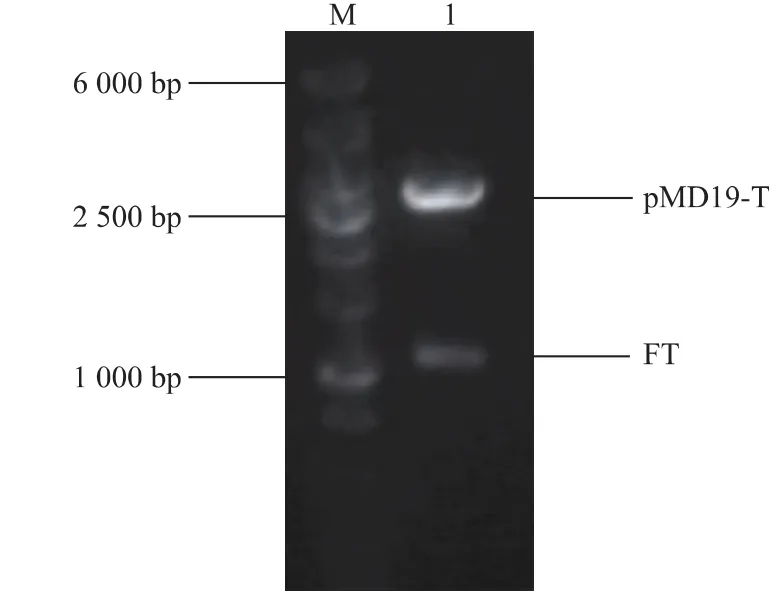
圖6 ②號菌質粒的雙酶切圖Fig.6 Restriction endonuclease of pMD19-FT
2.4 表達載體的構建及鑒定
將含有FT編碼區序列的重組質粒pMD19-FT和表達載體pPIC9K雙酶切后,取適量于16℃過夜連接,轉化到E.coil DH5α。用通用引物α-factor與3’AOX對挑取的菌落進行PCR鑒定,如圖7所示,①號菌、②號菌可能為陽性克隆。
將①號菌質粒進行雙酶切鑒定,測序驗證,結果為陽性,證明目的基因FT已正確連接到表達載體pPIC9K中,說明已成功構建表達載體pPIC9K-FT。
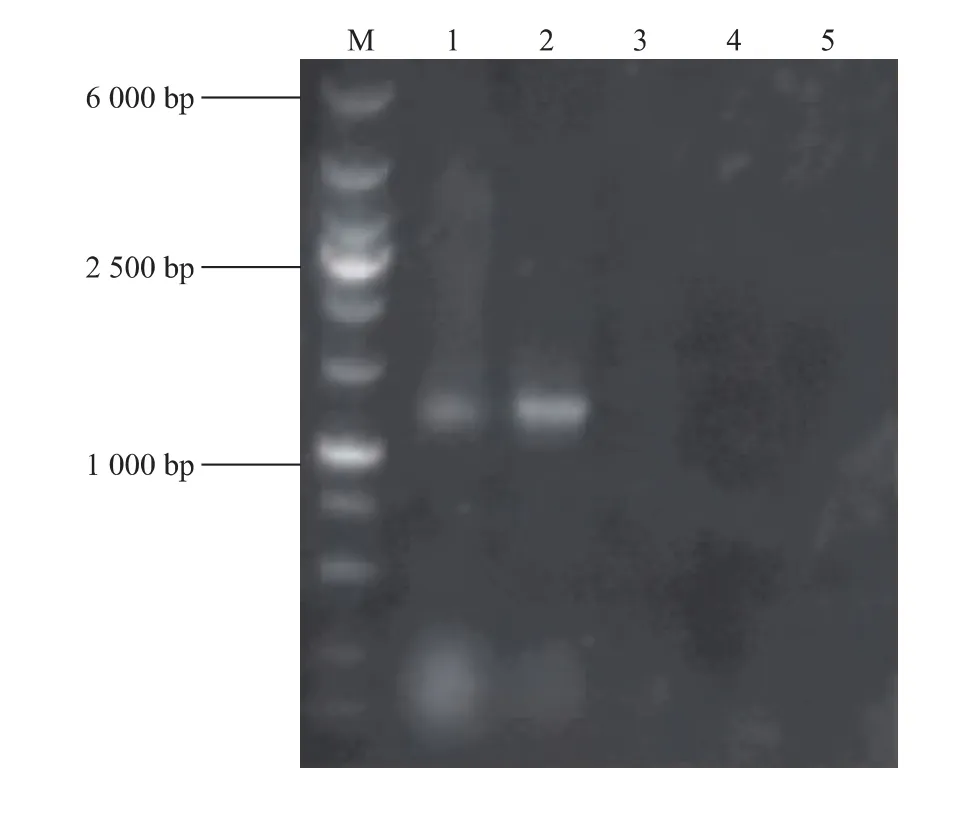
圖7 重組質粒的PCR鑒定Fig.7 PCR analysis of the DH5α
2.5 重組酵母的篩選鑒定及其誘導表達
大量制備pPIC9K-FT,用限制性內切酶Sac IHF將其線性化并濃縮至0.5~1.0 μg/μL。將線性化質粒電轉化至宿主菌P.pastoris GS115中,篩選有G418抗性的His+Mut+轉化子,并進行PCR鑒定。挑選在MD、MM平板上長得最快的G418抗性的His+Mut+轉化子,經G418終質量濃度為2 mg/mL的BMGY培養至OD600=2.0~6.0時,收集菌體用G418終質量濃度為2 mg/mL的BMMY稀釋至OD600=1.0,再進行誘導培養。并對目的基因蛋白質進行誘導表達,結果如圖8所示。在約45.0 kDa處,有一條明顯的蛋白質條帶,其相對分子質量與預測值相符,而此處陰性對照無此條帶,因此可以判斷此條帶為目的基因FT可以在宿主菌P.pastoris GS115中成功表達,并可以分泌至胞外,而且隨著培養時間的增加,目的蛋白質含量也不斷積累。
3 結語
依據NCBI上的相關序列,對奶牛FT基因進行了生物信息學的分析,通過在線預測可知,目的蛋白質FT為一種可溶性穩定蛋白質,具有糖基化、磷酸化、酰胺化等多個功能位點。利用蛋白質分析軟件SWISS-MODEL對其三維結構進行了建模預測。通過對FT基因的生物信息學分析,對主要理化性質及蛋白質結構進行了預測,這對其克隆表達、分離純化等后續相關工作的開展具有一定的指導意義。將FT編碼區序列連接到重組質粒pMD19-T載體上并轉化至E.coil DH5α中,對轉化子進行PCR鑒定、雙酶切鑒定及測序鑒定,證明了此段克隆基因的正確性。在此基礎上進一步構建了真核表達系統P.pastoris GS115-FT,并對其進行誘導表達,通過SDS-PAGE進行了目的檢測,結果顯示目的蛋白質的相對分子質量大小與預測值相符,且能夠成功分泌到胞外并隨之累積,證實了之前生物信息學分析的部分結果是可靠的。但是,有蛋白質的產生并不代表蛋白質就是有活性的。如果在表達過程中,重組蛋白質由于糖基化等后修飾需要或者經過折疊等導致了酶學性質的變化或者喪失,對其今后的開發利用都會產生巨大的影響。因此,后續的工作將重點研究所表達重組蛋白質的酶學性質,進一步證實其中一個點突變來源于個體差異的推測,及其他生物信息學的分析結果的可靠性。在證實重組蛋白質相關酶學性質后,進一步優化發酵條件,以期在發酵周期內獲得最大的生物量,為后續的開發利用創造條件。
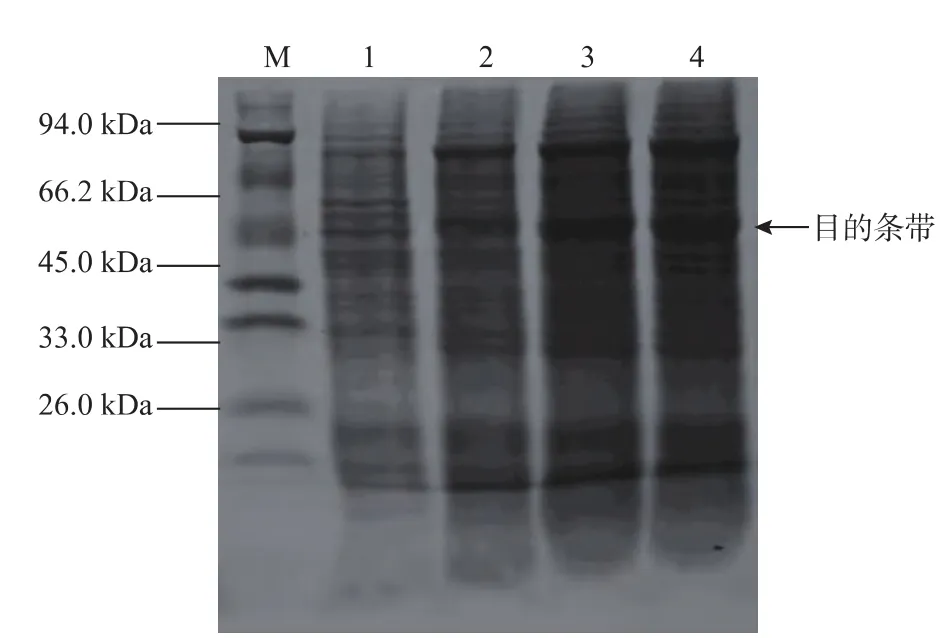
圖8 GS115-GT不同時間的SDS-PAGE圖譜Fig.8 SDS-PAGE analysis of expression by GS115-FT
作者利用分子生物學技術成功克隆了奶牛的FT基因,并在真核系統中可溶性表達,這為FT的大量制備提供了可能,為后續的科研工作,如酶學性質、Slex的生物合成及相關生理、病理機理等方面的深入研究打下了良好的基礎。
[1]陳惠黎.糖復合物的結構和功能[M].上海:上海醫科大學出版社,1997:54-270.
[2]Becker D J,Lowe J B.Fucose:Biosynthesis and biological function in mammals[J].Glycobiology,2003,13(7):41-53.
[3]Javaud C,Dupuy F,Maftah A,et al.The fucosyltransferase gene family:an amazing summary of the underlying mechanisms of gene evolution[J].Genetica,2003,118(2-3):157-170.
[4]Munro J M.Expression of sialyl-lewis X an E-selectin ligand in flammation,immune processes and lymphoid tissues[J].American Journal of Pathology,1992,141:1397-1408.
[5]Wang Q Y,Guo P,Duan L L,et al. α1,3-Fucosyltransferase-Ⅶ stimulates the growth of hepatocarcinoma cells via cyclin dependent kinase inhibitor p27Kip1[J].Cell Mol Life Sci,2005,62:171-178.
[6]Miyashiro M,Furuya S,Sugita T.Development of a sensitive separation and quantification method for sialyl Lewis X and Lewis X involving anion-exchange chromatography:biochemical characterization of alpha 1,3 fucosyltransferase Ⅶ [J].J Biochem,2004,136(5):723.
[7]吳興中,陳迎風.巖藻糖糖鏈與肝癌細胞的遷移作用[J].生物化學與生物物理進展,2002(6):932-937.WU Xingzhong,CHEN Yingfeng.Fucosylated ogligosaccharides in the migration of hepatoma cells[J].Progress in Biochemistry and Biophysics,2002(6):932-937.(in Chinese)
[8]李文樺,張文.α1,3巖藻糖基轉移酶Ⅶ和糖蛋白CD24與腫瘤轉移的研究進展[J].中國癌癥雜志,2008,18(10):790-795.LI Wenhua,ZHANG Wen.Advance in the study of α1,3 fucosy1transferase Ⅶ and glycoprotein CD24 in tumor metastasis[J].China Oncology,2008,18(10):790-795.(in Chinese)
[9]Elmgren A,Mollicone R,Costache M,et al.Significance of individual point mutations,T202C and C314T,in the human Lewis(FUT3) gene for expression of Lewis antigens by the human alpha (1,3/1,4)-fucosyltransferase,Fuc-TIII[J].The Journal of Biological Chemistry,1997,272(35):21994-21998.
[10]楊雪松,賈爽,張震波,等.人α1,3-巖藻糖基轉移酶IV熒光真核表達載體的構建及鑒定[J].世界華人消化雜志,2008,16(10):1064-1068.YANG Xuesong,JIA Shuang,ZHANG Zhenbo,et al.Construction and identification of human fucosyltransferase 4 eukaryotic fluorenscent expression vector[J].World Chinese Journal of Digestology,2008,16(10):1064-1068.(in Chinese)
[11]Elizabeth M S, Frances I S, Gherman W,et al.Cloning of a rat α1,3-fucosyltransferase gene:a member of the fucosyltransferase IV family[J].Glycoconjugate Journal,1997,14(2):249-258.
[12]哲名家,張淼濤,云濤,等.兔巖藻糖基轉移酶基因的克隆與原核表達[J].西北農林科技大學學報:自然科學版,2012,40(4):21-24.ZHE Mingjia,ZHANG Miaotao,YUN Tao,et al.Cloning and prokaryotic express of rabbit fucosyltransferases[J].Journal of Northwest A&F University:Nat Sci Ed,2012,40(4):21-24.(in Chinese)
[13]Baxevanis A D,Ouellette B F F.生物信息學[M].李衍達,孫之榮,譯.北京:清華大學出版社,2000:231-250.
[14]Arnold K,Bordoli L,Kopp J,et al.The SWISS-MODEL workspace:A Web-Based environment for protein structure homology modelling[J].Bioinformatics,2006,22:195-201.
[15]Schwede T,Kopp J,Guex N,et al.SWISS-MODEL:An automated protein homology-modeling server[J].Nucleic Acids Research,2003,31:3381-3385.
[16]Guex N,Peitsch M C.SWISS-MODEL and the Swiss-PdbViewer:An environment for comparative protein modelling[J].Electrophoresis,1997,18:2714-2723.
[17]Mount D W.生物信息學[M].鐘揚,王莉,張亮,譯.北京:高等教育出版社,2003:301-345.

