城市生活污水處理廠活性污泥中細菌群落結構組成研究
王 猛,李如剛,肖毅宏,邢夢雅,雷雙舟,徐佳寧,徐 欣,趙大勇
(1.河海大學水文水資源學院,江蘇 南京210098;2.無錫錢惠污水處理有限公司,江蘇 無錫214151)
活性污泥是微生物群體及它們所依附的有機物質和無機物質的總稱,具有由多種細菌組成并通過胞外聚合物和離子橋作用形成的絮狀結構。細菌是活性污泥組成和凈化功能的核心,也是生物法處理工業和城市生活污水的主體[1,2]。了解活性污泥中細菌的群落結構組成及多樣性,使其中的微生物更好地發揮凈化作用,對于進一步提高污水處理效率具有重要意義。
細菌群落多樣性的傳統研究方法是分離培養、顯微鏡觀察,這類方法不足以從整體上掌握細菌群落結構組成以及多樣性信息。隨著分子生態學技術的發展,新興的分子生物學技術如變形梯度凝膠電泳(Denaturing gradient gel electrophoresis,DGGE)、末端限制性片段長度多態性(Terminal restriction fragment length polymorphism,T-RFLP)、克隆文庫等的出現逐漸彌補了這一缺陷[3]。
T-RFLP依據微生物的基因組學信息,選取一段具有系統進化標記特征的DNA序列為分析目標,應用熒光物質標記1個或1對引物的5′端。擴增后的PCR產物經限制性內切酶消化處理,熒光標記末端片段可被測序儀識別,得到不同長度的末端片段峰,根據片段峰的大小和數量就可以檢測和分析環境樣品中微生物的群落結構組成和動態變化等信息[4,5]。1997年,Liu等[6]首先應用 T-RFLP技術研究微生物多樣性。隨后,該技術快速發展,已成為研究環境微生物多樣性的重要手段之一,與其它指紋圖譜技術相比,TRFLP具有更高的靈敏度和重復性[7]。目前,已有應用 T-RFLP技術研究土壤[8]或活性污泥[9]中微生物多樣性的研究報道。
作者在此以不同城市生活污水處理廠曝氣池中的活性污泥樣品作為研究對象,應用T-RFLP技術研究其中的細菌群落結構組成和多樣性,分析細菌群落組成與水處理工藝之間的關系,擬為提高污水處理效率提供理論依據。
1 實驗
1.1 樣品采集
分別從烏魯木齊、合肥、無錫和西安各選取一個大型生活污水處理廠采集活性污泥樣品,其基本信息如表1 所示。
從各污水處理廠曝氣池中采集活性污泥樣品2L,置于冰上迅速帶回實驗室。離心收集活性污泥至50 mL離心管,于-20℃保存。
1.2 樣品總DNA的提取與PCR擴增

表1 污水處理廠基本信息Tab.1 Information of different sewage treatment plants
將活性污泥樣品用真空冷凍干燥機(ALPHA1-2,德國Christ)凍干,取0.25g,利用DNA提取試劑盒(Power Soil DNA Isolation Kit,Mobio,USA)提取樣品總DNA。采用1%瓊脂糖凝膠電泳檢測所提取的DNA樣品,于-20℃保存。
PCR擴增反應采用細菌通用引物對8f(5′-AGAGTTTGATCCTGGCTCAG-3′)和 926r (5′-CCGTCAATTCCTTTGAGTTT-3′),前者5′端采用Cy5-N-羥基琥珀酰亞胺酯進行熒光標記。50μL的PCR反應體系包括:5μL 10×PCR緩沖溶液(Ex Taq Buffer,Takara,Otsu,Japan),4 μL dNTPs,4 μL MgCl2,0.1μL上、下游引物,0.4μL Ex Taq DNA 聚合酶和1μL提取的基因組DNA,加水補足至50μL。PCR反應條件為:95℃3min;94℃30s,53℃30s,72℃1min,循環30次;72℃7min。PCR擴增產物采用2%瓊脂糖凝膠電泳檢測。
1.3 T-RFLP分析
首先,用綠豆酶(Mung bean nuclease)在30℃消化PCR產物30min。用PCR產物純化試劑盒(Axygen Biotechnology Ltd.,Hangzhou,China)純化消化后的產物。然后,用限制性內切酶HhaI(Takara,Otsu,Japan)37℃消化3h。消化產物再次純化后,用CEQ8000(Beckman Coulter,Fullerton,CA,USA)核酸片段分析儀對酶切產物進行T-RFLP分析。進一步分析相對豐度超過1%、長度為60~600bp的TRFs片段。分別計算細菌群落的Shannon-Wiener指數(H′)、Simpson指數(D)和Pielou指數(J),以反映活性污泥樣品中細菌的多樣性。計算公式為:

式中:Pi表示峰面積占總面積的比例;S表示TRFs數量。
2 結果與討論
2.1 基于T-RFLP的細菌群落結構分析
2.1.1 細菌多樣性分析
根據T-RFLP圖譜中T-RFs的數量、種類和豐度,分別計算各樣品細菌多樣性指數,結果見表2 。
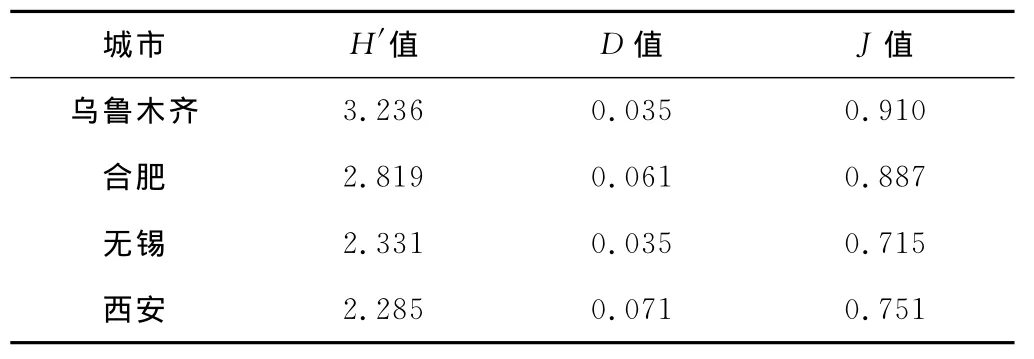
表2 不同活性污泥樣品細菌多樣性指數Tab.2 Bacterial diversity indexes of the activated sludge samples collected from different cities
從表2 可以看出:4個活性污泥樣品的H′值、D值均有顯著的差異;而J值則沒有顯著的差異,基本穩定在0.71~0.91之間;西安活性污泥樣品的H′值最低,表明西安活性污泥樣品中細菌多樣性最低;不同活性污泥樣品中細菌多樣性的順序為:烏魯木齊>合肥>無錫>西安。
2.1.2 細菌群落結構分析
通過對T-RFLP數據進行分析,得到相對豐度圖如圖1所示。

圖1 不同活性污泥樣品中各限制性片段的相對豐度Fig.1 Relative abundances of the T-RFs in the activated sludge samples collected from different cities
從圖1可以看出:長度為61~100bp T-RFs的總豐度在4個活性污泥樣品中都占到50%以上。長度為82bp、85bp、86bp、87bp、91bp、93bp、131bp、132bp、138bp、207bp的T-RFs在4個樣品中均有較高的相對豐度,如:長度為87bp的T-RF在4個樣品中的相對豐度均超過了15%,長度為86bp的T-RF在4個樣品中的相對豐度均超過了5%,可以判斷這2個片段為4個活性污泥樣品中的優勢種群;長度為62 bp的T-RF在烏魯木齊、合肥、無錫3個樣品中的相對豐度超過了5%;長度為88bp、95bp、132bp的T-RFs在合肥與無錫2個樣品中的相對豐度超過了5%。
2.2 T-RFs定性推斷
通過與數據庫 Microbial Community AnalysisⅢ(MiCA 3)(http://mica.ibest.uidaho.edu/pat.php)和Phylogenetic Assignment Tool(PAT)(http://secure.limnology.wisc.edu/trflp/)進行對比,可以鑒定特定長度的T-RF片段所對應的細菌的分類地位。與PAT數據庫比對結果表明:實驗所得到的優勢T-RFs所代表的細菌分別屬于芽孢桿菌綱(Bacilli)、α-變形菌綱(α-Proteobacteria)、δ-變形菌綱(δ-Proteobacteria)、γ-變形菌綱(γ-Proteobacteria),如表3 所示。

表3 PAT數據庫中與主要T-RFs對應的細菌的分類地位Tab.3 Phylogenetic assignment of the dominant T-RFs in PAT database
從表3 可以看出:62bp、95bp、96bp、211bp的T-RFs均為厚壁菌門(Firmicutes),具有嗜鹽特性[10],而檢索出的與其最相近的細菌類群卻各不相同,分別為玫瑰色鹽水球菌(Salinicoccus roseus)、芽孢桿菌屬(Bacillus arsenicoselenatis)、福賽類 擬 桿 菌 (Bacteroides forsythus)、氣球菌屬(Abiotrophia adiacens,在砷的地球化學循環中起重要作用[11])等;82bp、86 bp、88bp的 T-RFs均為變形菌門(Proteobacteria),其相近序列分別為芽單胞菌屬(Blastomonas natatoria)、紅螺菌屬(Rhodospirillum sodomense)、生脂固氮螺菌(Azospirillum lipoferum);91bp、93bp的 TRFs也屬于變形菌門(Proteobacteria),卻分別屬于δ-變形 菌 綱 (δ-Proteobacteria)與 γ-變 形 菌 綱 (γ-Proteobacteria),其相近序列也分別為蛭弧菌屬(Bdellovibrio stolpii)和布赫納式菌屬(Buchnera aphidicola,與蚜蟲共生[12])。
3 結論
研究了4個城市生活污水處理廠活性污泥中細菌的群落結構和多樣性。通過分析4個不同來源活性污泥樣品的Shannon-Wiener指數、Simpson指數和Pielou指數,得出4個樣品的多樣性順序為:烏魯木齊>合肥>無錫>西安;通過分析各樣品中T-RFs的相對豐度并與PAT數據庫進行對比,可以看出:4個活性污泥樣品中的優勢細菌屬于芽孢桿菌綱、α-變形菌綱、δ-變形菌綱和γ-變形菌綱;86bp、87bp的 T-RFs為4個活性污泥樣品中的優勢種群,其中86bp的TRF屬于α-變形菌綱(α-Proteobacteria)。
[1]余彬彬,李鈞敏,金則新.分子生物學技術在活性污泥微生物多樣性研究中的應用[J].江蘇農業科學,2009,(5):313-315.
[2]高平平,趙立平.可用于微生物群落分子生態學研究的活性污泥總 DNA提取方法研究[J].生態學報,2002,22(11):2015-2019.
[3]Theron J,Cloete T E.Molecular techniques for determining microbial diversity and community structure in natural environments[J].Critical Reviews in Microbiology,2000,26(1):37-57.
[4]Marsh T L.Terminal restriction fragment length polymorphism(T-RFLP):An emerging method for characterizing diversity among homologous populations of amplification products[J].Current Opinion in Microbiology,1999,2(3):323-327.
[5]Kitts C L.Terminal restriction fragment patterns:A tool for comparing microbial communities and assessing community dynamics[J].Curr Issues Intest Microbiol,2001,2(1):17-25.
[6]Liu W T,Marsh T L,Cheng H,et a1.Characterization of microbial diversity by determining terminal restriction fragment length polymorphisms of genes encoding 16SrRNA[J].Applied and Environmental Microbiology,1997,63(11):4516-4522.
[7]任南琪,趙陽國,高崇洋,等.TRFLP在微生物群落結構與動態分析中的應用[J].哈爾濱工業大學學報,2007,39(4):552-556.
[8]葛蕓英,陳松,胡蘭,等.土壤細菌DNA提取及多樣性分析的TRFLP方法[J].微生物學通報,2008,35(1):131-136.
[9]孫慶華,柏耀輝,趙翠,等.DGGE、T-RFLP、LH-PCR對兩種活性污泥的微生物種群多樣性分析的比較[J].環境工程學報,2009,3(8):1365-1370.
[10]Prasad M M,Seenayya G.Shelf-life of ribbonfish (Trichiurus haumela)cured in salt with red halophlic bacteria(Salinicoccus roseus)and chemical preservatives[J].Journal of Food Science and Technology(Mysore),2009,46(1):41-45.
[11]洪斌.微生物對砷的地球化學行為的影響——暨地下水砷污染機制的最新研究進展[J].地球科學進展,2006,21(1):77-82.
[12]苗雪霞,丁德誠.蚜蟲與其胞內共生細菌的相互作用[J].生命科學,2003,15(4):242-247.

