喉鱗癌中microRNA-197表達水平的研究
李俊 王建中 黃新生 白廣平 童華
(1.復旦大學附屬中山醫院青浦分院耳鼻咽喉科,上海 201700;2.復旦大學附屬中山醫院耳鼻咽喉科,上海 200032)
近年來發現,microRNAs(miRNAs)作為表觀遺傳修飾的手段和結果之一,很可能在人類癌癥的發生、發展和預后中起著重要作用[1]。喉癌是耳鼻咽喉科常見的惡性腫瘤,占全身惡性腫瘤的5.7%~7.6%,目前其發病率有明顯增長趨勢,其中喉鱗癌占全部喉癌的93%~99%。本研究采用miRNAs芯片技術檢測喉鱗癌細胞株 Hep-2中miRNAs的表達,并采用實時定量聚合酶鏈反應(realtime quantitative reverse-transcriptase polymerase chain reaction,RT-PCR)在喉鱗癌組織中驗證。
1 資料與方法
1.1 材料 20例喉鱗癌組織標本及10例癌旁正常組織標本均取自2010年7月—11月復旦大學附屬中山醫院耳鼻咽喉科手術標本。20例喉鱗癌病理診斷均為原發性喉鱗癌,所有患者均為首診首治,未行術前放化療。取材時,分別取癌旁正常組織(距癌組織大于3 cm)和癌組織,將組織塊剪至約1 cm大小,10 min內迅速置于液氮中儲存,后轉至-80℃冰箱保存。喉鱗癌細胞株Hep-2及永生化正常人支氣管上皮細胞株HBEAs-2B均購自中國科學院上海生物所細胞庫。采用miRCURY LNATM U-niversal RT microRNA PCR芯片(丹麥Exiqon公司,由上海康成生物工程有限公司檢測);RPMI 1640培養液和胎牛血清購自美國Hyclone公司;Trizol購自美國Invitrogen公司;SYBRTM Green master mix購自丹麥Exiqon公司;Taq聚合酶購自美國Promega公司;RNA酶抑制劑和MMLV反轉錄酶購自美國Epicentre公司;7900型PCR擴增儀購自美國Applied Biosystem公司;目的基因及內參引物均由上海英駿生物技術有限公司合成;其他常規試劑購自上海化學試劑有限公司。
1.2 方法
1.2.1 細胞培養 Hep-2和 HBEAs-2B細胞均用RPMI 1640培養液(10%胎牛血清、100 U/mL青霉素及100μg/mL鏈霉素)于37℃、5%CO2、飽和濕度下培養,每2~3 d換液,用0.25%胰酶消化傳代。取對數生長期的細胞用于實驗。
1.2.2 RNA提取及質量檢測 采用Trizol法提取細胞總RNA,具體方法按說明書進行。采用紫外分光光度計分別測定波長為230、260和280 nm處的吸光度值(A值),確定總RNA的純度和濃度;采用甲醛變性瓊脂糖凝膠電泳檢測總RNA樣品中28S RNA和18S RNA的比例,鑒定其純度和完整性。
1.2.3 miRNAs芯片檢測 采用 miRCURY LNATM Universal RT miroRNA PCR芯片,內設6個內參,包括3個 miRNAs(hsa-miR-103、hsamiR-191和hsa-miR-423-5p)以及3個小RNA(U6、SNORD38B和SNORD49A)。采用Universal RT反應,對Hep-2細胞及HBEAs-2B細胞進行高通量miRNAs定量檢測。將總RNA樣品濃度調整至1.492~1.786 ng/μL,按以下20μL體系合成cDNA:5×反應緩沖液4μL,酶混合液2μL,總RNA 14μL;反應條件:42℃60 min,95℃5 min。cDNA稀釋100倍后,將cDNA模板與SYBRTM Green Master Mix按1∶1的比例混合,以每孔10 μL加入384孔PCR板中。采用7900型PCR擴增儀進行反應,反應條件:95℃10 min;95℃10 s,擴增40個循環;60℃1 min;分析溶解曲線。采用GenEx qPCR分析軟件(www.exiqon.com/mirnapcr-analysis)對數據進行標準和深入分析。
1.2.4 RT-PCR 檢測 miRNA-197(miR-197)在喉鱗癌組織中的表達水平 內參U6引物序列:上游引物(F)為5′-GCTTCGGCAGCACATATACTAAAAT-3′; 下游引物 (R )為 5′-CGCTTCACGAATTTGCGTGTCAT-3′;目的基因 miR-197引物序列:F為5′-GGTTCACCACCTTCTCCA-3′;R為5′-CAGTGCGTGTCGTGGAGT-3′。RNA逆轉錄反應條件:16℃30 min,42℃40 min,85℃5 min。反應結束后,將反應產物cDNA放在冰上待用或-20℃保存。RT-PCR反應條件如下:95℃3 min;95℃15 s,60℃20 s,72℃20 s,40個循環;78℃20 s。為了建立溶解曲線,擴增反應結束后,按95℃15 s、60℃20 s、72℃20 s、99℃15 s,從72℃緩慢加熱至99℃。各樣品中的miR-197和內參U6分別進行RT-PCR反應。PCR反應結束后確認擴增曲線和溶解曲線,進行實驗結果的分析。
1.2.5 數據分析 采用ΔΔCt方法,miRNA PCR Array計算公式:ΔΔCt=ΔCt(Hep-2)-ΔCt(HBEAs-2B);通過2-ΔΔCt計算 Hep-2與 HBEAs-2B對應基因的表達差異。
1.3 統計學處理 采用SPSS 13.0統計軟件進行分析,組間比較采用t檢驗,P<0.05為差異有統計學意義。
2 結 果
2.1 RNA質檢結果 采用紫外吸收測定法測定從各細胞和組織樣本中分別提取的總RNA的濃度及純度,結果顯示,RNA純度和濃度均合格,A260/A280為1.8~2.1,A260/A230>1.8,濃度為600~2500 ng/μL。變性瓊脂糖凝膠電泳結果也提示,所提取RNA的濃度、純度和完整性均符合實驗要求。
2.2 miRNAs芯片結果 篩選條件為Fold Difference不低于2倍。與HBEAs-2B比較,Hep-2細胞中上調的miRNAs共166個,下調的miRNAs共47個。在 Hep-2細胞中,miR-197上調5.08倍,miR-133b上調21.79倍;miR-519d下調3.93倍,miR-20b下調21.49倍,經組間t檢驗比較,差異均有統計學意義(P均<0.05)。表達譜部分結果見表1。
2.3 RT-PCR檢測結果 miR-197在喉鱗癌組織及癌旁正常組織中的RT-PCR結果見圖1~4。目的基因miR-197和內參基因U6的擴增曲線從最大基線后開始出現指數增長期和平臺期,說明產物擴增較好。目的基因miR-197和內參基因U6的溶解曲線都只有單一銳利的峰,說明所設計的引物有較高特異性,只擴增出單一產物。設置為由系統自動調整基線和閾值,首先分別求出每個樣品3個重復孔中目的基因miR-197和內參基因U6的平均Ct值,然后用內參基因U6對各實驗組樣本進行校正(ΔCt),再取其對數,比較各樣本中 miR-197表達的差異,對對照組和實驗組樣本的ΔCt進行歸一化處理(2-△△Ct),最后根據2-△△Ct統計分析組間 miR-197的表達差異,見表2。結果顯示,miR-197在喉鱗癌組織中的表達水平顯著高于癌旁正常組織,差異有統計學意義(P<0.05)。

表1 Hep-2與HBEAs-2B中表達有差異的miRNAs譜



3 討 論
本研究采用miRNAs芯片技術檢測喉鱗癌細胞株Hep-2及正常人支氣管上皮細胞株HBEAs-2B中miRNAs的表達,首次獲得了喉鱗癌Hep-2細胞miRNAs表達譜,從中挑選出在Hep-2中顯著高表達的miR-197,進一步在20例喉鱗癌組織及10例癌旁正常組織中檢測其表達情況。本研究所用的丹麥Exiqon公司的miRCURY LNATM UniversalRT microRNA PCR芯片,實現了高通量高質量的miRNAs定量檢測工作,采用一次單鏈cDNA合成反應制備的cDNA可作為芯片所有miRNAs PCR反應的模板,避免了用混合RT引物對擴增特異性的影響,實現了引物Tm值均一;并能有效區分序列僅有一個堿基差異的miRNAs,還能區分成熟miRNAs和miRNAs前體;具有靈敏度高、特異性強、準確性高、質控嚴格、高重復性的特點。
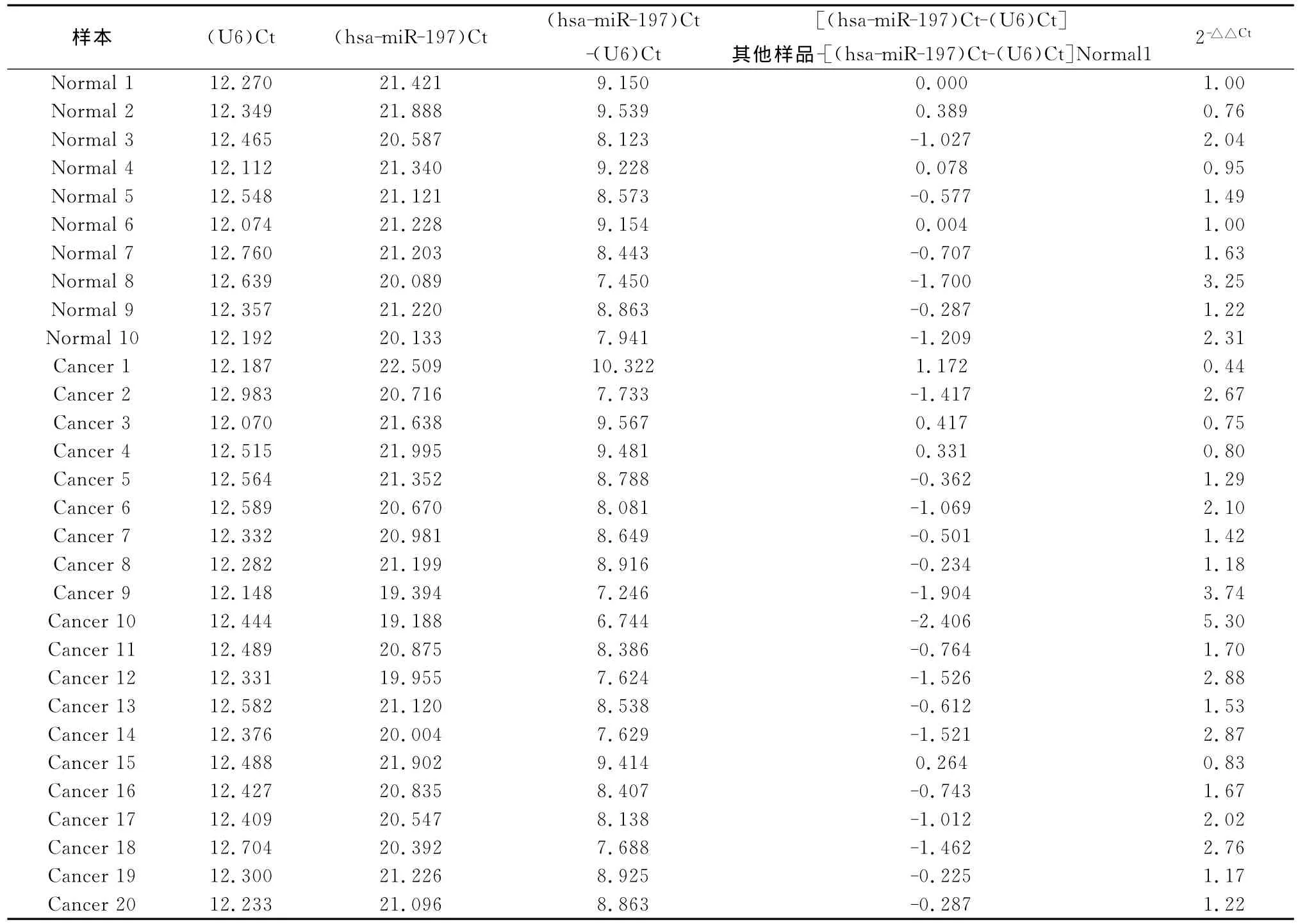
表2 RT-PCR檢測結果
本研究發現,miR-197在人喉磷癌組織中表達水平顯著升高。Du等[2]研究發現,miR-197在小細胞肺癌細胞株和非小細胞肺癌細胞株中表達水平高于人支氣管上皮細胞。Wong等[3]發現,miR-197、miR-184等24個miRNAs在舌鱗癌高表達,研究還發現,miR-184表達水平與舌鱗癌的發生有關。Nikiforova等[4]、Weber等[5]研究發現,miR-197在甲狀腺腫瘤中過表達,并且與其病理類型密切相關。Wang等[6]在婦女子宮肌瘤中發現 miRNA-21、miR-197等高表達。Hamada等[7]發現,miR-197在人胰腺癌呈顯著性高表達。
綜上所述,miR-197在體內外人喉鱗癌細胞中均呈現高表達狀態,揭示其可能在維持喉鱗癌細胞惡性表型中起重要作用,提示miR-197可能是喉鱗癌的特異性表達標志物之一,為進一步研究miR-197在喉癌中的作用機制奠定了實驗基礎。
[1] Zhang B,Pan X,Cobb GP,et al.microRNAs as oncogenes and tumor suppressors[J].Dev Biol,2007,302(1):1-12.
[2] Du L,Schageman JJ,Subauste MC,et al.miR-93,miR-98,and miR-197 regulate expression of tumor suppressor gene FUS1[J].Mol Cancer Res,2009,7(8):1234-1243.
[3] Wong TS,Liu XB,Wong BY,et al.Mature miR-184 as potential oncogenic microRNA of squamous cell carcinoma of tongue[J].Clin Cancer Res,2008,14(9):2588-2592.
[4] Nikiforova MN,Tseng GC,Steward D,et al.MicroRNA expression profiling of thyroid tumors:biological significance and diagnostic utility[J].J Clin Endocrinol Metab,2008,93(5):1600-1608.
[5] Weber F,Teresi RE,Broelsch CE,et al.A limited set of human MicroRNA is deregulated in follicular thyroid carcinoma[J].J Clin Endocrinol Metab,2006,91(9):3584-3591.
[6] Wang T,Zhang X,Obijuru L,et al.A micro-RNA signature associated with race,tumor size,and target gene activity in human uterine leiomyomas[J].Genes Chromosomes Cancer,2007,46(4):336-347.
[7] Hamada S,Satoh K,Miura S,et al.miR-197induces epithelialmesenchymal transition in pancreatic cancer cells by targeting p120 catenin[J].J Cell Physiol,2013,228(6):1255-1263.

