新疆低溫稠油油藏厭氧硝酸鹽還原菌與硫酸鹽還原菌富集產物菌群分析
馬莎莎,張 凡,舒福昌,侯讀杰,佘躍惠
(1.長江大學化學與環境工程學院,湖北 荊州 434023;2中國地質大學(北京)能源學院,北京 100080)
硝酸鹽還原菌(Nitrate reducing bacteria,NRB)和硫酸鹽還原菌(Sulfate reducing bacteria,SRB)是油田普遍存在的兩類微生物菌群。原油的酸化和管道的腐蝕主要是由SRB還原硫酸鹽產生H2S所導致的[1,2]。抑制SRB的活性、減少H2S的產生是油田生產的一項十分重要的工作。除了化學殺菌處理外,目前最為環保的抑制SRB活性的方法是生物競爭排除技術(Biocompetitive exclusion)[3],該技術是利用硝酸鹽還原菌、亞硝酸鹽還原菌以及反硝化微生物阻止SRB獲得所需營養物而抑制其產H2S的活性[4]。目前,大多研究僅針對單個菌株對H2S產生的影響,但油田微生物菌群較為復雜,菌群之間相互影響,因此,對微生物菌群進行全面的認識對生物競爭技術研究是很有必要的。隨著分子生物學和系統發育分析方法的發展,基于16S rRNA(rDNA)的分子生物學方法被用來分析復雜的生態系統,特別是構建16S rDNA PCR擴增片段克隆文庫來分析微生物菌群組成[5]。
作者在此直接采集油井井口樣品接種于NRB和SRB富集培養基中培養,通過克隆文庫分析富集培養物中的微生物菌群,以期為油田開發NRB抑制硫酸鹽還原作用新技術提供理論依據。
1 實驗
1.1 樣品
樣品采自新疆克拉瑪依油田六中區克下組油井T6191井口,該油層含油面積10.3 km2、地質儲量2.084×107t,屬克拉瑪依Ⅲ類礫巖油藏。該油井產出液平均含水量2.1%,原油相對密度0.899,地層原油粘度80 mPa·s,凝固點-49℃,含蠟量3%,含膠量58%,屬于低溫輕質稠油油藏。井口油水樣品被直接裝入無菌瓶,在進行分子實驗前于4℃保存。
1.2 NRB和SRB富集培養
在基礎培養基中加入不同的碳源和能源用來富集NRB和SRB菌群。
基礎培養基(1 L):10 g NaCl,3 g MgCl2·6H2O,0.15 g CaCl2·2H2O,0.25 g KCl,0.6 g KBr,0.5 g KH2PO4,pH值7.0。
NRB富集培養基:1 L基礎培養基中加入3 g KNO3,1 g乙酸鈉,1 g酵母膏,1 mL刃天青母液 (1%),pH值7.0。
SRB富集培養基:1 L基礎培養基中加入 4 g Na2SO4,1 g乙酸鈉,1 g酵母膏,1 mL刃天青母液 (1%),pH值7.0。
所有培養基被分裝入250 mL厭氧瓶中通入N2,高壓濕熱滅菌后,按30 mL·L-1加入1 mol·L-1NaHCO3、按1 mL·L-1加入微量元素母液和維生素母液[6]。培養基中接種2% (體積分數)產出水和5%(質量濃度)原油,按地層溫度25℃培養120 d。
1.3 NRB和SRB富集培養物中DNA的提取
1.3.1 富集產物DNA的提取
吸取2 mL富集培養液,于12 000 r·min-1離心10 min收集菌體細胞。根據FastDNA Spin Kit for Soil (Qbiogen,Carlsbad,CA.U.S.A)試劑盒說明提取基因組DNA。采用1%的瓊脂糖凝膠電泳檢測DNA。
1.3.2 16S rRNA基因全長擴增
使用16S rDNA片段擴增通用引物27f(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-GGCTACCTTGTTACGACTT-3′),從基因組總DNA中擴增16S rDNA片段。PCR擴增體系及程序參照文獻[7]。
1.4 克隆文庫的建立
用瓊脂糖凝膠回收試劑盒[Agarose Gel DNA Purification Kit (TianGen Biotech,Beijing,China)]對16S rDNA擴增產物進行割膠、純化。然后將回收產物16S rDNA片段pGEM T-easy克隆載體(Promega),通過化學法轉化到感受態細胞Trans1-T1 competent cells (TransGen Biotech,Beijing,China)中。從2個文庫各挑取100個白斑克隆,以少量菌體作為模板通過載體通用引物T7/SP6擴增來檢測陽性克隆。PCR產物先用限制性內切酶HaeⅢ 和HhaⅠ進行酶切,再用2%瓊脂糖凝膠電泳分析限制性片段,最后對電泳圖譜進行限制性酶切片段長度多態性(ARDRA)分析[8]。將酶切圖譜相同的克隆歸為一個發育類型(OTU)。
1.5 克隆文庫庫容
應用Rarefaction分析軟件(http://www.uga.edu/strata/software/software.html)對克隆文庫的庫容飽和度進行分析,以檢測建立的克隆文庫是否已經足夠全面地代表NRB和SRB菌群的多樣性。
1.6 測序和構建系統發育樹
挑取ARDRA分型歸為一類的代表性克隆接種于含有100 μg·mL-1氨芐的液體LB培養基中,37℃培養20 h后送測序公司(SinoGenoMax Co.,Ltd.,Beijing,China)測序。所得序列通過DNAMAN軟件進行處理,并在NCBI(National Center for Biotechnology,http://www.ncbi.nlm.nih.gov/BLAST/)數據庫中比對分析,尋找親緣關系最近的序列用DNAMAN軟件構建系統發育樹。
2 結果與討論
2.1 NRB和SRB微生物菌群分析
在NRB和SRB的16S rRNA基因克隆文庫DGS1和DGS2中各選擇100個陽性克隆子,通過HaeⅢ和HhaⅠ雙酶切分型分為了11和12個OTU。Rarefaction分析曲線(圖1)顯示,當陽性克隆子達到90個時曲線趨于水平,說明克隆文庫能夠反映樣品微生物菌群結構。
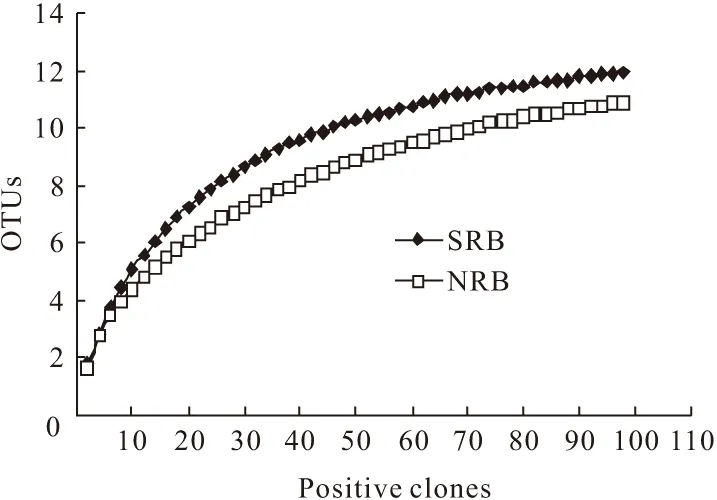
圖1 克隆文庫DGS1和DGS2的Rarefaction分析曲線
16S rRNA基因克隆文庫NRB和SRB的系統發育樹見圖2。
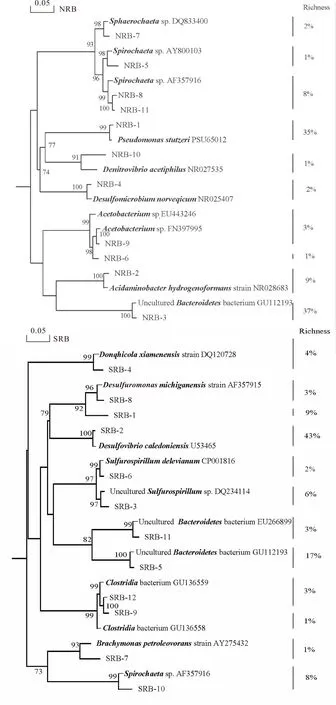
各分支后的數字代表該系統發育類型在克隆文庫中所占的比例
由圖2可知,克隆文庫NRB中優勢菌群為施氏假單胞菌(Pseudomonasstutzeri)(35%)、未培養擬桿菌(UnculturedBacteroidetesbacterium)(37%)、螺旋體(Spirochaeta)(9%)和產氨基酸桿菌(Acidaminobacterhydrogenoformans)(9%)。克隆文庫SRB中優勢菌群為脫硫弧菌(Desulfovibriocaledoniensis)(43%)、未培養擬桿菌(20%)、脫硫單胞菌(Desulfuromonasmichiganensis)(12%)和螺旋體(8%)。將所得序列在NCBI數據庫中對比分析,結果表明,大多數序列(>90%)和GenBank中已有的16S rDNA序列同源性大于97%,說明該富集培養物中微生物的可培養性。雖然NRB和SRB菌群為兩種不同培養基的富集產物(圖3),其優勢菌群不同,但是未培養擬桿菌和螺旋體在2個文庫中均出現了,說明這兩種菌能在兩種培養基中生長。特別是未培養擬桿菌在2個文庫中都為優勢菌。
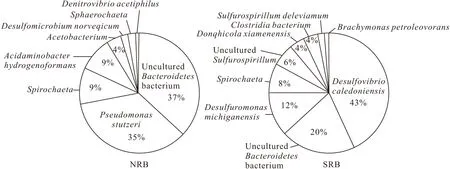
圖3 硝酸鹽還原菌和硫酸鹽還原菌富集培養物中菌群分布關系
2.2 討論
NRB和SRB富集產物微生物菌群結構存在差異。NRB富集培養物中施氏假單胞菌占35%,它是油田常見菌群,能將硝酸鹽還原成氮并且能代謝多種有機物[9];SRB富集培養物中脫硫弧菌、脫硫單胞菌、梭狀芽孢桿菌(Clostridiabacterium)都是典型的硫酸鹽還原菌,大約覆蓋了文庫的60%。由此可知富集培養產物為目的產物。
NRB和SRB富集產物微生物菌群雖然存在差異,但是未培養擬桿菌和螺旋體在2個文庫中都出現,特別是未培養擬桿菌在2個文庫中均為優勢菌,這可能是由于在富集培養基中加入了原油,而擬桿菌和螺旋體是一類能以烴為碳源的菌群[10,11]。因此在兩個加有原油的富集培養基中出現這類以烴為碳源的菌群并且形成一定優勢。因為原油儲層中不可避免地存在原油,為了能更好地模擬儲層環境,在富集培養基中加入原油是有必要的。
SRB富集產物中出現了一定量的硫磺單胞菌屬(Sulfurospirillum),這類菌的出現可能是由于培養基中硫化物含量增加所致,同時它們也是油田生物競爭排除技術的目的菌群,是后續研究工作的重點所在。
3 結論
基于16S rDNA分子克隆文庫方法,分析新疆克拉瑪依油田六中區采油井T6191井口樣品硝酸鹽還原菌(NRB)和硫酸鹽還原菌(SRB)富集產物的菌群多樣性。16S rRNA基因克隆建庫結果表明,NRB和SRB菌群存在明顯的差異,其優勢菌不同,前者主要為降烴菌,后者則與硫酸鹽還原作用密切相關;NRB和SRB菌群中同時存在未培養擬桿菌和螺旋體,這與原油儲層環境有關,它們均能降解石油烴,菌群分析結果為油田開發NRB抑制硫酸鹽還原作用新技術提供了理論依據。
[1] Caffrey S M,Park H S,Voordouw J K,et al.Function of periplasmic hydrogenases in the sulfate-reducing bacteriumDesulfovibriovulgarishildenborough[J].J Bacteriol,2007,189(17):6159-6167.
[2] Cornish Shartau S L,Yurkiw M,Lin S,et al.Ammonium concentrations in produced waters from a mesothermic oil field subjected to nitrate injection decrease through formation of denitrifying biomass and anammox activity[J].Appl Environ Microbiol,2010,76(15):4977-4987.
[3] Raji K,Sara E,Gray Murray R,et al.Molecular and cultivation-based analyses of microbial communities in oil field water and in microcosms amended with nitrate to control H2S production[J].Appl Microbiol Biotechnol,doi 10.1007/S00253-010-2974-8,published online,2010-11-06.
[4] Hulecki J C,Foght J M,Gray M R,et al.Sulfide persistence in oil field waters amended with nitrate and acetate[J].J Ind Microbiol Biotechnol,2009,36(12):499-511.
[5] Gittel A,Sorensen K B,Skovhus T L,et al.Prokaryotic community structure and sulfate reducer activity in water from high-temperature oil reservoirs with and without nitrate treatment[J].Appl Environ Microbiol,2009,75(22):7086-7096.
[6] Widdle F,Bak F.Gram-negative mesophilic sulfate-reducing bacteria[A].In: Balows A,Truper H G,Dworkins M,et al.The Prokaryotes(2nd ed)[C].New York:Springer,1992:3352-3378.
[7] Zhang F,She Y H,Zheng Y,et al.Molecular biologic techniques applied to the microbial prospecting of oil and gas in the Ban 876 gas and oil field in China[J].Appl Microbiol Biotechnol,2010,86(4):1183-1194.
[8] Lagacé L,Pitre M,Jacques M,et al.Identification of the bacterial community of maple sap by using amplified ribosomal DNA (rDNA) restriction analysis and rDNA sequencing[J].Appl Environ Microbiol,2004,70(4):52-60.
[9] Grigoryan Aleksandr A,Cornish Sabrina L,Buziak Brenton,et al.Competitive oxidation of volatile fatty acids by sulfate- and nitrate-reducing bacteria from an oil field in Argentina[J].Appl Environ Microbiol,2008,74(14):4324-4335.
[10] Baek K,Kim H S.Microbial community structure in hexadecane- and naphthalene-enriched gas station soil[J].J Microbiol Biotechnol,2009,19(7):651-657.
[11] Magot M,Fardeau M L,Arnauld O,et al.Spirochaetasmaragdinaesp.nov.,a new mesophilic strictly anaerobic spirochete from an oil field[J].FEMS Microbiol Lett,1997,155(2): 185-191.

