杜氏鹽藻寡糖基轉移酶亞基STT3a功能結構域的克隆與表達分析
王翠,李杰,柳麗平,曾磊,薛樂勛
鄭州大學生物工程系 細胞生物學研究室,鄭州 450001
杜氏鹽藻寡糖基轉移酶亞基STT3a功能結構域的克隆與表達分析
王翠,李杰,柳麗平,曾磊,薛樂勛
鄭州大學生物工程系 細胞生物學研究室,鄭州 450001
為了研究STT3a基因在杜氏鹽藻耐鹽及鞭毛再生方面的作用,根據衣藻、擬南芥等STT3a蛋白的氨基酸高度保守序列VCVFTA、DVDYVL設計一對簡并引物,采用RT-PCR及3'RACE的方法擴增杜氏鹽藻STT3a蛋白功能結構域的cDNA序列。序列分析顯示克隆的cDNA全長1 650 bp,具有一定保守性,與衣藻、擬南芥和人的相似性分別為48%、50%和46%。實時熒光定量PCR結果顯示杜氏鹽藻STT3amRNA水平隨著鹽濃度的升高而逐漸增加,其水平在3.5 mol/L NaCl濃度時比在1.5 mol/L NaCl濃度時升高了11倍(P<0.01)。另外,與沒有脫鞭毛的杜氏鹽藻相比,STT3amRNA在鞭毛再生過程中持續高表達。本研究顯示杜氏鹽藻STT3a基因的高表達可以增強其鹽適應和鞭毛再生能力。
杜氏鹽藻,STT3a基因,耐鹽,鞭毛再生
Abstract:To investigate the function ofSTT3agene in salt adaptation and flagellar regeneration ofDunaliella salina(D.salina), a pair of degenerate primers was designed according to conserved homologous amino acid sequences of VCVFTA and DVDYVL of STT3a fromChlamydomonas,Arabidopsis thalianaand other organisms.A cDNA sequence of 1 650 bp encoding a whole functional domain of STT3a was amplified fromD.salinaby RT-PCR and 3′ Rapid Amplification of cDNA Ends(RACE), which shared homology withChlamydomonas(48%),Arabidopsis thaliana(50%),Homo sapiens(46%), etc.Real-time fluorescence quantitative PCR(real-time Q-PCR)demonstrated that theSTT3amRNAs fromD.salinawere induced by increased concentration of NaCl, and increased to 11-fold higher by 3.5 mol/L NaCl than that by 1.5 mol/L NaCl(P<0.01).Also,STT3amRNA ofD.salinamaintained at a higher level in the process of flagellar regeneration with than without experiencing deflagellar treatment.In conclusion, the findings of this study demonstrate that the high expression of theSTT3agene enhances the capability of salt adaptation and flagellar regeneration inD.salina.
Keywords:Dunaliella salina,STT3agene, salt adaptation, flagellar regeneration
N-連接糖基化是內質網中最常見的蛋白質修飾,這個過程是由位于糙面內質網膜上的寡糖基轉移酶(OST)催化的。一系列的研究發現釀酒酵母的OST包含8個亞基:Ost1p,Ost2p,Wbp1,Stt3p,Swp1p,Ost4p,Ost5p,Ost3p/Ost6p[1],STT3p 在已知的OST亞基中是最保守的[2],它在酵母中編碼一個78 kDa的蛋白,為OST的催化亞基[3]。幾乎所有的真核生物基因組中都存在編碼酵母 Stt3p蛋白的同系物。哺乳動物細胞表達 Stt3p的兩種同系物STT3a和 STT3b,它們的表達具有組織特異性,并能調節OST的活性,且含有一個STT3a亞基的OST復合物比含有STT3b亞基的OST復合物對于選擇完全裝配的長鏈寡糖供體(Glac3Man9GlcNAc2-PP-Dol)更具有特異性[3]。最近的研究還發現擬南芥OST的STT3a亞基作為一種跨膜蛋白在高鹽應激反應中發揮一定作用[4]。
杜氏鹽藻Dunaliella salina(D.salina)是一種高度耐鹽的單細胞真核綠藻,它能在 0.5~5 mol/L NaCl鹽濃度下生存,但是目前關于其耐鹽機制還不清楚。另外,我們前期的研究發現:在高鹽條件下,鹽藻的運動性明顯降低,而鹽藻的鞭毛直接參與了鹽藻自身的運動。為了探討鹽藻的鹽耐受性和鞭毛介導的運動性之間是否有潛在的聯系,本研究根據已知物種的 STT3a高度保守序列設計一對簡并引物,通過RT-PCR及RACE方法獲得STT3a功能結構域的cDNA序列,并通過實時熒光定量PCR技術監測STT3a基因mRNA在杜氏鹽藻鹽適應及鞭毛再生過程中的動態變化,以期了解STT3a基因在杜氏鹽藻耐鹽及鞭毛再生中的作用。
1 材料與方法
1.1 杜氏鹽藻藻株、載體與菌株
杜氏鹽藻藻株為 UTEX-LB-1644,購自美國德州大學。載體 pMD18T-vector購自大連寶生物工程公司。大腸桿菌DH5α為本室保存。
1.2 主要試劑
EcoRⅠ、Hind Ⅲ、LATaq酶、rTaq酶、膠回收試劑盒、質粒提取試劑盒及反轉錄試劑盒均購自大連寶生物工程公司。實時定量PCR熒光染料購自天根生化科技(北京)有限公司。First Choice RLMRACE Kit購自美國Ambion公司。
1.3 杜氏鹽藻cDNA的制備及簡并引物的設計
杜氏鹽藻細胞按照 5×105/mL的接種量接種于改良的PKS液體培養基中,培養溫度為26℃,光照強度為4500 Lux,光/暗培養各12 h。取對數生長期的杜氏鹽藻細胞,用 Trizol法提取杜氏鹽藻細胞的總RNA,瓊脂糖凝膠電泳及分光光度計檢測所提取總RNA的質量和濃度。取6 μg RNA作為模板,按照反轉錄試劑盒說明書制備反轉錄反應液,于37℃反轉錄1 h后,70 ℃ 10 min ,置于?20℃保存。從GenBank上搜索近10個物種的STT3a蛋白的氨基酸序列進行比對,找出兩段相對比較保守的序列設計簡并引物,上游引物:5′-GTNTG(T/C)GTNTT(T/C)ACNGC-3′,下游引物:5′-A(A/G)NAC(A/G)TA(A/G)TCNAC(A/G)TC-3′,引物由上海博尚生物技術有限公司合成。
1.4 RT-PCR
以制備的 cDNA為模板,利用合成的簡并引物,用PCR的方法擴增STT3a的部分基因片段。PCR 反應體系為:cDNA 0.5 μL,10× LA Buffer 10 μL,dNTPs(10 mmol/L)8 μL,上、下游引物(20 μmol/L)各 1 μL,LATaq酶 0.5 μL,dH2O 補至100 μL。反應程序:95 ℃ 5 min;94℃ 30 s,50℃~60℃30 s,72 ℃ 2 min ,30個循環;72 ℃ 1 0 min,4℃終止反應。PCR反應結束后,1%的瓊脂糖凝膠電泳檢測擴增結果。將PCR產物膠回收,連接至pMD18T-vector中并轉化大腸桿菌 DH5α,用含有 Amp、IPTG和 X-Gal的平板進行藍白斑篩選,挑取陽性菌落擴大培養并提取質粒,將鑒定正確的菌樣送北京博尚生物技術有限公司測序,序列通過Primer Premier 5.0整理并用BLAST進行同源性分析。
1.5 STT3a基因的3′RACE擴增
根據簡并引物擴增得到的已知序列,設計STT3a的3′RACE引物(表1),提取杜氏鹽藻的總RNA,按照 FirstChoice RLM-RACE Kit說明進行操作。PCR反應程序:95 ℃ 5 min;94℃ 30 s,58℃ 30 s,72 ℃ 1 min ,30個循環;72℃ 1 0 min,4℃終止反應。巢式PCR反應產物經1%的瓊脂糖凝膠電泳檢測。對目的片段進行膠回收后連接于pMD18T-vector上,轉化大腸桿菌DH5α,挑取陽性菌落提取質粒,雙酶切鑒定,正確后由北京博尚生物技術有限公司進行測序,序列通過Primer Premier 5.0整理并用BLAST進行同源性分析。
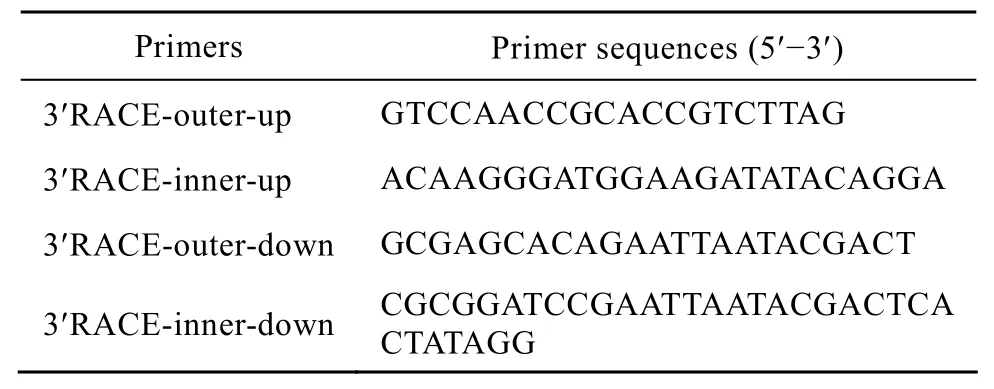
表1 設計的3′RACE引物序列Table 1 Primers used in 3′RACE
1.6 STT3a結構域的功能分析
通過Primer Premier 5.0在STT3a結構域中設計一對特異性引物,通過實時熒光定量 PCR分析STT3a在杜氏鹽藻鹽誘導及鞭毛再生過程中的作用。將對數生長期的杜氏鹽藻分別轉入0.75、1.0、1.5、2.0、3.0及3.5 mol/L NaCl的PKS液體培養基中光培養10 h后,收集不同鹽濃度下的杜氏鹽藻細胞;同時,將對數生長期的杜氏鹽藻分別轉入1.5、3.0 mol/L NaCl的PKS液體培養基中光照培養,分別收集0、2、4、6、8和10 h的杜氏鹽藻細胞,分別提取上述處理細胞的總RNA,取6 μg RNA作為模板反轉錄為cDNA。取0.8 μL cDNA作為模板,按照實時熒光定量 PCR說明書進行操作,同時以GAPDH為內參。反應程序:95 ℃ 5 min ;94℃ 30 s,60℃ 30 s,72℃ 30 s,35 個循環;95℃ 15 s,60℃30 s,95℃ 15 s。PCR產物通過瓊脂糖凝膠電泳檢測及測序,并運用 2-△△Ct方法[5]分析STT3a基因的相對表達量。
另外,取對數生長期的杜氏鹽藻細胞,在添加甘油的電擊緩沖液中用機械方法[6]去除鞭毛后,分別收集 0、15、30、45、60、90、120、150、180、210、240、270、300、360和420 min時的杜氏鹽藻細胞,同時以未經過去鞭毛處理的杜氏鹽藻細胞作為對照組。按上述方法制備 cDNA,進行實時熒光定量PCR。
1.7 杜氏鹽藻鞭毛再生不同時期的鞭毛銀染
運用改進的銀染法[7]對杜氏鹽藻鞭毛及藻體進行銀染。
1.8 杜氏鹽藻鞭毛再生不同時期鞭毛長度的測量
用熒光顯微鏡在相同條件下對鞭毛再生不同時期的杜氏鹽藻細胞照相,然后每個時期分別取30個杜氏鹽藻樣本,利用細線擬合鞭毛,進行鞭毛長度的測量,取其鞭毛生長平均值,根據公式V=(S2?S1)/t(其中V為相對生長速率,S2和S1分別為相鄰兩個時間點的鞭毛長度平均值,t代表間隔時間),最終求出杜氏鹽藻鞭毛不同時期的相對生長速率。
1.9 統計學分析
試驗中每項處理均重復 3次。數據運用 SPSS統計分析軟件對樣本進行方差檢驗,每次試驗中P<0.05,表明差異顯著。
2 結果
2.1 杜氏鹽藻總RNA的純度和質量鑒定
所提取的杜氏鹽藻總RNA質量完好,28S和18S rRNA帶比較清晰,無拖尾現象,其亮度比約為2∶1,無 RNA降解和 DNA、蛋白質污染,可以用于下步實驗。
2.2 RT-PCR產物鑒定
電泳結果如圖1所示,所得片段長度約為1 500 bp,與理論值相符。膠回收后連接于pMD18T-vector上,得到載體pMD-stt3,用EcoRⅠ和Hind Ⅲ進行雙酶切鑒定,可見一條載體片段和一條插入片段,插入片段與回收片段大小一致。
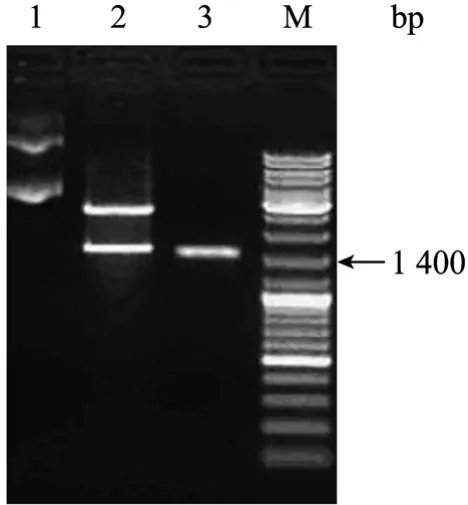
圖1 RT-PCR擴增杜氏鹽藻STT3a cDNAFig.1 Amplification ofSTT3acDNA fromD.salinaby RT-PCR. 1: pMD-stt3; 2: pMD-stt3 digested withEcoRⅠ andHind Ⅲ ; 3: PCR amplification product; M: DNA marker.
2.3 杜氏鹽藻STT3a基因3′下游序列的擴增
經過巢式PCR,電泳結果如圖2所示:所得片段長度約為 850 bp,膠回收后連接于 pMD18T-vector上,得到載體pSTT3-1,用EcoRⅠ和Hind Ⅲ進行雙酶切鑒定,可見一條載體片段和一條插入片段,插入片段與回收片段大小一致。
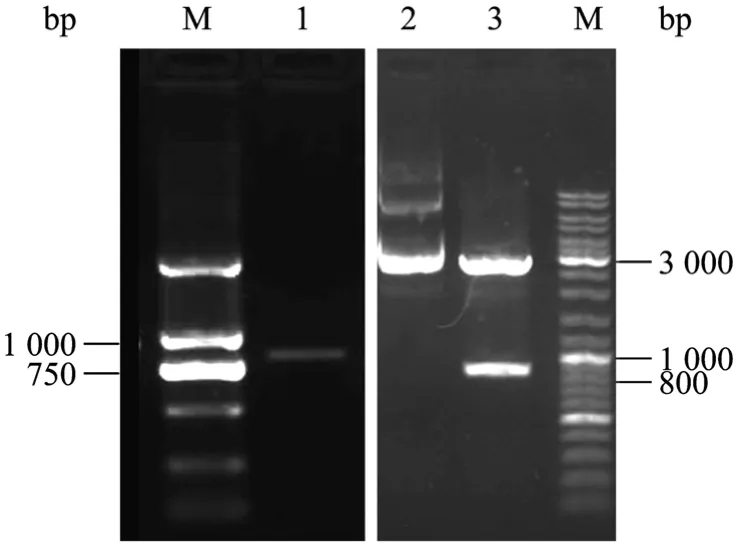
圖2 巢式PCR擴增杜氏鹽藻STT3a基因3′下游序列Fig.2 Amplification of the 3′ downstream sequence ofD.salinaSTT3agene by nested PCR.M: DNA marker; 1: 3′downstream sequence amplification product ofSTT3agene; 2:pSTT3-1; 3: pSTT3-1 digested withEcoRⅠandHind Ⅲ.
2.4 同源性分析
STT3a基因的同源性分析表明:在STT3a基因的3'下游序列中發現WWDYG與DK兩個保守的基序。克隆獲得STT3a基因1 869 bp,其中STT3a的功能結構域cDNA序列約為1 650 bp,編碼550個氨基酸(圖3),BLAST比對顯示它和衣藻、擬南芥、稻、高粱、毛果楊、玉米、人等的 STT3a同源性分別為 48%、50%、50%、50%、49%、50%和46%。GenBank登錄序列號:GU301076。
2.5 不同鹽濃度誘導下STT3a mRNA表達情況
如圖4所示:相對于1.5 mol/L NaCl鹽濃度的正常培養條件,杜氏鹽藻STT3amRNA水平在0.75、1.0、2.0、3.0和3.5 mol/L NaCl鹽濃度培養條件下明顯升高(P<0.01),且在3.5 mol/L NaCl濃度時比在1.5 mol/L NaCl濃度時升高了11倍。圖5中可以看出,在高鹽脅迫下(3.0 mol/L NaCl),STT3amRNA的水平高于正常鹽濃度條件(1.5 mol/L NaCl)下的STT3amRNA水平(P<0.05),在 10 h時,STT3amRNA的水平相對于0 h升高了8.5倍。這說明STT3a基因的表達與鹽誘導有關,STT3a基因可能直接或間接參與了杜氏鹽藻的耐鹽機制。
2.6 杜氏鹽藻鞭毛再生不同時期STT3a mRNA表達情況

圖3 STT3a蛋白推測的功能結構域Fig.3 Putative functional domains of STT3a fromD.salina.

圖4 不同鹽濃度誘導下 STT3a基因表達情況(**P<0.01)Fig.4 Expression ofD.salinaSTT3agene in different salt concentrations.**P<0.01.
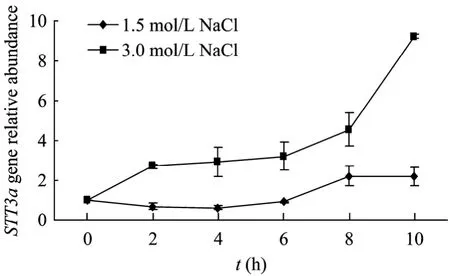
圖5 高鹽脅迫(3.0 mol/L NaCl)下杜氏鹽藻STT3a基因表達情況Fig.5 Expression ofD.salinaSTT3agene in high salt stress(3.0 mol/L NaCl).
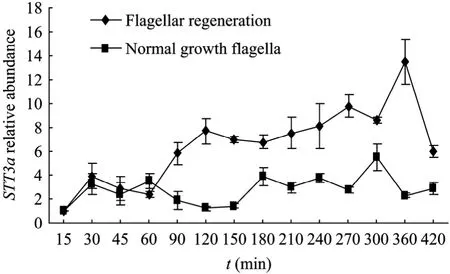
圖6 鞭毛再生不同時期STT3a基因表達情況Fig.6 Expression ofD.salinaSTT3agene in different states of flagellar regeneration.
如圖6所示:杜氏鹽藻鞭毛再生過程中STT3amRNA水平較正常培養條件下沒有脫鞭毛的持續高表達。鞭毛再生過程0~60 min中STT3amRNA的水平基本沒有變化,60~120 min中STT3amRNA的水平逐漸升高,說明杜氏鹽藻鞭毛在最初受損再生過程中延遲至 60 min才開始逐漸大量表達,120~300 min中STT3amRNA的水平處于平臺期,但相比較正常培養條件下的杜氏鹽藻,其STT3amRNA仍大量表達,這個時期的杜氏鹽藻鞭毛仍在逐漸生長,420 min后,STT3amRNA的水平逐漸趨于正常值(P<0.05)。
2.7 杜氏鹽藻鞭毛再生不同時期鞭毛長度變化及相對生長速率
從圖7中可以清楚地看出杜氏鹽藻鞭毛再生的前15 min鞭毛增長比較明顯,隨著時間的延長,杜氏鹽藻鞭毛逐漸延長,至420 min時基本上完成鞭毛再生。圖8中可以看出,在杜氏鹽藻鞭毛再生的初級階段,鞭毛生長較快,在前15 min內,其相對生長速率為 0.0835 μm/min,30~240 min 時鞭毛的相對生長速率逐步降低,300~360 min時鞭毛相對生長速率降低至0.0017 μm/min,表明鞭毛的生長基本完成。在杜氏鹽藻鞭毛再生的不同時期,其鞭毛相對生長速率的變化與STT3a基因mRNA水平的變化基本相一致。

圖8 杜氏鹽藻鞭毛再生不同時期的相對生長速率Fig.8 The relative growth rates ofD.salinain different times of flagellar regeneration.
3 討論
OST是蛋白進入分泌途徑的門戶,它負責催化完整的多萜醇結合前體聚糖向多肽接納體的轉移,這種酶的特異性決定需要有Asn-X-Ser/Thr(X是除脯氨酸以外的任何氨基酸)這一接納體序列的存在。在OST的作用下,寡糖鏈與膜上的磷酸多萜醇分離,而與Asn的N-端相連。完整的寡糖鏈向新生多肽鏈的轉移發生在內質網的腔內側。
以往對STT3a的研究多集中在對其結構方面的研究,但對其功能的研究還相對較少。研究發現STT3為 OST的催化亞單位,它有一個Ncyt-Clum拓撲結構,含有 11個跨膜螺旋[8],3′下游序列中有WWDYG與 DK兩個保守的基序[9]。Koiwa 等[4]發現擬南芥OST的STT3a亞基對鹽和滲透壓有反應,本研究中我們也證實了STT3a基因的表達與鹽誘導有關,隨著鹽濃度的逐漸升高,STT3a的mRNA表達也提高。
杜氏鹽藻的耐鹽機制比較復雜,到目前為止還沒有完全研究清楚。研究發現杜氏鹽藻的耐鹽機制除受到甘油和離子流動的調節外,還有一些蛋白質的參與[10]。Fisher等[11]在杜氏鹽藻細胞膜中發現了一種 p60蛋白質,它是一種雙拷貝的碳酸酐酶(DCA)基因,具有調節細胞滲透壓的作用,而且隨著鹽濃度的升高,其mRNA水平和細胞外DCA的活性也同時提高。本研究室也發現杜氏鹽藻細胞膜中的DCA基因mRNA水平隨著鹽濃度的升高而升高,并且在2.0 mol/L NaCl鹽濃度下杜氏鹽藻DCA活性達到最高。STT3a作為杜氏鹽藻一種新發現的跨膜蛋白,能直接感受培養基中鹽濃度的變化,本研究也證實杜氏鹽藻STT3a的 mRNA水平隨著鹽濃度的升高而升高。這些結果可能為我們研究杜氏鹽藻DCA與STT3a在耐鹽方面的協同作用提供理論依據。
鞭毛與纖毛是一種由細胞質膜延伸的細胞表面突起,主要由微管組成。鞭毛膜作為特化的細胞膜,其再生受到多種基因的精確調控,然而關于其調控機制知之甚少。為了解跨膜蛋白STT3a在杜氏鹽藻鞭毛再生中的作用,本研究監測了鞭毛再生不同時期STT3amRNA水平的變化。結果顯示,STT3a基因在杜氏鹽藻鞭毛最初受損再生過程中延遲至60 min才開始逐漸大量表達,之后直至鞭毛完成再生過程中都高表達。這可能是因為杜氏鹽藻藻體本身儲存的一些STT3a蛋白在鞭毛再生的初期起到作用,隨著鞭毛再生過程中對STT3a蛋白需求的增加,STT3a基因開始逐漸大量表達。這提示STT3a可能作為一種跨膜蛋白,直接或間接參與了杜氏鹽藻鞭毛膜的再生或者鞭毛再生過程中所需相關蛋白的糖基化修飾,這些結果可能為我們研究杜氏鹽藻鞭毛再生過程中基因的調控機制提供理論依據。
鞭毛作為杜氏鹽藻的運動細胞器直接參與鹽藻的游動,而高鹽條件下杜氏鹽藻運動遲緩是否和高鹽影響相關基因的表達有關,目前還沒有實驗證據。本研究發現跨膜蛋白STT3a既參與了杜氏鹽藻的耐鹽調控,又參與了杜氏鹽藻鞭毛的再生過程,提示杜氏鹽藻的鹽耐受性和鞭毛介導的運動性之間可能有潛在的聯系,值得進一步的研究。
REFERENCES
[1]Knauer R, Lehle L.The oligosaccharyltransferase complex from yeast.Biochim Biophys Acta, 1999, 1426(2):259?273.
[2]Burda P, Aebi M.The dolichol pathway of N-linked glycosylation.Biochim Biophys Acta, 1999, 1426(2):239?257.
[3]Kelleher DJ, Karaoglu D, Mandon EC.Oligosaccharyltransferase isoforms that contain differentcatalytic STT3 subunits have distinct enzymatic properties.Mol Cell, 2003, 12(1): 101?111.
[4]Koiwa H, Li F, McCully MG,et al.The STT3a subunit isoform of theArabidopsisoligosaccharyltransferase controls adaptive responses to salt/osmotic stress.Plant Cell, 2003, 15(10): 2273?2284.
[5]Livak KJ, Schmittgen TD.Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△Ctmethod.Methods, 2001, 25(4): 402?408.
[6]Rosenbaum JL, Moulder JE, Ringo DL.Flagellar elongation and shortening inChlamydomonas.The use of cycloheximide and colchicine to study the synthesis and assembly of flagellar proteins.J Cell Biol, 1969, 41(2):600?619.
[7]Jia YL, Xue LX, Li J,et al.Isolation and proteomic analysis of the halotolerant algaDunaliella salinaflagella using shotgun strategy.Mol Biol Rep, 2010, 37: 311?316.
[8]Kim H, von Heijne G, Nilsson I.Membrane topology of the STT3 subunit of the oligosaccharyl transferase complex.J Biol Chem, 2005, 280(21): 20261?20267.
[9]Igura M , Maita N, Kamishikiryo J,et al.Structureguided identi fi cation of a new catalytic motif of oligosaccharyltransferase.EMBOJ, 2008, 27(1):234?243.
[10]Li J, Lu YM, Xue LX,et al.A structurally novel salt-regulated promoter of duplicated carbonic anhydrase gene 1 fromDunaliella salina.Mol Biol Rep, 2010, 37:1143?1154.
[11]Fisher M, Gokhman I, Pick U,et al.A salt-resistant plasma membrane carbonic anhydrase is induced by salt inDunaliella salina.J Biol Chem, 1996, 271(30):17718?17723.
Characterization of the functional domain of STT3a of oligosaccharyltransferase from Dunaliella salina
Cui Wang, Jie Li, Liping Liu, Lei Zeng, and Lexun Xue
Laboratory for Cell Biology, Department of Bioengineering, Zhengzhou University, Zhengzhou 450001, China
Received:December 29, 2009;Accepted:April 21, 2010
Supported by:International Cooperative Project of the National Science and Technology Ministry(No.2007DFA01240), National Natural Science Foundation of China(No.30700014).
Corresponding author:Jie Li.Tel: +86-371-66658332; E-mail: lijiechen@zzu.edu.cn Lexun Xue.Tel: +86-371-66658332; E-mail: xuelx@371.net科技部國際科技合作項目(No.2007DFA01240),國家自然科學基金項目(No.30700014)資助。

