綠潮爆發后青島海域石莼屬綠藻的多樣性分析
梁成偉,張曉雯,宿烽,葉乃好
(1.青島科技大學化工學院,山東 青島 2660422; 2.中國水產科學研究院黃海水產研究所,山東 青島 266071)
綠潮爆發后青島海域石莼屬綠藻的多樣性分析
梁成偉1,張曉雯2,宿烽1,葉乃好2
(1.青島科技大學化工學院,山東 青島 2660422; 2.中國水產科學研究院黃海水產研究所,山東 青島 266071)
綠潮爆發后在青島沿海潮間帶采集了12株石莼屬綠藻,形態學結合分子生物學手段對這些綠藻進行多樣性鑒定。經過PCR擴增獲得了12株藻的核糖體轉錄間隔區(ITS)序列以及完整的5.8S rDNA序列。經過序列分析發現,不同藻株的ITS區域在序列長度上存在差異;G+C含量也存在差異,但是都處于較高的水平,最高的接近72%。ITS區域多序列比對結果及系統發育分析顯示這些藻株存在種的多樣性,而且由分子鑒定得出的多樣性與形態上呈現的多樣性并不完全一致。本研究結果顯示在青島海域綠潮爆發后石莼藻類仍然存在較好的多樣性。
石莼屬;ITS序列;多樣性
Abstract:Morphology combined with molecular biology were used to study the diversity of 12 strains of Ulva collected from Qingdao intertidal belt after largest macroalgal bloom in 2008.The nuclear-encoded ITS and associated 5.8S rDNA regions were obtained and sequenced.Sequence analysis revealed the ITS regions from different strains showed different patterns in length and G+C content.Furthermore,the frequencies of nucleotides G and C in ITS regions were at high level in all algal strains,and some were close to 72%.The results of alignment and phylogenetic tree showed these strains had diversity at species taxon.However,the polymorphism showed by morphology and molecular sequence was not consistent completely.In conclude,the results in this study revealed the diversity of Ulva algae in Qingdao belt after green tide.
Keywords:Ulva; ITS sequence; diversity
石莼屬(Ulva)屬于綠藻門 (Chlorophyta)、石莼科(Ulvaceae)的一屬,多數種類分布在溫帶至亞熱帶海洋中;生長在高潮帶至低潮帶和大干潮線附近巖石上或石沼中,目前發現約有100個種包括在這個屬內[1]。綠潮(Green tide)是世界沿海各國普遍發生的生態異常現象,在歐洲、亞洲、北美洲和澳大利亞均有記錄,主要發生在河口、內灣、潟湖和城市密集的海岸。造成綠潮的主要生物種類是石莼(Ulva sp.)和滸苔(Enteromorpha sp.),以石莼居多[2]。近兩年來在山東地區的海域也頻頻暴發綠潮。2008年6月在黃海中南海域爆發的綠潮藻,經鑒定為滸苔,并且造成本地綠潮藻并不是本地海域藻類的大量繁殖,而是由其他地區的海域漂浮而來的[3]。由于此次綠潮的爆發引起社會各界對漂浮滸苔高度關注,同時藻類學家在滸苔生理習性、繁殖周期及分子鑒定等方面也開展了一系列研究,并取得了一定的進展[4-6]。但是與此相比,對于石莼的研究主要集中其生長發育、營養成分及在漁業方面的應用[7-9],而對于某一特定海域中石莼的分類多樣性研究的較少;青島海域石莼綠藻種類多樣性也未見報道。因此,青島海域在綠潮爆發以后,當地相關藻類的多樣性如何引起我們的關注。
本研究通過形態學和分子生物生物學方法首次在綠潮爆發后對青島海域范圍內石莼藻類進行了系統發育關系和種質多樣性分析,為青島本土海域的綠藻資源提供一定的實驗數據,同時,為今后綠潮藻的來源分析和爆發機制的研究提供一定的幫助。
1 材料與方法
1.1 樣本的采集
在2009年4月對山東青島(市區、膠南、紅島、即墨)沿岸進行石莼藻的采集并制作標本。
1.2 模板DNA 的提取
將采集的藻類用無菌水清洗3遍,用吸水紙吸干,在液氮冷凍條件下將樣品研磨成粉末狀,利用天根植物全基因組提取試劑盒(DP320)提取全基因組DNA。
1.3 IST序列的擴增
用PCR方法進行從石莼樣本中獲得的ITS1-5.8S-ITS2序列。進行ITS 擴增的引物序列根據Leskinen 等[10]的結果設計,分別為T1(5'-TCGTAACAAGGTTTCCGTAGG-3')和 T2(5'-TTCCTTCCGCTTATTGATATGC-3'),由上海生物工程有限公司合成。PCR 反應總體系為25 μL,其中包括:10×PCR buffer 2.5 μL,2.0 mmol/L MgCl22.0 μL,10 mmol/L dNTPs 0.5 μL,10 μmol/L 引物T1 和T2 各1 μL,Taq DNA 聚合酶(Promega)0.2 μL,DNA 模版1 μL。反應條件為94 ℃預變性6 min;94 ℃ 變性1 min,54 ℃ 退火1 min,72 ℃ 延伸1 min,循環30 次;72 ℃繼續延伸10 min,4 ℃保存。PCR 產物進行瓊脂糖電泳,將目的條帶回收純化之后,送北京六合華大基因科技股份有限公司測序。
1.4 數據分析
將獲得ITS序列用BLAST與NCBI數據庫中序列進行比對。并從NCBI 數據庫中下載選代表性的物種的ITS 及5.8S rDNA 序列用于序列分析及系統進化分析,選用序列的登錄號及對應的物種如下:AY422521 (U.Procera)、AB097648 (U.linza)、FJ002301 (U.proliferna)、AB097662 (U.armoricena)、AJ234316 (U.fenestrate)、AY260560(U.californica)、AB097644 (U.flexuosa)、AB298457 (Ulva sp.)、FJ194957 (Ulva sp.)、AF013981 (U.Compressa)。另外,加入由本實驗室獲得的青島地區爆發的綠潮滸苔的ITS及5.8S rDNA 序列和外群序列AY360570 (P.percursa)。用Clustal X[11]軟件進行多序列比對及序列相似性分析。我們采用選取不同的方法(距離鄰接算法NJ、最大簡約性算法MP和最大似然性算法ML)分別構建系統發育樹,上述建樹過程是在MEGA 4.0[12]和(或)PHYLIP軟件包中實現的。系統發育樹的可靠性是通過Bootstrap方法重抽樣1 000次進行的。
2 結 果
2.1 不同藻株的形態及分布
從山東海域潮間帶采集石莼樣本在形態、色澤存在較大的差別,石莼樣本的形態見圖1。從外觀可以看出,所采集的樣本在形態上存在多樣性。有些樣本為膜狀體,呈寬葉片狀或裂成許多小葉片,如SH107、SH102、SH105;有些石莼樣本呈狹長的絲帶狀,如SH101、SH104、SH108、SH106; 有些石莼樣本呈管狀,并且分枝,藻體基部生出假根絲,形成固著器,如SH103、SH202、SH204;還有些石莼藻株成簇成長,如:SH201和SH203。
2.2 ITS序列分析
PCR產物經測序獲得600 bp左右的序列片段,包括部分 18S rDNA 和完整的 ITS1序列,5.8S rDNA序列、ITS 2 序列和部分28 S rDNA 序列。不同的藻株ITS1 長度不等(表1),SH105藻株的ITS1 序列最短,只有 152bp,而 SH104 、SH106和 SH108 三株藻的 ITS區域較長,有 196bp。SH202、SH204、SH101、SH102和SH107 具有相同長度的ITS1區域,長度為189 bp。 5.8S rRNA基因大小差別不大,除了SH203以外,其余藻株的5.8S rDNA的長度都是160 bp。通過比較ITS2區域的長度,發現具有最短ITS1區域的SH105藻株,卻具有最長的ITS2 區域,為199 bp,而其他的藻株ITS2區域長度范圍從175 bp到187 bp。
對ITS 1,5.8S r DNA 和ITS 2 區域的核苷酸組分分析,發現在ITS區域均具有較高的G+C含量,范圍大約在62% 到70% 之間。而5.8 S rDNA的G+C的含量基本上都接近50%(表1)。
2.3 多序列比對
從序列比對的結果來看(圖2),在核糖體RNA編碼區(包括 18S、5.8S、28S )是比較保守的,但是在ITS區域不同物種序列比對時會產生很多間隙(gaps)。并且,在ITS1 區域內序列差異較大,由比對產生的間隙也較多。在藻株SH105 ITS 1序列中,產生長達13個堿基的比對間隙。在ITS2中,雖然核苷酸之間的差異也較大,但是產生比對空隙的區域較少。

圖1:青島潮間帶采集的石莼藻株樣品的形態特征Fig 1.Morphological characteristics of Ulva colleted from the intertidal belt in Qingdao
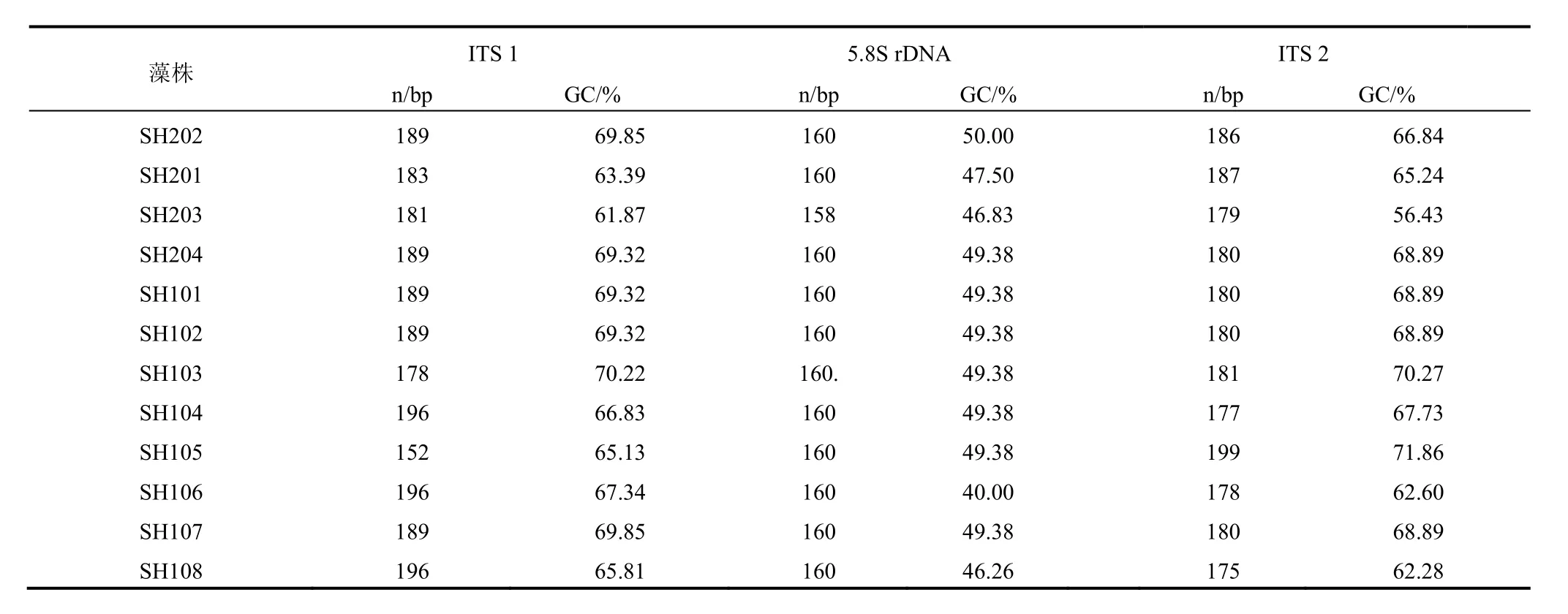
表1 不同藻株 ITS 1,5.8S rDNA 和ITS 2 區域的長度(n=核酸的數目)和GC含量Tab.1.Length (n=number of nucleotides)and GC% of ITS 1,5.8S rDNA and ITS 2 in different Ulva algae
2.4 藻株的分子鑒定與系統發育分析
為構建系統進化樹,從NCBI 數據庫中下載了10個石莼屬的ITS 和5.8S rDNA 的全序列,并以狹帶藻(Percursaria percursa)的ITS 和5.8S rDNA全序列作為外群[13]。采用Mega4.0 中的鄰接法及Phylip3.65 軟件中的最大簡約法構建了ITS 及5.8S rDNA 的系統進化樹,兩棵進化樹具有類似的拓撲結構,說明了系統進化樹的可靠性。文中給出的是NJ 法構建的系統樹,從該系統進化樹可以看出(圖3),采集到的石莼藻株在系統發育樹上聚為5個不同的類群中:其中藻株SH201,SH202,SH203 與U.compressa 聚為一大分支;藻株SH104、SH106和SH108 與U.califormica和U.flexuosa聚在一個大分支;藻株SH105與U.fenetrata 和U.armoricana聚為一個分支;藻株 SH101、SH107、SH102、SH204與U.proliferna、U.linza、U.Procera以及爆發綠潮藻聚為一大分支;SH103與兩個未確定物種的石莼屬藻類聚在一起。
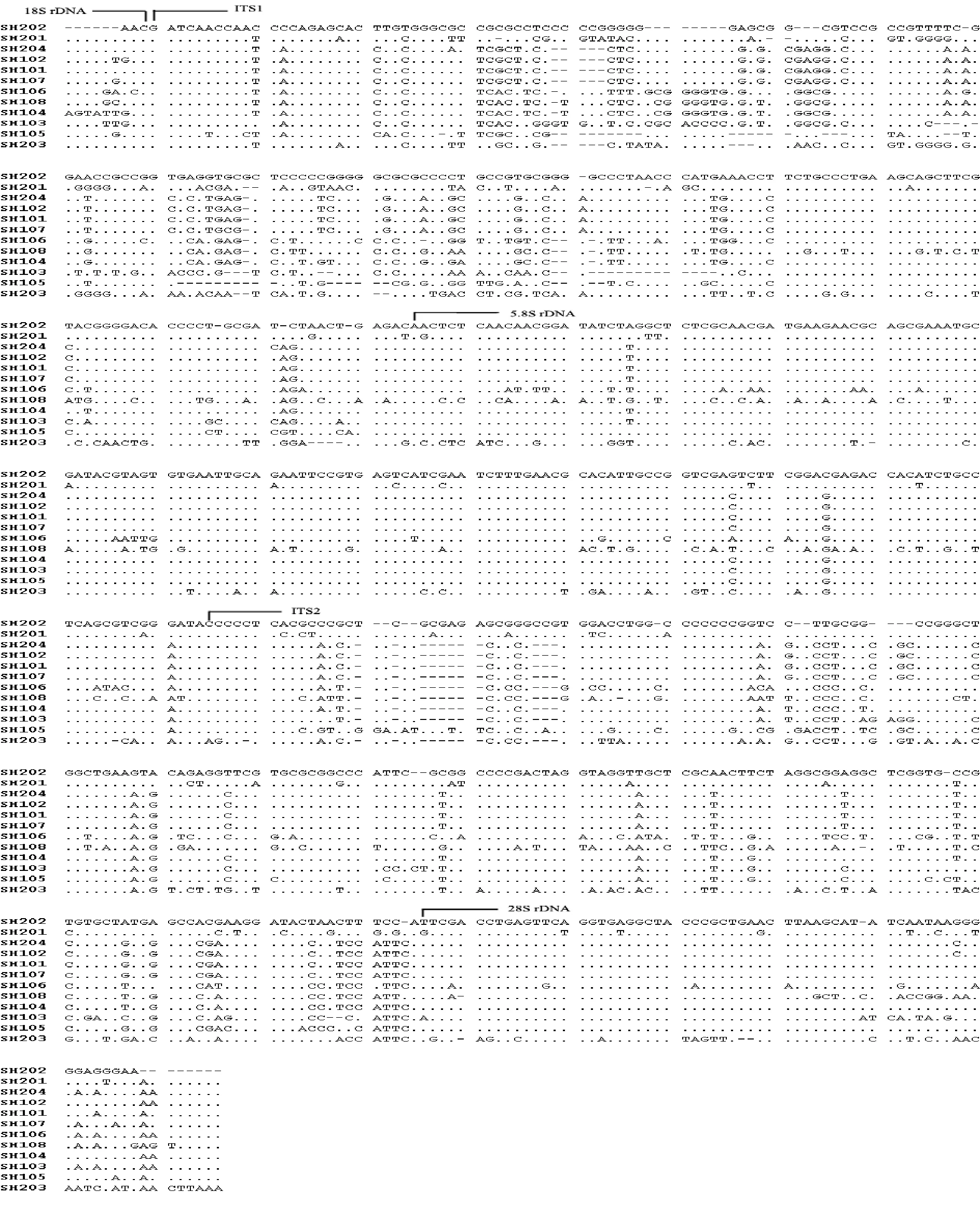
圖2 不同藻類樣品ITS1、5.8S和ITS2 全序列比對結果(“﹒”表示與最上面一行的核酸一致; “-”表示產生的比對空隙)Fig.2.The alignment of sequences of IST (including ITS1,5.8S and ITS2)from different Ulva algae.“﹒” this sign indicates a nucleotide indentical with that on the top row,and “-” represents an alignment gap.
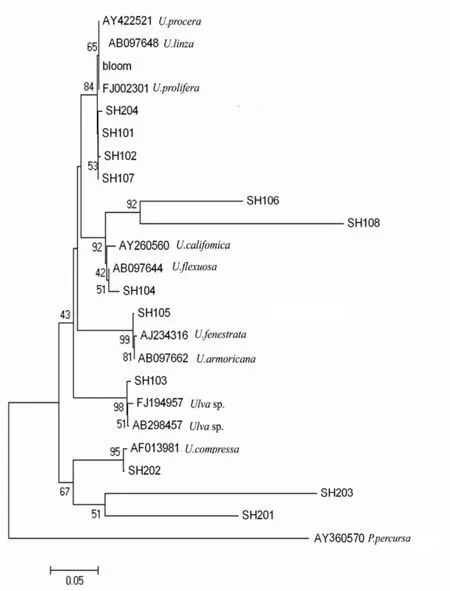
圖3 利用NJ法構建的ITS序列的系統進化樹Fig.3 Phylogenetic tree of ITS sequences constructed by Neighbor-Joining Method
3 討 論
真核生物基因組中編碼核糖體的基因包括 3種:28S rDNA,18S rDNA和5.8 S rDNA。它們在染色體上頭尾相連、串聯排列,相互之間由間隔區分隔。間隔區是位于核糖體大小亞基基因之間的核苷酸序列。位于18S rDNA的3′端與5.8S rDNA的5′端之間的序列稱為核糖體內轉錄間隔區 1(internal transcribed spacer 1,ITS1)位于5.8S rDNA的3′端與28S rDNA的5′端之間的序列稱為核糖體內轉錄間隔區 2 (internal transcribed spacer 2,ITS2)。28S rDNA的3′端與18S rDNA的5′端之間的序列稱為核糖體轉錄間隔區(internal genic spacer,IGS)。這 3種核糖體基因及間隔區有不同的進化程度,有的序列比較保守,有的序列進化較快,因此,可以根據它們的序列,將藻類鑒定到屬、種、亞種、變種的水平。
由于藻類隨著分布海域的不同而在形態、色澤以及藻體大小等方面會表現出明顯的差異,僅僅根據形態學判斷藻類的分類地位較為困難,所以現在通常借助于分子標記在種一級分類地位上進行確定。在本文中,我們擴增得到了青島海域石莼(Ulva)藻ITS序列包括5.8S rDNA 序列,通過比較ITS序列的多態性結合了藻類的形態特征首次研究了在生長在青島海域的石莼類藻的多樣性。通過比較ITS序列的長度和GC含量發現,這些不同的藻株在ITS區域序列長度差異較大,其中ITS 1序列長度差可達到 44 bp,通過多序列比對,也發現ITS1區域內存在較多的間隙,說明ITS區域在進化過程中插入或刪除事件是很普遍發生的。核苷酸組分分析發現,所采集的藻類的ITS區域的G+C含量也存在一定差異(表1),但是都處在較高水平。通常情況下,ITS1和ITS2 區域雖然中間間隔5.8S r DNA,但具有相近的G+C含量[14]。植物中,ITS區域的 G+C含量從 50% 左右到水稻中高達75%[15,16],在以前的文獻中也報道其他的綠藻 ITS中的G+C含量都低于60%,接近50%[17]。與ITS系列不同,在我們所測的樣品中,5.8S rDNA中G+C含量與其他的高等植物或綠藻中的含量較接近。在石莼屬綠藻中ITS區域的高G+C含量,可能是由于進化過程中受到堿基突變壓力,或受到高 G+C含量的選擇或者其他因素的影響。ITS序列長度及核苷酸組分都顯示了這些藻類在分子水平上呈現了多態性。
根據ITS序列構建系統發育樹結果顯示,采集的藻類在系統發育樹上聚在5個不同的分支上,說明它們在種的水平上存在多樣性,甚至存在種內多樣性,例如藻株SH106和SH108,很可能呈現出來是種內的多樣性。從系統發育進化樹來看,在分子水平上呈現的多樣性并不完全與形態上呈現的多樣性一致,對兩種方法存在的差異進行了比較(圖4)。例如,藻株SH103、SH202、SH204在形態上比較接近(圖1),但是它們分別位于不同的進化支上。SH104、SH108、SH106在形態上比較接近,它們在系統發育樹上也呈現出較近的親緣關系。SH101、SH102、SH107、SH204與青島地區綠潮爆發藻類具有較近的親緣關系。從分子鑒定結果可以看出在滸苔綠潮爆發之后,在青島海邊并沒有發現定生的黃海漂浮滸苔,而且對當地石莼屬藻類的多樣性影響不大。本研究首次對青島海域石莼屬藻類多樣性的進行研究,今后將會對綠潮藻的鑒定及來源分析提供一定的幫助。
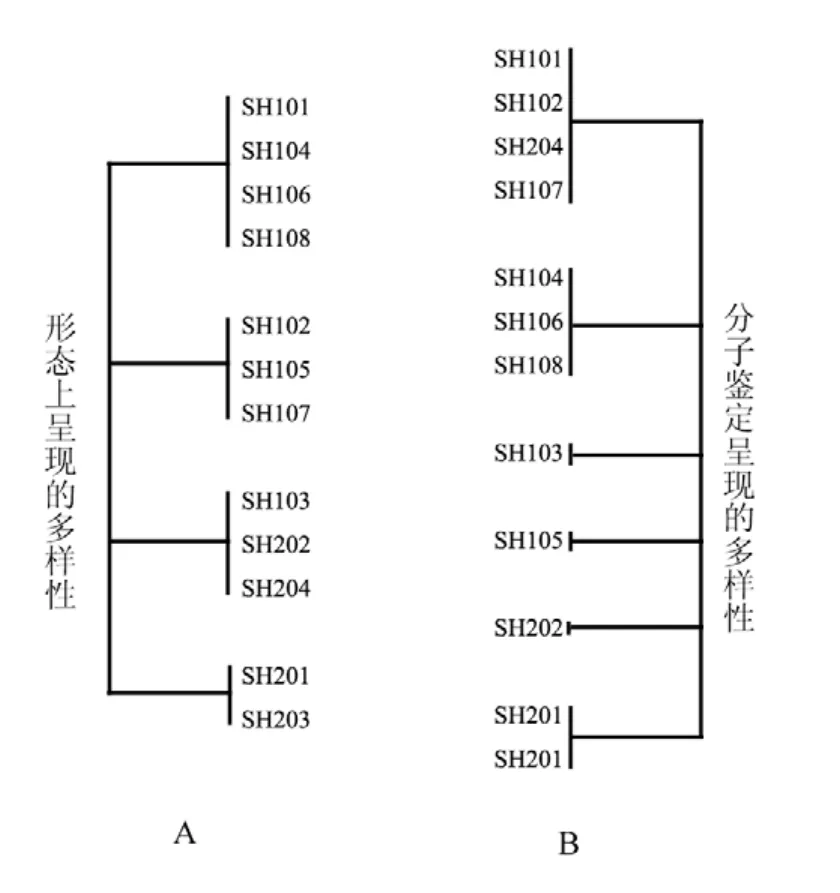
圖4 分子鑒定得出的多樣性與形態上呈現的多樣性的比較(A 圖中分支代表藻類具有相似的形態; B 圖中分支代表在系統發育樹中處于同于分支)Fig.4 Comparison of the morphology diversity and the diversity presented by molecular identification.The branch represents the similar morphology among different strains in figure A; the branch in figure B represents the different strains clade in the phylogenetic tree.
[1]Guiry M D,Guiry G M.AlgaeBase version 4.2.World-wide electronic publication,National University of Ireland,Galway.http://www.algaebase.org;search on 14 March 2007.
[2]Callow M E,Callow J A,Pickett-Heaps J D,et al.Primary Enteromorpha adhesion of Chlorophyta Ulvales propagules quantitative settlement studies and video microscopy [J].J Phycol,1997,33:938-947.
[3]Liu D Y,Keesing J K,Xing Q G,et al.World’s largest macroalgal bloom caused by expansion of seaweed aquaculture in China [J].Marine Pollution Bulletin,2009,58(6): 888-895.
[4]Leliaert F,Zhang X W,Ye N H,et al.Identity of the Qingdao algal bloom [J].Phycological Research,2009,57: 147-151.
[5]Lin A,Shen S D,Wang J W et al.Reproduction Diversity of Enteromor phaprolifera.Journal of Integrative Plant Biology[J].2008,50 (5): 622-629.
[6]張曉雯,毛玉澤,莊志鵬,等.黃海綠潮滸苔的形態學觀察及分子鑒定 [J].中國水產科學,2008,15: 822-829.
[7]Hiraoka M,Dan A,Shimada S,et al.Different life histories of Enteromorpha prolifera (Ulvales,Chorophyta)from four rivers on Shikoku Island [J].Phycologia,2003,42:275-284.
[8]Dan A,Hirosawa A,Makino K,et al.Observation on the effect of salinity and photon fluency rate on the induction of sporrulation and rhizoid formation in the green alga,Enteromorpha prolifera (Müller)J.Agardh (Chlorophyta,Ulvales)[J].Fish Sci,2002,68:1182-1188.
[9]部音利,馮亞非,孔東梅,等.南海條滸苔和裂片石莼的油脂提取及其脂肪酸分析 [J].食品科技,2008,33: 186-189.
[10]Leskinen E,Pamilo P.Evolution of the ITS sequence of Enteromorpha ribosomal DNA in Chlorophyceae [J].Hereditas,1997,126: 17-23.
[11]Thompson J D,Gibson T J,Plewniak F,et al.The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignmen aided by quality analysis tools [J].Nucleic Acids Res,1997,25: 4 876-4 882.
[12]Tamura K,Dudley J,Nei M,et al.MEGA4: Molecular Evolutionary Genetics Analysis (MEGA)Software Version 4.0 [J].Molecular Biology and Evolution,2007,24: 1 596-1 599.
[13]Shimada S,Hiraoka M,Nabata S,et al.Molecular phylogenetic analysis of the Japanese and Ulvales Ulvophyceae with special reference to the free-floating Ulva [J].J Phycol Res,2003,51:99-108.
[14]Baldwin B,Sanderson M J,Porter J M ,et al.The ITS region of nuclear ribosomal DNA: a valuable source of evidence on angiosperm phylogeny [J].Ann.Missourri Bot Gard,1995,82:247-277.
[15]Torres R A,Ganal M,Hemleben V.GC balance in the internal transcribed spacers ITS 1 and ITS 2 of muclear ribosomal RNA genes [J].J Mol Evol,1990,30: 170-181.
[16]Takaiwa R,Oono K,Sugira M.Nucleotide sequence of the 17S -25S spacer region from rice rDNA [J].Plant Mol.Biol,19 854:355-364.
[17]Leskinen E,Pamilo P.Evolution of the ITS sequences of ribosomal DNA in Enteromorpha (Chlorophyceae).[J].Hereditas,1997,126:17-23.
Analysis of diversity of Ulva in Qingdao after the largest macroalgal bloom
LIANG Cheng-wei1,ZHANG Xiao-wen2,SU Feng1,YE Nai-hao2
(1.Chemical Engineering College,Qingdao University of Science & Technology,Qingdao 266042,China;2.Yellow Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Qingdao 266071,China)
Q949.21+3; Q16 文獻識別碼:A
1001-6932(2010)05-0540-06
2009-08-25;
2009-12-26
青島市公共領域科技支撐計劃項目(09-2-5-8-hy);山東省自然科學基金面上項目(ZR2009EM013,Y2008D03);國家轉基因專項(2009ZX08009-019B);化學工程山東省強化建設學科(青島科技大學)開放基金;青島市科技計劃項目(08-1-3-27-jch)
梁成偉(1979-),女,講師,主要研究方向:藻類分子生物學。電子郵箱:liangchengwei@qust.edu.cn
葉乃好,電子郵箱:yehn@ysfri.ac.cn

