棘孢木霉TspyrG基因選擇標記轉化體系的建立



摘 要 旨在從棘孢木霉Ts93中克隆獲得尿嘧啶合成關鍵酶乳清酸核苷-5′-磷酸脫羧酶基因(TspyrG),該基因無內含子,開放式閱讀框大小為1 140 bp,編碼1個379氨基酸的蛋白。通過農桿菌介導技術,獲得棘孢木霉Ts93 TspyrG基因缺失突變株ΔTspyrG,突變株在不含尿嘧啶的培養基上無法生長。同時,以PCAMBIA1300質粒為骨架,構建轉化載體1300pyrG。以ΔTspyrG突變株為受體菌,成功構建以尿嘧啶營養缺陷為選擇標記的木霉菌農桿菌轉化系統。對綠色熒光蛋白(eGFP)的轉化試驗表明,該系統轉化效率與抗生素選擇標記轉化系統無明顯差異。
關鍵詞 棘孢木霉;營養缺陷型;農桿菌轉化系統
木霉是土壤中廣泛分布的一種絲狀真菌,經過近一個世紀的研究,已經發現這類菌株在促進植物生長、抑制病原菌、誘導植物抗性等方面都具有明顯效果,是一種應用廣泛的生防菌[1-2]。同時,木霉菌具有生長迅速、基因組小、分生孢子為單倍體、分子遺傳背景清晰等優點,有利于分子操作[3]。目前對木霉菌的分子操作主要選擇抗生素作為篩選標記,如潮霉素、G418[4]等。抗生素選擇標記由于成本高、環境污染大等缺點,不適于大規模突變株庫的構建[5]。營養缺陷型篩選標記是較好的代替方案[6-8],在酵母中已報道多種編碼營養物質代謝相關酶的基因,特別是氨基酸合成(如L-組氨酸、L-亮氨酸、L-色氨酸等)相關酶[9]。其中乳清酸核苷-5′-磷酸脫羧酶(EC:4.1.1.23)基因(pyrG)是常用的篩選標記。乳清苷-5′-磷酸脫羧酶是尿嘧啶核苷酸合成途徑中一種關鍵酶,缺失該酶的突變株無法合成尿嘧啶,需要外源補充;同時,該酶可以將5-氟乳清酸(5-FOA)催化為5-氟乳清苷酸(5-FUMP),5-氟乳清苷酸具有很強的細胞毒性,導致含有pyrG基因的野生株不能在含有5-FOA的培養基中存活。因此5′-氟乳清酸(5-FOA)和尿嘧啶可以作為遺傳轉化的正向反向篩選標記[10]。
隨著生物信息學技術在基因功能預測方面展示明顯優勢,功能基因的篩選已經有很強的預見性。但是,對于新功能基因的篩選,或者某些已知功能基因的新功能開發方面,突變株庫構建及篩選仍具有明顯優勢。目前,絲狀真菌中最常用的轉化方法是農桿菌介導轉化法(Agrobacterium-mediated transformation,ATMT)[11]。經過20多年的完善,ATMT已成功應用于50多種絲狀真菌的轉化[12]。而木霉菌的ATMT轉化具有效率高、單拷貝插入概念高等優勢。另外,突變株庫的構建需要廉價、高效、安全的選擇標記。本研究在前期以潮霉、G418作為篩選標記的木霉菌農桿菌轉化系統的基礎上,構建以TspyrG基因為篩選標記的農桿菌轉化體系,以棘孢木霉(Trichoderma asperellum)為受體,探討無抗生素篩選標記的突變株庫構建的可行性。
1 材料與方法
1.1 菌株與質粒
棘孢木霉Ts93,南昌師范學院生科院真菌分子遺傳實驗室分離純化,保藏在武漢中國典型培養物保藏中心(CCTCC NO:M2017147),該菌株已完成全基因組測序。質粒PCAMBIA1300、 eGFP、eYFP,南昌師范學院生科院真菌分子遺傳實驗室保存。
1.2 主要試劑與培養基
PCR相關酶、內切酶、T4連接酶等購自Takara公司。5-FOA和尿嘧啶購自Sigma公司。5-FOA用DMSO溶解,配制成600 mg/mL母液,備用;尿苷配制成6 mg/mL水溶液過濾除菌備用。
CA培養基:NaNO3 2 g/L,K2HPO4·3H2O 1 g/L,KCl 0.5 g/L,MgSO4·7H2O 0.5 g/L,FeSO4·7H2O 0.01 g/L,蔗糖 30 g/L,瓊脂粉 15 g/L,滅菌后按量加入尿苷至終濃度為60 mg/L。
PDA培養基:去皮土豆 200 g/L,葡萄糖20 g/L,瓊脂粉15 g/L。
1.3 試驗方法
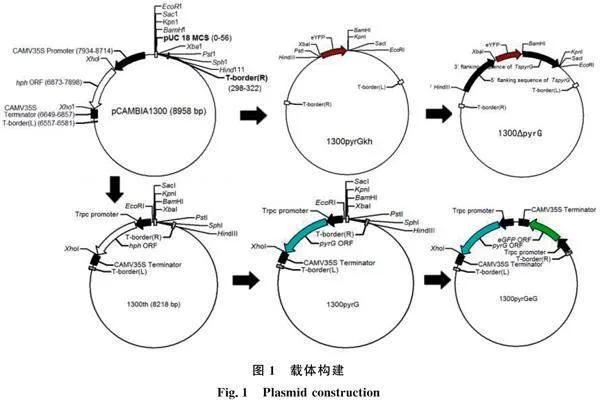
1.3.1 載體構建 以NCBI下載已知的pyrG基因序列,本地BLAST搜索Ts93全基因組序列,獲取TspyrG基因序列。圖1為載體圖,載體構建主要過程如下:以pCAMBIA1300載體為骨架,用XhoⅠ單切后自連,去除潮霉素基因。然后擴增473 bp eYFP基因序列(只取了部分eYFP基因序列,用于后期敲除突變株的PCR驗證,引物為XbeYFPU/BaeYFPL),通過XbaⅠ/BamHⅠ雙切后連接成1300pyrGkh;敲除載體按照同源置換方法,設計引物(表1)分別擴增基因5′端1 079 bp同源序列(引物HiTspyrGSU/XbTspyrGSL),3′端974 bp同源序列(引物BaTspyrGXU/KpTspyrGXL),酶切連接至1300pyrGkh載體,構建TspyrG基因敲除載體1300ΔpyrG。設計引物擴增TspyrG基因ORF(引物XhTspyrGU/XhTspyrGL),以1300th載體為骨架,通過酶切連結,將其中的潮霉素抗性基因ORF替換為pyrG基因的ORF,構建TspyrG基因重組載體1300pyrG。轉化效率檢測選用綠色熒光eGFP基因(引物XbeGFPU / BaeGFPL),構建載體1300pyrGeG(圖1)。eGFP基因重組過程如下:首先將eGFP與TspyrG兩個基因分別加上啟動子與終止子,然后在農桿菌介導轉化過程中,由于左(left border)右(right border)邊界內的序列都會隨機整合進目標菌的基因組中,所以eGFP與pyrG這兩個蛋白在各自的啟動子下分別表達。所以,如果以TspyrG敲除突變株為受體菌進行轉化,成功的轉化子就會因為重新獲得pyrG蛋白而恢復表型。而由于eGFP蛋白也成功轉入,就會使轉化子產生熒光現象。
1.3.2 ATMT轉化 PDA培養獲取木霉菌分生孢子,稀釋至5×105 CFU/mL,與等體積已活化含對應載體的農桿菌混勻,取200 μL混合液涂板,具體操作方法參見Fu等[13]。
2 結果與分析
2.1 棘孢木霉Ts93pyrG基因克隆
于NCBI下載10個pyrG基因序列,通過本地BLAST,在 Ts93全基因組中搜索相似基因,篩選獲得TspyrG基因,該基因無內含子,大小 1 140 bp,編碼1個379氨基酸的蛋白(NCBI登錄號OP186039),序列比對表明(圖2),該蛋白與T.asperellum的乳清苷-5-磷酸脫羧酶(NCBI登錄號GFP54067.1)相似度達到99.74%,蛋白質含有9個高度保守motif,與該酶的功能密切 相關。
2.2 5-FOA對Ts93菌株致死濃度確定
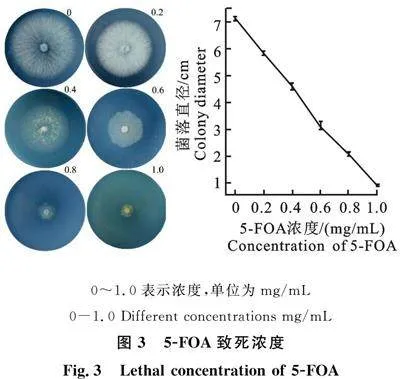
5-FOA按量添加至CA培養基,分別配制成終濃度為0、0.2、0.4、0.6、0.8、1.0、1.2 mg/mL的平板,接種直徑0.5 cm的Ts93菌餅,測定對野生株的致死濃度。結果見圖3,隨著濃度的升高,菌落直徑逐漸減小,至1.2 mg/mL時,Ts93完全無法生長,因此,為了降低突變株假陽性概率,將篩選濃度提高至1.5 mg/mL。
2.3 TspyrG基因敲除
通過ATMT方法進行基因敲除,其中TspyrG基因上游片段大小為1 080 bp,下游片段為946 bp,結果見圖4-A,構建成功的載體見圖4-B。共培養15個平板,挑選了33個可能的突變株。按照同源置換原理,TspyrG基因會被敲除載體中eYFP序列替換。因此,隨機挑選6個突變株提取DNA,以eYFPU/eYFPL引物,擴增eYFP序列。結果見圖4-C,其中1、3、5、6擴增條帶明顯。因此,挑選1號、3號兩個突變株進行單孢分離純化,挑取DNA,通過4輪PCR進一步確定是否為理想的TspyrG基因敲除突變株(圖4-D)。PCR驗證突變株的原理參考Fu等[13]。結果表明,TspyrG基因敲除突變株能夠擴增到eYFP序列,同時,由于eYFP準確替換了TspyrG基因,所以在該基因5′同源序列上游的引物YZTspyrGu結合eYFPL引物可以擴增出條帶。為了排除基因成功敲除的同時,會有額外的T-DNA隨機整合在基因組其他位置,從而影響其他基因的功能,通過設計T-DNA的側翼序列進行擴增,結果發現突變株未擴增出相應條帶。結合PCR驗證結果,確定TspyrG基因被成功敲除,同時無額外的T-DNA整合在基因組。
2.4 敲除突變株特性分析
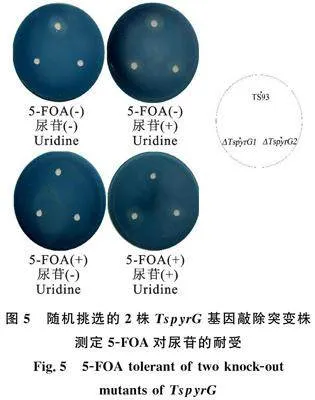
隨機挑選2個敲除突變株進行功能分析(圖5)。在不含5-FOA與尿苷的CA培養基上,Ts93生長正常,而兩突變株無法生長;添加尿苷后,Ts93與兩株突變株均能正常生長;而在添加了
5-FOA、不加尿苷的培養基上,3個菌株均無法生長;同時添加5-FOA與尿苷情況下,Ts93無法生長,而2個突變株生長正常。結果表明,敲除TspyrG基因后,菌株無法自主合成尿嘧啶,需要額外補充,但突變株同時具有抗5-FOA的能力(圖5)。
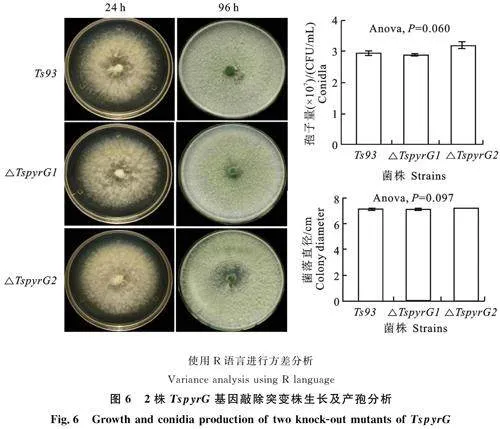
為了進一步確定該基因破壞后是否會影響菌株其他生理功能,分析突變株的生長與產孢能力(圖6)。在PDA培養基上,培養24 h后,Ts93與突變株菌落直徑均達到7.0 cm左右,三者生長速度無明顯差異(P>0.05);培養96 h后,菌株都產綠色孢子,孢子量均達到107CFU/mL,三者產孢量也無明顯差異(P>0.05)。綜合研究結果表明,TspyrG基因對菌株生長等功能無明顯影響。
2.5 尿苷營養缺陷型突變株庫的構建
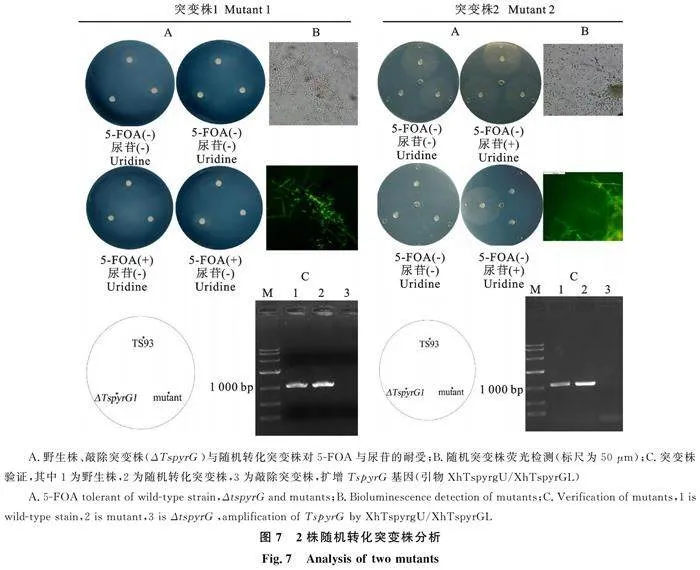
為驗證該基因作為選擇標記的可靠性,以ΔTspyrG突變株作為受體菌,以改造后的1300pyrGeG為轉化載體,通過農桿菌介導技術進行轉化。由于載體中含有TspyrG基因的完整表達框,成功轉化的菌株可以在不含尿苷的基礎
培養基上正常生長,以此作為篩選標記挑選出成功的轉化子。同時,用eGFP(enhanced green fluorescent protein,增強型綠色熒光蛋白)基因進一步檢測轉化效果。結果表明,共培養15個平板,篩選獲得30個可能的突變株,與基因敲除中30個板共獲得33個突變株無明顯差異。同時,30個突變株的生長速度有明顯差異,其中5個比野生菌Ts93快,7個比野生株慢,其他菌株無明顯差異(圖1)。隨機挑選2個轉化成功的突變株(生長速度與野生株無明顯差異)進行分析,其中突變株1在28 ℃培養12 h,突變株2培養 26 h。結果表明(圖7),在不含5-FOA與尿苷的CA培養基上,Ts93與回補株均能正常生長,但ΔTspyrG由于無法合成尿嘧啶而不能生長;在補充尿苷后,三者都能夠正常生長;而在只加5-FOA但不含尿苷的CA培養基上,三者均不能生長;當5-FOA與尿苷都補充后,ΔTspyrG可以生長,其他兩株均無法生長。eGFP基因成功轉化,菌絲與分生孢子梗有明顯熒光。同時,通過PCR對突變株進行驗證。選取引物XhTspyrgU/XhTspyrGL擴增TspyrG基因ORF(1 156 bp),其中野生株(泳道1)與回補突變株(泳道2)均可以擴增到該基因片段,但是敲除突變株無法擴增到基因片段。綜合上述結果表明,以ΔTspyrG為受體菌進行轉化后,由于TspyrG基因的回補,使ΔTspyrG回復了野生株特性。
3 討論與結論
在本研究中,建立了一種無抗生素篩選標記的木霉菌轉化系統,該系統能夠用于隨機突變株庫的構建,同時,TspyrG基因也可以作為轉基因過程的一種篩選標記,用于基因操作。該基因在酵母菌中研究很詳細,是常用的一種營養缺陷型選擇標記[14-15],在絲狀真菌中對該基因的研究主要集中在曲霉及部分植物病原菌[16-17]。木霉菌中關于該基因的研究最早是Berges等[18]在里氏木霉中(T.reesei)的突變株庫中發現兩個相關基因ura3和ura5。而本研究首次在棘孢木霉中克隆出該基因,同時,也分析了該基因作為篩選標記的可行性。基因分析表明,絲狀真菌中該基因的同源性很高,具有9個保守的Motif,蛋白質大小在300~500氨基酸。進一步研究表明,該基因破壞后,對于菌株的生長、產孢等能力無明顯影響,這也為今后用于構建隨機突變株并篩選功能基因提供保障。同時,以ΔTspyrG為受體菌構建突變株,只需要使用基礎培養基即可篩選獲得隨機突變株,大大節約了篩選成本,降低了抗生素的環境污染。對eGFP基因的轉化試驗表明,成功轉化后的突變株在性狀上回復了野生株的特性,而且該轉化系統獲得的突變株具備T-DNA的隨機整合特性,產生的突變株大部分生長無明顯影響,但也有部分菌株出現生長緩慢、產孢能力減弱現象,說明T-DNA的隨機整合造成了部分功能基因的缺失,從而影響菌株生長。木霉菌是傳統的生防菌,具備誘導植物抗性、拮抗病原菌、降解環境污染物等多種功能,而且具備基因組小,分生孢子單倍體等適合遺傳轉化特性,是很好的基因功能研究材料。而TspyrG基因作為一種遺傳選擇標記,可用于基因分子操作如雙敲除、多基因敲除,具備實際應用價值。
本研究從棘孢木霉Ts93中克隆獲得編碼乳清酸核苷-5′-磷酸脫羧酶基因TspyrG,該基因缺失后,菌株無法在基礎培養基上生長,需要額外補充尿嘧啶,但突變株能夠耐受1.0 mg/mL的5-FOA。以該基因缺失突變株為受體菌,通過回補試驗表明,回補突變株能夠恢復缺失功能,同時,轉化具備隨機整合特性,說明,該基因可以作為 Ts93基因操作的篩選標記。
參考文獻 Reference:
[1]SOOD M,KAPOOR D,KUMAR V,et al.Trichoderma:the “Secrets” of a multitalented biocontrol agent[J].Plants-Basel,2020,9(6):762-786.
[2]GUZMAN G P,PORRAS T M D,OLMEDO M V,et al.Trichoderma species:versatile plant symbionts[J].Phytopathology,2019,109(1):6-16.
[3]MUKHERJEE P K,HORWITZ B A,HERRERA E A, et al.Trichoderma research in the genome era[J].Annual Review of Phytopathology,2013,51:105-129.
[4]LEE H Y,PARK E H,KIM M D.Cloning of orotidine-5′-phosphate decarboxylase (URA3) gene from sourdough yeast Candida milleri CBS 8195[J].Food Science and Biotechnology,2012,21(5):1251-1255.
[5]PRONK J T.Auxotrophic yeast strains in fundamental and applied research[J].Applied and Environmental Microbiology,2002,68(5):2095-2100.
[6]URBELIENE N,KUTANOVAS S,MESKIENE R,et al.Application of the uridine auxotrophic host and synthetic nucleosides for a rapid selection of hydrolases from metagenomic libraries[J].Microbial Biotechnology,2019, 12(1):148-160.
[7]NOH M H,LIM H G,MOON D,et al.Auxotrophic selection strategy for improved production of coenzyme B-12 in Escherichia coli[J].Iscience,2020,23(3):1-8.
[8]IWASAKI T,MIYAJIMA N Y,FUKAZAWA R,et al.Escherichia coli amino acid auxotrophic expression host strains for investigating protein structure-function relationships[J].Journal of Biochemistry,2021,169(4):387-394.
[9]CAKAR Z P,SAUER U,BAILEY J E.Metabolic engineering of yeast:the perils of auxotrophic hosts[J].Biotechnology Letters,1999,21(7):611-616.
[10] VAN B I N A,DEM S L,DEVELTER D,et al.Development of a transformation and selection system for the glycolipid-producing yeast Candida bombicola[J].Yeast,2008,25(4):273-278.
[11]GROOT M J A,BUNDOCK P,HOOYKAAS P J J,et al.Agrobacterium tumefaciens-mediated transformation of filamentous fungi[J].Nature Biotechnology,1998, 16(9):839-842.
[12]HOOYKAAS P J J,VAN HEUSDEN G P H,NIU X L,et al.Agrobacterium-mediated transformation of yeast and fungi[J].Agrobacterium Biology:From Basic Science to Biotechnology,2018,418:349-374.
[13]FU K H,FAN L L,LI Y Y,et al. Tmac1,a transcription factor which regulated high affinity copper transport in Trichoderma reesei[J].Microbiological Research,2012,167(9):536-543.
[14]PARK E H,SEO J H,KIM M D.Cloning and characterization of the orotidine-5′-phosphate decarboxylase gene (URA3) from the osmotolerant yeast Candida magnoliae[J].Journal of Microbiology and Biotechnology,2012,22(5):642-648.
[15]TSANG P W K,WONG K S,CHU J K M.Isolation and characterization of Candida kefyr orotidine-5 ′-phosphate decarboxylase (URA3) gene[J].Yeast,2010,27(1):53-58.
[16]SUN X Y,ZHU J F,BAO L,et al.pyrG is required for maintaining stable cellular uracil level and normal sporulation pattern under excess uracil stress in Aspergillus nidulans[J].Science China-Life Sciences,2013,56(5):467-475.
[17]YING S H,FENG M G,KEYHANI N O.Use of uridine auxotrophy (ura3) for markerless transformation of the mycoinsecticide Beauveria bassiana[J].Applied Microbiology and Biotechnology,2013,97(7):3017-3025.
[18]BERGES T,BARREAU C.isolation of uridine auxotrophs from Trichoderma reesei and efficient transformation with the cloned ura3 and ura5 genes[J].Current Genetics,1991,19(5):359-365.
Establishment of Transformant System for Trichoderma asperellum Using TspyrG as Selected Marker
FAN Lili,WEI Runling,FU Kehe,LIU Wentao and SHI Jing
(Department of Biology,Nanchang Normal University,Nanchang 330032,China)
Abstract In this study,weTcqgh3FJQioCvUKLSL+GYg== cloned a TspyrG gene,which encodes orotidine-5′-phosphate decarboxylase,an essential enzyme in pyrimidine biosynthesis in T.asperellum Ts93 strain.The TspyrG gene,which does not contain introns,comprises an open reading frame of 1 140 codons and encode a 379 aa protein.The ΔtspyrG mutants were obtained via Agrobacterium-mediated transformation,which were unable to grow without the uracil.And the transformant plasmid 1300pyrG was constructed successfully based on the plasmid PCAMBIA1300.Agrobacterium transformation system with uracil auxotroph maker was successfully constructed using ΔTspyrG as recipient strain.Transformant of eGFP showed no significant difference between auxotrophic selection marker and antibiotic selection marker in the transformant system.
Key words Trichoderma asperellum; Auxotroph; Agrobacterium-mediated transformation
Received 2023-03-06 Returned 2023-04-06
Foundation item Project of Jiangxi Provincial Department of Education (No.151252,No.GJJ161233);Regional Project of National Natural Science Foundation of China(No.31660020).
First author FAN Lili,female,Ph.D,lecturer.Research area:molecular genetics of Trichoderma. E-mail:llfan31@163.com
Corresponding author FU Kehe,male,Ph.D,lecturer.Research area:microbial soil remediation. E-mail:khfu0112@163.com
(責任編輯:郭柏壽 Responsible editor:GUO Baishou)
基金項目:江西省教育廳項目(151252,GJJ161233);國家自然科學基金地區項目(31660020)。
第一作者:范莉莉,女,博士,講師,主要從事木霉菌分子遺傳研究。E-mail:llfan31@163.com
通信作者:傅科鶴,男,博士,講師,主要從事微生物土壤修復研究。E-mail:khfu0112@163.com

