癌癥標志本體構建與應用
吳萌 康宏宇 徐曉巍 侯麗 李姣

摘要:目的 構建一種可溯源的癌癥標志知識本體,支持基因突變、癌癥標志和細胞系等相關數據整合、標準化和關聯,并基于該本體實現新知識發現。方法 基于“七步法”和現階段本體的構建方法,明確癌癥標志本體覆蓋范圍、框架結構、可復用的術語和新增術語;以結直腸癌為例,利用文本挖掘和文本分類技術對PubMed文獻中結直腸癌標志相關知識進行提取,利用癌癥標志本體對提取的知識進行規范化表達。結合癌癥體細胞突變目錄中已有的癌癥標志證據和進一步語義檢索,實現新知識的發現。結果 最終構建癌癥標志本體包含類9910個,實例6138個,實現2310篇涉及結直腸癌標志文獻摘要和26個結直腸癌標志相關證據的語義表示。與癌癥體細胞突變目錄數據集相比,本研究發現與結直腸癌相關的多個基因的癌癥標志的新證據。結論 本研究有助于整合癌癥分子層面的致病機制,了解基因突變在癌癥發生過程中的具體作用,實現癌癥標志知識的快速發現。
關鍵詞:結直腸癌;本體;癌癥標志
中圖分類號: R735.3;Q756文獻標志碼: A文章編號:1000-503X(2023)04-0618-09
DOI:10.3881/j.issn.1000-503X.15386
Development and Application of Ontology of Cancer Hallmarks
WU Meng,KANG Hongyu,XU Xiaowei,HOU Li,LI Jiao
Center of Medical Information Innovation Research,Institute of Medical Information/Library,
CAMS and PUMC,Beijing 100020,China
Corresponding author:HOU Li Tel:010-52328750,E-mail:hou.li@imicams.ac.cn
ABSTRACT:Objective To develop a traceable cancer hallmark ontology with terminology including gene mutation,cancer hallmark,and cell line for knowledge integration,standardization,correlation,and discovery.Methods The Ontology Development 101 and the current ontology development methods were employed to determine the content coverage,structural layers,reusable terms,and new terms of the cancer hallmark ontology.Taking colorectal cancer as a study case,we extracted the knowledge related with colorectal cancer hallmarks using text mining and text classification technology from PubMed,and then formalized the extracted knowledge into the cancer hallmark ontology.Moreover,we made use of existing cancer hallmark evidence in Catalogue of Somatic Mutations in Cancer and further semantic retrieval to discover new knowledge.Results The established cancer hallmark ontology comprised 9910 classes and 6138 instances,which realized the semantic representation of 2310 article abstracts about colorectal cancer and 26 pieces of evidence about genes and their cancer hallmarks.Compared with the Catalogue of Somatic Mutations in Cancer,new evidence for more genes associated with colorectal cancer hallmarks was found based on cancer hallmark ontology.Conclusion This study is of great significance to the research on the cancer pathogenesis at the molecular level,the revealing of specific roles of genes and mutations in the occurrence of cancer,and the rapid knowledge discovery of cancer hallmarks.
Key words:colorectal cancer;ontology;hallmarks of cancer
Acta Acad Med Sin,2023,45(4):618-626
隨著癌癥靶向治療技術的發展和精準醫學概念的提出,越來越多的研究關注于癌癥發生的內部分子機制,以及其對細胞表型特征變化的影響。其中,癌癥標志(hallmarks of cancer,HOC)[1]的提出為癌癥發生過程中細胞特征的變化提供了重要標識、分類體系和相關詞匯[2-3]。從基本的原理層面捕捉癌癥發生的復雜內因,為人們理解和探索癌癥的表型與機制提供了清晰易懂的邏輯框架[4]。2011年版的癌癥標志包含10大類,分別是持續的增殖信號、逃避生長抑制、抵抗細胞死亡、無限復制、血管新生、侵襲轉移、基因組不穩定和突變、能量代謝異常、免疫逃逸、促瘤炎癥[5]。有相關研究已將癌癥標志作為癌癥研究的重要部分。例如癌癥體細胞突變目錄(Catalogue of Somatic Mutations in Cancer,COSMIC)推出的癌癥基因普查項目[6]利用人工的方式從文獻中進行HOC相關知識的審編,對癌癥相關基因的功能進行描述。LnCeCell[7]對競爭性內源RNA相關的HOC進行標識。Baker等[8]構建了HOC語料庫,將HOC的10大標志擴展為37類,借助文本分類技術對1852篇PubMed文獻摘要的句子進行分類。并在后續研究中,提供了HOC自動分類工具CHAT[2],以及基于文獻的HOC瀏覽工具LION LBD[9],通過實體識別工具PubTator[10]識別基因和疾病等生物醫學實體,展示HOC與基因和疾病的共現關系。此外,隨著預訓練模型在自然語言處理領域的發展,一些預訓練模型在HOC語料庫已達到較好的分類效果,如BioLinkBERT[11]、NCBI_BERT[12]、PubMedBERT[13]等。這些方法為HOC的自動審編提供幫助。現階段還缺乏一種系統的、結構化的、機器可理解的知識表示框架對HOC知識進行梳理,對HOC與其他生物醫學實體之間具體關聯進行描述。在生物醫學領域,本體已經成為多源數據融合、語義表示、標準化和鏈接的重要技術,廣泛用于表示領域數據之間的語義關聯[14],為文本挖掘和知識推理提供助力。因此,本研究以結直腸癌為例,提出一種可溯源的HOC本體語義表示框架,實現基因突變、HOC和細胞系等相關數據整合、標準化和關聯,并提供相關文獻來源,將HOC本體與文本挖掘技術結合,提供更全面更詳細的HOC知識,實現進一步的數據發現。
資料和方法
本體構建 基于“七步法”[15]和現階段本體的構建方法[16-17],設計HOC本體構建方法和知識發現實驗流程(圖1)。
確定本體范圍:本體是一個特定領域的模型,為特殊的目的而構建。HOC本體構建的目的是為基因突變在癌癥發生過程中的具體作用提供統一的結構化表示和整合方案。在HOC相關的科學文獻表述中,研究人員針對癌癥病患的組織樣本或相關細胞系做全面的組學數據分析和癌細胞特征檢測。探明例如在哪種細胞系中,發生了哪些突變從而引起了哪些HOC的變化。其后,通過專家咨詢的方式明確HOC本體需要回答的勝任力問題[18]:(1)在某種癌癥中,哪些基因參與了某種癌癥特征的發生?(2)在某個癌癥中,某個基因參與了哪些癌癥特征的發生?(3)哪些文獻可能表明,某個突變參與了某個癌癥特征的發生?(4)哪些證據表明,某個基因與某種癌癥的發生有關?(5)哪些證據表明,某個細胞系中發生了某個癌癥特征的改變?(6)在某個癌細胞系中,哪些基因可能參與了癌癥特征的發生?
依據HOC本體的構建目標、應用場景及勝任力問題,本研究最終確定HOC本體的范圍涵蓋癌癥、細胞系、基因、突變、HOC、文獻、證據等多種類型生物醫學實體,并定義這些實體之間的語義關系(圖2)。利用中間實體類型證據和引文,對多種生物醫學實體進行鏈接。引文與HOC和各種生物醫學實體之間的關系為共現關系。證據表示已確認的證據信息,其與HOC和各種生物醫學實體之間的關系為確定的相關關系。此外,一些實體類型可以復用已有的本體,在圖2中用曲線表示出子類的關系。
復用本體類和公理:隨著語義網技術的發展,在生物醫學領域,已經存在一些被研究人員充分認可的本體,復用或引用已有的本體是生物醫學領域本體構建的常用方法。在HOC本體的構建過程中,選擇OntoFox[19]工具復用相關本體的術語、屬性和關系。OntoFox是密西根大學何勇群團隊研發的一款在線的本體術語提取工具,可以便捷地對開放生物醫學本體倉儲中的本體進行獲取和復用。
Chen等[3]發現基因本體(gene ontology,GO)[20]、京都基因和基因組百科全書[21]和分子特征數據庫[22]等數據庫和本體對HOC術語進行了標準化描述。參考Chen等[3]的研究,HOC相關術語主要復用自GO,盡可能選擇明確表示正負調控的GO術語與HOC建立映射(表1)。基因突變術語復用自序列本體(sequence ontology,SO)中的gene(SO_0000704)和sequence_alteration(SO_0001059)。疾病術語復用自疾病本體(disease ontology,DO)中的cancer(DOID_162)。細胞系術語復用自細胞系本體(cell line ontology,CLO)的immortal cell line cell(CLO_0000019)。同時復用其上下位術語、相關術語、術語之間的語義關系(對象屬性)和術語屬性(注釋屬性)(表2)。
本體對齊:為保障復用不同本體時術語的一致性,本研究選取ROBOT(V1.8.3)[23]工具將這4個本體中抽取的術語進行融合與對齊。ROBOT的Merge工具可以對多個本體進行融合,并對資源標識符相同的術語進行對齊,包括屬性和關系。完成融合后,利用ROBOT的Report工具對融合結果進行進一步檢驗,主要包括:(1)術語缺少名稱;(2)1個術語有多個不同的名稱、多個不同的定義;(3)不同的類有相同的名稱、相同的定義等不一致錯誤;(4)關系的名稱相同但含義和用法不同。其后,借助protégé工具和人工方式,對檢驗發現的不一致問題進行修正。人工方式通過兩名相關領域專家進行輪流修正校對,保證本體對齊的準確性和正確性。
構建關系和屬性:在本體復用和融合的基礎上,新構建癌癥標志和證據等類,包含10個癌癥標志子類,對其屬性和相關關系進行豐富,新增語義關系相關癌癥標志、有引文等12個,標題、年份等屬性5個,以上文中確定本體范圍部分的語義模型設計為基礎,利用protégé本體編輯軟件構建新的類和語義關系。
填充實例:結直腸癌作為目前世界上新發癌癥發病率第3、死亡率第2的惡性腫瘤,其防治形勢十分嚴峻[24]。本研究以結直腸癌為實例,對構建的HOC本體進行填充和驗證。首先,在生物醫學文獻網站PubMed上搜索結直腸癌和細胞相關的文獻,檢索語句為“(colorectal cancer[Title/Abstract]) AND (cell[Title/Abstract])”,檢索到1975年1月1日至2022年9月2日發表的相關文獻共29 986篇。導出文獻的PMID,利用PubTator[10]工具的API,獲取每篇文獻的實體識別結果,包括基因、疾病、化學藥品、突變、物種和細胞系。
利用HOC語料庫[8]和BioLinkBERT[11]模型,對每篇PubMed文獻的摘要進行HOC分類。BioLinkBERT模型是目前已知在HOC數據集上分類效果最佳的預訓練模型,其base和large規模在HOC數據集的Micro F1分別達到84.35%和84.87%。本研究利用其在GitHub網站上共享的預訓練模型和微調代碼[25],在HOC數據集上對base規模的BioLinkBERT模型進行微調,遵循與其原文相同的微調方法和評測指標[11],最終Micro F1達到84.31%。利用微調后的模型對29 986篇結直腸癌相關的摘要進行分類預測。通過模型預測,分類結果中包含10大類HOC的摘要共23 081篇。根據本研究需求,利用PubTator識別結果篩選出包含基因和細胞系,但不包含化學藥品的摘要共2355篇,將其作為引文的實例填充進HOC本體中,建立引文與HOC和各種生物醫學實體的共現關系。此外,利用COSMIC Cancer Gene Census[6]中與結直腸癌相關的數據對證據的實例進行構建。其提供了多種癌癥相關的基因和HOC信息,其中與結直腸癌相關的基因66個,有HOC信息的基因12個,相關文獻25篇,證據26個(其中1篇文獻有2個證據)。
基于HOC本體的知識發現 基于已構建的HOC本體,通過SPARQL多跳查詢,利用已知的HOC證據可以實現新證據和新基因的發現(圖3)。圖3上半部分表示利用COSMIC中已有結直腸癌HOC的基因,發現其新的HOC;下半部分表示利用COSMIC中已有結直腸癌相關的基因和HOC,發現與其共同出現在文獻標題中的新基因。由于實例數據量較大,選擇Python的owlready2包,實現實例的填充和SPARQL查詢。
新證據發現:COSMIC對癌癥相關的基因和HOC進行了人工審編,具有較高的準確性。以AKT1為例,在COSMIC中,其HOC包括抵抗細胞死亡(來源文獻PMID:28339062)及侵襲轉移(來源文獻PMID:24297510)。在HOC本體中,對文獻標題包含AKT1的文獻和HOC進行SPARQL查詢,并對只包含AKT1的文獻進行篩選,其中AKT1在HOC本體中ID為gene_8(圖4)。
新基因發現:在結直腸癌中,許多基因與經典的通路和基因相互調控,從而引發HOC的發生,因此,選擇在文獻標題中出現次數最多的幾個基因,并找到在文獻標題中與這些基因共同出現的其他基因,發現新的癌癥靶點及相應證據,對基因在文獻標題中的出現情況進行SPARQL查詢(圖5),對結果進行統計(圖6)。
發現與結直腸癌相關并在文獻標題中出現的基因共1435個,其中,CTNNB1、AKT1、CD8A為在文獻標題中出現數量最多的3個基因。結合COSMIC中這些基因的結直腸癌標志審編結果數量,最終選擇CTNNB1(審編HOC證據為2個)和KRAS(審編HOC證據為4個)2個基因,利用SPARQL查詢找到與其共同出現在標題中的其他基因,其中CTNNB1在HOC本體的ID為gene_0(圖7)。根據COSMIC數據集已知CTNNB1在結直腸癌中的HOC為基因組不穩定和突變、免疫逃逸。KRAS在結直腸癌中的HOC為無限復制、侵襲轉移和能量代謝異常。
結果
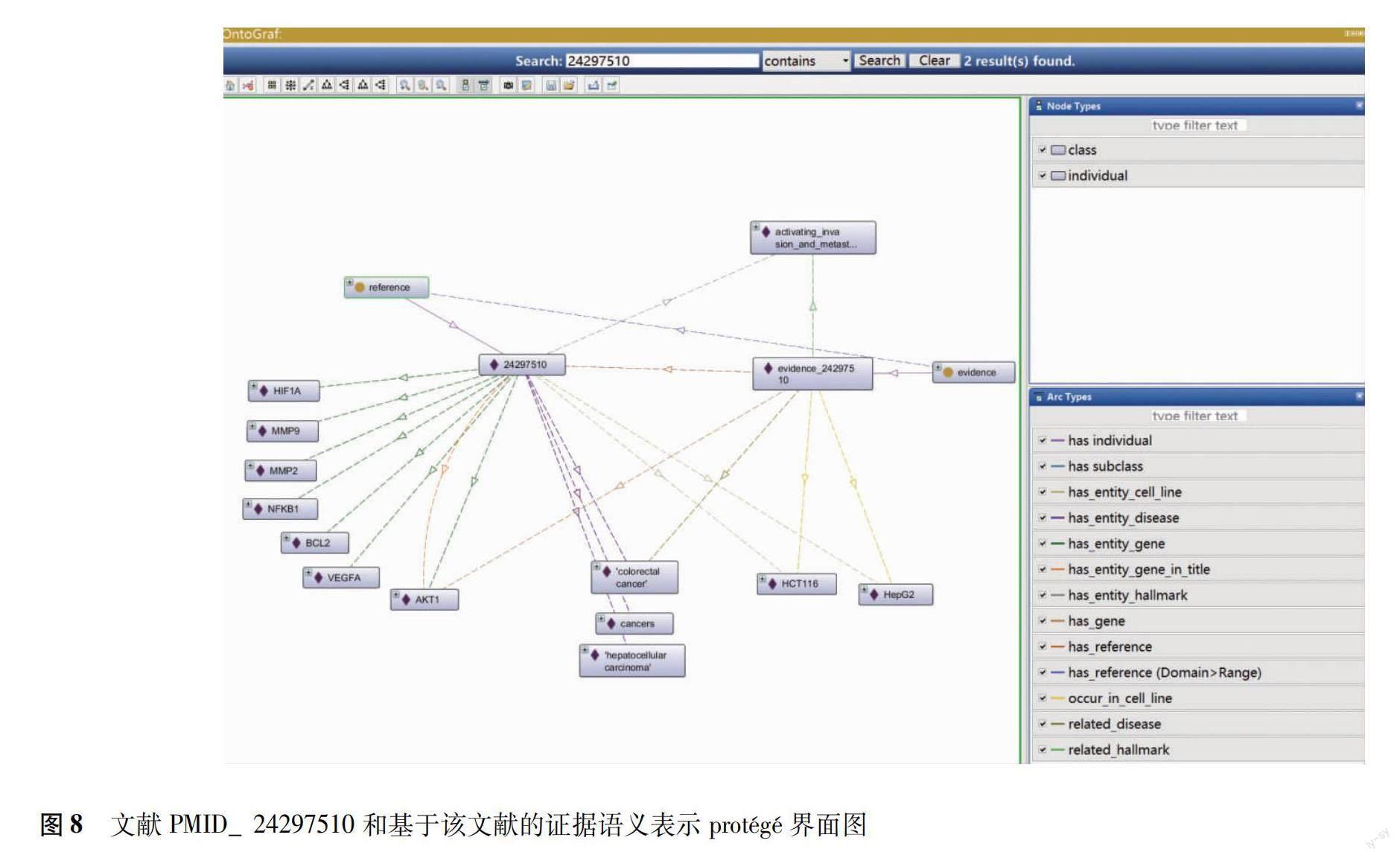
本體構建結果 最終構建HOC本體包含類9910個,實例6138個,對象屬性45個,注釋屬性36個。其中引文的實例為2310個,證據的實例為26個,序列變異的實例為116個,基因的實例為2913個,疾病的實例為380個,細胞系的實例為345個。例如,利用protégé軟件對文獻PMID24297510和基于該文獻的證據進行語義表示(圖8)。圖8左半部分表示引文PMID24297510,以及在該引文中出現的HIF1A等基因、出現在標題的AKT1基因、HCT116等細胞系、結直腸癌等疾病和癌癥標志侵襲轉移;右半部分表示來源于該引文的證據,即在COSMIC中確定的基因、HOC、疾病和細胞系等之間的關聯。
知識發現
新證據發現:通過SPARQL檢索后,HOC本體中文獻標題包含AKT1的文獻和HOC,為持續的增殖信號(PMID:18570457、31164794、35860583)、侵襲轉移(PMID:24297510、18570457、31164794)。其中,在文獻PMID24297510中AKT1基因的HOC為侵襲轉移,與COSMIC中數據相符。通過人工審核后,新發現的證據包括:在文獻PMID18570457、PMID31164794中AKT1的HOC為侵襲轉移。而持續的增殖信號相關的文獻經過審核后,未發現AKT1與其描述相關。
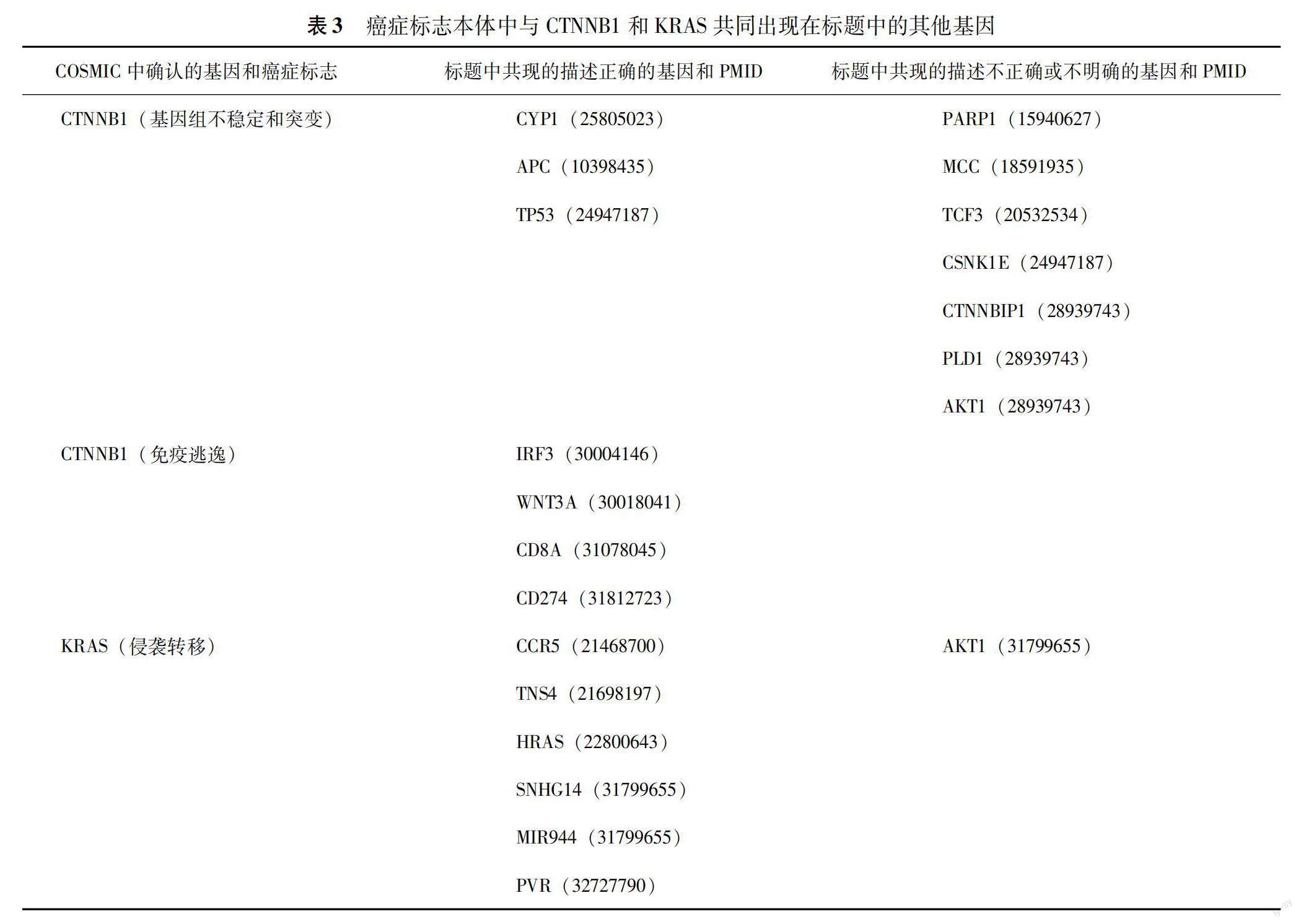
新基因發現:利用SPARQL查詢找到與CTNNB1和KRAS共同出現在標題中的其他基因,并對結果進行人工審核。最終發現13個基因明確與相應的HOC相關,可以作為結直腸癌標志的新證據,8個基因在相關文獻中未明確表明與相關HOC有關(表3)。此外,通過審核PVR(PMID:32727790),發現KRAS基因新的HOC及證據為免疫逃逸(PMID:32727790)。而基于KRAS的另外兩個HOC:無限復制和能量代謝異常,未發現新的相關基因。
討論
本研究提出一種可溯源的HOC本體語義表示框架,有助于整合與分析癌癥分子層面的致病機制,了解基因突變在癌癥發生過程中的具體作用,助力癌癥靶向治療的研究。本體評估主要有基于金標準的評估、基于應用的評估、數據驅動的評估以及人工評估4種方式[26]。HOC本體的構建過程充分借鑒“七步法”的方法體系,通過復用盡可能滿足專業性和行業內認可的標準,并通過一致性檢驗及專家進一步審核,保證本體的一致性、準確性和正確性。并在后續知識表達和知識發現的具體應用中,驗證HOC本體的應用能力。在HOC術語構建過程中,通過明確HOC術語與GO術語的映射關系對HOC術語進行擴展,有助于進一步理解HOC的內涵。此外,HOC本體為人和機器都提供了較強的可解釋性,利用中間節點引文和證據有效表達了摘要和句子中多種實體共現的情況。與已有HOC相關工作相比,HOC本體的構建過程更節省人力,利用生物醫學實體識別工具和文本分類技術實現癌癥標志知識的自動提取,并通過HOC本體為文獻中的HOC知識提供結構化的語義表示,其可溯源的框架設計為HOC證據提供了相應的文獻來源。基于已構建的HOC本體,本研究提出了一種利用SPARQL檢索和已有HOC證據快速發現新HOC證據的方法,為臨床和科研工作提供便捷的服務。對知識發現結果進行分析顯示,通過初步篩選的幾個結直腸癌基因進行檢索,與COSMIC數據集人工審編結果相比,發現新的HOC相關文獻和基因,證實HOC本體在知識發現方面的有效性。基因和基因之間存在相互調節關系,基因可能不會直接作用于改變HOC。因此,選擇與已知HOC相關的基因在標題中共現的其他基因,有助于發現新的基因與HOC。
本研究目前只采用了機器處理的方式,未對處理結果進行大批量人工審核,應考慮PubTator實體識別工具和BioLinkBERT分類模型存在的誤差。后續將研究人工和機器結合的方式對HOC證據進行進一步審核和擴充,借鑒證據本體對證據相關的術語進行標準化,提供更高質量的證據服務。此外,充分融合生物醫學知識庫中已有的關聯關系如基因-突變、基因-疾病、疾病-細胞系等,為HOC知識發現提供更多的知識來源,也是本研究后續探索的研究方向。在證據來源方面,本研究只納入PubMed文獻中的HOC信息,未納入其他HOC信息來源如臨床試驗等。
參 考 文 獻
[1]Hanahan D,Weinberg RA.The hallmarks of cancer[J].Cell,2000,100(1):57-70.DOI:10.1016/S0092-8674(00)81683-9.
[2]Baker S,Ali I,Silins I,et al.Cancer Hallmarks Analytics Tool (CHAT):a text mining approach to organize and evaluate scientific literature on cancer[J].Bioinformatics,2017,33(24):3973-3981.DOI:10.1093/bioinformatics/btx454.
[3]Chen Y,Verbeek FJ,Wolstencroft K.Establishing a consensus for the hallmarks of cancer based on gene ontology and pathway annotations[J].BMC Bioinformatics,2021,22(1):178.DOI:10.1186/s12859-021-04105-8.
[4]陶青,石毓君.解讀癌癥研究的里程碑:Hallmarks of Cancer[J].中國普外基礎與臨床雜志,2022,29(4):431-433.DOI:10.7507/1007-9424.202202033.
[5]Hanahan D,Weinberg RA.Hallmarks of cancer:the next generation[J].Cell,2011,144(5):646-674.DOI:10.1016/j.cell.2011.02.013.
[6]Sondka Z,Bamford S,Cole CG,et al.The COSMIC cancer gene census:describing genetic dysfunction across all human cancers[J].Nat Rev Cancer,2018,18(11):696-705.DOI:10.1038/s41568-018-0060-1.
[7]Wang P,Guo Q,Hao Y,et al.LnCeCell:a comprehensive database of predicted lncRNA-associated ceRNA networks at single-cell resolution[J].Nucleic Acids Res,2021,49(D1):D125-D133.DOI:10.1093/nar/gkaa1017.
[8]Baker S,Silins I,Guo Y,et al.Automatic semantic classification of scientific literature according to the hallmarks of cancer[J].Bioinformatics,2016,32(3):432-440.DOI:10.1093/bioinformatics/btv585.
[9]Pyysalo S,Baker S,Ali I,et al.LION LBD:a literature-based discovery system for cancer biology[J].Bioinformatics,2019,35(9):1553-1561.DOI:10.1093/bioinformatics/bty845.
[10]Wei CH,Allot A,Leaman R,et al.PubTator central:automated concept annotation for biomedical full text articles[J].Nucleic Acids Res,2019,47(W1):W587-W593.DOI:10.1093/nar/gkz389.
[11]Michihiro Y,Jure L,Percy L.LinkBERT pretraining language models with document links[EB/OL].[2022-11-11].https://arxiv.org/abs/2203.15827.
[12]Yifan P,Shankai Y,Zhiyong L.Transfer learning in biomedical natural language processing:an evaluation of BERT and ELMo on ten benchmarking datasets[EB/OL].[2022-11-11].https://arxiv.org/abs/1906.05474.
[13]Yu G,Robert T,Hao C,et al.Domain-specific language model pretraining for biomedical natural language processing[EB/OL].[2022-11-11].https://arxiv.org/abs/2007.15779.
[14]Haendel MA,Chute CG,Robinson PN.Classification,ontology,and precision medicine[J].N Engl J Med,2018,379(15):1452-1462.DOI:10.1056/NEJMra1615014.
[15]Natalya FN,Deborah LM.Ontology development 101:a guide to creating your first ontology[EB/OL].[2022-11-11].http://www.ksl.stanford.edu/people/dlm/papers/ontology-tutorial-noy-mcguinness-abstract.html.
[16]Lee JH,Park HA,Song TM.A determinants-of-fertility ontology for detecting future signals of fertility issues from social media data:development of an ontology[J].J Med Internet Res,2021,23(6):e25028.DOI:10.2196/25028.
[17]Reyes-Pe?a C,Tovar M,Bravo M,et al.An ontology network for diabetes mellitus in Mexico[J].J Biomed Semantics,2021,12(1):19.DOI:10.1186/s13326-021-00252-2.
[18]Bravo M,Hoyos Reyes LF,Reyes-Ortiz JA.Methodology for ontology design and construction[J].Contaduría y Administración,2019,64(4):134.DOI:10.22201/fca.24488410e.2020.2368.
[19]Xiang Z,Courtot M,Brinkman RR,et al.OntoFox:web-based support for ontology reuse[J].BMC Res Notes,2010,3:175.DOI:10.1186/1756-0500-3-175.
[20]The Gene Ontology Consortium.The gene ontology resource:20 years and still GOing strong[J].Nucleic Acids Res,2019,47(D1):D330-D338.DOI:10.1093/nar/gky1055.
[21]Kanehisa M,Furumichi M,Sato Y,et al.KEGG:integrating viruses and cellular organisms[J].Nucleic Acids Res,2021,49(D1):D545-D551.DOI:10.1093/nar/gkaa970.
[22]Liberzon A,Birger C,Thorvaldsdóttir H,et al.The Molecular Signatures Database (MSigDB) hallmark gene set collection[J].Cell Syst,2015,1(6):417-425.DOI:10.1016/j.cels.2015.12.004.
[23]Jackson RC,Balhoff JP,Douglass E,et al.ROBOT:a tool for automating ontology workflows[J].BMC Bioinformatics,2019,20(1):407.DOI:10.1186/s12859-019-3002-3.
[24]Sung H,Ferlay J,Siegel RL,et al.Global cancer statistics 2020:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA Cancer J Clin,2021,71(3):209-249.DOI:10.3322/caac.21660.
[25]Michihiro Y,Jure L,Percy L.LinkBERT:a knowledgeable language model pretrained with document links[EB/OL].[2022-11-11].https://github.com/michiyasunaga/LinkBERT.
[26]Brank J,Grobelnik M,Mladenic′ D.A survey of ontology evaluation techniques[EB/OL].[2022-11-11].https://www.researchgate.net/publication/228857266_A_survey_of_ontology_evaluation_techniques.
(收稿日期:2022-11-17)

