豚鹿SSR標記開發與遺傳多樣性分析
耿廣耀 由玉巖 劉群秀
(1.上海動物園,上海,200335;2.北京動物園,圈養野生動物技術北京市重點實驗室,北京,100044)
微衛星DNA(microsatellite DNA)又稱簡單重復序列(simple sequence repeat,SSR),廣泛分布在真核、原核生物和病毒基因組中,是基因組中變化最快、多態性較高和兩端較為保守的一段序列[1],具有易檢測、分型重復性好和統計方法多樣等優點,可做為一種有效的遺傳標記,廣泛應用于種群遺傳多樣性研究、物種保護、物種鑒定和親權分析等方面[2-5]。不同物種的微衛星標記可在近緣物種中相互借用[6],但不同物種有時會表現出特異性,進而出現引物通用性較差,不能確定擴增產物是否為期望序列,因此在應用微衛星進行遺傳多樣性研究時需要針對特定物種進行標記開發[7-8]。
豚鹿(Axisporcinus)隸屬鹿科(Cervidae)花鹿屬(Axis),是國家一級重點保護野生動物,分布在南亞次大陸至東南亞的中南半島區域,我國境內野生種群在20世紀60年代已滅絕,圈養種群僅分布在成都動物園、上海動物園和北京動物園。目前關于豚鹿分子標記開發的研究有限,余建秋等[9]利用磁珠富集法獲取7個微衛星位點對成都動物園27只豚鹿個體進行親權分析;Wang等[10]利用RAD-seq對成都動物園圈養豚鹿進行遺傳多樣性研究。本研究基于Illumina MiSeq測序平臺對上海動物園圈養豚鹿進行微衛星標記開發,篩選出7個SSR標記對該種群進行遺傳多樣性分析,探究這些標記在圈養豚鹿種群中的適用性,為豚鹿種群保護研究提供科學依據。
1 材料與方法
1.1 樣品及總DNA提取
1.2 SSR分子標記開發
將提取的DNA通過超聲隨機打斷,制備測序文庫。利用p(AG)10、p(AC)10、p(AAC)8、p(ACG)8、p(AAG)8、p(AGG)8、p(ACAT)6和p(ATCT)68種探針富集基因組文庫中的SSR片段。利用Illumina MiSeq平臺測序富集得到的SSR片段,并利用Primer 3 V2.3.6對Cluster內簡單序列長度多態性(simple sequence length polymorphism,SSLP)大于2的SSR序列進行引物設計。
1.3 引物驗證與分型
選取80個SSR引物熒光標記后,對10個隨機樣品進行擴增和毛細管電泳檢測,篩選出擴增效率高和穩定性好的多態性SSR引物。利用篩選出的SSR引物對22只豚鹿個體進行PCR擴增和毛細管電泳檢測。PCR反應體系為20.0 μL:DNA模板1.0 μL(濃度為15 ng/μL),Taq酶0.5 μL,10 μmol/L的上、下游引物各1.0 μL,超純水14.0 μL,10×buffer 2.0 μL,dNTP 0.5 μL。反應程序:95 ℃預變性5 min;95 ℃變性30 s,52~62 ℃退火30 s,72 ℃延伸30 s,10個循環;95 ℃變性30 s,52 ℃退火30 s,72 ℃延伸30 s,25個循環;72 ℃延伸7 min。
1.4 數據分析
利用Cervus 3.0和PopGene 3.2[11]分別計算各位點等位基因數(number of alleles,Na)、有效等位基因數(effective number of alleles,Ne)、多態信息含量(polymorphism information content,PIC)、觀測雜合度(observed heterozygosity,Ho)、期望雜合度(excepted heterozygosity,He)和哈迪-溫伯格平衡(Hardy-Weinberg equilibrium,H-W)等。
2 結果與分析
2.1 豚鹿基因組中SSR位點信息
利用微衛星識別工具(MIcroSAtellite identification tool,MISA)采用搜索參數(mono-10、di-6、tri-5、Tetra-5、penta-5和hexa-5,復合序列中兩個不同SSR之間允許的最大間隔為100 bp)在7 639 467條序列(總長度2 571 197 198 bp)中,檢測到SSR片段序列5 684 557條,含1個以上SSR片段的序列為1 396 521條,以復合形式存在的SSR序列為2 412 186條。單堿基重復到六堿基重復在豚鹿基因組中均有分布,其中雙堿基重復占比最大,其次是單堿基重復,各堿基重復信息見表1。
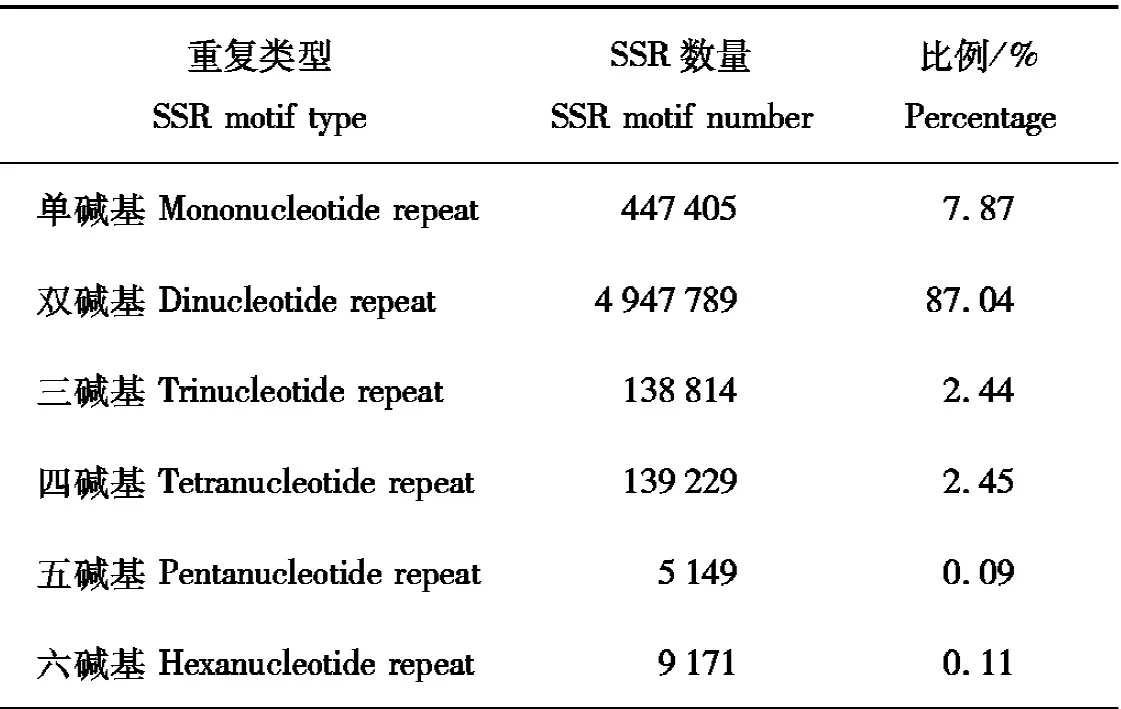
表1 不同重復類型SSR統計
單堿基重復SSR位點以A/T重復為主,占比為96.27%。雙堿基重復中,AC/GT重復數為3 944 705,占比最大,為79.73%;AG/CT重復數為844 429,占比為17.07%;AT/AT重復數為156 843,占比為3.16%;CG/CG重復數為1 812,占比為0.04%。三堿基重復中主要重復為AAC/GTT、AAG/CTT和AGG/CCT。四堿基重復中主要重復為ACTC/GAGT。五堿基重復中主要重復為AACAC/GTGTT。六堿基重復中主要重復為ACACCC/GGGTGT。
2.2 多態性SSR引物篩選與遺傳多樣性分析
試驗篩選出7個擴增效率高和穩定性好的多態性SSR引物,引物序列信息見表2。7個SSR標記在22只圈養豚鹿個體中共檢測到19個等位基因,單個標記的等位基因數為2~5個,平均值為2.714個,觀測雜合度為0.227~1.000,平均值為0.680,期望雜合度為0.206~0.672,平均值為0.513。多態性信息含量為0.181~0.590,平均值為0.421。哈迪-溫伯格平衡檢驗經Bonferroni校正后,有2個位點不符合哈迪-溫伯格平衡(表3)。

表2 引物序列信息
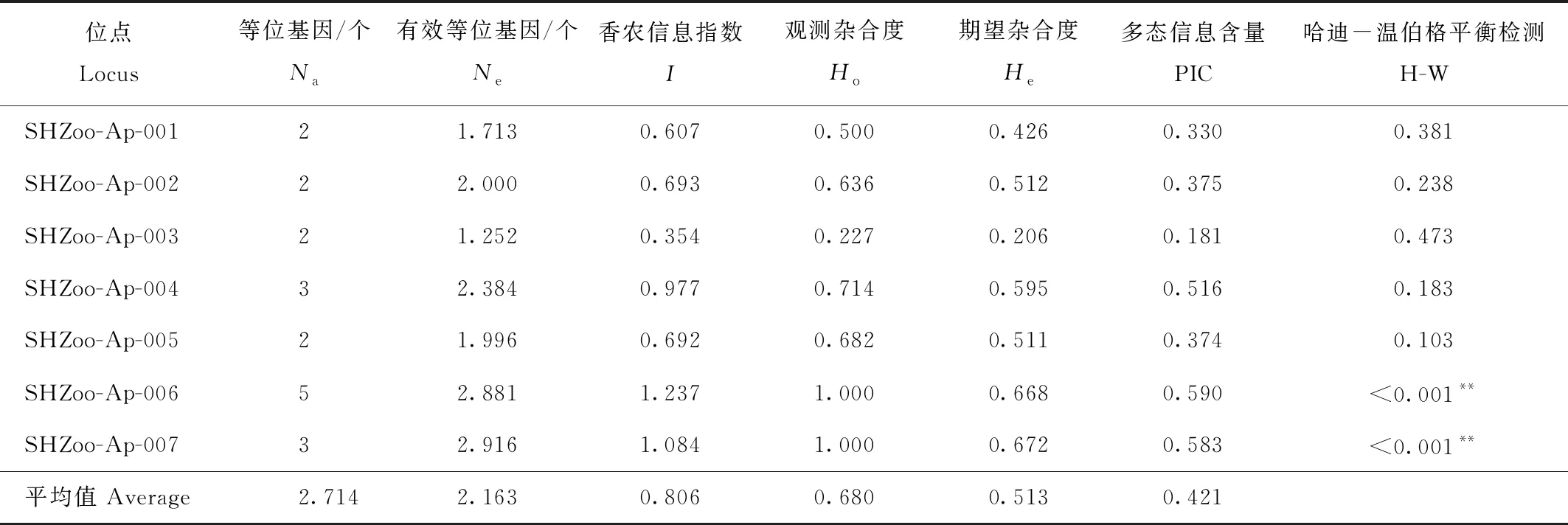
表3 豚鹿7個多態性基因位點分析
3 討論
通過近緣物種間引物借鑒、公共數據庫和相關文獻[12]查找,小片段DNA克隆法和傳統微衛星富集文庫法得到的微衛星標記可以在一定程度上滿足遺傳多樣性研究、遺傳結構分析和親緣關系鑒定等工作[9,13],但因工作量大、成本高無法滿足連鎖圖譜構建、基因定位和QTL分析等需要大量標記的研究。利用通量大和周期性短的高通量測序技術,將通過探針富集得到的SSR序列測序獲取微衛星位點,是目前大量獲取微衛星序列的一種常用方法,該技術也是目前從gDNA、cDNA和EST中篩選微衛星位點的一種方式。本研究利用高通量測序技術對探針富集所得序列測序,得到的SSR標記對豚鹿物種構建遺傳連鎖圖譜和分子標記的開發等具有重要意義。
本研究發現豚鹿SSR位點以雙堿基重復為主,其次是單堿基重復,與梅花鹿(Cervusnippon)[14]和大鼠[15]等物種的微衛星優勢重復類型一致,與牛、綿羊[16]和豬[17]等以單堿基重復為主的類型不同,這可能是由豚鹿自身基因決定的,也可能與本研究采取的SSR開發方法有關。研究發現,選用不同的搜索參數會出現不同優勢的堿基重復類型[18-19],也可能是此種原因導致了豚鹿SSR分子標記以雙堿基重復為主,但大部分研究認為雙堿基重復是物種優勢重復堿基類型[20],本研究也證明了此種觀點。其他類型的堿基重復所占比例總體上呈現隨重復單位的增加所占比例減少的趨勢,符合有關研究規律[21]。在單堿基重復中,以A/T重復為主,是物種中普遍存在的現象。在雙堿基重復中,AC/GT重復占比遠大于另外3種重復,這與動植物中SSR DNA中AC/GT含量豐富的研究結果[22-24]相一致,此種現象可能是由于在DNA復制過程中,連接A、T的雙鍵比連接G、C的三鍵更容易斷裂導致。
群體雜合度的高低可以反映群體在多個基因座上的遺傳變異程度。本研究所選種群的平均觀測雜合度為0.680,平均期望雜合度為0.513,表明該群體的遺傳多樣性較高,觀測雜合度與期望雜合度有差異說明該種群存在雜合子缺失或存在純合子過剩的現象。本研究中有2個位點不符合哈迪-溫伯格平衡,這可能與近親雜交、存在無效等位基因、種群退化和圈養管理模式等因素有關。多態性信息含量(PIC)是衡量群體遺傳多樣性的一個重要參數,依據其值高低分為高度多態位點(PIC>0.50)、中度多態位點(0.25≤PIC≤0.50)和低度多態位點(PIC<0.25)。本研究篩選出的7個SSR標記,高度多態位點有3個,中度多態位點有3個,低度多態位點有1個,這可能是因樣本量小于25所致[25]。
本研究采用Illumina MiSeq測序平臺結合SSR富集文庫測序方法開發的豚鹿SSR分子標記,能夠為豚鹿種群的遺傳多樣性和連鎖圖譜構建等研究提供技術基礎。

