散射共振光譜法優化DNA去除溴化乙錠污染物
張 杰 李軍生 覃 懿 黃國霞 閻柳娟 馬 紀
散射共振光譜法優化DNA去除溴化乙錠污染物
張杰1,2李軍生1,2覃懿3黃國霞1,2閻柳娟1,2馬紀1,2
(1.廣西科技大學,廣西 柳州 545006;2.廣西糖資源綠色加工重點實驗室,廣西 柳州 545006;3.中國科技開發院廣西分院,廣西 南寧 530022)
文章利用紫外吸收光譜研究了DNA與溴化乙錠 (EtBr)的相互作用、散射共振光譜研究了二者的相互作用強度及影響因素,最后通過磁珠分離EtBr-DNA復合物以去除EtBr污染物。紫外可見吸收光譜結果表明DNA與EtBr的結合模式為嵌插結合。散射共振光譜和EtBr去除實驗結果表明,CaCl2、SDS、CTAB、葡萄糖和尿素能夠促進二者的結合,使DNA結合飽和值變大,提高EtBr的去除效率;NaCl抑制二者的結合,使DNA結合飽和值變小,降低EtBr的去除效率。研究為高效去除EtBr污染物提供新見解。
溴化乙錠;DNA;去除
引言
溴化乙錠(ethidium bromide,EtBr)具有抗病毒功能,為獸醫領域中一種抗錐蟲的藥物,可用于治療或預防由錐蟲亞目原生動物引起的感染[1,2]。然而,自20世紀60年代Lepecq和Paoletti首次報道EtBr與DNA結合熒光顯著增強(約25倍)現象后,使其成為了一種不可替代的核酸染料,并廣泛應用于實驗室核酸染色[3]。但是,越來越多的研究結果表明,EtBr可能是一種非常強的誘變劑、致癌物或致畸物,因為它可以干擾體內核酸的合成[4]。盡管有這樣的潛在危害,EtBr仍舊大范圍地在實驗室中使用。水溶性極強且在自然環境中較穩定的EtBr不經過特殊處理直接排放至下水道,無疑會給環境帶來巨大威脅。到目前為止,去除EtBr的常用方法是吸附和降解,但這些常規處理方法產成的復合物可能會給污染物的處理帶來更大的負擔,因此亟需探索一種簡單高效環保的去除EtBr污染物的方法。
DNA是具有高度特異性功能的生物聚合物,已在環境保護領域得到廣泛應用。有學者提出基于嵌入原理,利用DNA俘獲消除黃曲霉毒素、吖啶、多環芳烴等有害物質。這些已有的研究成果為構建選擇性消除EtBr污染物提供了新見解。已有大量實驗結果證明EtBr是經典的DNA嵌入[5,6],但研究其相互作用的強度及其影響因素的報道很少見。因此,探究DNA與EtBr的結合強度可為基于嵌插原理消除EtBr提供理論基礎。
紫外-可見光譜法是探究小分子與DNA相互作用模式的常用方法。此前,小分子與DNA的紫外-可見光譜中可觀察到紅移和減色現象,以確定兩者的相互作用模式。共振光散射(RLS)分析法能高靈敏度地檢測到核算、蛋白質等生物大分子的結構變化,已應用于探究DNA和小分子體外相互作用的影響。磁珠表面豐富的活性基團,可分離出溶液中的DNA。因此本文利用紫外-可見光譜法研究EtBr與DNA的相互作用模式,共振光散射法研究EtBr與DNA的相互作用強度,并計算EtBr的DNA結合飽和值反映其結合DNA能力的大小,最后通過磁珠分離出EtBr-DNA復合物,為選擇性去除EtBr污染物構建一種高效環保的新途徑。
1 實驗部分
1.1 儀器及試劑
Gary-60紫外可見分光光度計(安捷倫科技公司);RF-5301PC熒光分光光度計(日本島津);pH計(PHS-25CW,上海思龍科學儀器有限公司);數控水浴鍋(HH-4,國華電器有限公司)。
EtBr(上海源葉,10 mg/mL,純度>99.9%),用pH 7.40的Tris-HCl緩沖溶液稀釋成所需濃度。DNA參照劉潤芝[7]的方法從鯉魚魚白中提取,提取得到的DNA在紫外分光光度計中測量其在230 nm(A230)、260 nm(A260)和280 nm(A280)處的吸光度值,當A230/A280>2.0且A260/A280=1.8時表明DNA純度達標,方可用二次水配置成相應濃度用于實驗。DNA綁定液(2 M KCl,0.01 M Tris,pH 6.3)用超純水配制;Tris-HCl緩沖溶液的配制方法參照生化試劑指導手冊。
1.2 方法
1.2.1EtBr與DNA相互作用的紫外可見吸收光譜
在1.0 cm×1.0 cm的石英比色皿中加入2 mL EtBr溶液(3.0×10-5mol/L)。接著逐次往EtBr溶液中滴加10 μL DNA溶液(1.0×10-3mol/L),混合均勻后靜置15 min,掃描其在350 nm~600 nm范圍內的紫外吸收光譜。
1.2.2EtBr與DNA相互作用的散射共振光譜
在1.0 cm×1.0 cm的石英比色皿中加入3 mL EtBr溶液 (1.138×10-5mol/L),設定熒光分光光度計λem=λex進行同步掃描。接著逐次往EtBr溶液中滴加10 μL DNA溶液(3.6×10-5mol/L),混合均勻后靜置15 min,進行同步掃描并記錄。取362 nm處的共振峰值記為RLS強度值。
1.2.3EtBr與DNA的結合飽和值
在掃描散射共振光譜時,當EtBr溶液中加入DNA,其RLS強度值會不斷增大;當兩者結合達到飽和時,隨著DNA的繼續加入RLS強度值開始減小。EtBr的RLS強度值最大時對應的DNA濃度即為飽和DNA濃度,因此DNA結合飽和值可由以式(1)計算:
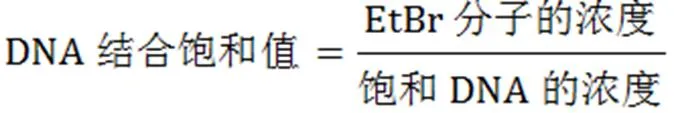
1.2.4EtBr去除實驗
將1.0 mL EtBr溶液(0.5 μg/mL)和0.1 mL DNA溶液(0.2 mg/mL)轉移到EP管中,于35 ℃的搖床中反應15 min,然后加入磁珠(5.0 mg)和DNA綁定液(1 mL),在搖床中繼續反應45 min。反應結束后,將EP管放入磁力分離器中靜置5 min,轉移上清液至新的EP管中,于紫外分光光度計中測定其在480 nm處的吸光度,根據濃度和吸光度換算出樣品的EtBr殘留濃度。根據以式(2)、式(3)計算EtBr去除效率(R,%)和DNA吸附量(q,mg/g)。
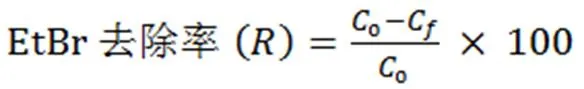
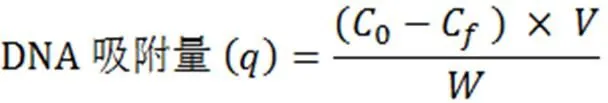
式中C0是EtBr的初始濃度(μg/mL),Cf是EtBr的殘留濃度(μg/mL),V是溶液的體積(mL),W是DNA的重量(μg)。
2 結果與討論
2.1 EtBr與DNA相互作用的紫外可見吸收光譜
如圖1所示,EtBr溶液中不含DNA時,在480 nm出現明顯的紫外吸收峰,然而隨著DNA濃度的增加,EtBr溶液的紫外-可見光譜觀察到明顯的紅移,特征峰的位置從480 nm移動到495 nm(Δλmax = 15 nm),吸光度從0.34減小為0.19(H = 44.12%)。這可以解釋為EtBr分子中的稠環結構與DNA發生相互作用[8],其π*軌道與DNA堿基對的π電子軌道耦合,產生π-π疊加效應,導致π→π*的躍遷能量和躍遷概率降低,從而使吸收光譜出現減色效應。因此,EtBr與DNA之間發生了嵌插相互作用,形成復合物。
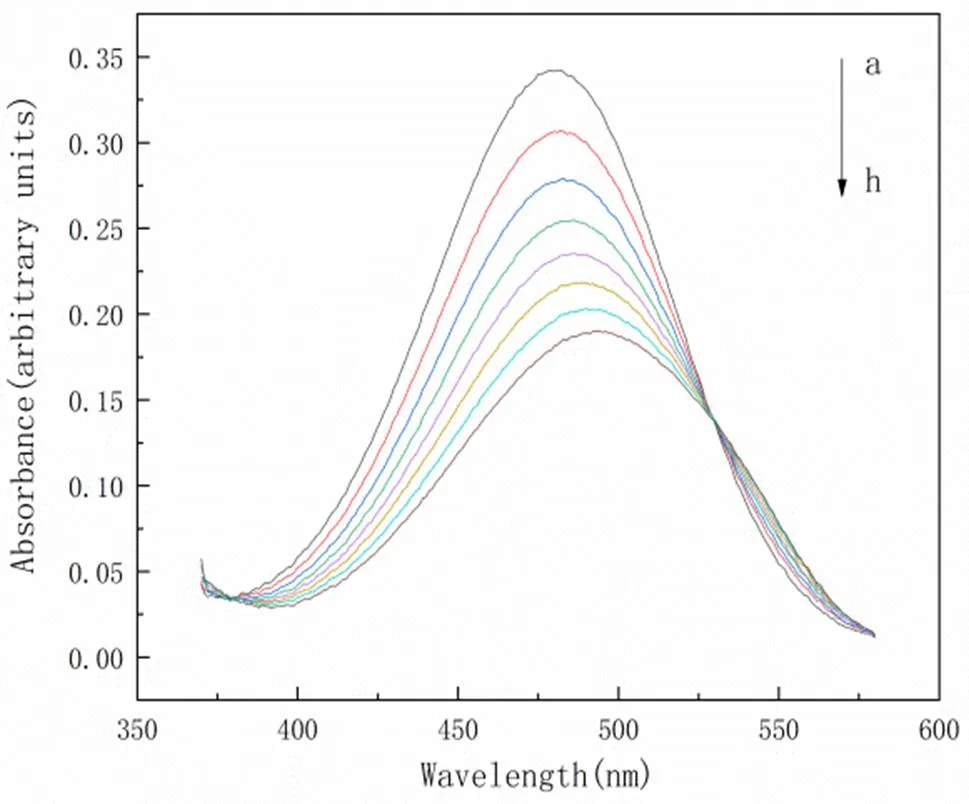
圖1 EtBr分別在有和沒有DNA情況下的紫外吸收光譜變化(室溫,pH7.4). a-h: [EtBr]= 1.9009×10-5 mol/L, [DNA]= 0, 0.39, 0.72, 1.04, 1.37, 1.70, 2.02,2.41×10-4 mol/L.
2.2 EtBr與DNA相互作用的散射共振光譜
2.2.1溫度對EtBr與DNA相互作用的影響
如圖2所示,EtBr-DNA復合物的RLS強度在35 ℃達到最大。當體系溫度低于35 ℃時,分子熱運動速度變慢導致EtBr與DNA的碰撞機會減少,不利于兩者的結合;當溫度高于35 ℃時,DNA的雙螺旋結構受到輕微的影響,導致兩者的結合強度受到影響,使RLS強度下降。因此,EtBr與DNA相互作用的最佳溫度35 ℃。
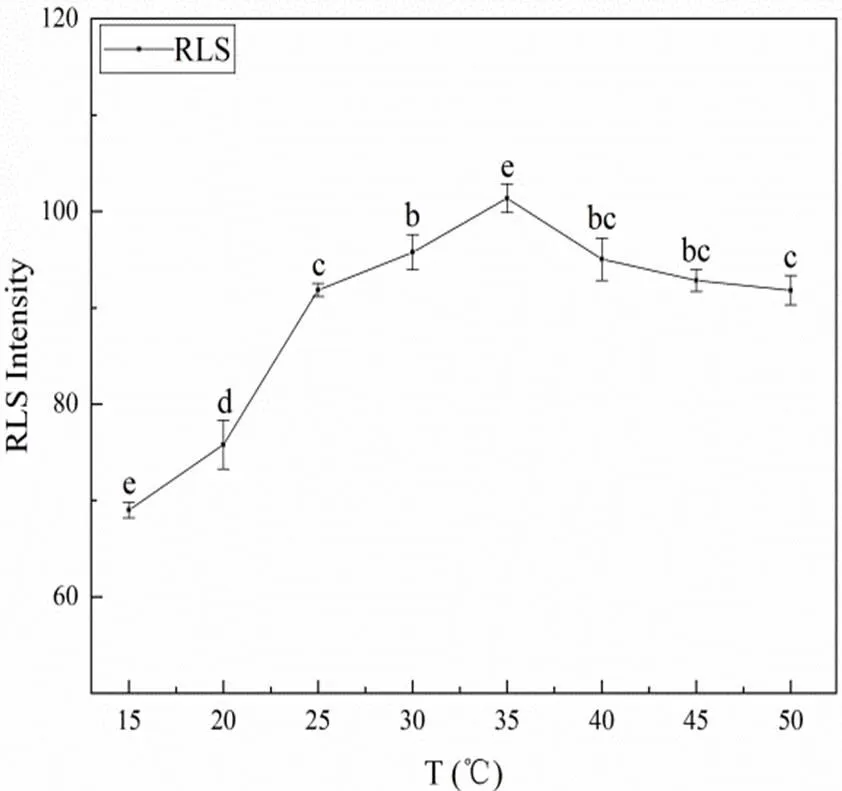
圖2 不同溫度下EtBr-DNA的RLS強度變化(pH7.4),[EtBr]=1.138×10-5 mol/L,[DNA]=0.25×10-5 mol/L(P<0.05)
2.2.2pH對EtBr與DNA相互作用的影響
從圖3可知,pH從5.8增加至7.4時,EtBr與DNA的RLS強度逐漸增大,pH在7.4~9.0范圍內迅速減小。這是因為低于pH為7.4時,溶液中的H+與帶正電荷的EtBr發生競爭作用,減弱了EtBr和帶負電荷的DNA之間的靜電相互作用[9],不利于兩者結合,導致RLS強度減小。當pH高于7.4時,溶液中的OH-削弱了DNA表面基團的質子化,干擾了DNA與非電離EtBr的結合。因此,當pH為7.4時EtBr與DNA相互作用強度最大。
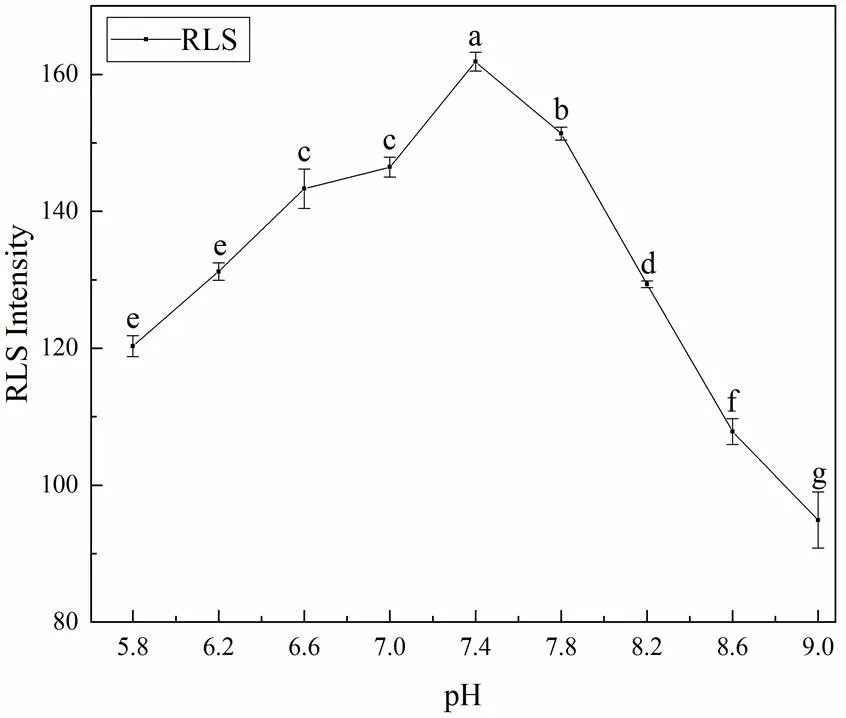
圖3 不同pH條件下EtBr-DNA的RLS強度變化(35 ℃),[EtBr]=1.138×10-5 mol/L,[DNA]=0.25×10-5 mol/L(P<0.05)
2.2.3環境共存物對EtBr與DNA相互作用的影響
如圖4(A)所示,在溫度為35 ℃、溶液的pH 為7.4時,隨著NaCl濃度的增大,EtBr-DNA復合物的RLS強度逐漸減小。這可以解釋為EtBr與DNA之間存在靜電相互作用,體系中引入的Na+富集在DNA結構附近對EtBr產生斥力,打破了電荷平衡[10]。因此,NaCl的加入,不利于EtBr與DNA的結合。
如圖4(B)所示,在溫度為35 ℃、溶液的pH 為7.4時,隨著CaCl2濃度的增加,EtBr-DNA復合物的RLS強度呈先增大后減小的趨勢,當CaCl2濃度為0.03 mol/L時RLS強度達到最大。適量的Ca2+會增加DNA結構的穩定性,有利于EtBr與DNA的結合;而Ca2+過多時會產生與Na+同樣的作用,抑制兩者的結合。因此,體系中加入0.03 mol/L CaCl2會增強EtBr與DNA的結合。
如圖4(C)所示,在溫度為35 ℃、溶液的pH 為7.4的條件下,當SDS濃度增加至0.6×10-3mol/L時,EtBr-DNA復合物的RLS強度急速增加;繼續加入SDS溶液時,體系的RLS強度出現降低的趨勢。SDS為陰離子表面活性劑,它的引入會減弱DNA的親水性,從而EtBr更容易與DNA相結合[11];當SDS濃度過大時則會增加DNA的疏水性,DNA結構發生卷曲使得堿基對不能完全裸露,不利于EtBr與DNA結合。因此,體系中加入0.6×10-3mol/L SDS可以促進EtBr與DNA的結合。
如圖4(D)所示,在溫度為35 ℃、溶液的pH 為7.4時,1.6×10-4mol/L CTAB能夠使EtBr-DNA復合物的RLS強度達到最大。CTAB是一種陽離子表面活性劑,少量的CTAB可以增加溶液的表面活性,使DNA雙鏈更好地完全展開,有助于EtBr與DNA的結合;而過多CTAB會沉淀DNA,使DNA成為高度卷曲狀態,阻礙其與EtBr的結合。因此,體系中加入1.6×10-4mol/L CTAB可以促進EtBr與DNA的結合。
如圖4(E)所示,設定溫度為35 ℃、溶液的pH 為7.4,當溶液中葡萄糖的濃度為0.0016 mol/L時,EtBr-DNA復合物的RLS強度達到峰值;繼續增大葡萄糖的濃度時,RLS強度逐漸下降。結果說明溶液中加入少量的葡萄糖會促進EtBr與DNA的結合;當葡萄糖濃度過高時,溶液的黏度增大,不利于分子間的運動,從而導致EtBr與DNA的結合強度下降[12]。因此,體系中加入0.0016 mol/L 葡萄糖可以促進EtBr與DNA的結合。
如圖4(F)所示,設定溫度為35 ℃、溶液的pH 為7.4,當溶液中尿素的濃度增加至0.013 mol/L時,EtBr-DNA復合物的RLS強度有所增強,而尿素的濃度超過0.013 mol/L時,RLS強度逐漸變低。這是因為濃度過高的尿素誘導DNA變性后結構發生變化,EtBr與DNA的相互作用受到影響,從而出現RLS強度下降的情況。因此,體系中加入0.013 mol/L 尿素有利于EtBr與DNA的結合。
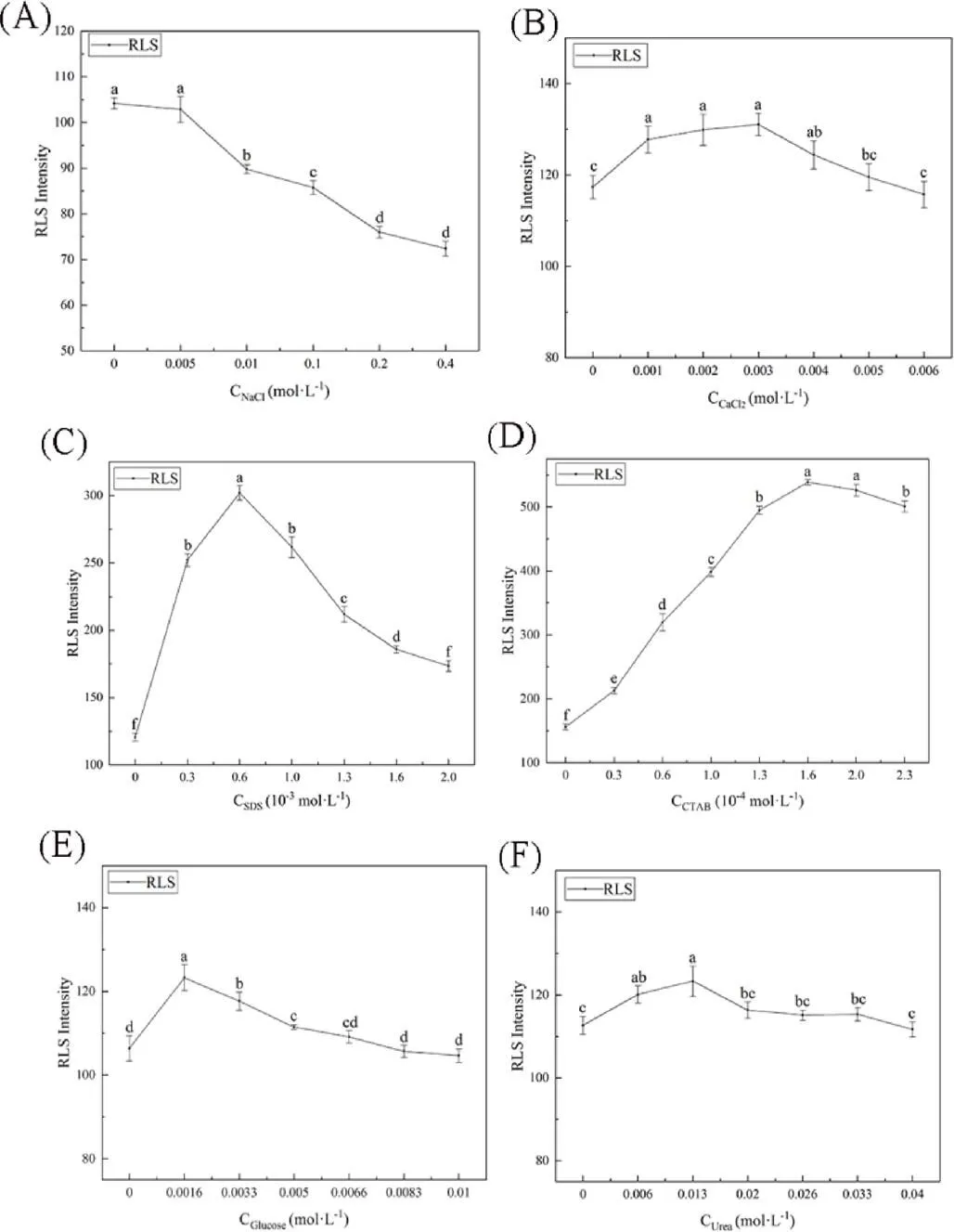
圖4 (A)、(B)、(C)、(D)、(E)、(F)分別為在不同NaCl、CaCl2、SDS、CTAB、葡萄糖、尿素濃度下,EtBr-DNA復合物RLS強度的變化,[EtBr]=1.138×10-5 mol/L,[DNA]=0.25×10-5 mol/L(P<0.05)
2.3 EtBr與DNA的結合飽和值
常溫下DNA與EtBr的散射共振光譜如圖5所示。EtBr在362 nm出現穩定且明顯的共振峰。隨著DNA的不斷加入,EtBr-DNA復合物的RLS強度不斷增大,直至DNA過量時,體系的RLS強度出現下降的趨勢。這是因為散射共振光譜能靈敏地檢測到體系中粒子大小的變化[10],當EtBr溶液中加入DNA后,EtBr迅速地與DNA發生嵌插相互作用形成體積較大的EtBr-DNA復合物,使得EtBr-DNA的共振信號急劇增加。當加入過量DNA時,由于溶液中EtBr分子與DNA的結合達到飽和而不再發生相互作用,此時的DNA相對EtBr-DNA復合物的體積明顯變小,導致共振信號強度下降。所以當體系的共振強度出現下降的情況,即可視為DNA已經達到飽和狀態。根據公式(1)可計算出常溫下EtBr的DNA結合飽和值為13.87。
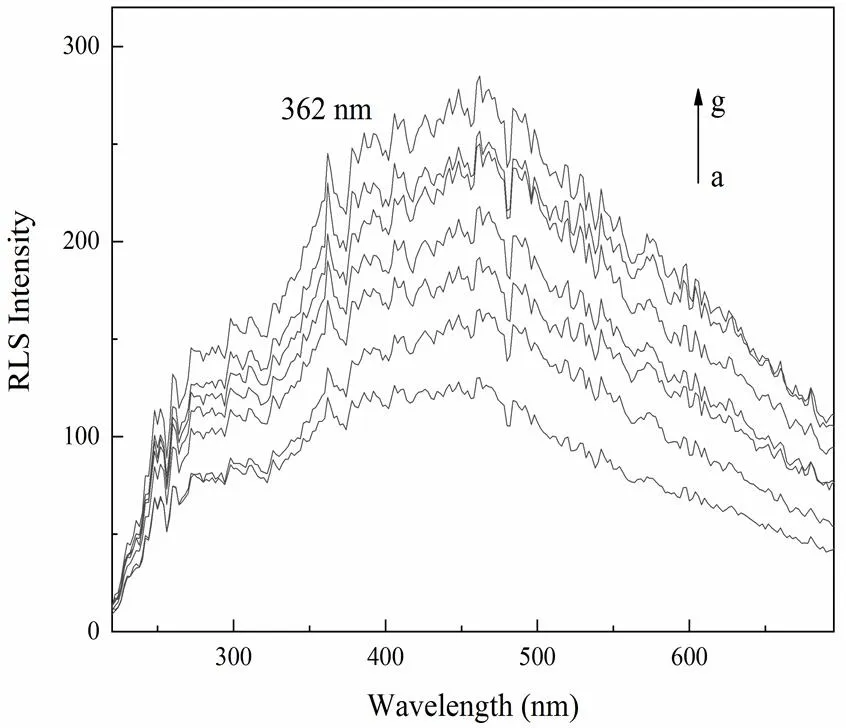
圖5 EtBr在加入DNA前后的共振光散射光譜變化(室溫,pH 7.4),[EtBr]=1.138×10-5 mol/L, a-e:[DNA]= 0,0.12,0.24,0.36,0.49,0.61,0.73,0.97,0.82×10-6 mol/L
DNA結合飽和值能客觀地反映出EtBr與DNA結合能力的大小,將EtBr溶液處于散射共振光譜探索出來的最佳結合條件,探究各條件下的DNA結合飽和值,結果如表1所示。與溫度為30 ℃、pH為7.4時相比,NaCl(0.01 mol/L)的引入會使EtBr的DNA結合飽和值減小11%,而CaCl2(0.03 mol/L)的加入會使DNA結飽和值增11%。溶液體系中加入表面活性劑SDS(0.6×10-3mol/L)、CTAB(1.6×10-4mol/L)時,EtBr的DNA結合飽和值有所提高,其中CTAB增大DNA結合飽和值的幅度要大于SDS。葡萄糖(0.0016 mol/L)和尿素(0.013 mol/L)均能促進EtBr與DNA的結合,DNA結合飽和值分別提高9%和20%。

表1 EtBr在各條件下的DNA結合飽和值
2.4 DNA去除EtBr
表2列出了DNA在散射共振光譜技術探究出來的最佳條件下對EtBr的去除效率及吸附能力。與EtBr-DNA體系相比,加入NaCl會降低DNA對EtBr的去除率,去除能力降低4.16%;加入0.03 mol/L CaCl2、0.6×10-3mol/L SDS、1.6×10-4mol/L CTAB、0.0016 mol/L葡萄糖和0.013 mol/L尿素時,能增強DNA對EtBr的去除率,去除能力分別增大4.16%、1.12%、5.04%、5.19%、2.39%和1.20%,這與之前計算得到的DNA結合飽和值的趨勢一致。以上結果表明,基于EtBr與DNA嵌插相互作用去除環境中的EtBr污染物時,溶液體系中應避免NaCl,加入0.03 mol/L CaCl2、0.6×10-3mol/L SDS、1.6×10-4CTAB、0.0016 mol/L葡萄糖或者0.013 mol/L尿素可提高去除效率。
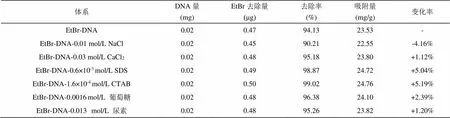
表2 不同體系下DNA對EtBr的去除能力
3 結論
本文通過光譜學實驗研究了EtBr與DNA的相互作用模式、結合強度及影響因素,并通過磁珠分離EtBr-DNA復合物以去除EtBr污染物。紫外可見吸收光譜結果證實了EtBr與DNA發生嵌插相互作用,并形成穩定的復合物。散射共振光譜結果表明35 ℃、pH7.4為EtBr與DNA結合的最條件,0.03 mol/L CaCl2、0.6×10-3mol/L SDS、1.6×10-4CTAB、0.0016 mol/L葡萄糖和0.013 mol/L尿素能使EtBr-DNA復合物的RLS強度增大,NaCl可降低EtBr-DNA復合物的RLS強度。此外,EtBr的DNA結合飽和值的計算結果表明,適宜濃度下的CaCl2、SDS、CTAB、葡萄糖和尿素對EtBr與DNA的結合有促進作用,DNA結合飽和值分別提高了9%、9%、20%、9%和20%;NaCl對二者的結合起阻礙作用,DNA結合飽和值降低了11%。DNA去除EtBr實驗結果表明, NaCl可使DNA對EtBr的去除率降4.16%;當體系中加入0.03 mol/L CaCl2、0.6×10-3mol/L SDS、1.6×10-4CTAB、0.0016 mol/L葡萄糖或0.013 mol/L尿素能使DNA對EtBr的去除率分別增強4.16%、1.12%、5.04%、5.19%、2.39%和1.20%。以上結果為基于DNA與EtBr的嵌插作用去除EtBr污染物提供一種新的方法。
[1] Woolfe, G. The Trypanocidal action of phenanthridine compounds[J]. Annals of Tropical Medicine and Parasitology, 1952, 46(4): 285-288.
[2] Woolfe G. Trypanocidal action of phenanthridine compounds: effect of changing the quaternary groups of known trypanocides[J]. British Journal of Pharmacology, 2012, 11(3): 330-333.
[3] Lepecq J B, Paoletti C C. A fluorescent complex between ethidium bromide and nucleic acids. Physical-Chemical characterization[J]. Journal of Molecular Biology, 1967, 27(1): 87-106.
[4] 石立杰. 金屬卟啉催化的致癌、致突變劑無害化處理技術研究-氯化血紅素對溴化乙錠的催化轉化[J]. 2007(8): 46915.
[5] 馬立波,石昕,牛淑妍,等. 溴化乙錠與DNA相互作用的電化學及紫外-可見光譜研究[J]. 青島科技大學學報(自然科學版),2003,24(5): 398-400.
[6] Vardevanyan P O, Arakelyan V B, Parsadanyan M A, et al. Analysis of experimental binding curves of EtBr with single-and double-stranded DNA at small fillings[J]. Modern Physics Letters B, 2014, 28(22): 1450178.
[7] 劉潤芝. 從鯉魚精巢中提取脫氧核糖核酸(DNA)的研究[J]. 湖南師范大學自然科學學報, 1988(3): 95-98.
[8] Chilina A R, Ming C, Ives D H, et al. A new method for the isolation of eukaryotic nuclear proteins[J]. Analytical Biochemistry, 1976, 72(1-2): 552-565.
[9] Nguyen V T, Nguyen T B, Chen C W, et al. Influence of pyrolysis temperature on polycyclic aromatic hydrocarbons production and tetracycline adsorption behavior of biochar derived from spent coffee ground [J]. Bioresource technology, 2019, 284: 197-203.
[10] Rajabi H R, Razmpour S. Spectrochimica acta part A: molecular and biomolecular spectroscopy[J]. Spectrochimica Acta-Part A: Molecular and Biomolecular Spectroscopy, 2009, 115(1-2): 269-274.
[11] Dias R L, Basso B, Donadio M V F, et al. Leucine reduces the proliferation of MC3T3-E1 cells through DNA damage and cell senescence[J]. Toxicology in Vitro: An International Journal Published in Association with Bibra, 2018, 48: 1-10.
[12] 李蔚侖,李軍生,黃國霞,等. 乙酰紫草素的潛在毒性機理研究及毒性評價[J]. 食品工業,2015, 36(5): 174-178.
Optimization of DNA removal of ethidium bromide pollutants by scattering resonance spectroscopy
In this paper, the intercalation between DNA and ethidium bromide (EtBr) was studied by UV absorption spectroscopy, and the interaction intensity and influencing factors were studied by scattering resonance spectroscopy. Finally, the ETBR-DNA complex was separated by magnetic beads to remove EtBr contaminants. The UV-vis absorption spectroscopy results showed that the binding mode of DNA and EtBr was intercalation binding. The results of the resonance light scattering spectrometry and EtBr removal experiments showed that CaCl2, SDS, CTAB, glucose and urea can promote the combination of the two, increase the saturation value of DNA binding and improve the removal efficiency of EtBr; NaCl inhibits the combination of the two, reduces the saturation value of DNA binding and reduces the removal efficiency of EtBr. This study provides new insights for the efficient removal of EtBr pollutants.
ethidium bromide; DNA; removal
O65
A
1008-1151(2022)06-0042-05
2022-04-01
廣西自然科學基金項目(2018GxNsFDA281030,2020GXNSFAAl59021);國家自然科學基金項目(21966008)。
張杰(1993-),廣西科技大學生物與化學工程學院研究生,研究方向為生物分子的化學修飾與功能研究。
李軍生,廣西科技大學生物與化學工程學院教授,碩士研究生導師,研究方向為生物分子的化學修飾與功能研究;覃懿,中國科技開發院廣西分院工程師,研究方向為制藥工程研究。

