HLLA1基因單核苷酸多態性與高發區食管鱗癌患者術后生存分析
謝葉真,趙學科,宋 昕,徐瑞華,魏夢霞,王 苒,韓雪娜,孫 琳,2,陳亞杰,2,馬琳琳,2,喬佳欣,2,白合林,任景麗,周勝理,王立東
食管癌(esophageal cancer, EC)是世界多發惡性腫瘤之一,位居中國癌癥死亡的第四位[1]。我國是世界食管癌高發國家之一,新增病例數位于世界前列[2]。近幾年將早期檢測、手術、放化療等運用于食管癌的防治,取得了不錯的成效,但食管癌的預后不良,5 a生存率僅有15%~25%[3]。在中國,97%的食管癌是食管鱗癌,具有顯著高發區和家族聚集現象兩大流行病學特征[4]。已有研究表明,食管癌可能是環境和遺傳相互作用的結果[5],其發生發展是由多種因素、多個階段、多個過程決定的,環境是主導,通過基因起作用[6-7]。基因組變異是DNA的堿基對改變,包括胚系突變(單核苷酸多態性,即SNP)和體細胞突變[8-9]。目前,用全基因組測序(whole genome sequencing, WGS)和全基因組外顯子測序(whole exome sequencing, WES)已發現了很多促進腫瘤發生發展的體細胞突變[10-11],但食管癌多發生在亞種人群,而有關食管鱗癌胚系突變的研究很少,本次實驗以199例高發區食管癌患者的癌組織細胞和癌旁組織細胞的全外顯子測序數據,運用順式表達數量性狀基因座的分析方法,得到了與基因表達水平相關的SNP,將其中rs2280851 SNP的3個基因型與199例高發區食管癌患者術后總生存期進行生存分析,運用LC-MS/MS蛋白表達定量測得CTR9蛋白表達量,探究食管癌的可疑致病SNP。現報道如下。
1 材料與方法
1.1 研究對象
納入的199例患者均來自鄭州大學第一附屬醫院省部共建食管癌防治國家重點實驗室50萬例食管癌患者臨床信息數據庫[4],患者均來自于河南省食管癌高發區的某市級醫院,經醫院確診并行手術治療,確診時間為2019年11月至2020年1月。其中男132例(66.33%),女67例(33.67%),每位患者對本研究均知情同意,并且此項研究已獲得鄭州大學倫理委員會的批準。
收集199位食管癌患者的癌組織和癌旁組織,均為新鮮組織樣本,在手術切除后30 min內進行宏觀解剖,并保存在-80℃下。樣本采集前未接受放化療等其他治療,以半定量的方式對腫瘤細胞進行視覺評分,僅選擇惡性細胞>70%的腫瘤進行全基因組外顯子測序。癌旁正常組織是距腫瘤邊緣至少2 cm遠的鄰近上皮正常組織(生殖系對照),用于取樣和分子分析。本研究主要通過電話、家訪、村醫與患者或患者家屬直接聯系以及通過新合作醫療數據庫、醫療保障局數據庫和公民死亡信息登記管理等系統查詢方式進行隨訪,每0.5 a進行一次隨訪,記錄去世時間和主要死因等。本研究隨訪截止到2021年12月31日,隨訪成功199 例。
1.2 組織樣本的制備
將199例食管癌患者的癌組織和癌旁組織配對,分裝到準備好的已經標記的凍存管內,樣本編號為001~199號,在樣本類型中,食管癌組織用字母T標記(Tumor),癌旁正常組織用字母N標記(Normal)。收集到的樣品在液氮中快速冷凍并保存,同時保證每份樣品有足夠的DNA可供提取。
1.3 多組學SNP位點捕獲、檢測與篩選
1.3.1 DNA樣品檢測利用瓊脂糖凝膠電泳分析DNA樣本是否有蛋白,RNA污染及其降解程度。并用Qubit 3.0測出DNA樣本濃度,選擇0.6 μg以上的樣品進行后續建庫。
1.3.2 建庫捕獲及上機測序采用Agilent液相芯片捕獲系統對人全外顯子區的DNA進行富集,Agilent SureSelect Human All Exon V6試劑盒進行建庫及捕獲。后在Illumina平臺上進行高通量測序。
1.3.3 數據質量控制高通量測序獲得的原始圖像數據經過轉化后形成原始Raw Data測序序列。將原始Raw Data數據進行過濾,并且控制平均堿基位置測序錯誤率低于0.1%,樣本的測序reads達到95%以上的對比率,且堿基覆蓋深度達到10×以上時,該點檢測的SNP比較可信。
1.3.4 多組學SNP位點篩選實驗方法為WES(由諾禾致源測序公司進行),基于WES數據中所檢測到的SNP位點,經過將正常與癌組織中都有的SNP進行比對,共有62萬個SNP;其中以編碼區非同義突變、P<0.2為條件篩選出的SNP位點有3 897個;在Mutation表中都存在且生存曲線有差異的有797個位點。
1.4 蛋白表達的測定
采用液相色譜—質譜(liquid chromatography-tandem mass spectrometry,LC-MS/MS)技術對199例食管癌的癌組織和癌旁組織樣本進行分析。
2 結果
2.1 rs2280851 SNP與食管癌患者生存期的關聯分析
rs2280851與生存期的生存分析曲線表明,rs2280851的A突變為G,199例食管癌患者中,有109例G/G基因型個體,54例A/G基因型個體,36例A/A基因型個體;A/A基因型個體生存率顯著高于G/G基因型個體,A/A基因型個體生存率高于A/G基因型個體,A/G基因型個體生存率高于G/G基因型個體(P<0.001)。見圖1。
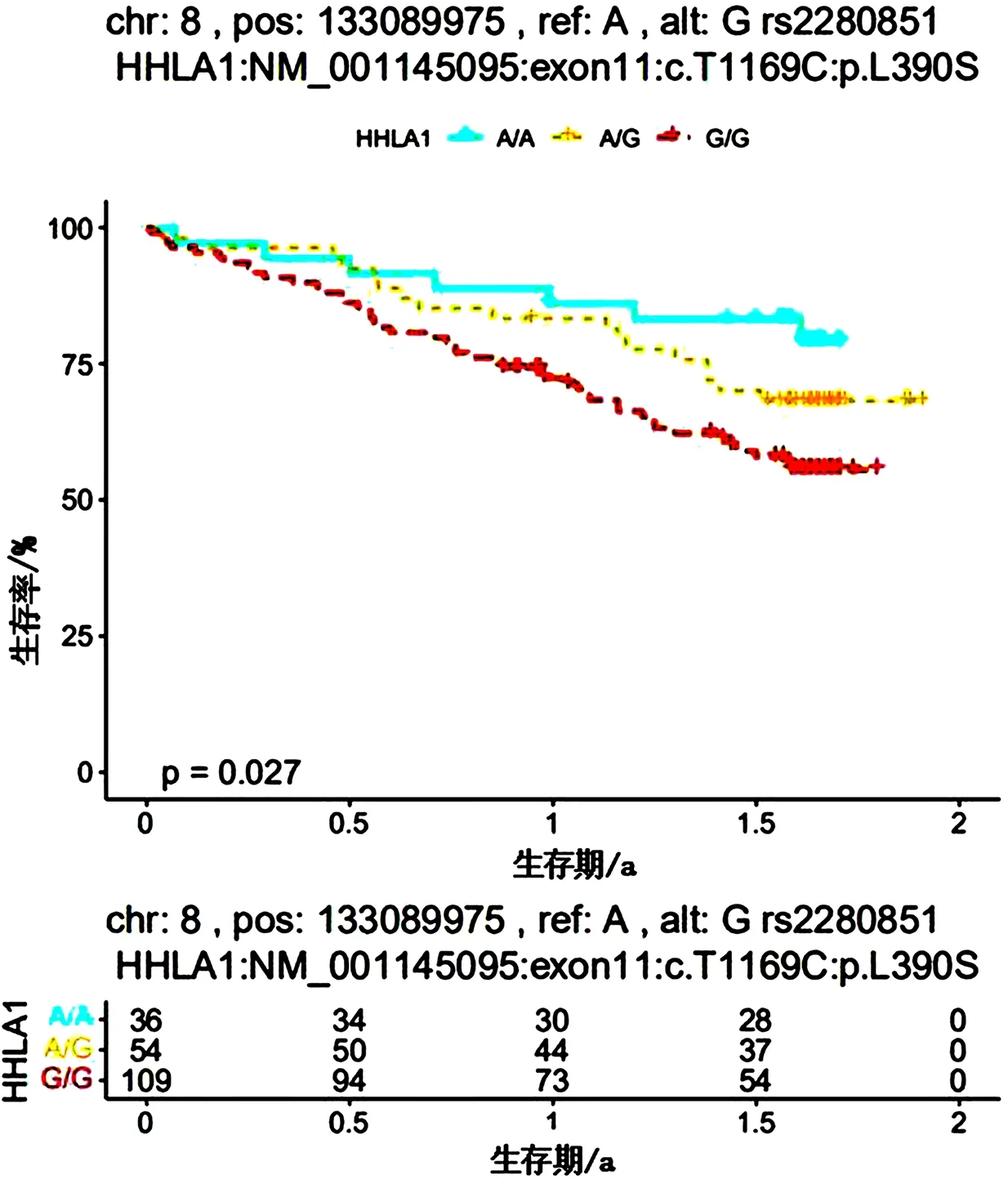
圖1 3種基因型術后生存分析比較
2.2 rs2280851與CTR9蛋白表達量的關系
eQTL分析表明,eQTL距離調控基因非常遠,大于20 Mb以上,是通過調控HHLA1基因產生的轉錄子調控蛋白來介導靶標目的基因的表達。作者做了rs2280851與CTR9蛋白表達量關系的研究。相對于rs2280851的參考型A/A,突變型G/G型蛋白表達量最少,rs2280851突變后,通過調控HHLA1基因產生的轉錄子,顯著降低CTR9的表達。見圖2。

圖2 rs2280851的3種基因型CTR9蛋白表達
3 討論
食管癌是常見的消化道腫瘤之一,我國食管癌主要是鱗癌[12],高發于河南、山西、太行山、閩粵等地區[13]。食管癌是環境和遺傳長期相互作用的結果,有關研究已發現p53、BRCA2等基因與食管癌相關[14-15],但是所用方法通量低,不能鑒別易感基因和變異位點。以高通量方法,系統地闡明食管癌相關候選基因非常必要。本實驗對199例高發區食管鱗癌患者進行全外顯子測序,通過樣本檢測、數據質控及與患者術后生存期進行生存分析,發現rs2280851位點突變型G/G對食管鱗癌患者的生存影響較大,為可疑致病SNP。通過NCBI數據庫查找,rs2280851位點關聯的基因為HLLA1。通過LC-MS/MS蛋白表達定量和eQTL相關分析,發現本組患者SNP rs2280851等位基因G的攜帶量與CTR9蛋白的表達呈負相關。
rs2280851 SNP影響HLLA1所調控的CTR9蛋白的表達,rs2280851 SNP與食管癌患者的生存預后相關。正常人與HLLA1基因相關的rs2280851的堿基是A,當A突變后,CTR9蛋白的表達量降低,但預后變差。由此可見,HLLA1基因SNP rs2280851G/G型可能是致病SNP。已有研究表明,CTR9是PAF1復合物重要的組成蛋白,PAF1在RNA聚合酶Ⅱ轉錄時起到輔助功能,參與染色質中組蛋白的甲基化修飾,在細胞內的轉錄延伸中發揮功能[16]。另外一個重要的蛋白是paf1,paf1和ctr9的突變會致癌或促進癌相關疾病的發展[17]。現有研究表明CTR9蛋白在肝癌患者的腫瘤組織中表達上調,CTR9的下調顯著降低了肝癌細胞的增殖、侵襲和遷移[18]。
目前普遍認為食管癌的發生機制是環境—基因—遺傳相互作用,高發區是環境因素,高發區食管癌患者術后生存期低,可能與HLLA1基因的SNP rs2280851 A突變為G,CTR9蛋白表達降低有關。本研究運用全外顯子測序技術和LC-MS/MS蛋白表達定量,首次提出SNPrs2280851對應的基因HLLA1的突變型GG是食管癌變的重要分子事件,提示HLLA1可能與腫瘤的發生和發展有關。同時為“環境-遺傳-基因互作”食管癌變機制提供了新的證據和高危人群分子分型標記,有利于預警篩查早期發現和防治食管癌高危人群,確定用于早期發現、早期診斷和個體化的分子靶標,為進一步建立食管癌高危人群預警篩查、早期發現和防治提供理論和技術支撐。

