水開菲爾和水開菲爾粒細菌多樣性分析及乳酸菌分離鑒定
高芫超,肖愛波,王 成,王天琪,于 冰,許云賀,張莉力
(錦州醫科大學 食品科學與工程學院,遼寧 錦州 121000)
水開菲爾粒,由胞外多糖組成,是由乳酸菌、醋酸菌等共生微生物組成的穩定菌落,被限制在多醣體和蛋白質基質中,含具有保健功能的次級代謝產物[1]。水開菲爾,也叫作中國菌菇、緹比水晶和天山雪蓮等,它是由有機糖溶液和水開菲爾粒自然發酵而成的一種發酵飲料,含多種益生菌。水開菲爾中微生物種類很多,主要含乳酸菌、醋酸菌和酵母菌,它們是由多種細菌和真菌組成的共生菌系[1],在細菌組成中能觀察到乳酸菌的顯著控制[2]。在水開菲爾發酵過程中,水開菲爾粒會“消化”掉糖,制作出富含益生性乳酸菌的水開菲爾[3]。水開菲爾中含多種酶,能水解多糖,產生果香。其發酵劑水開菲爾粒經發酵后,會得到粗纖維等不溶性沉淀成分,這些成分可以用于制作膳食纖維[4-5]。若經常喝水開菲爾,乳酸菌可以在人體內存活并生長繁殖,水開菲爾的保健功能包括:促進消化、增強肝臟功能、調節內分泌系統、調節血壓、降低膽固醇、幫助減肥[1-2]。益生菌若想成功定植并作用于胃腸道,需要能夠適應胃腸道的低 pH及高滲透壓的膽鹽環境[6]。因此,篩選耐酸、耐膽鹽的益生菌株具有重要研究意義和應用價值。目前,國內外對水開菲爾的研究包括水開菲爾粒發酵時的微生物菌落動力學和代謝組學,研究表明水開菲爾發酵過程中的優勢菌是乳酸菌,但是水開菲爾形成穩定結合體的分子背景尚不清楚,細菌區系的綜合組成也尚未科學界定[7]。近年來,公眾普遍開始關注食品營養及保健功效,以牛奶為基礎的奶開菲爾現已被廣泛研究并應用于各種發酵酸奶、益生性乳酸菌飲料中,可作為良好益生菌來源,具有許多潛在的健康益處。然而目前水開菲爾只限制于家庭小作坊式內部生產,其未得到廣泛使用、未能投入工業化生產,主要是因為缺乏發酵水開菲爾的菌種。
所以,本實驗利用高通量測序技術結合平板菌落計數法,分析水開菲爾和水開菲爾粒的細菌區系及優勢菌屬。然后以耐酸、耐膽鹽能力做指標,從中篩選出具有潛在益生性的菌株。本研究為水開菲爾工業發酵提供備選菌株以及為水開菲爾產品的研發奠定基礎。
1 材料與方法
1.1 實驗材料
水開菲爾、水開菲爾粒:湛江市非歌食品店;MRS肉湯培養基、MRS培養基、營養瓊脂培養基:北京陸橋有限公司;細菌基因組 DNA提取試劑盒:沈陽立信生物有限公司。
1.2 儀器與設備
壓力蒸汽滅菌鍋(DGL-50B)、紫外可見分光光度計(752N):常州鴻運實驗儀器廠;恒溫培養箱(303-4B):浙江尚誠儀器廠;超凈工作臺(JB-VS-1300):上海力辰儀器廠;精密 pH 計(PHS-25)、電子分析天平(M4-AL204):上海越平有限公司;生物顯微鏡(BM-500T):福建睿鴻光電科技有限公司。
1.3 實驗方法
1.3.1 傳統平板計數法測定細菌數量
對水開菲爾和水開菲爾粒中的細菌和乳酸菌計數,細菌參照 GB 4789.2—2016《食品安全國家標準 食品微生物學檢驗 菌落總數測定》,乳酸菌參照GB 4789.35—2016《食品安全國家標準 食品微生物學檢驗 乳酸菌檢驗》,每種樣品做三次平行實驗,記錄結果。
1.3.2 高通量測序分析細菌區系
主要步驟包括:對水開菲爾、水開菲爾粒中總DNA提取和16SrDNA的V3-V4可變區PCR擴增,利用的擴增引物為正向引物 520F: 5′-AYTGGGYDTAAAGNG-3′,反向引物 802R: 5′-TACNVGGGTATCTAATCC-3′,然后檢測擴增序列,將合格序列進行文庫擴建并上機進行高通量測序。測序平臺為NovaSeq 6000[8]。得到高通量測序結果后,去除原始序列中的Barcode和引物序列,再拼接各樣本序列獲得Raw Tags,對其進行質量控制和過濾處理,去除不合格序列,用UCHIME軟件識別并去除嵌合序列,最終獲得有效數據[9]。選用Greengenes數據庫(Release 13.8,http://greengenes.secondgenome.com/)進行注釋。
1.3.3 ASV聚類及物種組成分析
用Uparse軟件對全部Effective Tags做ASVs聚類,參照相似性為100%,出現頻數最高的序列作為代表序列,利用分類數據庫分析其物種注釋,得到從門至屬的分類學信息,在門和屬水平上統計樣品的物種組成。
1.3.4 潛在益生性乳酸菌的篩選
對水開菲爾和水開菲爾粒取樣后進行梯度稀釋,取 10-5、10-6、10-7、10-8四個梯度各 1 mL用MRS固體培養基進行傾注,37 ℃培養48~72 h,挑取微白色、呈圓形或梭形的菌落,劃線接種到MRS固體培養基上,37 ℃培養 48~72 h,劃線2~3次,觀察其個體形態,直至確定是單菌落后編號[10]。
1.3.4.1 初篩 編號菌株進行 H2O2酶及革蘭氏染色實驗,初步篩選出H2O2酶陰性及革蘭氏染色陽性的菌株,對符合上述條件的菌株進行復篩。
1.3.4.2 復篩 以菌株的耐酸性,膽鹽耐受力作為菌株的潛在益生性篩選指標[11-13]。將初篩所得菌株接種至 MRS肉湯培養基中,37 ℃培養24 h活化菌株,用于接種耐酸性和膽鹽耐受力實驗。
耐酸性:活化初篩所得菌株,用鹽酸調節MRS肉湯培養基的pH值至3.0,4.0,5.0,以pH值為6.0的MRS肉湯培養基為對照組,分裝于各試管中。接種5%(v/v)菌種至上述試管中,37 ℃培養3 h。以未接種的MRS肉湯培養基為參比,在波長 600 nm處利用紫外分光光度計測定不同培養基中的 OD值,每種樣品做三次平行實驗并取其平均值。按照下列公式計算菌株的耐酸性:菌株的耐酸性存活率(%)=(實驗組的OD600 nm值/對照組的OD600 nm值)*100%。上述步驟中耐酸性好的菌株進行膽鹽耐受力的測定。
膽鹽耐受力:參考 Dunne[15]等的方法,配置含0%和0.3%(m/v)牛膽鹽的兩種MRS液體培養基,分裝于各試管。接種 5%(v/v)菌種至上述試管,無牛膽鹽為對照組,37 ℃培養24 h。在波長 600 nm處利用紫外分光光度計測定不同培養基中的OD值,每種樣品做三次平行實驗并取其平均值。按照下列公式計算菌株的膽鹽耐受力:菌株的膽鹽耐受力(%)=(實驗組的OD600 nm值/對照組的OD600 nm值)*100%
1.3.5 個體、群體形態觀察
對復篩所得菌株進行革蘭氏染色,顯微鏡下觀察菌體形態,記錄結果。
1.3.6 生理生化實驗
參照《伯杰細菌鑒定手冊》中相關的乳酸菌實驗方法[16]
1.3.7 菌種的16SrDNA鑒定
主要過程包括:提取菌種基因組DNA,PCR擴增16SrDNA序列,擴增引物為 27F及1492R通用引物。用1%瓊脂糖凝膠電泳檢測擴增及純化后 PCR產物的 DNA序列。測序所得菌株的16SrDNA序列在NCBI的GeneBank中進行比對,得到相似度最高的菌株序列[17],通過MEGA-X軟件繪制系統發育樹[18]。
1.4 數據分析
數據采用SPSS 22.0版本,包括ANOVA單因素方差分析、LSD以及SNK檢驗,結果用x± sem表示。使用Origin繪制圖形。
2 結果與分析
2.1 水開菲爾和水開菲爾粒中菌落總數
水開菲爾和水開菲爾粒細菌計數結果見表1。水開菲爾細菌總數為(7.32±0.01 lg) cfu/mL,乳酸菌總數為(7.08±0.02 lg) cfu/mL;水開菲爾粒細菌總數為(8.90±0.05 lg) cfu/mL,乳酸菌總數為(8.69±0.08 lg) cfu/mL。表明在水開菲爾和水開菲爾粒細菌區系中,乳酸菌為兩種樣品的優勢菌,有利于后續乳酸菌篩選實驗的進行。接下來結合高通量測序分析水開菲爾和水開菲爾粒的細菌區系。
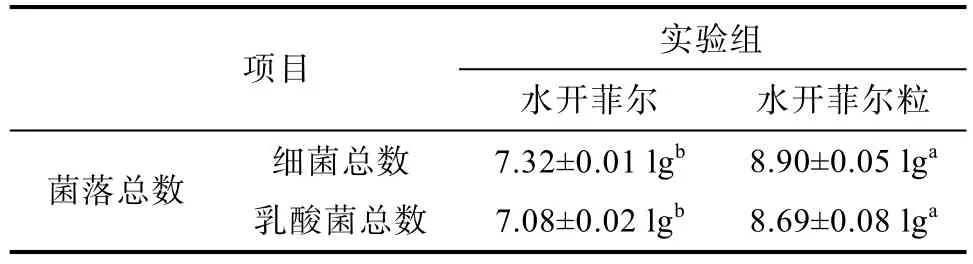
表1 水開菲爾和水開菲爾粒細菌計數結果Table 1 Results of bacterial counting of water kefir and water kefir granules cfu/mL
2.2 高通量測序技術分析水開菲爾和水開菲爾粒細菌區系及優勢菌屬
2.2.1 ASVs聚類
高通量測序分析水開菲爾和水開菲爾粒細菌區系,結果分別獲得60 151和64 256條有效序列。有效序列按100%一致性進行ASVs聚類,出現頻次多的序列為代表性序列,并注釋其物種。其花瓣圖如圖1所示,結果共得到438個ASVs,水開菲爾中含 168個 ASVs,水開菲爾粒中含 270個ASVs,其中兩組共有的 ASVs數為 93個,兩組特有的ASVs分別為75和177個。
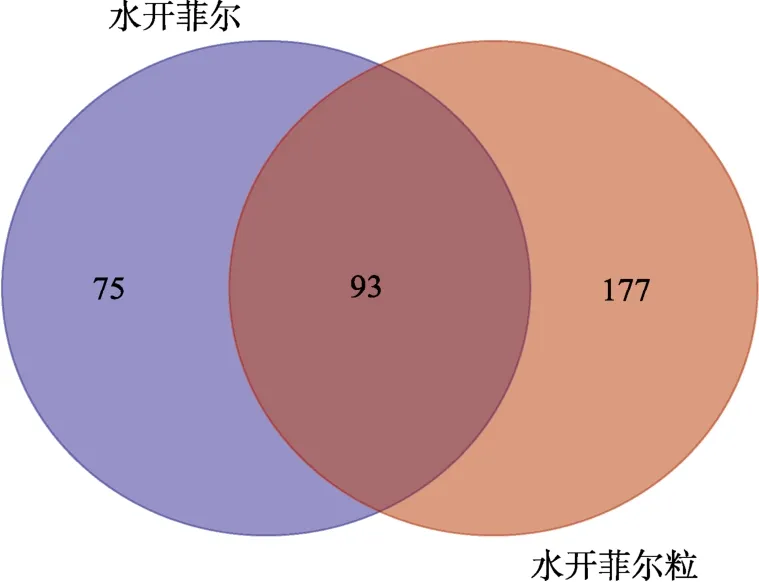
圖1 水開菲爾和水開菲爾粒的ASVs聚類Fig.1 ASVs clustering of water kefir and water kefir granules
2.2.2 細菌α多樣性分析
兩種樣品多樣性指數分析見表2。如表所示,水開菲爾和水開菲爾粒的覆蓋率均為0.999,表明測序深度能夠代表樣本的真實組成[19]。水開菲爾粒的Chao1、ACE、Shannon、Simpson指數顯著(P<0.05)高于水開菲爾粒。上述結果表明,發酵劑水開菲爾粒的細菌豐富度及多樣性更高。
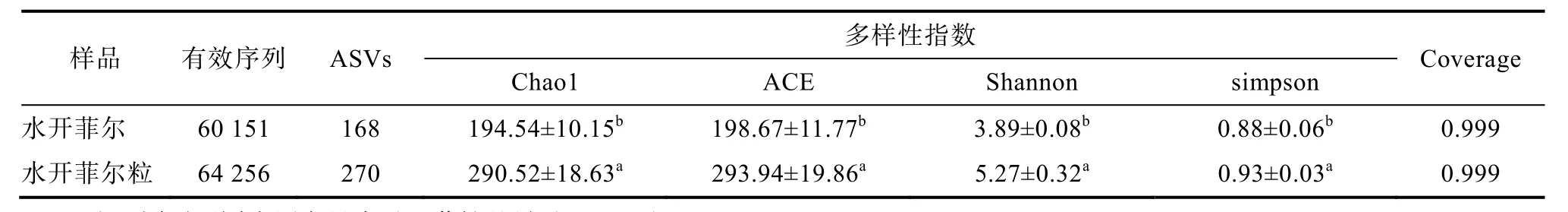
表2 兩種樣品多樣性指數分析Table 2 Diversity index analysis of two samples
2.2.3 細菌的組成分布
基于門水平的細菌結構見圖2,在門水平上,水開菲爾中厚壁菌門(79.58%)和變形菌門(19.88%)占總細菌的99.46%;水開菲爾粒中厚壁菌門(86.67%)和變形菌門(13.32%)占總細菌的99.99%。結果表明,水開菲爾與水開菲爾粒細菌區系相似,優勢菌門均為厚壁菌門和變形菌門,其中厚壁菌門是最豐富的門,水開菲爾粒中厚壁菌門的含量高于水開菲爾。
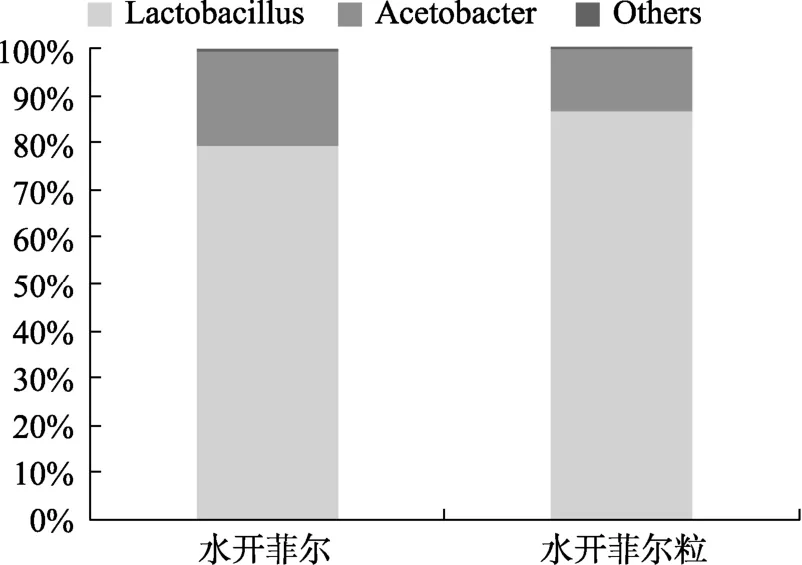
圖2 基于門水平的細菌結構Fig.2 Bacterial structure based on gate level
兩種樣品的屬水平結構組成見圖3,水開菲爾中優勢菌屬是乳桿菌屬和醋桿菌屬,相對含量分別為79.42%、16.38%,上述兩個屬共占細菌群體的95.80%;水開菲爾粒中乳桿菌屬(86.64%)和醋桿菌屬(13.32%)占總細菌的99.96%。結果表明,水開菲爾與水開菲爾粒細菌區系相似,優勢菌屬均為乳桿菌屬和醋桿菌屬,其中乳桿菌屬是最豐富的屬,且其在水開菲爾粒中含量高于水開菲爾。有實驗表明一些有害菌在pH<4.5環境中很難存活,但乳桿菌屬耐酸性較強,能在較低pH值下正常生長繁殖,抑制有害菌生長,占據優勢地位[20]。由此實驗結果可知,樣品中主要存在的菌為本實驗后續篩選所需的乳酸菌,更進一步為本實驗順利進行奠定理論基礎。
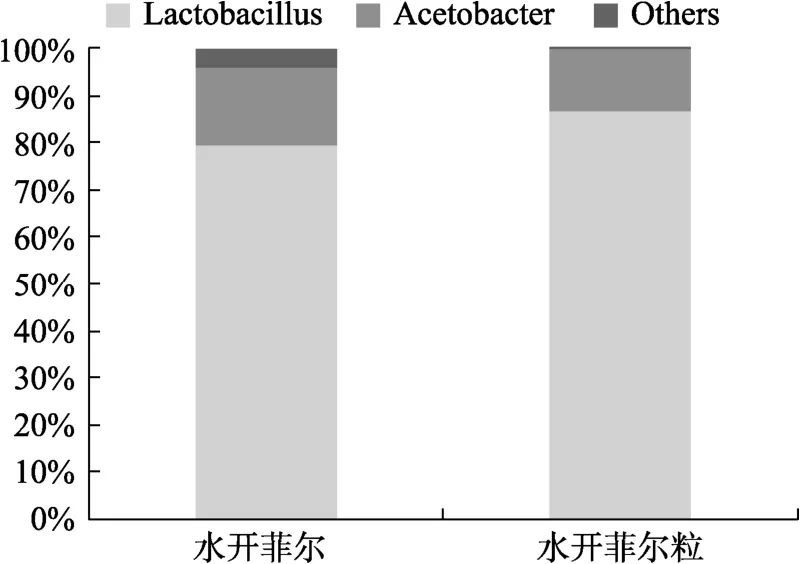
圖3 基于屬水平的細菌結構Fig.3 Bacterial structure based on genus level
2.3 水開菲爾和水開菲爾粒中潛在益生性乳酸菌的篩選
2.3.1 初篩
不同菌株的形態特征如表3所示,從水開菲爾和水開菲爾粒中共篩選出 30株革蘭氏染色呈陽性,過氧化氫酶實驗呈陰性,不含芽孢的菌株。通過油鏡觀察,多數菌株為桿狀,少數為球形或卵圓形。

表3 初步篩選的不同菌株的形態特征Table 3 Morphological characteristics of different strainspreliminarily screened
2.3.2 復篩
初篩所得30株菌接種到MRS肉湯培養基中,37 ℃培養24 h,分別對其培養液進行耐酸性、膽鹽耐受力的測定。
菌株耐酸性見表4,30株菌株菌體密度隨pH值升高而不斷上升,且存在差異。當pH值為3.0時,30株菌株均可以生長,但生長情況不同。王俊國[21]、Wang[22]等研究乳酸菌耐酸性,測定其活菌數時得到同樣結論。一般認為,正常人在進食后,胃液pH值在3.0上下波動,消化時間需3 h,作為益生性乳酸菌需要有耐酸性而且可以在酸性環境中生長[23],所以這 30株菌可以隨食物到達小腸。其中,cc9菌株的耐酸性最好,cc1、cc4、cc5、cc7、cc8、cc9、cc10、cc12、cc15、cc16、cc17、cc22、cc25、cc26、cc27的菌體密度均比較高,按大小依次排列。上述結果說明這15株菌株有很好的耐酸性,可以很好的存活在酸度低的環境中,對上述15株菌株做進一步的篩選。

表4 不同菌株的耐酸性Table 4 Acid tolerance of different strains
菌株膽鹽耐受性:不同菌株的膽鹽耐受能力存在差異[24-25],一般情況下,體內小腸膽鹽濃度范圍是0.03%~0.3%[23]。因此,作為益生性乳酸菌,必須能在0.3%膽鹽濃度環境中存活并生長。菌株的膽鹽耐受性見表5,同樣培養24 h,15株菌的菌體密度在無膽鹽培養基中很高,在含0.3%膽鹽培養基中均下降。其中,cc1膽鹽耐受性最強,為35.68%,高于郭均[23]等從水開菲爾中分離出的益生性乳酸桿菌,cc1>cc9>cc5>cc22>cc12>cc4>cc8,這7株菌株膽鹽耐受性好,能夠耐受消化道的高膽鹽環境,順利通過小腸到達大腸。
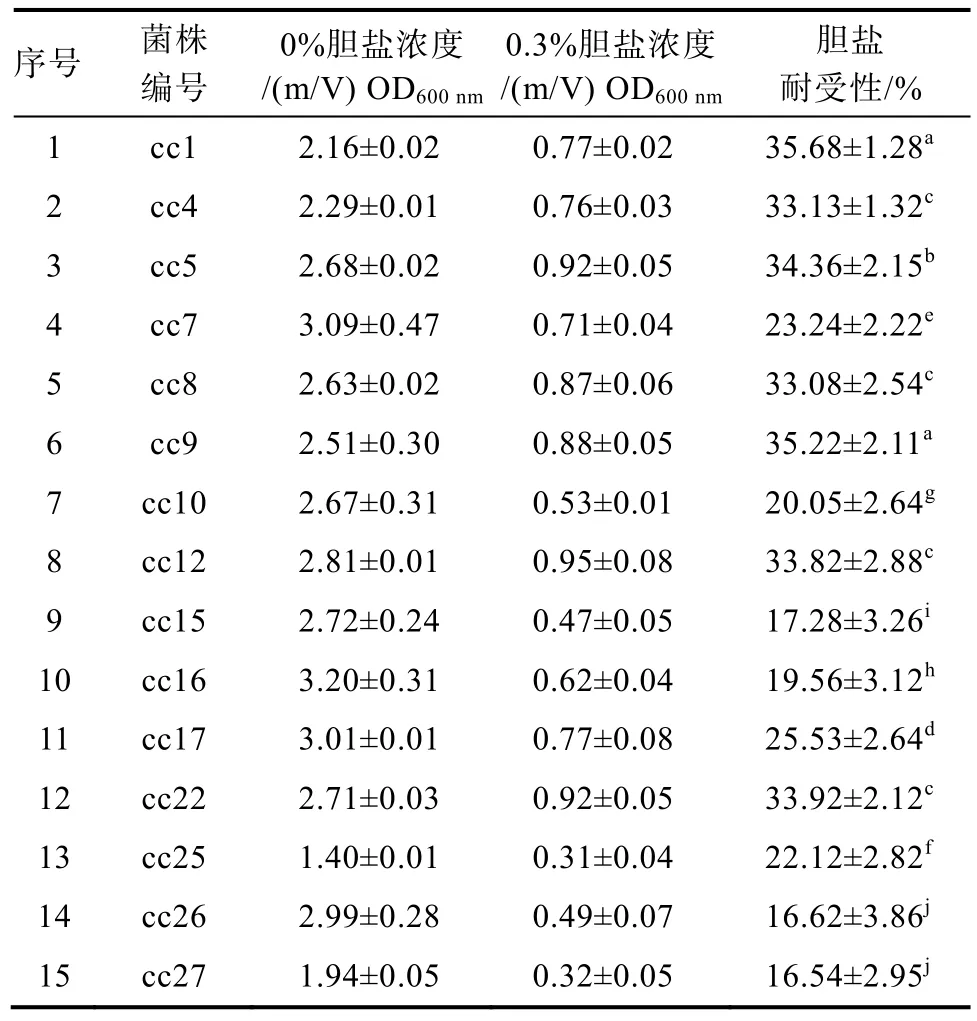
表5 不同菌株的膽鹽耐受性Table 5 Bile salt tolerance of different strains
2.4 菌株的生理生化特征
2.4.1 個體、群體形態特征
通過油鏡觀察,7株菌株形態一致,呈桿狀,菌落大小為1~3 mm,形態呈乳白色、圓形、邊緣整齊、表面光滑,細胞分裂后排列方式為鏈狀,革蘭氏染色呈陽性,不含芽孢。
2.4.2 生理生化檢測
生理生化結果見表6,其結果表明,這 7株菌不產吲哚、硫化氫,接觸酶實驗為陰性,硝酸鹽實驗呈陽性,不運動,具有一定耐酸耐鹽性,將其初步歸屬為乳桿菌屬。
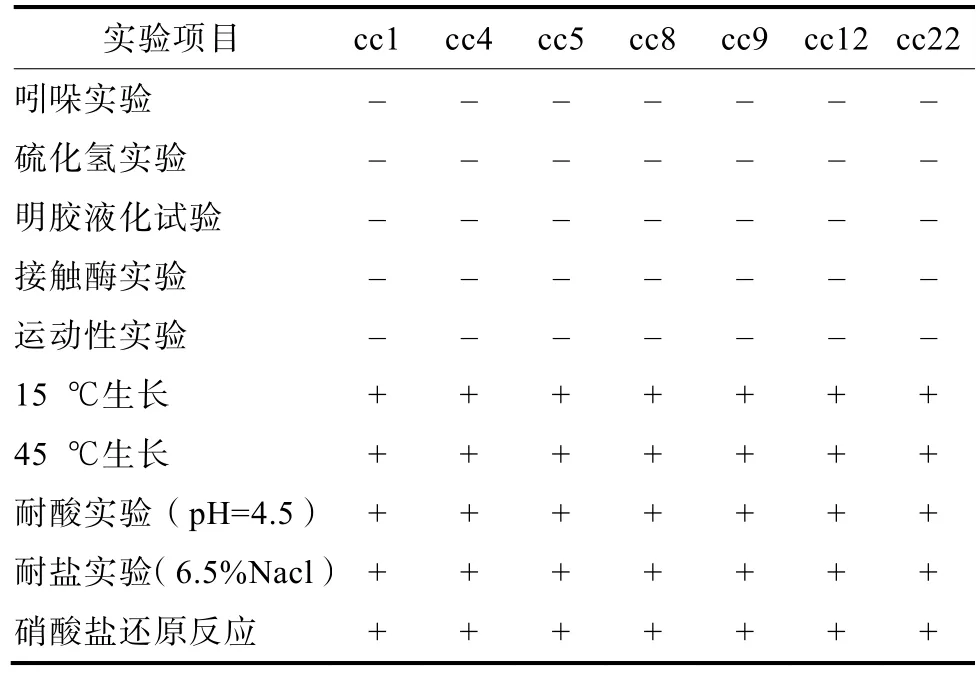
表6 菌株生理生化特性Table 6 Physiological and biochemical characteristics of strains
2.5 菌株的16SrDNA鑒定
篩選出7株乳酸菌cc1,cc4,cc5,cc8,cc9,cc12,cc22,其16SrDNA序列長度分別為1 454 bp,1 468 bp,1 457 bp,1 467 bp,1 467 bp,1 450 bp,1 439 bp。將其提交到Genbank經BLAST比對,系統發育樹結果見圖 4,cc1的親緣性與Lactobacillus paracasei(MT613527.1)最接近,cc4的親緣性與LimosiLactobacillus fermentum(MW405853.1)最接近,cc5的親緣性與Lactobacillus harbinensis(MW405854.1)最接近,cc8的親緣性與Lactobacillus fermentum(EU419595.1)最接近,cc9的親緣性與Lactobacillus fermentum(MT611890.1)最接近,cc12的親緣性與Lactobacillus satsumensis(MW405857.1)最接近,cc22的親緣性與Lactobacillus fermentum(MT641196.1)最接近,相似度均>99%。結合個體和群體形態、生理生化實驗及16S rDNA的分子測序結果,初步鑒定cc4、cc8、cc9、cc22為發酵乳桿菌,cc1為副干酪乳桿菌、cc5為哈爾濱乳桿菌、cc12為紅條乳桿菌,GenBank號分別為MW405853、MW405855、MW405856、MW405858、MW405852、MW405854、MW405857,分別命名為Lactobacillus fermentumcc4、Lactobacillus fermentumcc8、Lactobacillus fermentumcc9、Lactobacillus fermentumcc22、Lactobacillus paracaseicc1、Lactobacillus harbinensiscc5、Lactobacillus satsumensiscc12。
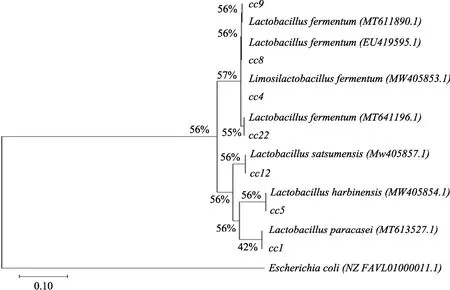
圖4 菌株系統發育樹Fig.4 Phylogenetic tree of strains
3 結論
采用高通量測序技術結合平板菌落計數法,檢測分析了水開菲爾和其發酵劑水開菲爾粒兩種樣品的細菌組成中,優勢門均為厚壁菌門和變形菌門,優勢屬均為乳桿菌屬和醋桿菌屬。為尋找具有潛在益生性的菌株,以耐酸性和膽鹽耐受力為指標,從自然發酵水開菲爾和水開菲爾粒中篩選出 7株乳酸菌。經鑒定,4株為發酵乳桿菌:L. fermentumcc4、L. fermentumcc8、L. fermentumcc9、L. fermentumcc22,1株為副干酪乳桿菌:L. paracaseicc1,1株為哈爾濱乳桿菌:L.harbinensiscc5,1株為紅條乳桿菌:L. satsumensiscc12。這些菌株可作為水開菲爾工業化發酵生產的備選菌株。

