山東省栽培靈芝屬菌株的分子特征
蘭玉菲 孔 怡* 于清偉 馬為勇 崔 曉
(1泰安市農業科學院,山東泰安271000;2泰安市寧陽縣華豐鎮農技站,山東泰安271413)
靈芝屬Ganoderma真菌是重要的藥用真菌,隸屬于真菌界(Fungi),擔子菌門(Basidiomycota),傘菌亞門(Agaricomycetes),多孔菌目(Polyporales),多孔菌科(Polyporaceae),在我國已有悠久的應用歷史[1]。
近年來山東省靈芝生產發展迅速,種質資源也在不斷豐富,但目前對靈芝屬種質資源的遺傳背景和遺傳多樣性缺少系統研究。因此,筆者對山東省栽培靈芝菌株的遺傳多樣性進行研究,明確了27個栽培靈芝屬菌株的分類地位,為靈芝屬種質資源保護、遺傳改良和新品種選育提供理論依據。
1 材料與方法
1.1 供試靈芝菌株
供試的27個靈芝菌株中ZL81來自四川中醫藥科學院,紫芝、園芝1號來自壽光食用菌研究所,其他24個菌株均來自泰安市農業科學院。具體編號見表1。

表1 試驗靈芝屬菌株及編號
1.2 主要試劑與儀器
真菌總DNA提取試劑盒(E.Z.N.A.?Fungal DNA Kit)、2×EasyTaq PCR SuperMix等購自北京全式金生物技術有限公司;常規試劑為進口分裝或國產分析純。My Cycle PCR儀,美國伯樂儀器公司。
1.3 供試培養基
PDA加富培養基:配方參照文獻[2]。
1.4 菌絲體制備
切取保存在PDA加富斜面培養基上的菌絲純培養5 mm2,接種于PDA加富平板培養基中央,25℃暗培養5~7 d,待菌絲接近生長至平板邊緣后,刮取菌絲體用于基因組DNA的提取。
1.5 DNA的提取
利用DNA提取試劑盒提取真菌總DNA。具體步驟參照文獻[3]。提取的總DNA于-20℃保存備用。
1.6 ITS擴增
參照文獻[4]用于真菌ITS區域擴增的通用引物ITS1/ITS4進行PCR擴增。PCR反應體系和PCR反應程序參照文獻[3]。

表2 PCR擴增ITS所用引物的核苷酸序列
1.7 PCR產物測序
PCR擴增結束后,取4μL反應液進行瓊脂糖凝膠電泳,將有目的條帶的PCR產物送山東省農業科學院生物技術研究中心測序。
1.8 構建系統發育樹
將所得DNA序列輸入GenBank進行Blast檢索,采用Clustalx1.81、DNASTAR、DNAMAN和MEGA 6.06軟件對所得到的核苷酸序列與GenBank中收錄分離物的相應序列進行比較和分析,并構建系統進化樹進行遺傳距離分析。
2 結果與分析
2.1 ITS擴增
以提取的27個靈芝菌株總DNA為模板,用真菌ITS區域擴增的通用引物ITS1/ITS4對其進行PCR擴增,獲得27個靈芝屬菌株的ITS序列,長度約為600 bp的目的片段,與預期片段大小一致。

圖1 部分靈芝菌株核糖體ITS區PCR擴增結果
2.2 ITS序列分析
測序獲得27個菌株的ITS序列片段,測得的菌株序列提交到GenBank并獲得登錄號(表3)。根據GenBank中已有靈芝屬菌株的ITS序列資料,確定ITS序列的ITS1、5.8S和ITS2的序列范圍。供試靈芝屬菌株的ITS序列的長度變異較大,G.lingzhisp.nov.ITS序列長度為542 bp,G.applanatumITS序列長度為560 bp,G.sienseITS序列長度為555 bp。

表3 27個靈芝屬菌株ITS序列的登錄號
2.3 聚類分析
根據MEGA6.06軟件中的Neighbour-Joining methods,以樟芝(Autrodia comphorata)作為外類群,對27個測得的和26個來自GenBank的ITS序列進行聚類分析,建立基于ITS序列的系統進化樹(圖2)。結果表明,山東省栽培靈芝菌株分布于3個聚類組(Ⅰ組、Ⅳ組和Ⅴ組)。Ⅰ組為G.lingzhisp.nov.,包括35個菌株,均來自東亞地區(其中2個菌株來自日本,其余菌株分別來自中國的遼寧省、河南省、安徽省、湖北省、廣東省、浙江省、四川省、山東省、福建省和江蘇省),23個供試菌株均聚于該組,為山東省主要栽培菌株,組內各菌株間核苷酸一致率在99.26%~100.00%。Ⅱ組為G.multipileum,包括3個菌株,均來自亞洲熱帶地區。Ⅲ組為G.lucidum,包括4個菌株,來自歐洲大陸。Ⅳ組為G.applanatum,包括5個菌株,供試菌株PCSS、FCSS和YXSS聚于該組,該組內菌株核苷酸一致率非常高,PCSS、YXSS與GenBank中的2個菌株(DQ425009、DQ424996)核苷酸一致率達100%,FCSS與其他4個菌株的核苷酸一致率為99.82%;Ⅴ組為G.siense,包括3個菌株,供試ZZ菌株聚于該組,與來自上海市(DQ424982)、福建省(HQ235633)的2個菌株核苷酸一致率分別為99.28%、99.64%。結果表明,利用ITS區能夠有效準確地將山東省栽培靈芝菌株分成G.lingzhisp.nov.、G.applanatum和G.siense三大類群,而與來自歐洲的G.lucidum親緣關系較遠,明確了山東省栽培靈芝菌株的分類地位。
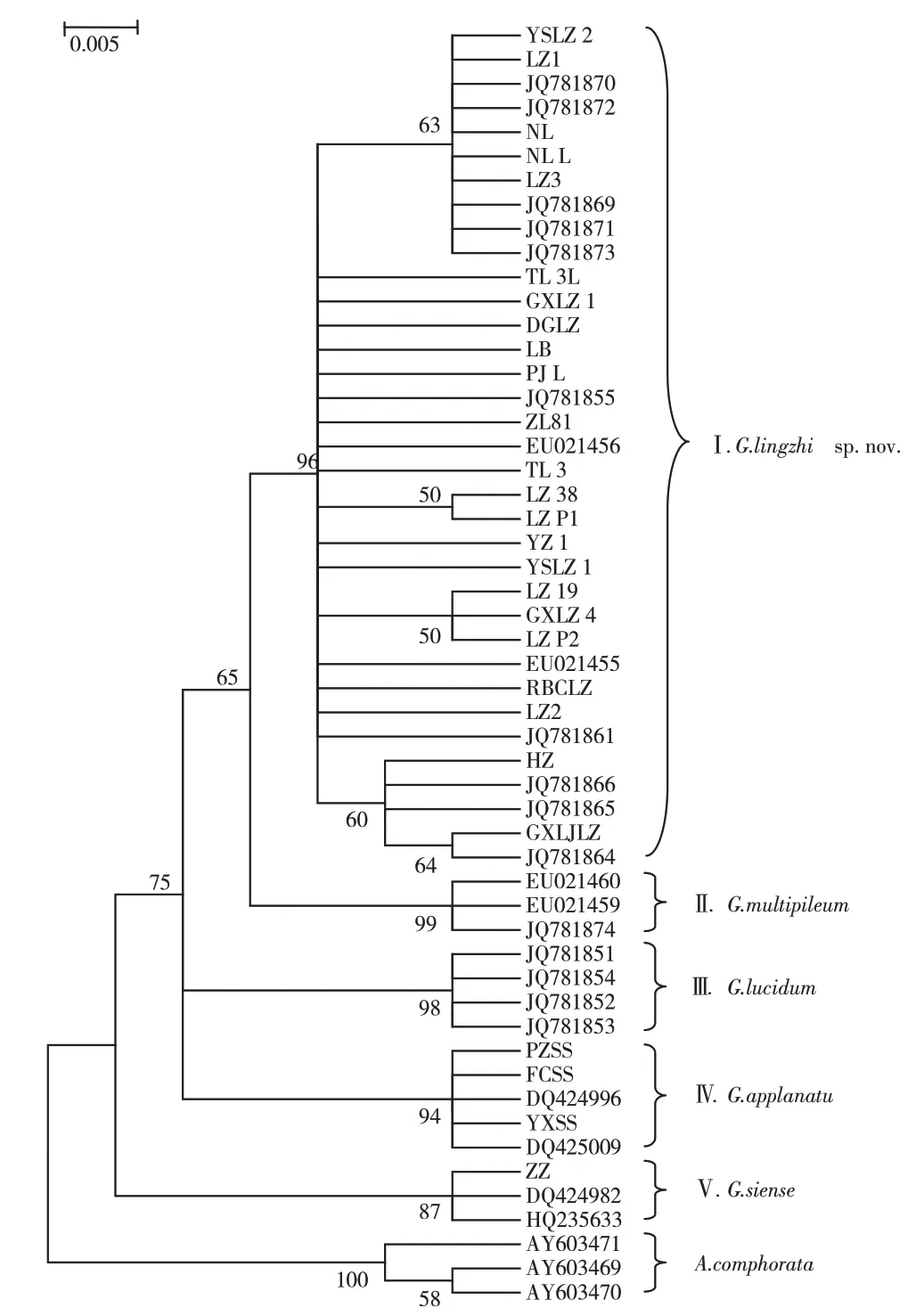
圖2 基于ITS序列構建的NJ系統發育樹
2.4 基于DNA序列的種間種內遺傳距離分析
根據MEGA6.06軟件構建的系統進化樹聚類情況,對所有菌株進行分組,計算種間種內遺傳距離。由表4可以看出,靈芝屬5個不同種種間遺傳距離的變化在0.039~0.101,表明他們之間的遺傳關系比較遠;而種內遺傳距離的變化在0.000~0.003,說明種內不同菌株間的遺傳距離都比較小,表明種內菌株之間的遺傳關系較近,并且遺傳進化過程中遺傳信息流動可能發生較頻繁。

表4 基于DNA序列的種間種內遺傳距離分析
3 小結
靈芝屬Ganoderma真菌為中國傳統的藥用真菌,具有扶正固本,提高機體免疫力等多種功能。靈芝因其突出的保健作用、不斷發現新藥用功效而成為科學家的研究熱點。然而靈芝屬的分類比較混亂,過去一直將來自東亞的靈芝“Lingzhi”歸為G.lucidum(起源于歐洲)。2012年,Cao等利用rDNA nuc-ITS,結合mt-SSU,RPB1,RPB2和TEF1-α對來自世界不同地區的靈芝菌株進行系統發育樹分析,明確“Lingzhi”的分類地位,并提出來自東亞的“Lingzhi”屬于一個新種G.lingzhi,同時進行形態學鑒別[5]。
筆者對獲得的27個ITS序列、26個來自GenBank的ITS序列進行聚類分析,建立基于ITS序列系統進化樹,結果表明,23個供試菌株均聚于G.lingzhi,其余4個菌株聚于G.applanatum和G.siense,結果與Cao等(2012)一致。
研究從DNA水平上對山東省栽培靈芝菌株的遺傳多樣性進行研究,明確了靈芝屬種質資源的親緣關系和分子特征,為靈芝屬種質資源遺傳改良、新品種的選育奠定分子基礎,同時為特異種質的鑒定提供了有效方法。
——山東省濟寧市老年大學之歌

