HOXD基因簇內一系列CTCF位點反轉揭示絕緣子功能
何象龍,李金環,吳強
研究報告
基因簇內一系列CTCF位點反轉揭示絕緣子功能
何象龍,李金環,吳強
上海交通大學系統生物醫學研究院比較生物醫學研究中心,系統生物醫學教育部重點實驗室,上海 200240
CTCF (CCCTC-binding factor)是一種重要的染色質架構蛋白,其與絕緣子的方向性結合在哺乳動物基因組三維空間結構形成和維持中起著至關重要的作用。正向–反向相對方向的CTCF結合位點(簡稱CTCF位點)可以在染色質黏連蛋白(cohesin)的協助下,形成染色質環,介導遠距離DNA元件之間的相互作用;而在染色質拓撲結構域邊界區域的CTCF位點呈現反向–正向相背方向分布,發揮絕緣子的功能。為進一步研究CTCF介導染色質環的形成與其絕緣功能之間的關系,本研究采用DNA片段編輯方法通過設計成對sgRNA (dual sgRNA)構建了一系列基因簇區域CTCF位點反轉的單細胞克隆。定量高分辨率染色質構象捕獲實驗顯示邊界區域CTCF位點反轉會改變原有的染色質環方向,通過環擠出模型(loop extrusion)形成新的染色質環,引起染色質拓撲結構域邊界漂移至新形成的一對反向–正向CTCF位點處。此外,串聯排列的CTCF位點可以通過阻礙反方向滲透的黏連蛋白繼續滑動發揮絕緣子的功能。RNA-seq實驗發現CTCF位點反轉引起的局部基因組空間結構變化會進一步影響基因的表達。上述研究表明相鄰兩個染色質拓撲結構域邊界區域的反向-正向CTCF位點可以通過與各自所在拓撲結構域內相向的CTCF位點形成染色質環,阻礙黏連蛋白滑動,該發現為進一步研究CTCF的絕緣功能和其對基因組拓撲結構的影響提供了參考。
CTCF;染色質環;;絕緣子;CTCF位點反轉
大約2 m長的人類基因組通過多次盤曲折疊,最終被包裹在直徑約5 μm的細胞核中,形成了復雜的染色質三維空間結構[1~3]。特定的染色質空間結構對于DNA復制[4]、基因轉錄調控[5,6]和DNA修復[7,8]等生物學過程有著重要作用。早期通過光學顯微鏡研究發現,有絲分裂間期染色體有規律的分布在細胞核的不同區域,被稱為染色質領地(chromosome territories)[9];隨著染色質構象捕獲(chromosome con-formation capture, 3C)[10]、環狀染色質構象捕獲(circular chromosome conformation capture, 4C)[11]、Hi-C[12]和配對末端標簽測序分析染色質相互作用(chromatin interaction analysis by paired-end tag sequencing, ChIA-PET)[13]等技術的發展,A/B隔室(compartment)、拓撲相關結構域(topologically asso-ciated domain, TAD)和染色質環(chromatin loop)等概念被相繼提出[2,12,14~18],促進人類從多個層次對染色質高級結構的認識。基于Hi-C數據,基因組上平均大小約為1 Mb、內部DNA相互作用緊密的區域,被定義為TAD[13,14]。在人的不同細胞系中,TAD表現出較好的保守性[15]。通常認為在TAD內部,增強子(enhancer)、絕緣子(insulator)與沉默子(silencer)等順式作用元件可以通過CTCF(CCCTC-binding factor)和YY1等染色質架構蛋白介導的染色質環與基因的啟動子(promoter)發生相互作用[5,19~22],來調控基因的表達。
CTCF蛋白是一種參與染色質折疊的重要架構蛋白。CTCF蛋白最早發現于對雞()基因的研究,由于其與DNA結合的序列含有高度保守的CCCTC序列,因而被稱為CCCTC結合因子[23]。CTCF蛋白通過11個串聯的鋅指結構與人類基因組上成千上萬的絕緣子調控元件結合[19,24~27]。在哺乳動物中,CTCF結合位點(CTCF-binding site, CBS)是最主要的絕緣子調控元件,包含4個模塊(module),即module 1~4,其具有方向性,module 1~4的方向是正向,module 4~1的方向則為反向[5]。CTCF在黏連蛋白的協助下,通過環擠出模型介導遠距離的DNA相互作用[16,18]。在TAD邊界處分布著大量反向-正向的CBS,通過阻斷cohesin的滑動,起到絕緣子的功能[2,5,28~30]。刪除CBS導致CTCF無法結合,會破壞原有DNA之間的染色質環,引起局部基因組構象的改變[28,31~33]。反之,插入異位CBS會形成新的染色質環,同時作為絕緣子阻礙原有染色質環的形成[34,35]。在基因簇的增強子和啟動子之間插入正向或者反向的CBS,都可以起到絕緣子的作用,顯示絕緣子沒有方向性[34]。在原鈣粘蛋白(protocadherin,)、免疫球蛋白 (immunoglobulin,)、等基因簇存在著成串同向的CBS,這些CBS均有機會形成染色質環[5,36,37],顯示正向-反向CBS對cohesin的阻礙作用是有滲透性的,這可能是由于CTCF與DNA的動態結合[34,38]。然而,當前對于成串CBS絕緣子功能的研究主要側重于啟動子與增強子的相互作用,在染色質拓撲結構域邊界處的成串CBS的絕緣子功能還缺少系統性的研究。
基因簇是控制四肢發育時空順序的重要基因簇,被廣泛應用于對基因組三維空間結構的研究[19,39]。基因簇區域存在一系列的CBS,與兩側調控區域內的CBS通過環擠壓模型分別形成兩個TAD[19,39~42]。基因有序的表達受到兩側TAD內增強子簇的調控,其表達分為兩個階段;在四肢發育早期,3′端粒側TAD (telomeric domain, T-DOM)內由CS38-41 (conserved sequence elements 38-41)和CS65 (conserved sequence element 65)組成的下游增強子簇或超級增強子(downstream enhancer cluster or super-enhancer)共同調節的表達,并在四肢近端維持這種表達模式;在肢端發育后期,5′中心粒側TAD (centromeric domain, C-DOM)內由Island 1-5、GCR和Prox組成的上游增強子簇或超級增強子(downstream enhancer cluster or super-enhancer)協同調節四肢遠端的表達[19,42,43]。基因簇區域大范圍的片段反轉破壞原來的基因組三維結構,影響基因簇的正常表達[39,41,44],但這些大范圍的片段反轉主要針對增強子調控元件,針對絕緣子CTCF位點的DNA片段反轉研究較少。
成簇規律間隔短回文重復系統(clustered regularly interspaced short palindromic repeats/CRISPR-asso-ciated nuclease 9, CRISPR/Cas9)源于古菌和細菌中的一種適應性免疫系統,近年來被廣泛應用于DNA片段反轉、DNA片段刪除和染色體異位等編輯實驗[22,45~52]。HEK293T細胞系是一種常用的模式細胞系,常被用于對染色質三維空間結構的研究[5,53,54]。本實驗室之前通過對HEK293T細胞系以及HEC-1B等細胞系中CBS位點反轉的研究發現,CBS方向性是哺乳動物細胞中普遍存在的規律[5]。本研究利用CRISPR DNA片段編輯技術在HEK293T細胞系中對基因簇所在區域中串聯排列的CBS元件進行一系列組合性的反轉,利用定量高分辨率染色質構象捕獲(quantitative high-resolution chromosome co-nforma-tion capture copy, QHR-4C)技術[34]研究CBS反轉后染色質空間構象的變化,發現CBS反轉會改變該CBS原有的遠距離DNA相互作用,引起染色質拓撲結構域邊界漂移至新形成的一對反向–正向的CBS處;同時,相鄰兩個染色質拓撲結構域邊界區域的反向-正向CBS可以通過與各自所在拓撲結構域內的相向CBS形成染色質環,發揮絕緣子功能,這種絕緣子的作用有助于穩定基因組三維空間構象。
1 材料與方法
1.1 實驗材料
人胚腎細胞系HEK293T細胞從中國科學院細胞庫購買;pcDNA3.1-Cas9-WT質粒由北京大學席建忠教授惠贈;pGL3-U6-sgRNA質粒由上海科技大學黃行許教授惠贈;DMEM培養基從美國Hyclone公司購買;胎牛血清購自上海ExCell公司;Ⅰ內切酶、Ⅱ內切酶、T4 DNA連接酶、蛋白酶K和ChIP-Seq建庫試劑盒均購自美國NEB公司;DNA凝膠回收試劑盒和質粒DNA小量試劑盒購自美國AXYGEN公司;嘌呤霉素、NP-40、Triton X-100、DMSO、Tris base、SDS、醋酸鈉購自美國SIGMA公司;Lipofectamine 3000、RNaseA、糖原、16%甲醛溶液購自美國Thermo Fisher Scientific公司;胰酶和青鏈霉素購自美國Gibco公司;Streptavidin Magnetic Beads購自美國Invitrogen公司;RNA-seq試劑盒和Phanta酶購自南京諾維贊生物科技有限公司;TRIzol購自美國Ambion公司;AMPure XP beads購自美國Beckman Coulter公司;CTCF抗體從英國Abcam公司購買;三氯甲烷、硼酸和乙醇購自上海滬試實驗室器材股份有限公司;異丙醇和氯化鈉購自上海凌峰化學試劑有限公司;引物均由上海生物工程股份有限公司和北京擎科新業生物技術有限公司提供。
1.2 HOXD基因簇TAD分析
從SRA數據庫下載HEK293細胞的Hi-C數據[54]:SRR710075、SRR710076、SRR710077,以hg19為參考基因組,利用HiC-Pro[55]軟件進行分析,獲得標準化矩陣;截取chr2:175,760,000~178,200,000范圍的矩陣,利用TADtool繪制熱圖[56]。
1.3 CTCF結合序列分析
分析CTCF蛋白ChIP-seq數據,確定基因簇區域CBS位點分布情況;利用MEME[57]軟件分析獲得CTCF的結合基序(motif),以此基序作為參考,使用FIMO[57]軟件查找基因簇區域CTCF結合序列,并確定CBS方向。
1.4 細胞培養
HEK293T細胞用89% DMEM、10%胎牛血清和1%青鏈霉素雙抗配制的完全培養基培養,在5% CO2濃度、37℃恒溫培養箱中培養。
1.5 構建sgRNA質粒
使用I酶將pGL3-U6-sgRNA質粒線性化,并用瓊脂糖凝膠進行分離純化。將購買的sgRNA正反脫氧核苷酸單鏈溶解并稀釋到20 μmol/L (表1),配制引物退火溶液,體系如下:正反單鏈sgRNA各2 μL,2 μL NEB buffer2,14 μL ddH2O,并置于水浴鍋中95℃ 5 min,然后自然冷卻至室溫。帶有懸掛端的雙鏈sgRNA與獲得的線性化載體連接,然后用感受態細胞轉化搖菌。利用質粒小量試劑盒提取質粒,并用桑格測序(Sanger sequencing)確認質粒是否正確連接。
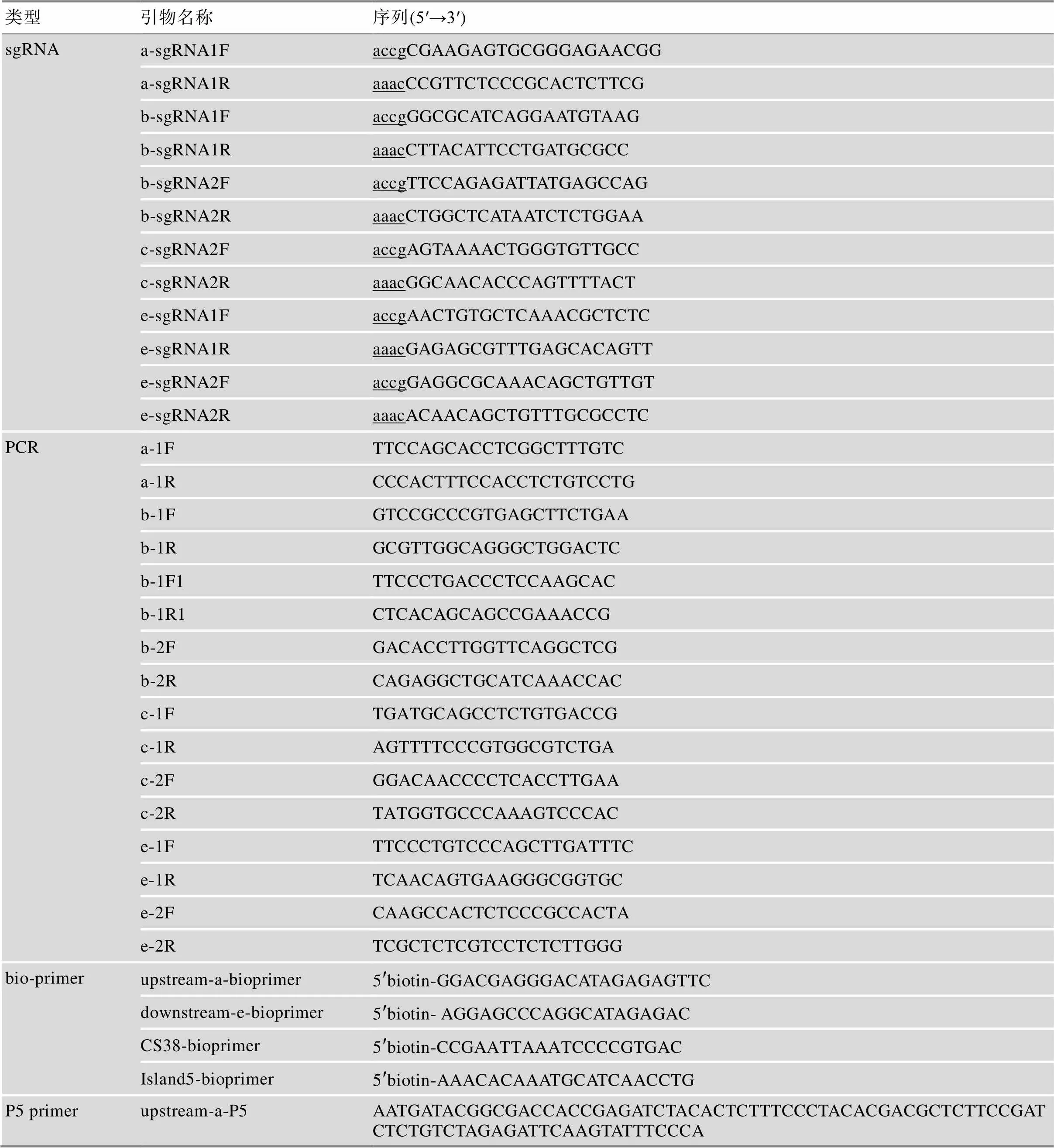
表1 引物序列
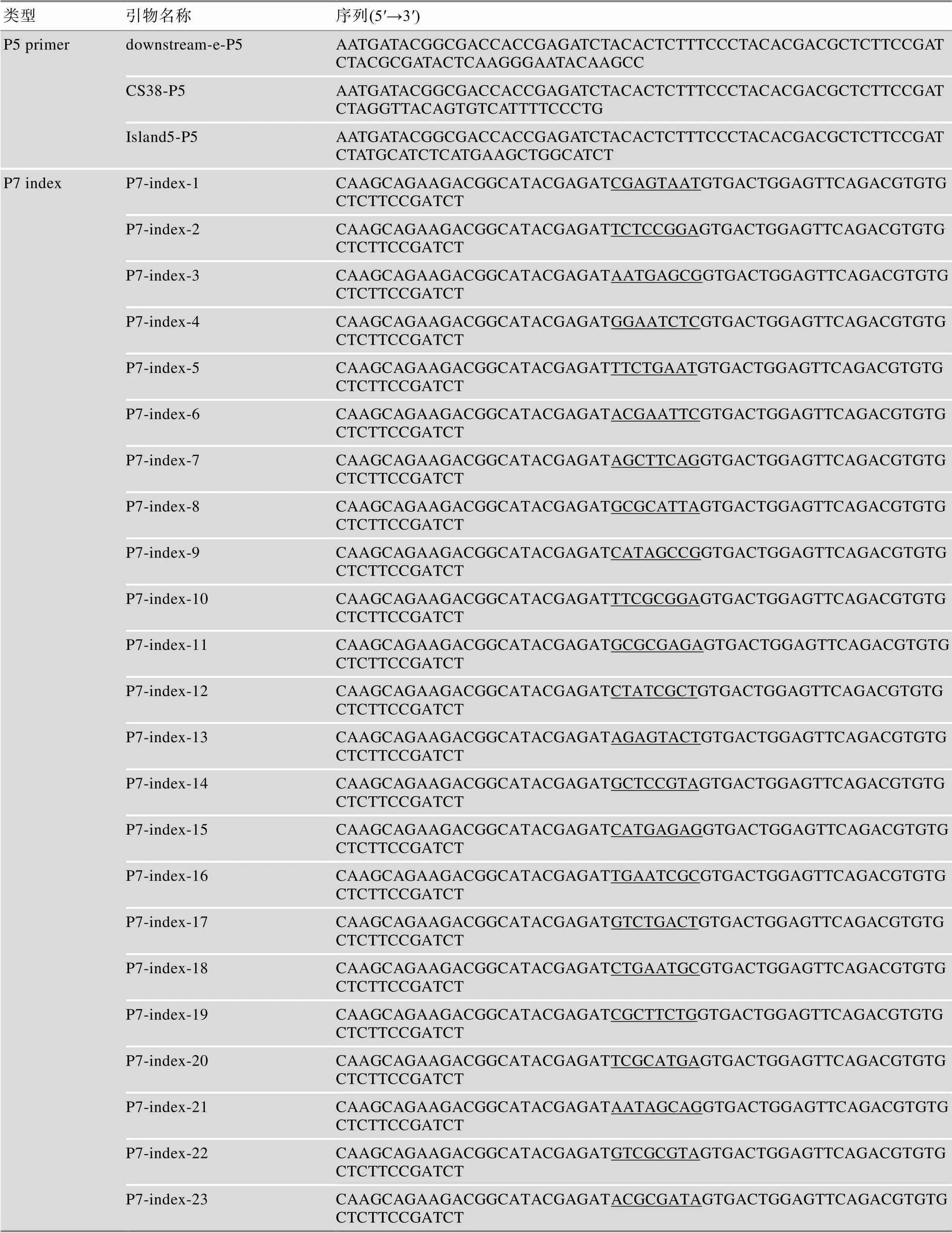
續表

續表
sgRNA中下劃線標示小寫字母部分為構建質粒所需的粘性末端,P7 index中下劃線標示部分為index。
1.6 篩選CTCF位點反轉的單細胞克隆
待到12孔板中HEK293T細胞長到約60%時,利用Lipofectamine 3000試劑將Cas9質粒、sgRNA質粒一起轉染到細胞中。2天后,用含有2 ng/μL嘌呤霉素的DMEM培養基培養4天。換用普通DMEM完全培養基培養2天后,進行單克隆鋪板。
約10天后,用胰酶消化單克隆細胞,取部分細胞使用堿裂法裂解獲得基因組。使用特異性引物進行PCR鑒定,鑒定各單細胞克隆類型,鑒定引物如表1所示。PCR產物經TA克隆后,進行桑格測序,確定單細胞克隆基因型。
1.7 定量高分辨率染色質構象捕獲實驗QHR-4C
QHR-4C實驗方法參考本實驗室之前的研究[34],步驟如下:培養鑒定好的單細胞克隆,收取處于對數生長期的細胞500萬個左右,用4C裂解液(配方為:50 mmol/L Tris-HCl, 150 mmol/L NaCl, 5 mmol/L EDTA, 0.5% NP-40, 1% Triton X-100)裂解細胞兩次。獲得的細胞核沉淀經Ⅱ內切酶過夜酶切后,用T4 DNA連接酶在16℃過夜連接,之后利用超聲將DNA片段打斷為200~1000 bp長度范圍。超聲后的DNA片段利用酚氯仿進行純化,后使用帶有biotin的引物進行線性擴增。擴增產物加上接頭后進行PCR擴增和產物純化,獲得QHR-4C文庫。文庫用Illumina HiSeq測序獲得原始數據,根據index和barcode拆分數據。用fastuniq去重,通過bowtie2比對到hg19參考基因組。比對文件經過samtools排序,用r3Cseq進行計數和標準化,獲得每個酶切片段的RPKM值。每個單細胞克隆株至少做兩個重復實驗,共計99個QHR-4C文庫,每個文庫測序約800萬條序列。所用index見表1。
1.8 RNA-seq實驗
待細胞長到12孔板約80%~90%時,使用TRIzol處理細胞,并提取細胞總RNA。隨后用諾維贊RNA-seq建庫試劑盒進行RNA-seq文庫構建。步驟如下:用beads捕獲mRNA,熱打斷至150~200 bp,使用隨機引物逆轉錄獲得cDNA,加A和加接頭,最后進行PCR擴增。文庫用Illumina HiSeq進行測序,數據經index拆分后,利用trim_galore去除低質量reads,使用STAR比對到hg19參考基因組,利用cufflinks對轉錄本進行計數和標準化,得到每個基因的FPKM值。每個單細胞克隆株做三個重復實驗,共計33個RNA-seq文庫,每個文庫測序約1000萬條序列。
1.9 ChIP-seq實驗
待細胞匯合度達到10 cm培養皿約80%~90%時收取細胞,用甲醛交聯固定,用超聲將基因組DNA打斷為100~1000 bp,孵育protein G凝膠珠。4℃過夜孵育一抗后,再次加入protein G凝膠珠孵育,經洗脫DNA和用酚氯仿純化DNA后,利用DNA建庫試劑盒進行建庫,包括末端修復、末端加A、加接頭和PCR擴增等步驟,獲得ChIP-seq文庫。文庫用Illumina HiSeq進行測序,測序數據經過index拆分獲得雙端測序序列,使用trim_galore進行數據過濾,bowtie2比對到hg19參考基因組。之后用samtools對比對結果進行排序,使用macs2成峰。實驗做兩個重復,每個重復測序約2000萬條序列。
1.10 高通量測序數據信息
該研究的相關結果數據已收錄在國家基因庫生命大數據平臺(CNGBdb)[58]的國家基因庫序列歸檔系統(CNSA)[59],項目編號:CNP0001772。
2 結果與分析
2.1 HOXD基因簇區域CTCF位點序列分析
為了研究TAD邊界處成串CBS的絕緣子作用,本研究選定基因簇為研究對象。Hi-C數據顯示基因簇位于兩個TAD邊界處,CTCF ChIP-seq數據證實基因簇以及兩側的C-DOM和T-DOM分布著大量CTCF結合位點(圖1A)。為了判斷CBS的方向性,對每一個CTCF結合峰進行序列分析(圖1B)。這些CBS基序共包括4個module,其中module 2~4包含最為保守的20 bp核心基序,少部分在核心基序上游含有module 1,正向CBS方向為module 1到module 4 (圖1B)。在module 2~3中最為保守的C20、G25和G28是判斷CBS存在的重要參考,而module 1和位于24位的堿基是否為A則可以用于判斷CBS方向性;這些CBS序列特性也與之前發表文章相符[2,5]。在HEK293T細胞中,基因簇分布著5個反向CBS和2個正向CBS,分別命名為CBS a-e和f-g (圖1A)。在C-DOM中大部分是正向CBS,只有3個反向CBS (圖1B);T-DOM中CBS呈反向串聯排布(圖1B)。
2.2 構建HOXD基因簇一系列CTCF位點反轉的單細胞克隆株
利用CRISPR/Cas9 DNA片段編輯技術,針對預反轉的DNA片段設計一對sgRNA,將Cas9和sgRNA質粒轉染到HEK293T細胞中,Cas9核酸酶在成對sgRNA的介導下對目的DNA片段進行切割。斷裂的雙鏈DNA在細胞DNA修復系統的作用下,可以產生DNA片段原位反轉,反轉后的DNA片段具有兩個不同的接口(上游接口和下游接口)(圖1C)。利用此策略,我們對基因簇內CBS a-e進行組合反轉(圖1D)。共獲得5種CBS反轉單細胞克隆株,分別為e反轉、b-e反轉、a-e反轉、a-c反轉和a-b反轉(圖1D),由于不同sgRNA具有不同的編輯效率,單克隆篩選數相差較大(圖2A),其中a-b反轉共篩選1307個單細胞克隆只獲得了一個預期反轉克隆。最后,e反轉獲得5個純合子克隆株,b-e反轉、a-e反轉、a-c各獲得2個純合子克隆株,a-b反轉獲得1個純合子克隆株(圖2A)。全部單細胞克隆株通過PCR進行鑒定,DNA片段反轉的接口還進行TA克隆和桑格測序確定其具體的基因型,大部分接口為兩種基因型,除了在PAM位點上游3 bp處精確切割后的連接產物外,單堿基插入占據相當的比例(圖2,B~J),推測Cas9核酸酶在PAM位點上游4 bp處切割導致[48,60]。在b-e#127單細胞克隆株的一個等位基因下游接口存在518 bp的堿基刪除,但刪除區域不包括CBS核心基序(圖2G);在a-c#81單細胞克隆株的一個等位基因上游接口存在258 bp的堿基插入,經比對發現插入序列為質粒序列(圖2I),因此這兩個細胞株仍是純合的CBS反轉單細胞克隆株。
2.3 CTCF位點反轉引起染色質拓撲結構域邊界漂移
為了研究CBS的絕緣子作用,對獲得的CBS反轉單細胞克隆株進行QHR-4C實驗。在野生型細胞中,位于T-DOM中的增強子CS38主要與正向CBS f-g有較強的DNA相互作用(圖3A),而位于C-DOM中的增強子Island5主要與反向的CBS a-e具有DNA相互作用(圖3B),與Hi-C結果一致(圖1A)。CBS e反轉后,CBS e與CS38的遠距離DNA相互作用增強(圖3A),顯示反轉后CBS e可以作為絕緣子阻礙CBS f-g滲透的cohesin繼續滑動;與之相反,CBS e與Island5的遠距離DNA相互作用減弱(圖3B)。CBS d和反轉后的CBS e呈反向-正向分布,形成了一個新的染色質拓撲結構域邊界。
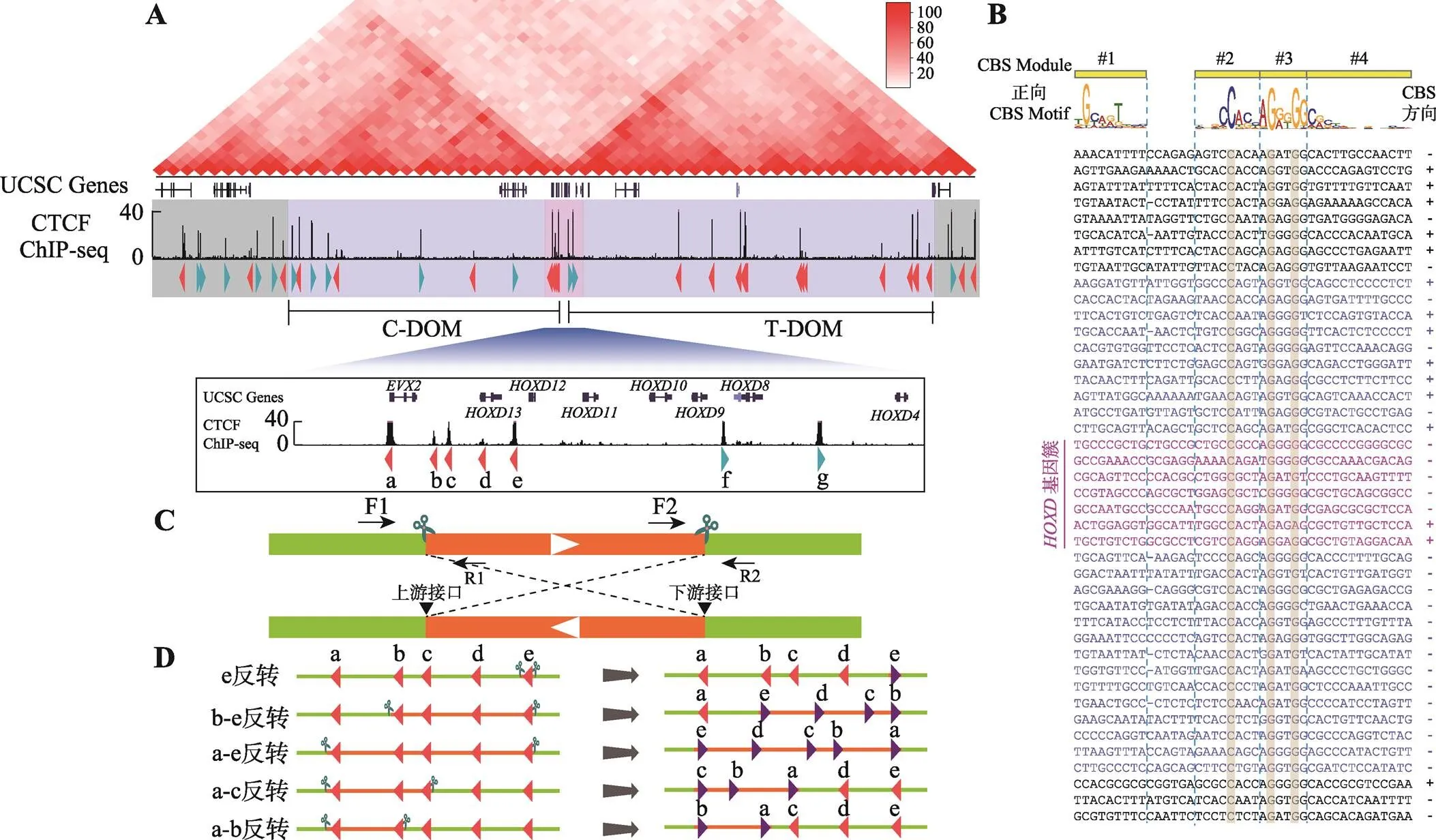
圖1 人類HOXD基因簇區域CTCF位點序列分析
A:基因簇區域基因組三維空間結構示意圖。基因簇位于C-DOM和T-DOM兩個TAD邊界處,位于基因簇附近的CBS a-e與C-DOM中的正向CBS形成染色質環,CBS f-g與T-DOM中的成串反向CBS形成染色質環。B:基因簇區域CBS序列比對圖。正向CBS的方向為module 1~4,其中module 1與module 2之間相隔5 bp或者6 bp。DNA序列中的“?”表示該CBS的module 1與module 2間隔為5 bp,故用“?”補齊。右側CBS方向列,“+”表示CBS為正向,“?”表示該CBS為反向且顯示DNA序列是反向互補序列。C:利用CRISPR/Cas9系統進行DNA片段反轉示意圖。針對預反轉片段設計一對sgRNA進行片段編輯,會產生上游接口和下游接口;在切割點附近設計引物,用引物F1+F2、R1+R2進行PCR鑒定,若出現目的條帶且F1+R1、F2+R2和F1+R2無目的條帶,則為純合反轉單細胞克隆。D:基因簇內CBS組合性反轉示意圖,共構建五種反轉單細胞克隆株。
CBS b-e反轉后,CBS a與CBS e形成一對反向-正向的CBS (圖1D),CBS b-e與Island5的遠距離DNA相互作用下降(圖3B),與CS38形成新的遠距離DNA相互作用(圖3A),阻礙從CBS f-g方向滲透的cohesin繼續滑動。CBS a和反轉后的CBS e形成了新的染色質拓撲結構域邊界,與CBS e反轉結果相似,即邊界CBS反轉引起染色質拓撲結構域邊界的漂移。CBS a-e全部反轉后,CBS a-e與Island5的DNA相互作用明顯減弱(圖3B),與CS38形成新的DNA相互作用(圖3A),由于缺乏成對反向-正向CBS,破壞了區域的染色質拓撲邊界。
為了進一步確定這些染色質空間構象的改變,選用CBS a和CBS e附近的DNA片段作為觀測點(upstream-a和downstream-e,upstream-a觀測點位于CBS a的上游,downstream-e觀測點位于CBS e的下游),為了保證觀測點不隨CBS反轉而改變,觀測點的位置不在反轉片段內。在CBS e反轉的細胞中,CS38與CBS a的遠距離DNA相互作用沒有變化(圖4A);而CBS b-e反轉后,CS38與CBS a的遠距離DNA相互作用有輕微增強(圖4A);反轉CBS a-e后CBS a主要與CS38形成較強的DNA相互作用(圖4A)。從downstream-e為觀測點來看,所有反轉克隆均與CS38有較強的DNA相互作用(圖4B),與上述結果相同。這些結果說明,反轉CBS后,該CBS原有的遠距離DNA相互作用被破壞,即CBS方向可以決定染色質環的形成,反轉的CBS可以阻礙反方向滲透的cohesin繼續滑動,發揮絕緣子的功能;同時,邊界CBS元件反轉會引起染色質拓撲結構域邊界的漂移。
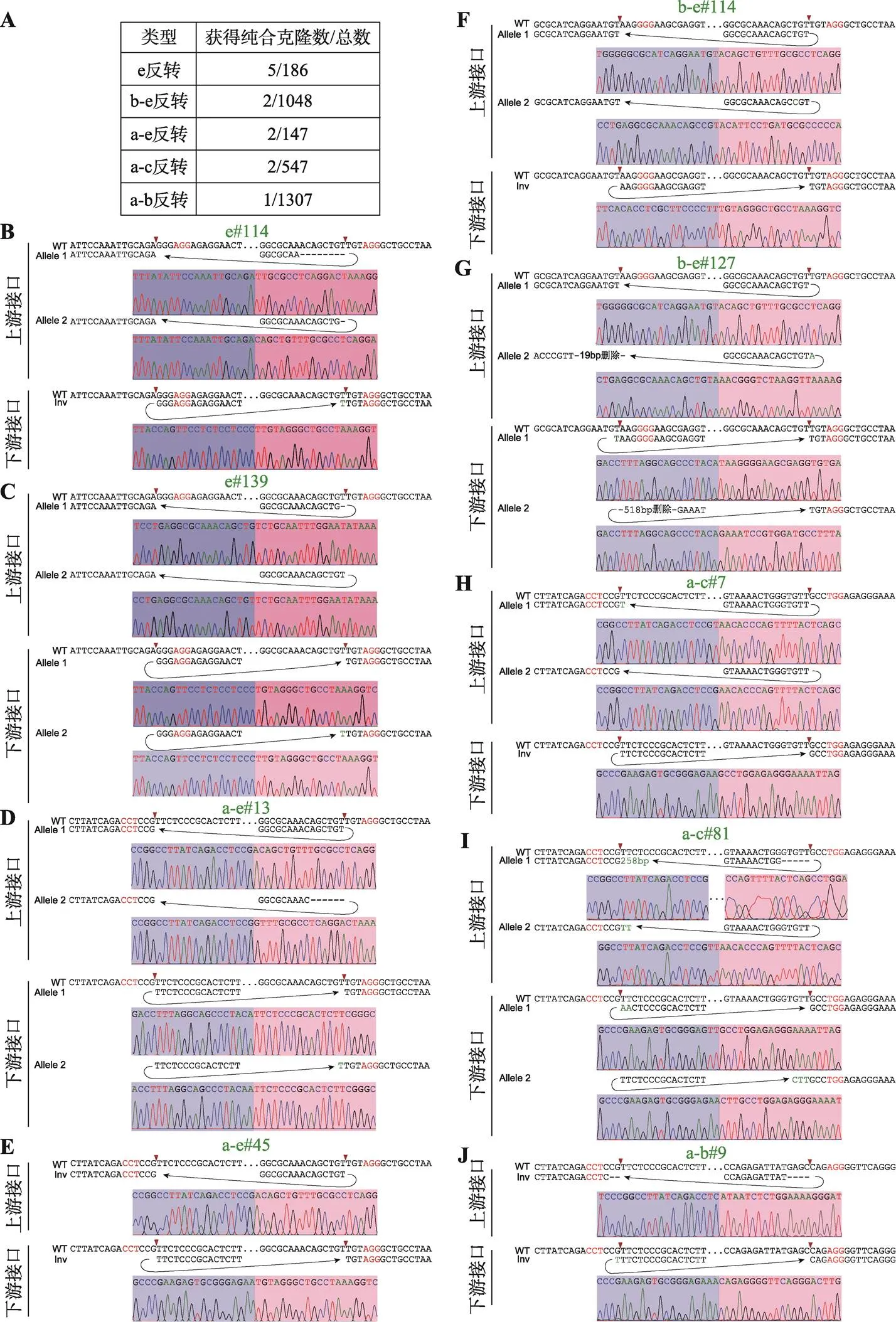
圖2 反轉單細胞克隆株基因型鑒定
A:獲得純合CBS反轉單細胞克隆株個數以及篩選克隆總數目。B~J:每一個純合反轉單細胞克隆株的基因型鑒定(TA克隆桑格測序圖)。序列中紅色表示PAM所在位置,紅色箭頭指示PAM上游–3位。序列中“–”表示存在堿基刪除,綠色表示存在堿基增加。

圖3 從兩側增強子觀察CBS反轉后局部三維基因組構象變化
A:以CS38為觀測點(viewpoint, VP)顯示,CBS反轉后與CS38的染色質相互作用顯著增強。CBS反轉單細胞克隆株數據分別與野生型(WT)進行log2轉化,獲得兩者差異數據。B:以Island5為觀測點顯示,CBS反轉后與Island5的原有染色質相互作用減弱。
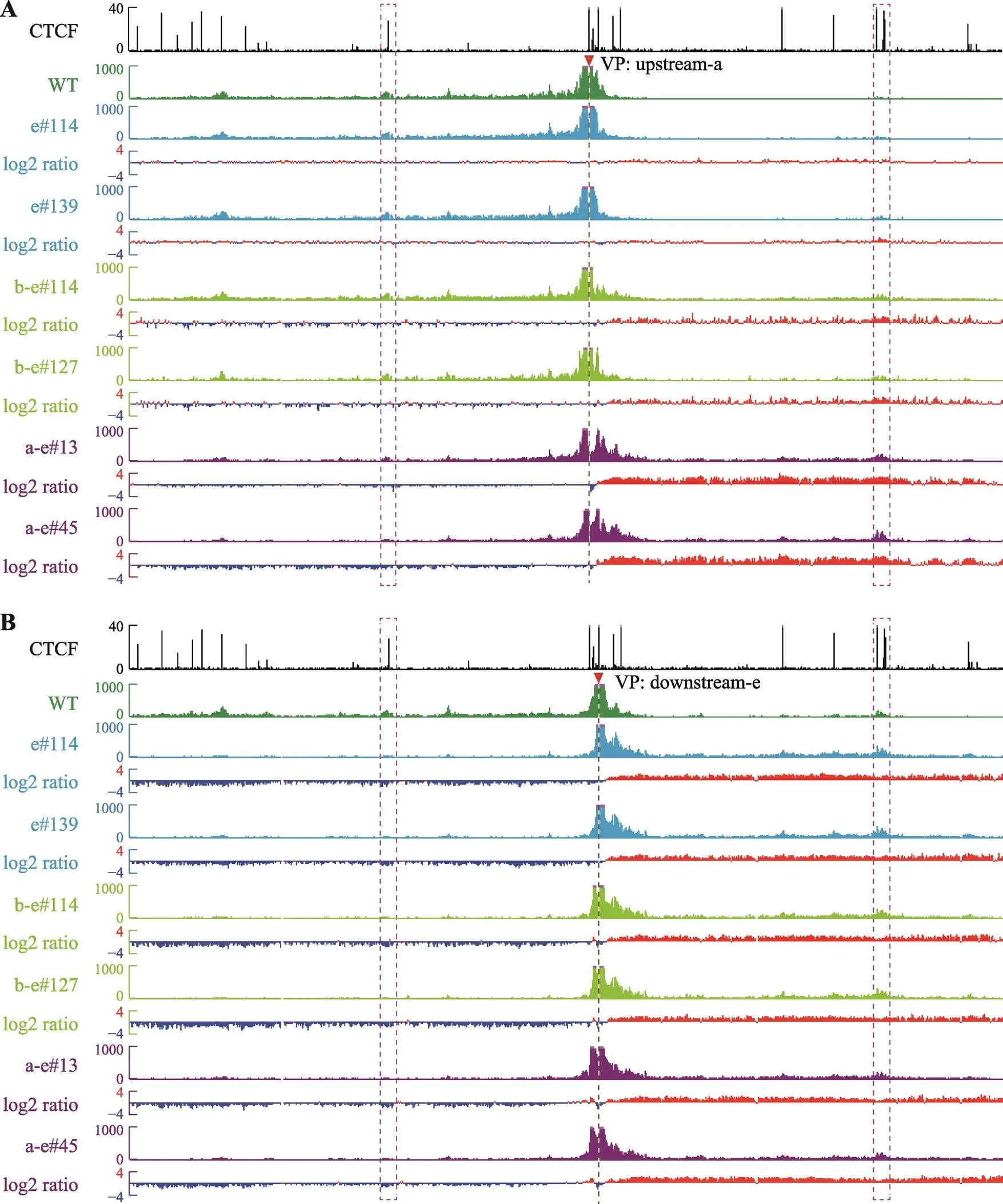
圖4 從反轉位點附近觀察CBS反轉后局部三維基因組構象變化
A:以upstream-a為觀測點,在CBS e反轉細胞中觀測點與兩側遠距離DNA相互作用無明顯變化,在CBS b-e反轉細胞中觀測點與CS38的遠距離DNA相互作用增強,在CBS a-e反轉細胞中觀測點與CS38相互作用明顯增強,伴隨著與Island5的遠距離DNA相互作用減弱。B:以downstream-e為觀測點,在三種包含CBS e反轉的細胞株中,觀測點與CS38的染色質相互作用均增強,伴隨著與Island5染色質相互作用減弱。
2.4 非邊界CTCF位點也發揮絕緣子功能
本實驗室之前的研究發現,位于基因簇啟動子區域的正向CBS可以與位于遠端的增強子HS5-1形成染色質環,阻礙更遠處的啟動子CBS與增強子HS5-1的遠距離DNA相互作用,具有絕緣子功能[34]。為了研究基因簇區域CBS是否同樣具有絕緣子功能,本研究進一步反轉了CBS a-b和CBS a-c (圖1D)。以Island5為觀測點顯示,反轉CBS a-b或CBS a-c后,Island5與CBS e的遠距離DNA相互作用增強(圖5A),而以CS38為觀測點顯示CS38與CBS a-e的遠距離DNA相互作用無明顯變化(圖5B)。相應地,upstream-a與上游的Island5的遠距離DNA相互作用減弱(圖5C),downstream-e與Island5的遠距離DNA相互作用增強(圖5D),與Island5為觀測點的結果相同。但是,染色質拓撲結構域邊界并沒有發生變化。因此,CBS a-b或a-c可以作為絕緣子,通過與Island5形成染色質環,分別阻礙CBS c-e或d-e與Island5的遠距離DNA相互作用。反轉細胞株的RNA-seq實驗顯示基因簇區域局部染色質構象變化后,基因表達水平會發生改變(圖6,A~D)。其中,在所有的CBS反轉細胞株中,的表達水平均顯著下降;除CBS b-e反轉細胞株外,與的表達水平均下降;有趣的是,在CBS e反轉和CBS a-e反轉的細胞中,的表達水平顯著上升(圖6,A~D)。
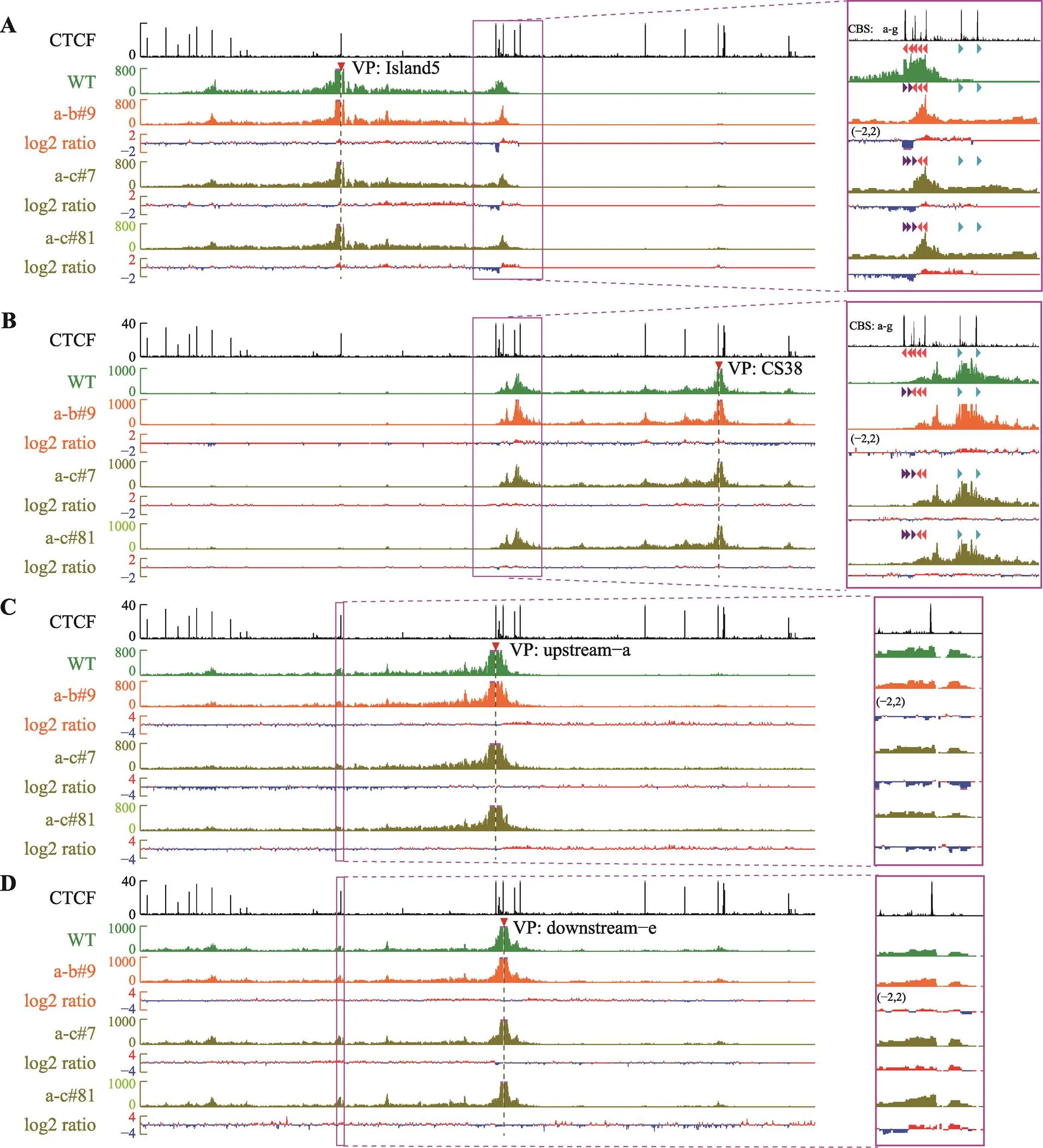
圖5 非邊界的成串CTCF位點也能發揮絕緣子功能
A:在CBS a-b或CBS a-c反轉細胞株中,Island5與被反轉CBS原有的遠距離DNA相互作用減弱,與CBS e的遠距離DNA相互作用增強。B:以CS38為觀測點顯示,觀測點與被反轉CBS的染色質相互作用無明顯變化。C:以upstream-a為觀測點,在CBS a-b或CBS a-c反轉細胞中,可見CBS a與Island5的染色質相互作用減弱。D:以downstream-e為觀測點,在CBS a-b或CBS a-c反轉細胞中,可見CBS e與Island5的染色質相互作用增強。
3 討論
CTCF通過阻礙cohesin蛋白在染色質上滑動形成染色質環,介導遠距離的DNA相互作用,對于基因組三維空間結構的形成與維持有重要作用[29,61,62]。多個位點的CBS反轉和插入實驗顯示染色質環形成依賴于成對正向-反向CBS[5,28,29],而CBS的絕緣子功能是不依賴方向的[34]。研究CBS的方向性有助于理解CBS在染色質高級拓撲結構中的功能,但是在成串CBS中,理解CBS的功能則更加復雜。原鈣粘蛋白基因簇的CBS研究顯示,成串CBS可能通過環擠出模型,呈現類似于“葫蘆”結構的平衡分布模式[34]。這顯示成對正向-反向CBS對于cohesin的阻礙不是完全的,存在cohesin滲透出正向-反向CBS對(CBS pair)限制的染色質范圍的情況,可能是由于CTCF與DNA結合是動態的[38],因而,在成串CBS中,CBS呈“葫蘆”狀分布[34]。但是,位于染色質拓撲結構域邊界的成串CBS如何發揮絕緣子功能,目前仍缺少系統研究。
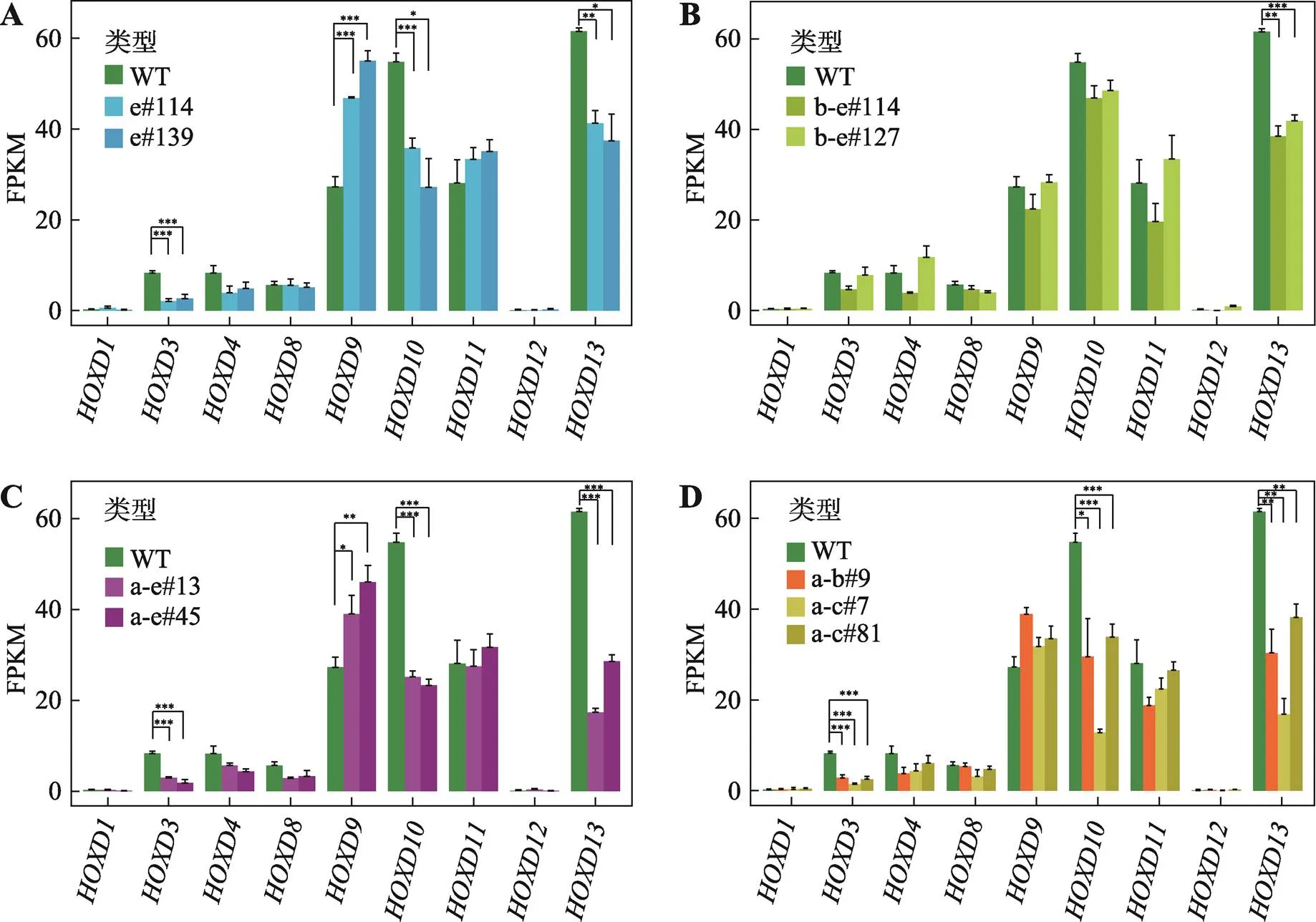
圖6 CTCF位點反轉改變HOXD基因表達
A:CBS e反轉后,、和的表達水平明顯降低,的表達水平升高;*:<0.05,**:<0.01,***:< 0.001;FPKM,fragments per kilobase of transcript per million mapped reads。B:CBS b-e反轉后,表達水平顯著降低。C:CBS a-e反轉后,、和的表達水平降低,而的表達水平上升。D:CBS a-b或CBS a-c反轉后,、和的表達水平降低。
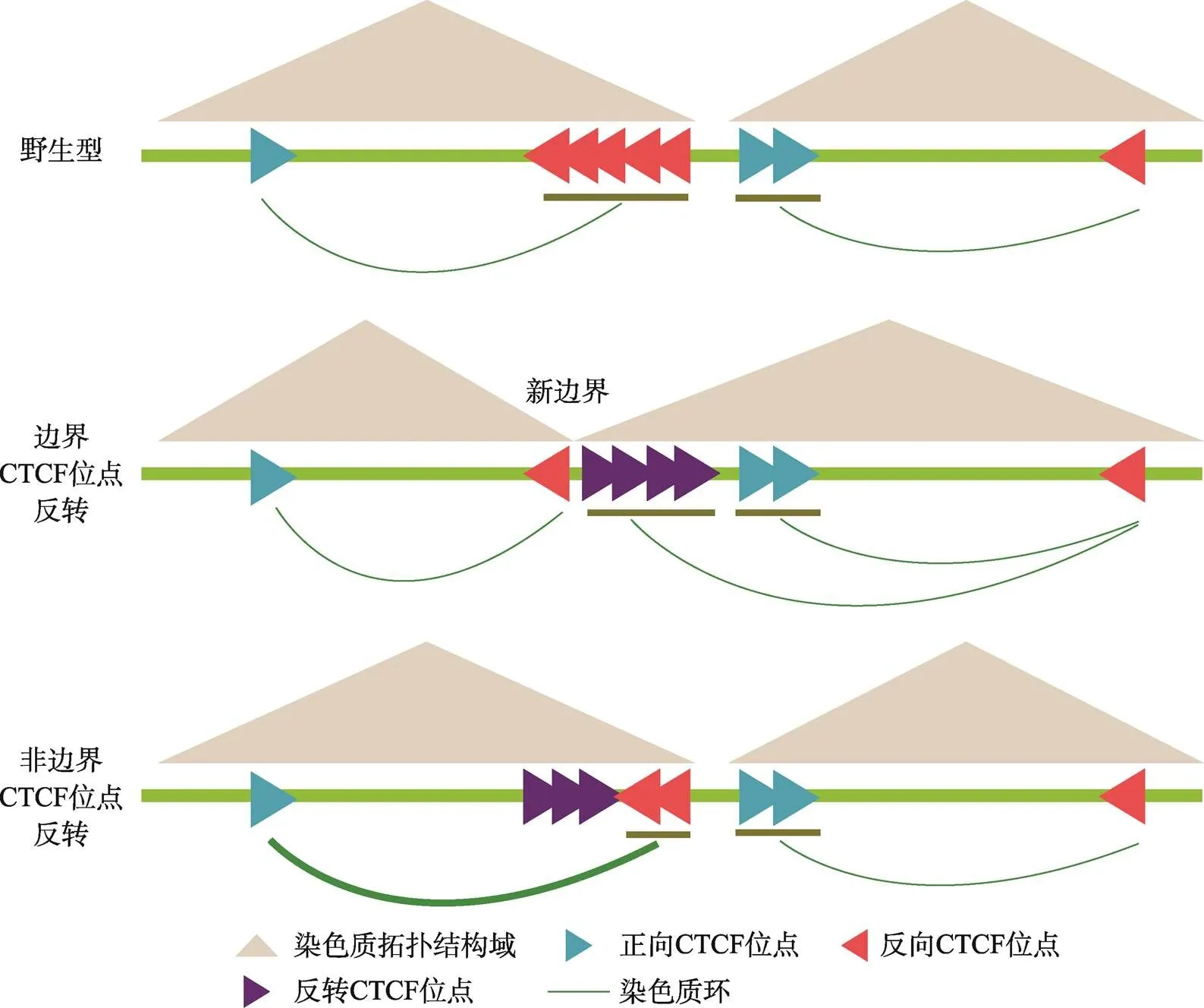
圖7 染色質拓撲結構域邊界成串CTCF位點發揮絕緣子功能的模式圖
A:在野生型細胞中,位于染色質拓撲結構域邊界成串反向-正向排列的CBS分別與兩側拓撲結構域內的CBS形成染色質環。邊界CBS反轉后,可以阻礙反方向滲透的cohesin滑動,形成染色質環;染色質結構域邊界也漂移至形成的一對反向-正向CBS處,而非邊界CBS反轉不引起染色質結構域邊界漂移。非邊界CBS反轉后邊界CBS與拓撲結構域內的相向CBS的遠距離DNA相互作用增強,顯示非邊界CBS也能發揮絕緣子功能。
本研究通過對基因簇區域的CBS進行CRISPR組合反轉,獲得了五種純合反轉單細胞克隆株。QHR-4C實驗顯示,在CBS e、CBS b-e、CBS a-e反轉細胞中,由于包含了TAD邊界的CBS元件,反轉的CBS引起了遠距離DNA互作的巨大變化,證實了邊界單個CBS反轉可以改變染色質環形成方向[29]。在這三種反轉細胞中,多個觀測點的QHR-4C數據顯示,反轉后CBS可以阻礙滲透的cohesin繼續滑動,發揮絕緣子功能,染色質拓撲結構域邊界也漂移至新形成的一對反向-正向CBS處(圖7)。其次,CBS a-b和CBS a-c反轉結果顯示,CBS a-b 或a-c可以作為絕緣子,分別阻礙CBS c-e或d-e與增強子Island5的遠距離DNA相互作用,證實在成串CBS a-e中,非邊界的CBS也可以作為絕緣子阻礙邊界CBS與增強子的染色質環的形成;反轉非邊界CBS不改變染色質拓撲結構域邊界(圖7)。CBS反轉引起的局部染色質構象變化會進一步影響基因簇的表達,但是對于不同的基因,引起的表達水平變化不盡相同,顯示出基因簇存在復雜的表達調控機制。綜上所述,本研究驗證了環擠出模型,同時進一步完善了對CTCF絕緣功能的理解。
[1] Kumar Y, Sengupta D, Bickmore W. Recent advances in the spatial organization of the mammalian genome.,2020, 45: 18.
[2] Wu Q, Liu PF, Wang LY. Many facades of CTCF unified by its coding for three-dimensional genome architecture., 2020, 47(8): 407–424.
[3] Ning CY, He MN, Tang QZ, Zhu Q, Li MZ, Li DY. Advances in mammalian three-dimensional genome by using Hi-C technology approach., 2019, 41(3): 215–233.
寧椿游, 何夢楠, 唐茜子, 朱慶, 李明洲, 李地艷. 基于Hi-C技術哺乳動物三維基因組研究進展. 遺傳, 2019, 41(3): 215–233.
[4] Marchal C, Sima J, Gilbert DM. Control of DNA replication timing in the 3D genome., 2019, 20(12): 721–737.
[5] Guo Y, Xu Q, Canzio D, Shou J, Li JH, Gorkin DU, Jung I, Wu HY, Zhai YN, Tang YX, Lu YC, Wu YH, Jia ZL, Li W, Zhang MQ, Ren B, Krainer AR, Maniatis T, Wu Q. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function., 2015, 162(4): 900–910.
[6] de Laat W, Duboule D. Topology of mammalian developmental enhancers and their regulatory landscapes., 2013, 502(7472): 499–506.
[7] Sanders JT, Freeman TF, Xu Y, Golloshi R, Stallard MA, Hill AM, San Martin R, Balajee AS, McCord RP. Radiation-induced DNA damage and repair effects on 3D genome organization., 2020, 11(1): 6178.
[8] Arnould C, Rocher V, Finoux AL, Clouaire T, Li K, Zhou F, Caron P, Mangeot PE, Ricci EP, Mourad R, Haber JE, Noordermeer D, Legube G. Loop extrusion as a mechanism for formation of DNA damage repair foci., 2021, 590(7847): 660–665.
[9] Cremer T, Cremer M. Chromosome territories., 2010, 2(3): a003889.
[10] Dekker J, Rippe K, Dekker M, Kleckner N. Capturing chromosome conformation., 2002, 295(5558): 1306–1311.
[11] Zhao ZH, Tavoosidana G, Sj?linder M, G?nd?r A, Mariano P, Wang S, Kanduri C, Lezcano M, Sandhu KS, Singh U, Pant V, Tiwari V, Kurukuti S, Ohlsson R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions., 2006, 38(11): 1341–1347.
[12] Lieberman-Aiden E, van Berkum NL, Williams L, Imakaev M, Ragoczy T, Telling A, Amit I, Lajoie BR, Sabo PJ, Dorschner MO, Sandstrom R, Bernstein B, Bender MA, Groudine M, Gnirke A, Stamatoyannopoulos J, Mirny LA, Lander ES, Dekker J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome., 2009, 326(5950): 289–293.
[13] Fullwood MJ, Liu MH, Pan YF, Liu J, Xu H, Mohamed YB, Orlov YL, Velkov S, Ho A, Mei PH, Chew EGY, Huang PYH, Welboren WJ, Han YY, Ooi HS, Ariyaratne PN, Vega VB, Luo YQ, Tan PY, Choy PY, Wansa KDSA, Zhao B, Lim KS, Leow SC, Yow JS, Joseph R, Li HX, Desai KV, Thomsen JS, Lee YK, Karuturi RKM, Herve T, Bourque G, Stunnenberg HG, Ruan XA, Cacheux- Rataboul V, Sung WK, Liu ET, Wei CL, Cheung E, Ruan YJ. An oestrogen-receptor-α-bound human chromatin interactome., 2009, 462(7269): 58–64.
[14] Nora EP, Lajoie BR, Schulz EG, Giorgetti L, Okamoto I, Servant N, Piolot T, van Berkum NL, Meisig J, Sedat J, Gribnau J, Barillot E, Bluthgen N, Dekker J, Heard E. Spatial partitioning of the regulatory landscape of the X-inactivation centre., 2012, 485(7398): 381–385.
[15] Dixon JR, Selvaraj S, Yue F, Kim A, Li Y, Shen Y, Hu M, Liu JS, Ren B. Topological domains in mammalian genomes identified by analysis of chromatin interactions., 2012, 485(7398): 376–380.
[16] Rao SSP, Huntley MH, Durand NC, Stamenova EK, Bochkov ID, Robinson JT, Sanborn AL, Machol I, Omer AD, Lander ES, Aiden EL. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping., 2014, 159(7): 1665–1680.
[17] Guo Y, Monahan K, Wu HY, Gertz J, Varley KE, Li W, Myers RM, Maniatis T, Wu Q. CTCF/cohesin-mediated DNA looping is required for protocadherin α promoter choice., 2012, 109(51): 21081– 21086.
[18] Sanborn AL, Rao SSP, Huang SC, Durand NC, Huntley MH, Jewett AI, Bochkov ID, Chinnappan D, Cutkosky A, Li J, Geeting KP, Gnirke A, Melnikov A, McKenna D, Stamenova EK, Lander ES, Aiden EL. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes., 2015, 112(47): E6456–E6465.
[19] Andrey G, Montavon T, Mascrez B, Gonzalez F, Noordermeer D, Leleu M, Trono D, Spitz F, Duboule D. A switch between topological domains underliesgenes collinearity in mouse limbs., 2013, 340(6137): 1234167.
[20] Dixon JR, Gorkin DU, Ren B. Chromatin domains: The unit of chromosome organization., 2016, 62(5): 668–680.
[21] Weintraub AS, Li CH, Zamudio AV, Sigova AA, Hannett NM, Day DS, Abraham BJ, Cohen MA, Nabet B, Buckley DL, Guo YE, Hnisz D, Jaenisch R, Bradner JE, Gray NS, Young RA. YY1 is a structural regulator of enhancer- promoter loops., 2017, 171(7): 1573–1588.e28.
[22] Guo Y, Wu Q. Inversion of CTCF binding sites by DNA fragment editing alters genome topology and enhancer/ promoter functions., 2015, 37(10): 1073–1074.
郭亞, 吳強. 采用DNA片段編輯技術反轉CTCF結合位點改變基因組拓撲結構和增強子與啟動子功能. 遺傳, 2015, 37(10): 1073–1074.
[23] Lobanenkov VV, Nicolas RH, Adler VV, Paterson H, Klenova EM, Polotskaja AV, Goodwin GH. A novel sequence-specific DNA binding protein which interacts with three regularly spaced direct repeats of the CCCTC- motif in the 5'-flanking sequence of the chickengene., 1990, 5(12): 1743–1753.
[24] Yin ML, Wang JY, Wang M, Li XM, Zhang M, Wu Q, Wang YL. Molecular mechanism of directional CTCF recognition of a diverse range of genomic sites., 2017, 27(11): 1365–1377.
[25] Nanni L, Ceri S, Logie C. Spatial patterns of CTCF sites define the anatomy of TADs and their boundaries., 2020, 21(1): 197.
[26] Ghirlando R, Felsenfeld G. CTCF: Making the right connections., 2016, 30(8): 881–891.
[27] Phillips JE, Corces VG. CTCF: Master weaver of the genome., 2009, 137(7): 1194–1211.
[28] de Wit E, Vos ESM, Holwerda SJB, Valdes-Quezada C, Verstegen MJAM, Teunissen H, Splinter E, Wijchers PJ, Krijger PHL, de Laat W. CTCF binding polarity determines chromatin looping., 2015, 60(4): 676–684.
[29] Lu YJ, Shou J, Jia ZL, Wu YH, Li JH, Guo Y, Wu Q. Genetic evidence for asymmetric blocking of higher-order chromatin structure by CTCF/cohesin., 2019, 10(12): 914–920.
[30] Wu Q, Jia ZL. Wiring the brain by clustered protocadherin neural codes., 2021, 37(1): 117–131.
[31] Narendra V, Rocha PP, An DS, Raviram R, Skok JA, Mazzoni EO, Reinberg D. CTCF establishes discrete functional chromatin domains at theclusters during differentiation., 2015, 347(6225): 1017–1021.
[32] Ren G, Jin WF, Cui KR, Rodrigez J, Hu GQ, Zhang ZY, Larson DR, Zhao KJ. CTCF-mediated enhancer-promoter interaction is a critical regulator of cell-to-cell variation of gene expression., 2017, 67(6): 1049–1058.e6.
[33] Gómez-Marín C, Tena JJ, Acemel RD, López-Mayorga M, Naranjo S, de la Calle-Mustienes E, Maeso I, Beccari L, Aneas I, Vielmas E, Bovolenta P, Nobrega MA, Carvajal J, Gómez-Skarmeta JL. Evolutionary comparison reveals that diverging CTCF sites are signatures of ancestral topological associating domains borders., 2015, 112(24): 7542–7547.
[34] Jia ZL, Li JW, Ge X, Wu YH, Guo Y, Wu Q. Tandem CTCF sites function as insulators to balance spatial chromatin contacts and topological enhancer-promoter selection., 2020, 21(1): 75.
[35] Zhang D, Huang P, Sharma M, Keller CA, Giardine B, Zhang HY, Gilgenast TG, Phillips-Cremins JE, Hardison RC, Blobel GA. Alteration of genome folding via contact domain boundary insertion., 2020, 52(10): 1076–1087.
[36] Zhai YN, Xu Q, Guo Y, Wu Q. Characterization of a cluster of CTCF-binding sites in a protocadherin regulatory region., 2016, 38(4): 323–336.
翟亞男, 許泉, 郭亞, 吳強. 原鈣粘蛋白基因簇調控區域中成簇的CTCF結合位點分析. 遺傳, 2016, 38(4): 323–336.
[37] Zhang XF, Zhang Y, Ba ZQ, Kyritsis N, Casellas R, Alt FW. Fundamental roles of chromatin loop extrusion in antibody class switching., 2019, 575(7782): 385– 389.
[38] Xu DF, Ma RS, Zhang JH, Liu ZJ, Wu B, Peng JH, Zhai YN, Gong QG, Shi YY, Wu JH, Wu Q, Zhang ZY, Ruan K. Dynamic nature of CTCF tandem 11 zinc fingers in multivalent recognition of DNA as revealed by NMR spectroscopy., 2018, 9(14): 4020–4028.
[39] Amandio AR, Lopez-Delisle L, Bolt CC, Mascrez B, Duboule D. A complex regulatory landscape involved in the development of mammalian external genitals., 2020, 9: e52962.
[40] Bianco S, Annunziatella C, Andrey G, Chiariello AM, Esposito A, Fiorillo L, Prisco A, Conte M, Campanile R, Nicodemi M. Modeling single-molecule conformations of theregion in mouse embryonic stem and cortical neuronal cells., 2019, 28(6): 1574–1583.e4.
[41] Fabre PJ, Leleu M, Mormann BH, Lopez-Delisle L, Noordermeer D, Beccari L, Duboule D. Large scale genomic reorganization of topological domains at thelocus., 2017, 18(1): 149.
[42] Noordermeer D, Leleu M, Splinter E, Rougemont J, De Laat W, Duboule D. The dynamic architecture ofgene clusters., 2011, 334(6053): 222–225.
[43] Montavon T, Soshnikova N, Mascrez B, Joye E, Thevenet L, Splinter E, de Laat W, Spitz F, Duboule D. A regulatory archipelago controlsgenes transcription in digits., 2011, 147(5): 1132–1145.
[44] Rodríguez-Carballo E, Lopez-Delisle L, Willemin A, Beccari L, Gitto S, Mascrez B, Duboule D. Chromatin topology and the timing of enhancer function at thelocus., 2020, 117(49): 31231– 31241.
[45] Li JH, Shou J, Guo Y, Tang YX, Wu YH, Jia ZL, Zhai YN, Chen ZF, Xu Q, Wu Q. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9., 2015, 7(4): 284–298.
[46] Li JH, Shou J, Wu Q. DNA fragment editing of genomes by CRISPR/Cas9., 2015, 37(10): 992– 1002.
李金環, 壽佳, 吳強. CRISPR/Cas9系統在基因組DNA片段編輯中的應用. 遺傳, 2015, 37(10): 992–1002.
[47] Liu PF, Wu Q. Probing 3D genome by CRISPR/Cas9., 2020, 42(1): 18–31.
劉沛峰, 吳強. CRISPR/Cas9基因編輯在三維基因組研究中的應用. 遺傳, 2020, 42(1): 18–31.
[48] Shou J, Li JH, Liu YB, Wu Q. Precise and predictable CRISPR chromosomal rearrangements reveal principles of Cas9-mediated nucleotide insertion., 2018, 71(4): 498–509.e4.
[49] Wang LY, Huang HY, Wu Q. The diversity of DNA fragment editing by CRISPR/Cas9 in highly homologous or repetitive sequences., 2017, 39(4): 313–325.
汪樂洋, 黃海燕, 吳強. 利用CRISPR/Cas9對基因組中高度同源DNA片段編輯多樣性的遺傳學研究. 遺傳, 2017, 39(4): 313–325.
[50] Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas9 systems., 2013, 339(6121): 819–823.
[51] Wu Q, Shou J. Toward precise CRISPR DNA fragment editing and predictable 3D genome engineering., 2021, 12(11): 828–856.
[52] Wang N, Jia ZL, Wu Q. RFX5 regulates gene expression of thecluster., 2020, 42(8): 760–774.
王娜, 甲芝蓮, 吳強. RFX5調控原鈣粘蛋白α基因簇的表達. 遺傳, 2020, 42(8): 760–774.
[53] Wu YH, Jia ZL, Ge X, Wu Q. Three-dimensional genome architectural CCCTC-binding factor makes choice in duplicated enhancers atlocus., 2020, 63(6): 835–844.
[54] Zuin J, Dixon JR, van der Reijden MIJA, Ye Z, Kolovos P, Brouwer RWW, van de Corput MPC, van de Werken HJG, Knoch TA, van IJcken WFJ, Grosveld FG, Ren B, Wendt KS. Cohesin and CTCF differentially affect chromatin architecture and gene expression in human cells.2014, 111(3): 996–1001.
[55] Servant N, Varoquaux N, Lajoie BR, Viara E, Chen CJ, Vert JP, Heard E, Dekker J, Barillot E. HiC-Pro: An optimized and flexible pipeline for Hi-C data processing.2015, 16: 259.
[56] Kruse K, Hug CB, Hernández-Rodríguez B, Vaquerizas JM. TADtool: Visual parameter identification for TAD- calling algorithms., 2016, 32(20): 3190– 3192.
[57] Bailey TL, Johnson J, Grant CE, Noble WS. The meme suite., 2015, 43(W1): W39–W49.
[58] Chen FZ, You LJ, Yang F, Wang LN, Guo XQ, Gao F, Hua C, Tan C, Fang L, Shan RQ, Zeng WJ, Wang B, Wang R, Xu X, Wei XF. CNGBdb: China National GeneBank DataBase., 2020, 42(8): 799–809.
陳鳳珍, 游麗金, 楊帆, 王麗娜, 郭學芹, 高飛, 華聰, 談聰, 方林, 單日強, 曾文君, 王博, 王韌, 徐訊, 魏曉鋒. CNGBdb: 國家基因庫生命大數據平臺. 遺傳, 2020, 42(8): 799–809.
[59] Guo XQ, Chen FZ, Gao F, Li L, Liu K, You LJ, Hua C, Yang F, Liu WL, Peng CH, Wang LN, Yang XX, Zhou FY, Tong JW, Cai J, Li ZY, Wan B, Zhang L, Yang T, Zhang MW, Yang LL, Yang YW, Zeng WJ, Wang B, Wei XF, Xu X. CNSA: a data repository for archiving omics data., 2020, 2020: baaa055.
[60] Shi X, Shou J, Mehryar MM, Li JW, Wang LY, Zhang M, Huang HY, Sun XF, Wu Q. Cas9 has no exonuclease activity resulting in staggered cleavage with overhangs and predictable di- and tri-nucleotide CRISPR insertions without template donor., 2019, 5: 53.
[61] Gligoris TG, Scheinost JC, Burmann F, Petela N, Chan KL, Uluocak P, Beckou?t F, Gruber S, Nasmyth K, L?we J. Closing the cohesin ring: structure and function of its Smc3-kleisin interface., 2014, 346(6212): 963– 967.
[62] Nishiyama T. Cohesion and cohesin-dependent chromatin organization., 2019, 58: 8–14.
Combinatorial CRISPR inversions of CTCF sites incluster reveal complex insulator function
Xianglong He, Jinhuan Li, Qiang Wu
CTCF (CCCTC-binding factor) is a zinc-finger protein which plays a vital role in the three-dimensional (3D) genome architecture. A pair of forward-reverse convergent CTCF binding sites (CBS elements) mediates long-distance DNA interactions to form chromatin loops with the assistance of the cohesin complex, while CBS elements at the chromatin domain boundaries show reverse-forward divergent patterns and function as insulators to discriminate against DNA interaction between chromatin domains. However, there are still many unresolved problems regarding CTCF-mediated insulation function. In order to study the connections between chromatin loops and the insulation function of CBS elements, we combinatorically inverted CBS elements at thelocus by using CRISPR/Cas9 DNA-fragment editing methods in the HEK293T cell line and obtained five different kinds of single-cell CRISPR clones. By performing quantitative high-resolution chromosome conformation capture copy (QHR-4C) experiments, we found that boundary CBS inversions abolish original chromatin loops and establish new loops from the opposite direction, thus shifting the insulator boundary to the new divergent CTCF sites. Furthermore, tandem CBS elements block cohesin permeated from the opposite orientation to function as insulators. RNA-seq experiments showed that alterations of local three-dimensional genome architecture would further influence gene expression of thecluster. In conclusion, a pair of divergent CBS elements function as insulators by forming chromatin loops within chromatin domains to block cohesin sliding.
CTCF; chromatin loop;; insulator; CBS inversion
2021-04-11;
2021-05-12
國家自然科學基金項目(編號:31800636, 31630039, 91940303)和上海市科學技術委員會項目(編號:19JC1412500)資助[Supported by the National Nature Science Foundation of China (Nos. 31800636, 31630039, 91940303) and Science and Technology Commission of Shanghai Municipality program (No. 19JC1412500)]
何象龍,在讀碩士研究生,專業方向:生物學。E-mail: esroom@163.com
吳強,博士,教授,研究方向:三維基因組架構與功能。E-mail: qwu123@gmail.com
10.16288/j.yczz.21-131
2021/7/7 08:47:52
URI: https://kns.cnki.net/kcms/detail/11.1913.r.20210706.1111.001.html
(責任編委: 谷峰)

