小開放閱讀框編碼微肽的研究進展
陳相穎,李夢瑋,王穎,陳權,徐寒梅
綜 述
小開放閱讀框編碼微肽的研究進展
陳相穎,李夢瑋,王穎,陳權,徐寒梅
中國藥科大學江蘇省合成多肽藥物發現與評價工程中心,南京 211198
已有的研究表明,生命體中存在著大量的非編碼RNA (non-coding RNA, ncRNA),先前被錯誤注釋為ncRNA的分子序列中實際上包含小的開放閱讀框(short open reading frame, sORF),部分sORF可轉錄并翻譯成進化保守的微肽(micropeptide),這些sORF由于序列較短和研究技術手段的限制而被忽略。迄今為止,已在生命體中發現一些sORF編碼的功能各異的微肽,它們對生命活動的調控起著重要作用。本文對近年來發現的功能性微肽進行綜述,介紹了本課題組發現新型微肽MIAC (micropeptide inhibiting actin cytoskeleton)的過程,同時總結了研究潛在微肽的相關技術,以期為研究人員利用相關技術發現新微肽提供借鑒和參考。
非編碼RNA;小開放閱讀框;微肽
隨著科學技術的發展,人們對于生物復雜性有了更進一步的認識。中心法則指出,遺傳信息通常經歷由脫氧核苷酸轉錄到核糖核苷酸再翻譯為蛋白質的過程[1]。人類基因組計劃證明,人類基因組的3/4能夠被轉錄,但只有約1.5%的基因具有編碼蛋白的能力[2]。這就引發了人們對基因組中剩余的大量非蛋白編碼基因的思考,這些非蛋白編碼基因是否包含更多的遺傳信息。DNA元件百科全書(ency-clopedia of DNA elements, ENCODE)的數據表明,80%的基因具有特定的生物學功能,而大部分基因處于非蛋白質編碼區域,這部分基因轉錄產生大量的ncRNA[3]。隨著高通量測序技術的發展,越來越多的實驗證明ncRNA分子序列上含有小的開放閱讀框序列(short open reading frame, sORF),可編碼小于100個氨基酸的微小蛋白,被人們稱為微肽(micropeptide),加工修飾后的微肽可通過與其他蛋白相互作用而發揮其生理或病生理的作用[4]。研究表明,包括果蠅()、小鼠()、人()在內的許多動物基因組中包含數百萬個sORF,其中一些具有關鍵的生理或病生理功能,如鈣離子穩態、代謝、成肌細胞融合和肌肉發育、胚胎發育、物質降解、癌癥等[5~24]。本文主要介紹了近年來發現的功能性微肽、本課題組發現新型微肽MIAC (micropeptide inhibiting actin cytoskeleton)的過程以及研究潛在微肽的技術,期望為進行相關微肽研究的科研人員提供新思路。
1 sORF和微肽簡介
開放閱讀框(open reading frame, ORF)最初被定義為起始密碼子與終止密碼子間的潛在翻譯序列[25]。可翻譯的ORF通常是指mRNA上的編碼序列(coding sequences, CDS),該序列翻譯產生具有生物學功能的蛋白質[26]。由于ORF編碼蛋白質的可能性隨著其長度的增加而增加,查找ORF的算法大多都以300個密碼子或100個氨基酸為閾值作為最短的檢測長度[27]。sORF在序列長度上區別于ORF,理論上sORF的大小可以從最低限制的2個密碼子到100個密碼子,sORF由于其極短的長度在最初被認為是非編碼的[28]。最近研究發現,真核基因組中存在數百萬個sORF序列,并且有些sORF序列可以定位到轉錄本,這部分sORF具有編碼并翻譯產生蛋白的能力[28,29]。因此,微肽被定義為長度小于100個氨基酸的蛋白質。
根據果蠅和哺乳動物中sORF的位置、大小、保守性和翻譯方式等特性,sORF可分為五類(圖1):基因間ORF (intergenic ORF)、上游ORF (upstream ORF, uORF)、長非編碼ORF (long non-coding ORF, lncORF)、短編碼序列(short coding sequence, short CDS)和短同工型ORF (short isoform ORF)。其中,基因間ORF占sORF的96%,但其并不會進行轉錄和翻譯。uORF位于5′端非翻譯區(5′ untranslated region, 5′ UTR),具有較低效率的翻譯功能并能調節轉錄本中下游的ORF。lncORF存在于lncRNA (long non-coding RNA)中,與uORF相似具有較低的翻譯效率。最近發現,幾種lncRNA可編碼和翻譯為具有生物學功能的微肽,并且在進化中高度保守。短CDS是在單順反子轉錄本中發現的,具有與ORF類似的翻譯效率,在果蠅和哺乳動物中存在著數百種短CDS。短同工型ORF是sORF中占比最少的一類,由mRNA的選擇性剪接產生[4]。
關于微肽的翻譯機制,目前有如下幾種可能的解釋:根據核糖體的掃描模型,mRNA的5′端帽子結構與核糖體40S小亞基結合以復合物形式向3′端掃描,若遇起始密碼子,核糖體40S小亞基便與60S大亞基形成80S核糖體,從而介導5′UTR中sORF的翻譯,遇到終止密碼子時翻譯結束,大小亞基解離;而40S小亞基則繼續向前掃描,當遇到ORF的起始密碼子時重新結合核糖體60S大亞基。第二種可能的機制是只有部分40S小亞基結合在5' UTR中sORF的起始密碼子處,另一部分繼續向前掃描至ORF的起始密碼子,這種機制被稱為核糖體的泄漏掃描[30]。但上述兩種機制只適用于uORF產生的微肽,關于其他類型微肽的翻譯機制還有待研究,還有一種猜想是RNA編輯促成了微肽的翻譯,即在轉錄后將A-C/G/A-G修改為A-U-G[31]。未來可以通過基因敲除RNA編輯的關鍵酶來研究RNA編輯在RNA水平上產生sORF起始密碼子的作用。
2 功能性微肽的發現
基于生物信息學及高通量測序技術對微肽的深入研究,越來越多的微肽被證明在生命活動的許多過程中起著重要的調節作用,包括鈣離子穩態、代謝、成肌細胞融合和肌肉發育、胚胎發育、物質降解、癌癥等(圖2)[5~24]。下面將對近年來功能性微肽的發現進行介紹,并將其總結在表1中。
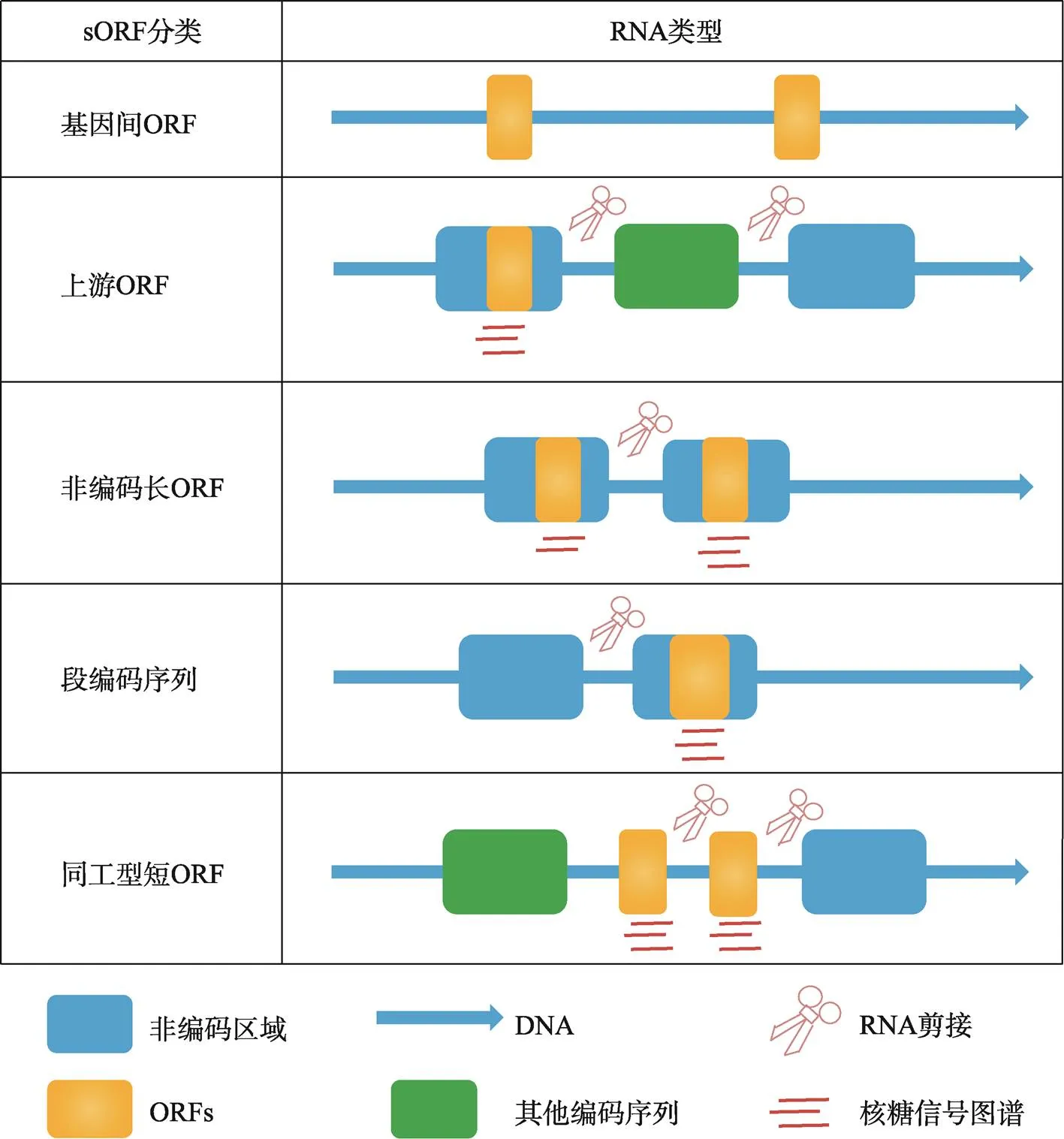
圖1 sORF的分類
2.1 鈣離子穩態相關微肽
Ca2+是肌肉收縮的主要調節因子,控制著肌肉的生長、代謝和病理重塑[32]。美國德克薩斯大學西南醫學中心Eric N. Olson實驗室的研究結果顯示,微肽對于Ca2+穩態的調節起重要作用[5,6,8]。肌調素(myoregulin, MLN)是由骨骼肌特異性lncRNA編碼的46個氨基酸的微肽,它可直接與肌漿網Ca2+-ATP酶(sarcoplasmic reticulum Ca2+-ATPase, SERCA)相互作用以降低SERCA對Ca2+的親和力,從而減少Ca2+攝入肌漿網和肌細胞的收縮性[6]。因此,將MLN鑒定為骨骼肌中的SERCA抑制性微肽。相反,DWORF (dwarf open reading frame)可解除SERCA抑制性微肽的作用而增強肌漿網攝取Ca2+的能力,它是由心肌特異性lncRNA編碼的34個氨基酸的微肽[8]。隨后,Anderson等[5]在非肌肉細胞中發現兩種SERCA抑制性微肽ELN (endoregulin)和ALN (another-regulin),這兩種微肽具有與MLN相似的結構和功能,表明Ca2+相關微肽在不同的細胞類型中保守,Ca2+穩態的調節對于許多細胞功能具有重要意義。
另外,Magny等[7]在果蠅的基因中也發現編碼SERCA抑制性微肽的序列,該微肽可影響果蠅心肌中Ca2+的運輸。跨物種的氨基酸相關序列分析表明,Ca2+相關微肽的結構和功能在果蠅到脊椎動物中具有高度保守性,與其在SERCA中調節Ca2+攝取的生物學功能相關[5,7]。
2.2 線粒體代謝相關微肽
線粒體作為一種功能性細胞器,在新陳代謝及能量供應方面起著重要的作用,大量的研究表明線粒體DNA中也存在sORFs[9,33]。Makarewich等[10]在線粒體內膜中發現由lncRNA編碼的微肽MOXI (micropeptide regulator of β-oxidation),MOXI與催化長鏈脂肪酸氧化的線粒體三功能蛋白(mitochon-drial trifunctional protein, MTP)結合,可增強脂肪酸的β氧化作用。Stein等[11]在骨骼肌和心臟中發現由lncRNA編碼的線粒體跨膜蛋白Mtln (mitoregulin),Mtln作為粘性分子可通過增強線粒體蛋白復合物的裝配和穩定性從而提高線粒體的呼吸效率。此外,在線粒體中還發現由編碼的16個氨基酸的微肽MOTS-c (mitochondrial open reading frame of the 12S rRNA-c),它可抑制葉酸循環及嘌呤核苷酸的從頭合成途徑而導致AMPK (AMP-activated protein kinase)活化,從而調節胰島素的敏感性[9]。這些結果表明,線粒體可通過微肽在細胞和機體水平上主動調控代謝穩態。
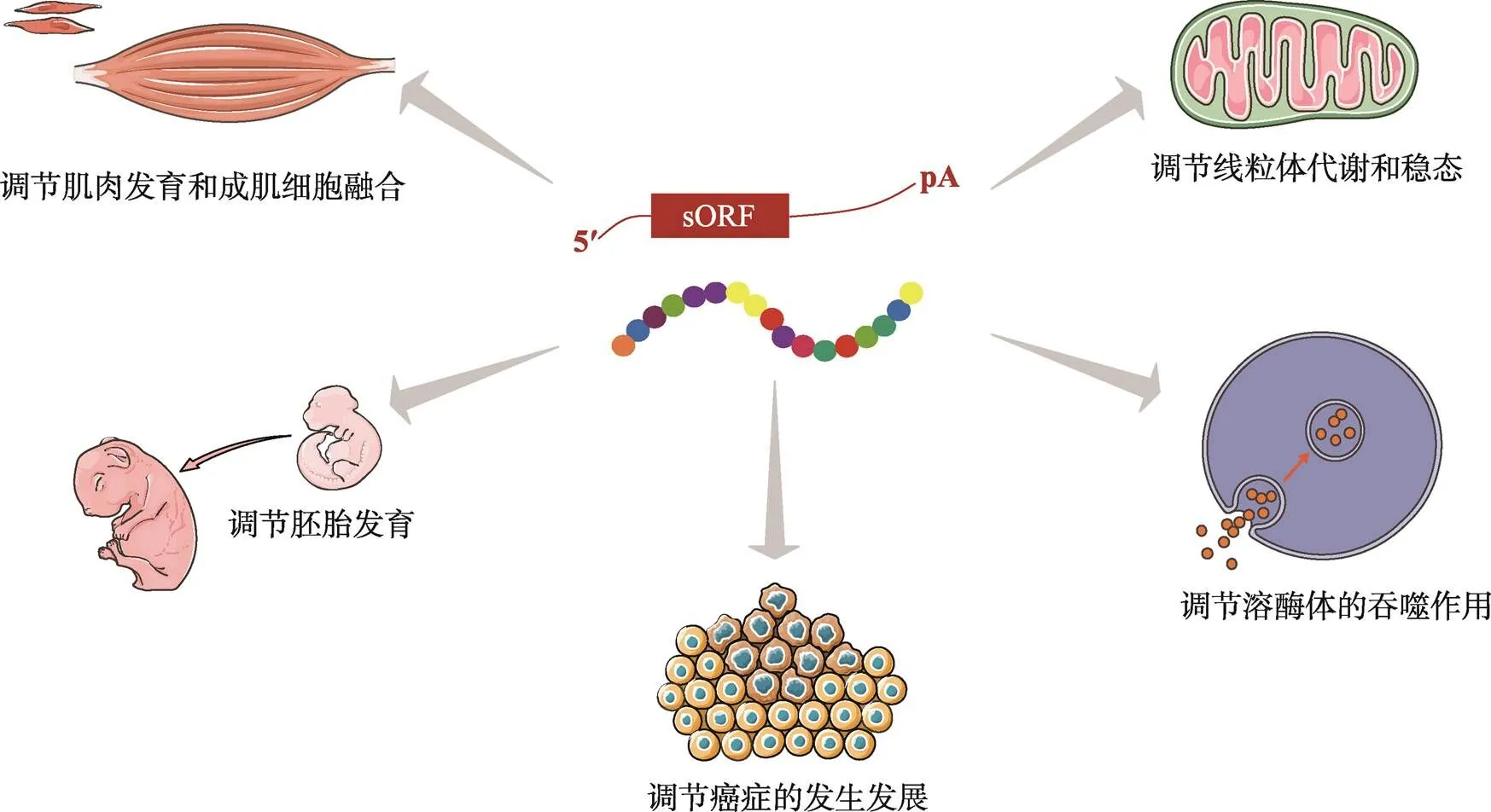
圖2 微肽的生理與病生理功能
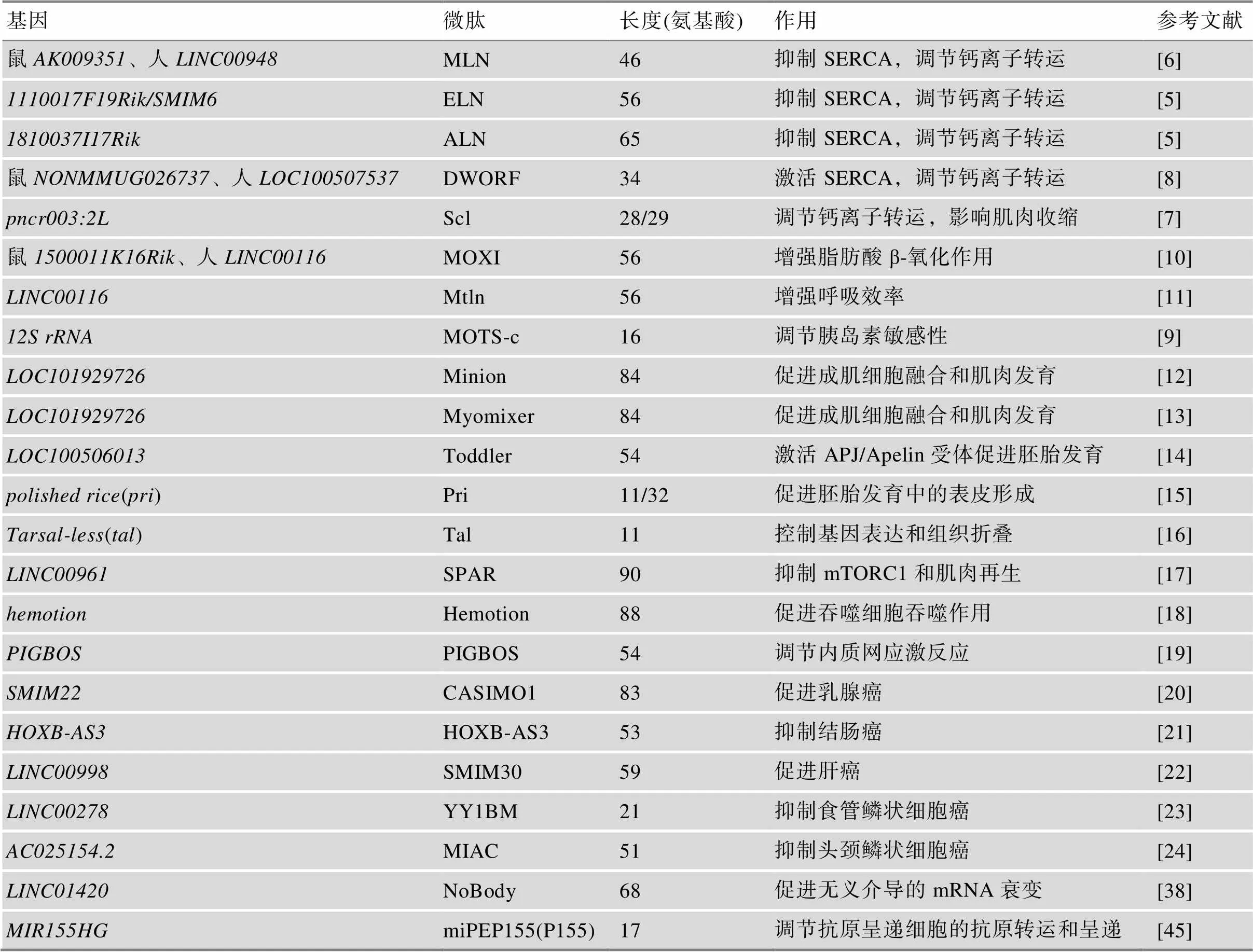
表1 功能性微肽的發現
2.3 成肌細胞融合和肌肉發育相關微肽
骨骼肌的形成需要單核成肌細胞融合形成多核細胞肌管以產生收縮性肌纖維,Myomaker是成肌細胞融合所需的肌肉特異性蛋白[34,35]。最近研究發現,有多種肌肉特異性的微肽在哺乳動物成肌細胞融合的過程中也起著關鍵作用[12,13]。Zhang等[12]發現一種sORF編碼的新微肽Minion (microprotein inducer of fusion),Minion與Myomaker共表達可誘導細胞融合和細胞骨架的快速重排。Myomixer是長為84個氨基酸的肌肉特異性微肽,可促進成肌細胞融合,Myomixer與Myomaker結合還可誘導成纖維細胞間的融合及成纖維細胞和成肌細胞的融合[13]。因此,sORF編碼的微肽對于肌肉發育過程中的肌纖維形成具有重要調控作用。
2.4 胚胎發育相關微肽
Toddler是在斑馬魚()中發現的由lncRNA編碼的長為58個氨基酸的微肽,研究發現它作為APJ/Apelin受體信號轉導的激活劑,可促進原腸胚的形成[14]。先前的研究表明APJ/Apelin受體信號轉導在心血管發育和生理調節等多種生物過程中發揮著重要作用[36]。Pauli等[14]發現Toddler功能缺失的斑馬魚沒有正常的心臟和血液循環,這些研究表明Toddler在早期胚胎發育過程中是不可或缺的。
另外,在果蠅中還發現與胚胎發育相關的微肽[15,16]。Kondo等[15]在果蠅的上皮組織中發現lncRNA()實際上被轉錄成多順反子mRNA,可編碼長為11或32個氨基酸的微肽(Pri)。Pri通過調節F-actin在上皮形態的發生中起重要作用,而Pri功能的喪失可完全消除果蠅的表皮結構。Galindo等[16]在果蠅中發現基因()對果蠅的胚胎發育和形態發生至關重要,可翻譯為短至11個氨基酸的微肽,控制著果蠅的基因表達和組織折疊。這些結果表明,極短的sORF具有翻譯功能并在發育過程中具有重要調控作用。
2.5 物質降解相關微肽
近年來,在生物技術的驅動下,與物質降解作用相關的微肽也不斷被發現,它們在廢物和毒素的降解方面發揮著重要的作用[17~19]。SPAR (small regu-latory polypeptide of amino acid response)是由lncRNA編碼的長為90個氨基酸的保守性微肽,定位于晚期內體及溶酶體。SPAR與溶酶體表面v-ATPase復合物的四個亞基相互作用,負性調節mTORC1的活化而抑制肌肉再生[17]。
此外,Pueyo等[18]在果蠅中發現一個組織特異性的sORF基因,其編碼果蠅巨噬細胞中長為88個氨基酸的跨膜微肽(Hemotin)。實驗研究表明,Hemotin peptide結合并抑制銜接蛋白14-3-3ζ,進而促進磷脂酰肌醇的磷酸化而調節吞噬作用中的內體成熟。并且,研究人員在脊椎動物中還發現Hemotin的功能同源物Stannin,表明這種吞噬作用的新型調節因子具有物種間保守性[18]。
未折疊蛋白反應(unfolded protein response, UPR)是真核細胞內質網(endoplasmic reticulum, ER)中的一個基本過程,在ER中只有正確組裝和折疊的蛋白質才能分泌到胞外或展示在細胞表面,而不折疊的蛋白將被內質網相關蛋白所降解[37]。位于線粒體外膜的微肽PIGBOS與ER蛋白CLCC1結合從而調節內質網中的UPR,而PIGBOS的缺失會導致UPR升高和細胞死亡[19]。由此可見,微肽對細胞器間的通訊、體內的平衡以及細胞的存亡至關重要。
2.6 癌癥相關微肽
微肽在癌癥的發生發展中也具有重要的調控作用[20~24]。CASIMO1 (cancer-associated small integral membrane open reading frame 1)是第一個被發現具有致癌作用的功能性微肽,它與膽固醇合成的關鍵酶角鯊烯環氧化酶(squalene epoxidase, SQLE)相互作用從而調節癌細胞的代謝穩態,敲低可導致乳腺癌細胞的增殖減少[20]。在結腸癌(colon cancer, CRC)方面,Huang等[21]發現由lncRNA編碼的長為53個氨基酸的保守微肽(HOXB-AS3)的缺失是CRC代謝中的關鍵致癌因素,HOXB-AS3能抑制結腸癌的生長。Pang等[22]還發現由lncRNA編碼的59個氨基酸的微肽SMIM30可通過調節細胞增殖和遷移促進肝癌的發生發展。Wu等[23]通過對281對男性食管鱗狀細胞癌(esophageal squamous cell carcinoma, ESCC)組織樣本中lncRNA的差異表達分析發現與癌旁組織相比,在ESCC組織中顯著下調,進一步研究發現編碼微肽YY1BM (Yin Yang 1 (YY1)-binding micropeptide),YY1BM可與雄激素受體(androgen receptor, AR)結合并下調的表達從而導致癌細胞凋亡,由此可見,YY1BM可作為一種潛在的抗癌微肽。另外,Li等[24]證明MIAC能抑制頭頸鱗狀細胞癌(head and neck squamous cell carcinoma, HNSCC)的生長和轉移。此外,還有一些微肽與癌癥并不直接相關,例如NoBody (-annotated P-dissociating polypeptide)是由編碼的68個氨基酸的微肽,它與mRNA脫帽蛋白相互作用,促進無義介導的mRNA衰變(nonsense mediated decay, NMD),癌細胞可能利用此過程降解抑制腫瘤的mRNA[38]。總而言之,這些新發現的轉錄本豐富了腫瘤調控分子,并為癌癥的臨床診斷和治療提供新的潛在靶標。
3 微肽MIAC的發現
本課題組研究發現,lncRNA可能編碼長為51個氨基酸的內源性微肽[24]。在驗證這段序列的翻譯編碼能力時,我們通過體外翻譯實驗和體內細胞構建實驗加以證明,結果表明lncRNA能夠編碼一種新型微肽,我們將其命名為MIAC。在微肽MIAC的功能研究中,我們構建了MIAC穩定過表達和敲除的CAL27細胞系,實驗發現MIAC通過負調控癌細胞的增殖和轉移而抑制HNSCC的發生發展。
接下來,我們對MIAC抑制HNSCC相關機制做了進一步的研究。通過質譜鑒定與MIAC相互作用的蛋白,并結合50個HNSCC臨床樣本和50個正常樣本中蛋白的表達情況,最后聚焦于其中的這三種蛋白:水通道蛋白2 (aquaporin 2, AQP2)、ITGB4 (integrin beta 4)和SEPT2 (septin 2)。進一步的機制探究表明MIAC直接與AQP2相互作用,通過調控SEPT2/ITGB4抑制骨架蛋白重排,最終抑制HNSCC的生長和轉移。由此可見,MIAC在HNSCC中具有調控作用,為開發治療HNSCC的藥物提供新的研究方向,而AQP2作為MIAC的作用靶點對于研究HNSCC的藥物同樣存在重要意義。
為進一步探究MIAC的臨床和治療意義,我們分析TCGA數據庫中500個HNSCC臨床樣本和44個正常樣本中MIAC的相對表達情況,發現MIAC在HNSCC中呈下調趨勢,并且MIAC表達水平的降低與HNSCC患者的整體生存率差呈正相關。我們進一步分析94對HNSCC臨床樣本中MIAC的相對表達量,分析結果也與數據庫中的情況一致,相比于在正常樣本中,MIAC在HNSCC樣本中的表達量降低。因此,MIAC是由lncRNA編碼的一種新型內源性微肽,對于HNSCC的發生發展起著重要的調控作用。
創新型藥物作為自主研發和具有自主知識產權的藥物,對于我國建設創新型國家的進一步發展具有重要意義。MIAC作為HNSCC的調控分子,在創新型HNSCC藥物的開發中具有重大的研究意義:MIAC可作為潛在診斷標志物來制備診斷HNSCC的試劑盒,為HNSCC的診斷和預防提供新的途徑;而MIAC作為調控HNSCC的小分子多肽,也可通過偶聯化學藥物的方式來提高治療HNSCC藥物的靶向性和穩定性。此外,MIAC在其他腫瘤和疾病中的意義還有待探究。
4 研究潛在微肽的相關技術
當前的研究表明,在動物基因組中約1.2%的sORF可被轉錄,其中只有約1/3能被翻譯[4]。這些占比很小的功能性sORF理論上也可產生成千上萬個未被表征的微肽,即使這些微肽中只有小部分具有生物活性,仍意味著可能存在數百甚至數千種有生物學功能的微肽。因此,當前面臨的挑戰是如何識別具有生物活性的sORF及其微肽。下面將總結介紹研究潛在微肽的相關技術,這些技術可用于鑒定可能編碼微肽的sORF。
4.1 生物信息學分析
生物信息學(bioinformatics)是利用生物數據來開發算法和軟件的交叉學科,目前運用生物信息分析技術,基于保守序列可從非編碼區域預測具有編碼蛋白能力的sORFs,生物信息分析技術還依據sORFs序列中的密碼子含量和編碼特征以區分sORFs編碼區與非編碼區[39]。我們可以利用生物信息數據庫挖掘相關數據,如ATCG、UCSC等,而常用于預測sORFs的分析軟件有CPAT、ORFfinder、PhyloCSF、uPEPperoni[40~44]等。Niu等[45]運用ORFfinder在人源基因中預測到一條54個堿基的sORF,后續實驗證明該sORF可編碼長為17個氨基酸的功能性微肽miPEP155。miPEP155可調節抗原呈遞細胞(antigen-presenting cells, APC)中的抗原轉運和呈遞,可作為自身免疫性疾病的候選藥物[45]。
4.2 核糖體圖譜分析
核糖體圖譜分析(ribosome profiling)可用來識別具有翻譯潛力的sORF,該技術的原理是翻譯核糖體可保護長為20~30個核苷酸的mRNA片段免受核酸酶的消化[46]。然而,Wilson等[47]的研究表明某些sORF雖然與核糖體結合但并不進行翻譯。于是,在Ribo-Seq的基礎上改良而開發了多聚核糖體分析(Poly-Ribo-Seq),使用這種技術可以分離由多個核糖體結合并被主動翻譯的mRNA,由此可將不進行翻譯的單核糖體-mRNA復合物區分開[48]。此外,Guttman等[49]還開發了核糖體釋放分數(ribosome release score, RRS)作為翻譯的度量指標,相比于終止密碼子下游的非編碼區,編碼區與核糖體具有更高的相關性,由此可區分編碼轉錄本和非編碼轉錄本。Chen等[26]利用核糖體圖譜分析發現了3455個非經典CDS,其中的96%是編碼小于100個氨基酸的微肽。
4.3 質譜和蛋白質組學
最近基于質譜(mass spectrometry, MS)的蛋白質組學(proteomics)也用于發現和驗證內源表達的微肽。該技術的基本原理是通過測量氣態的離子化肽或蛋白質的質荷比來研究蛋白質的表達和相互作用,因此MS通過檢測從sORF翻譯的微肽,從而直接驗證轉錄產物編碼蛋白質的潛力[50]。基于MS的蛋白質組學在研究和鑒定新型微肽方面已取得實質性的進展,Chen等[26]通過基于MS的HLA-I肽組學,發現240個微肽可被HLA-I提呈,表明這些肽會進入HLA-I呈遞途徑并可能擁有免疫原性。但MS在技術上仍然存在一定限制,樣品制備過程中的消化酶決定微肽片段化的方式,片段過小不能產生足夠的檢測信號,片段過大則無法用于MS分析,小片段的微肽在樣品制備過程中還存在丟失的可能[39]。因此,需要進一步結合核糖體圖譜分析等其他分析方法以確定新型微肽的存在。
4.4 蛋白質基因組學
蛋白質基因組學(proteogenomics)是在基于蛋白質組學分析的基礎上結合基因組學和轉錄組學的分析方法,通過追溯基因組和轉錄本中的蛋白質/微肽的預測序列,來鑒定基因的翻譯和表達情況[51]。在蛋白質基因組學研究中,Slavoff等[31]從人白血病細胞系K562細胞中發現了86個未報道過的微肽。
4.5 其他相關技術
為證實sORF是否具有編碼蛋白產生微肽的能力,可以使用以下幾種方法來進行驗證。在理想的狀態下,可以設計目的微肽的抗體并通過免疫組化或蛋白質印記來驗證其特異性[52]。例如,Li等[24]通過制備MIAC的單克隆抗體以檢測MIAC的內源表達。對于不能產生抗體的目的微肽而言,也可采用CRISPR/Cas9基因編輯技術。該技術通過同源定向修復將FLAG/MYC或其他標簽添加到預測的sORF,從而產生融合蛋白,再通過檢測融合蛋白以驗證目的微肽的存在[52]。為確定轉錄本中的sORF是否翻譯為微肽,Polycarpou-Schwarz等[20]在CASIMO1編碼序列的C端插入了一個Flag標簽,并通過anti-Flag抗體檢測到了CASIMO1-Flag的表達。此外,還可通過體外翻譯來評估sORF編碼蛋白的能力,通過多方面的驗證以確定sORF是否具有編碼能力。
5 結語與展望
大規模基因組測序的迅速發展促進人們對基因組的深入研究,揭示sORF序列的復雜性。微肽的發現使人們認識到這些重要小肽的生物學作用,它們在生命活動及疾病的發展進程中起著重要調控作用。微肽可以以配體或信號分子的形式發揮作用,也可與其他蛋白質相結合,通過遮蔽受體蛋白的關鍵位點或影響受體蛋白的活性從而發揮調控作用,如前所述的HOXB-AS3 peptide[21]通過競爭性結合hnRNP A1中RGG基序的精氨酸殘基,阻斷精氨酸殘基與丙酮酸激酶M(pyruvate kinase M, PKM)的結合,從而抑制結腸癌細胞的葡萄糖代謝進程。SMIM30[22]與非受體酪氨酸激酶SRC/YES1結合,驅動其膜錨定和磷酸化,激活下游絲裂原活化蛋白激酶(mitogen-activated protein kinase, MAPK)信號通路,通過調節細胞增殖和遷移促進肝癌的發生發展。
然而,對于功能性微肽及其作用機制的探索仍處于起步狀態,雖然已經存在許多挖掘未知微肽的生物技術,但由于微肽本身分子量小、表達豐度低等特點,這些生物技術的應用仍然存在局限性,生命體中仍有大量的微肽等待被發現。相信在未來的研究中,能克服檢測障礙,進一步拓展和優化挖掘微肽的技術與方法。另一方面,還需要進行大量的工作以闡明微肽的生物學作用,并對其作用機制開展進一步的研究,以便應用于正常生理功能的探索及疾病的臨床診療。
[1] Crick F. Central dogma of molecular biology., 1970, 227(5258): 561–563.
[2] Djebali S, Davis CA, Merkel A, Dobin A, Lassmann T, Mortazavi A, Tanzer A, Lagarde J, Lin W, Schlesinger F, Xue CH, Marinov GK, Khatun J, Williams BA, Zaleski C, Rozowsky J, R?der M, Kokocinski F, Abdelhamid RF, Alioto T, Antoshechkin I, Baer MT, Bar NS, Batut P, Bell K, Bell I, Chakrabortty S, Chen X, Chrast J, Curado J, Derrien T, Drenkow J, Dumais E, Dumais J, Duttagupta R, Falconnet E, Fastuca M, Fejes-Toth K, Ferreira P, Foissac S, Fullwood MJ, Gao H, Gonzalez D, Gordon A, Gunawardena H, Howald C, Jha S, Johnson R, Kapranov P, King B, Kingswood C, Luo OJ, Park E, Persaud K, Preall JB, Ribeca P, Risk B, Robyr D, Sammeth M, Schaffer L, See LH, Shahab A, Skancke J, Suzuki AM, Takahashi H, Tilgner H, Trout D, Walters N, Wang HE, Wrobel J, Yu YB, Ruan XA, Hayashizaki Y, Harrow J, Gerstein M, Hubbard T, Reymond A, Antonarakis SE, Hannon G, Giddings MC, Ruan YJ, Wold B, Carninci P, Guigó R, Gingeras TR. Landscape of transcription in human cells., 2012, 489(7414): 101–108.
[3] ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome., 2012, 489(7414): 57–74.
[4] Couso JP, Patraquim P. Classification and function of small open reading frames., 2017, 18(9): 575–589.
[5] Anderson DM, Makarewich CA, Anderson KM, Shelton JM, Bezprozvannaya S, Bassel-Duby R, Olson EN. Widespread control of calcium signaling by a family of SERCA-inhibiting micropeptides., 2016, 9(457): ra119.
[6] Anderson DM, Anderson KM, Chang CL, Makarewich CA, Nelson BR, McAnally JR, Kasaragod P, Shelton JM, Liou J, Bassel-Duby R, Olson EN. A micropeptide encoded by a putative long noncoding RNA regulates muscle performance., 2015, 160(4): 595–606.
[7] Magny EG, Pueyo JI, Pearl FMG, Cespedes MA, Niven JE, Bishop SA, Couso JP. Conserved regulation of cardiac calcium uptake by peptides encoded in small open reading frames., 2013, 341(6150): 1116–1120.
[8] Nelson BR, Makarewich CA, Anderson DM, Winders BR, Troupes CD, Wu FF, Reese AL, McAnally JR, Chen XW, Kavalali ET, Cannon SC, Houser SR, Bassel-Duby R, Olson EN. A peptide encoded by a transcript annotated as long noncoding RNA enhances SERCA activity in muscle., 2016, 351(6270): 271–275.
[9] Lee C, Zeng J, Drew BG, Sallam T, Martin-Montalvo A, Wan JX, Kim SJ, Mehta H, Hevener AL, de Cabo R, Cohen P. The mitochondrial-derived peptide MOTS-c promotes metabolic homeostasis and reduces obesity and insulin resistance., 2015, 21(3): 443–454.
[10] Makarewich CA, Baskin KK, Munir AZ, Bezprozvannaya S, Sharma G, Khemtong C, Shah AM, McAnally JR, Malloy CR, Szweda LI, Bassel-Duby R, Olson EN. MOXI is a mitochondrial micropeptide that enhances fatty acid beta-oxidation., 2018, 23(13): 3701–3709.
[11] Stein CS, Jadiya P, Zhang XM, McLendon JM, Abouassaly GM, Witmer NH, Anderson EJ, Elrod JW, Boudreau RL. Mitoregulin: a lncRNA-encoded microprotein that supports mitochondrial supercomplexes and respiratory efficiency., 2018, 23(13): 3710–3720.e8.
[12] Zhang Q, Vashisht AA, O'Rourke J, Corbel SY, Moran R, Romero A, Miraglia L, Zhang J, Durrant E, Schmedt C, Sampath SC, Sampath SC. The microprotein Minion controls cell fusion and muscle formation., 2017, 8: 15664.
[13] Bi PP, Ramirez-Martinez A, Li H, Cannavino J, McAnally JR, Shelton JM, Sánchez-Ortiz E, Bassel-Duby R, Olson EN. Control of muscle formation by the fusogenic micro-peptide myomixer., 2017, 356(6335): 323–327.
[14] Pauli A, Norris ML, Valen E, Chew GL, Gagnon JA, Zimmerman S, Mitchell A, Ma J, Dubrulle J, Reyon D, Tsai SQ, Joung JK, Saghatelian A, Schier AF. Toddler: an embryonic signal that promotes cell movementApelin receptors., 2014, 343(6172): 1248636.
[15] Kondo T, Hashimoto Y, Kato K, Inagaki S, Hayashi S, Kageyama Y. Small peptide regulators of actin-based cell morphogenesis encoded by a polycistronic mRNA., 2007, 9(6): 660–665.
[16] Galindo MI, Pueyo JI, Fouix S, Bishop SA, Couso JP. Peptides encoded by short ORFs control development and define a new eukaryotic gene family., 2007, 5(5): e106.
[17] Matsumoto A, Pasut A, Matsumoto M, Yamashita R, Fung J, Monteleone E, Saghatelian A, Nakayama KI, Clohessy JG, Pandolfi PP. mTORC1 and muscle regeneration are regulated by the-encoded SPAR polypeptide., 2017, 541(7636): 228–232.
[18] Pueyo JI, Magny EG, Sampson CJ, Amin U, Evans IR, Bishop SA, Couso JP. Hemotin, a regulator of phago-cytosis encoded by a small ORF and conserved across metazoans., 2016, 14(3): e1002395.
[19] Chu Q, Martinez TF, Novak SW, Donaldson CJ, Tan D, Vaughan JM, Chang TN, Diedrich JK, Andrade L, Kim A, Zhang T, Manor U, Saghatelian A. Regulation of the ER stress response by a mitochondrial microprotein., 2019, 10(1): 4883.
[20] Polycarpou-Schwarz M, Gro? M, Mestdagh P, Schott J, Grund SE, Hildenbrand C, Rom J, Aulmann S, Sinn HP, Vandesompele J, Diederichs S. The cancer-associated microprotein CASIMO1 controls cell proliferation and interacts with squalene epoxidase modulating lipid droplet formation., 2018, 37(34): 4750–4768.
[21] Huang JZ, Chen M, Chen D, Gao XC, Zhu S, Huang HY, Hu M, Zhu HF, Yan GR. A peptide encoded by a putative lncRNAsuppresses colon cancer growth., 2017, 68(1): 171–184.e6.
[22] Pang YN, Liu ZY, Han H, Wang BL, Li W, Mao CB, Liu SR. Peptide SMIM30 promotes HCC development by inducing SRC/YES1 membrane anchoring and MAPK pathway activation., 2020, 73(5): 1155–1169.
[23] Wu SQ, Zhang LY, Deng JQ, Guo BB, Li F, Wang YR, Wu R, Zhang SH, Lu JC, Zhou YF. A novel micropeptide encoded by Y-linkedlinks cigarette smoking and AR signaling in male esophageal squamous cell carcinoma., 2020, 80(13): 2790–2803.
[24] Li MW, Li X, Zhang YN, Wu HM, Zhou HZ, Ding X, Zhang XM, Jin XR, Wang Y, Yin XQ, Li CC, Yang PW, Xu HM. Micropeptide MIAC inhibits HNSCC progression by Interacting with aquaporin 2., 2020, 142(14): 6708–6716.
[25] Sieber P, Platzer M, Schuster S. The definition of open reading frame revisited., 2018, 34(3): 167– 170.
[26] Chen J, Brunner AD, Cogan JZ, Nu?ez JK, Fields AP, Adamson B, Itzhak DN, Li JY, Mann M, Leonetti MD, Weissman JS. Pervasive functional translation of noncanonical human open reading frames., 2020, 367(6482): 1140–1146.
[27] Jackson R, Kroehling L, Khitun A, Bailis W, Jarret A, York AG, Khan OM, Brewer JR, Skadow MH, Duizer C, Harman CCD, Chang L, Bielecki P, Solis AG, Steach HR, Slavoff S, Flavell RA. The translation of non-canonical open reading frames controls mucosal immunity., 2018, 564(7736): 434–438.
[28] Orr MW, Mao YH, Storz G, Qian SB. Alternative ORFs and small ORFs: shedding light on the dark proteome., 2020, 48(3): 1029–1042.
[29] Ladoukakis E, Pereira V, Magny EG, Eyre-Walker A, Couso JP. Hundreds of putatively functional small open reading frames in drosophila., 2011, 12(11): R118.
[30] Andrews SJ, Rothnagel JA. Emerging evidence for functional peptides encoded by short open reading frames., 2014, 15(3): 193–204.
[31] Slavoff SA, Mitchell AJ, Schwaid AG, Cabili MN, Ma J, Levin JZ, Karger AD, Budnik BA, Rinn JL, Saghatelian A. Peptidomic discovery of short open reading frame- encoded peptides in human cells., 2013, 9(1): 59–64.
[32] Dufresne SS, Dumont NA, Boulanger-Piette A, Fajardo VA, Gamu D, Kake-Guena SA, David RO, Bouchard P, Lavergne é, Penninger JM, Pape PC, Tupling AR, Frenette J. Muscle RANK is a key regulator of Ca2+storage, SERCA activity, and function of fast-twitch skeletal muscles., 2016, 310(8): C663– C672.
[33] Gusic M, Prokisch H. ncRNAs: new players in mitochondrial health and disease?, 2020, 11: 95.
[34] Krauss RS, Joseph GA, Goel AJ. Keep your friends close: cell-cell contact and skeletal myogenesis., 2017, 9(2): a029298.
[35] Millay DP, Gamage DG, Quinn ME, Min YL, Mitani Y, Bassel-Duby R, Olson EN. Structure-function analysis of myomaker domains required for myoblast fusion., 2016, 113(8): 2116–2121.
[36] Read C, Nyimanu D, Williams TL, Huggins DJ, Sulentic P, Macrae RGC, Yang PR, Glen RC, Maguire JJ, Davenport AP. International union of basic and clinical pharmacology. CVII. structure and pharmacology of the Apelin receptor with a recommendation that Elabela/Toddler is a second endogenous peptide ligand., 2019, 71(4): 467–502.
[37] Walter P, Ron D. The unfolded protein response: from stress pathway to homeostatic regulation., 2011, 334(6059): 1081–1086.
[38] D'Lima NG, Ma J, Winkler L, Chu Q, Loh KH, Corpuz EO, Budnik BA, Lykke-Andersen J, Saghatelian A, Slavoff SA. A human microprotein that interacts with the mRNA decapping complex., 2017, 13(2): 174– 180.
[39] Makarewich CA, Olson EN. Mining for micropeptides., 2017, 27(9): 685–696.
[40] Li X, Li MW, Zhang YN, Xu HM. Common cancer genetic analysis methods and application study based on TCGA database., 2019, 41(3): 234–242.
李鑫, 李夢瑋, 張依楠, 徐寒梅. 常用腫瘤基因分析方法及基于TCGA數據庫的分析應用. 遺傳, 2019, 41(3): 234–242.
[41] Wang LG, Park HJ, Dasari S, Wang SQ, Kocher JP, Li W. CPAT: coding-potential assessment tool using an alignment- free logistic regression model., 2013, 41(6): e74.
[42] Hanada K, Akiyama K, Sakurai T, Toyoda T, Shinozaki K, Shiu SH. sORF finder: a program package to identify small open reading frames with high coding potential., 2010, 26(3): 399–400.
[43] Lin MF, Jungreis I, Kellis M. PhyloCSF: a comparative genomics method to distinguish protein coding and non- coding regions., 2011, 27(13): i275–i282.
[44] Skarshewski A, Stanton-Cook M, Huber T, Al Mansoori S, Smith R, Beatson SA, Rothnagel JA. uPEPperoni: an online tool for upstream open reading frame location and analysis of transcript conservation., 2014, 15: 36.
[45] Niu LM, Lou FZ, Sun Y, Sun LB, Cai XJ, Liu ZY, Zhou H, Wang H, Wang ZK, Bai J, Yin QQ, Zhang JX, Chen LJ, Peng DH, Xu ZY, Gao YY, Tang SB, Fan L, Wang HL. A micropeptide encoded by lncRNAsuppresses autoimmune inflammationmodulating antigen pre-sentation., 2020, 6(21): eaaz2059.
[46] Ingolia NT. Ribosome footprint profiling of translation throughout the genome., 2016, 165(1): 22–33.
[47] Wilson BA, Masel J. Putatively noncoding transcripts show extensive association with ribosomes., 2011, 3: 1245–1252.
[48] Aspden JL, Eyre-Walker YC, Phillips RJ, Amin U, Mumtaz MAS, Brocard M, Couso JP. Extensive translation of small open reading frames revealed by Poly-Ribo-Seq., 2014, 3: e03528.
[49] Guttman M, Russell P, Ingolia NT, Weissman JS, Lander ES. Ribosome profiling provides evidence that large noncoding RNAs do not encode proteins., 2013, 154(1): 240–251.
[50] Aebersold R, Mann M. Mass-spectrometric exploration of proteome structure and function., 2016, 537(7620): 347–355.
[51] Menschaert G, Feny? D. Proteogenomics from a bioinformatics angle: a growing field., 2017, 36(5): 584–599.
[52] Housman G, Ulitsky I. Methods for distinguishing between protein-coding and long noncoding RNAs and the elusive biological purpose of translation of long noncoding RNAs., 2016, 1859(1): 31–40.
[53] Yang J, Meng XD, Pan JC, Jiang N, Zhou CW, Wu ZH, Gong ZH. CRISPR/Cas9-mediated noncoding RNA editing in human cancers., 2018, 15(1): 35–43.
Progress on sORF-encoded micropeptides
Xiangying Chen, Mengwei Li, Ying Wang, Quan Chen, Hanmei Xu
Existing research has shown that there are a large amount of non-coding RNAs (ncRNAs) in organisms. Short open reading frames (sORFs) abundantly exist in molecular sequences inaccurately annotated as ncRNAs. Several sORFs can be transcribed and translated into evolutionarily conserved micropeptides, which were ignored in previous studies due to short sequence lengths and the limitations of research techniques. To date, sORF-encoded micropeptides with various functions have been found to play important roles in regulating vital biological activities. This article reviews the functional micropeptides which have been found in recent years, introduces the new micropeptide designated as MIAC that we have discovered and describes the related technologies for mining potential micropeptides, thereby providing insights and references for new micropeptide discovery for researchers.
non-coding RNA; small open reading frames; micropeptides
2021-05-08;
2021-07-06
中國藥科大學天然藥物活性組分與藥效國家重點實驗(編號:SKLNMZZCX201821, SKLNMZZ202028),國家科技重大新藥開發項目(編號:2019ZX09301124, 2019ZX09201001, 2019ZX09301-110)和中國博士后科學基金資助項目(編號:2017M621884, 2020M681787) 資助[Supported by the Project Program of State Key Laboratory of Natural Medicines (Nos. SKLNMZZCX201821, SKLNMZZ202028), the National Science and Technology Major Projects of New Drugs (Nos. 2019ZX09301124, 2019ZX09201001, 2019ZX09301-110) and China Postdoctoral Science Foundation (Nos. 2017M621884, 2020M681787)]
陳相穎,在讀碩士研究生,專業方向:微生物與生化藥學。E-mail: cxy000111@qq.com
徐寒梅,博士,教授,研究方向:多肽類藥物研究與開發。E-mail: 13913925346@126.com
10.16288/j.yczz.21-167
2021/7/23 16:26:21
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210723.0937.002.html
(責任編委: 薛宇)

