人工模擬條件下環境DNA 宏條形碼技術的定量分析初探*
牟 銘 李 昂 趙新寧 柳淑芳① 莊志猛
(1. 上海海洋大學水產與生命學院 上海 201306;2. 中國水產科學研究院黃海水產研究所 青島海洋科學與技術試點國家實驗室海洋漁業科學與食物產出過程功能實驗室 山東 青島 266071)
環境 DNA 宏條形碼技術(environmental DNA metabarcoding)可以通過高通量測序對環境 DNA(eDNA)進行多物種分類鑒定。隨著eDNA 宏條形碼技術的應用和發展,該技術對群落中單一或組合物種的定量評估逐漸顯現出優勢。無論是水族箱或中觀實驗等小型生態系統(Minamotoet al; Moyeret al, 2014;Pilliodet al, 2013; Thomsenet al, 2012; Doiet al,2017),還是江河湖泊等自然淡水系統(Doiet al, 2017;Lacoursiererousselet al, 2016),甚至是開放式的海洋系統(Joet al, 2017),研究人員都嘗試過使用eDNA宏條形碼技術測算種群相對數量。大量研究表明,特定環境中物種的個體數量和基于eDNA 宏條形碼技術獲得的高通量測序reads 數之間具有明顯的相關性。Evans 等(2016)設計了一個包含魚類和兩棲類的中觀實驗,證明水體中物種個體數或生物量相對豐度與eDNA 宏條形碼技術獲得的高通量測序reads 數呈正相關;Hanfling 等(2016)對比較了一個天然湖泊的長期監測魚類豐度結果與eDNA 宏條形碼技術得到的高通量測序reads 數之間的相關性,結果表明二者呈正相關;Thomsen 等(2016)研究發現,當將魚類的eDNA 高通量測序reads 數聚類到“科”的分類等級時,eDNA 宏條形碼技術測得的深海生境中的生物量與傳統拖網捕撈數據比較吻合。可以預見,eDNA 宏條形碼技術具有快速測算群落中物種豐度的潛能,將成為資源保護和管理中具有應用前景的調查工具。
然而,通過對已發表的相關文獻進行梳理和歸納,發現當前大多數利用eDNA 高通量測序結果的reads 數來評估自然環境中生物相對數量的研究并不能得到明確的量化關系結果(Limet al, 2016)。究其原因,是因為從生物到eDNA 以及從eDNA 再到高通量測序結果的2 個過程中的數學關系難以理清。其中,實驗室操作過程中的主要問題是:(1)自然水體中,不同濃度 eDNA 富集效率不同(Kellyet al, 2016);(2)PCR 擴增過程中,通用引物對不同物種DNA 存在偏倚性(Elbrechtet al, 2015; Pinolet al, 2015)。假定水體中的eDNA 全部回收,且PCR 擴增時不存在引物偏倚性,這種理想狀態下的水體中eDNA 組成與其高通量測序reads 數是否完全呈線性關系?為了探究這個問題,本研究在實驗室可控條件下,選擇2 個同屬近緣種,對其DNA 樣品進行不同比例混合,模擬從自然水體中富集到的eDNA 復合樣品,既保證了樣品的回收率,又降低了引物偏倚的干擾。以此為模板,探究eDNA 宏條形碼技術檢測種群相對數量的準確性,旨在為驗證eDNA 宏條形碼技術監測水生生物資源量的可行性提供直接證據。
1 材料與方法
1.1 實驗方案設計
1.1.1 實驗樣品的選擇 鑒于通用引物對不同物種的擴增效率有所不同,本研究實驗樣品選取2 個同屬近緣物種用于制備模擬eDNA 樣品,以確保將PCR過程中因引物對物種DNA 的偏移引起的實驗誤差降低到最小程度。
實驗樣品選擇了2 種常見對蝦:凡納濱對蝦(Penaeus vannamei)和墨吉對蝦(Penaeus merguiensis),它們同為節肢動物門(Arthropoda)、軟甲綱(Malacostraca)、十足目(Decapoda)、對蝦科(Penaeidae)、對蝦屬(Penaeus),是較為常見的同屬近緣種。
1.1.2 模板DNA 的提取 采用傳統的酚-氯仿-異戊醇方法分別提取2 種對蝦肌肉組織DNA,經純化后使用超微量核酸蛋白分析儀(GE,美國)檢測濃度,稀釋DNA 濃度至20 ng/μL (±1%),-20℃保存備用。
1.1.3 模擬eDNA 的制備 人工模擬eDNA 復合樣品,共設7 個實驗組:將凡納濱對蝦和墨吉對蝦的DNA 模板分別按照1∶1、1∶2、1∶4、1∶8、1∶10、1∶50 和1∶100 的比例均勻混合,且混合后DNA 終濃度一致。設陽性對照2 組:分別選用凡納濱對蝦和墨吉對蝦DNA 樣品。設陰性對照1 組:用純水代替DNA 樣品。每組樣品設3 個重復。
將上述制備的eDNA 復合樣品作為eDNA 宏條形碼檢測的模板DNA。
1.2 通用引物的選取
考慮到后續高通量測序及DNA 條形碼數據庫信息資源的豐富度,本研究選取了Leray 等(2013)推薦的長度為313 bp 的線粒體COⅠ基因序列作為待測eDNA 目標片段,該片段長度適中、數據資源豐富、物種鑒定特異性好,且引物通用性強。通用引物序列見表1,由華大基因有限公司合成。
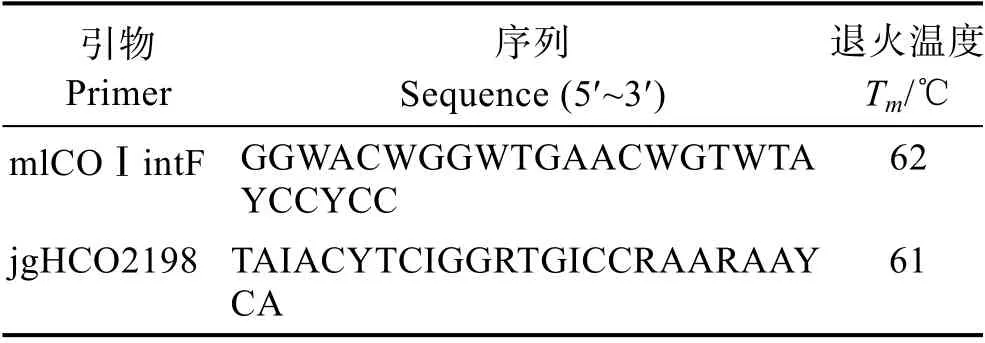
表1 COⅠ基因PCR 擴增引物信息Tab.1 Primer sets of COⅠ used for PCR amplification
1.3 PCR 擴增
PCR 擴 增 體 系:2×RapidTaqMaster Mix(Vazyme) 25 μL,上下游引物(mlCOⅠintF/jgHCO2198)(10mmol/L)各2 μL,DNA 模板5 μL,無菌水補齊總體積至50 μL。
為了增強擴增特異性,使用Touch down PCR 反應程序:95℃預變性10 min;95℃變性30 s,62℃退火30 s,72℃延伸30 s,16 個循環;95℃變性30 s,50℃退火30 s,72℃延伸30 s,25 個循環;72℃延伸3 min;4℃保存。
用1%瓊脂糖凝膠電泳檢測PCR 產物質量,選用DL2000 DNA marker,目標條帶約為300 bp。挑選電泳條帶單一且明亮的PCR 產物用于高通量測序。
1.4 測序及結果注釋
將所得PCR 產物送至青島歐易生物科技有限公司,進行高通量測序。采用Illumina Miseq 平臺測序。
按照97%相似性對非重復序列(不含單序列)進行OTU(operational taxonomic units)聚類,在聚類過程中去除嵌合體(chimera,融合了2 個不同序列信息的序列)、異源雙鏈序列等干擾序列,得到OTU 聚類結果,然后對不同OTU 的代表序列進行結果注釋。在結果注釋時,參照 NCBI 公共數據庫(https://www.ncbi.nlm.nih.gov/)來進行分類學信息的標注,同時使用中國重要漁業生物DNA 條形碼信息平臺(http://www.fisherybarcode.cn)對注釋結果進行輔助校正。
1.5 數據處理
統計每個樣品高通量測序的原始reads 數,以所有樣本中reads 數最低值為基準,進行抽平,使每個樣品測序結果的reads 數相同,用于后續數據分析。
在推導數學關系時,設:eDNA 樣品來自2 個物種,分別為A、B;實際參與PCR 反應的底物模板DNA 的量為n;參與PCR 反應的底物模板DNA 理論量為x;每個循環中,參與擴增的2 個物種底物模板DNA 比率為λ;擴增的循環數為c;PCR 產物的高通量測序reads 數為y。假定理想情況為:在PCR 擴增的不同循環中,參與擴增的2 個物種底物模板DNA比率固定;PCR 擴增全程都保持底數為2 的指數增長型(反應物足夠),則高通量測序reads 數可以反應eDNA 樣品中2 個物種的相對量,即推得公式(1):
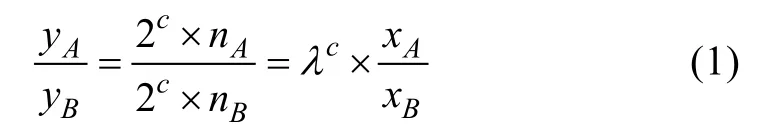
經變形得公式(2):

2 結果與分析
2.1 PCR 擴增結果
以人工模擬的7 組eDNA 復合樣品及對照組樣品為模板,使用eDNA 宏條形碼通用引物進行PCR 擴增,經1%瓊脂糖凝膠電泳檢測,結果顯示,陽性對照和7 個實驗組均成功擴增出300 bp 左右的目標片段,陰性對照無擴增產物(圖1)。
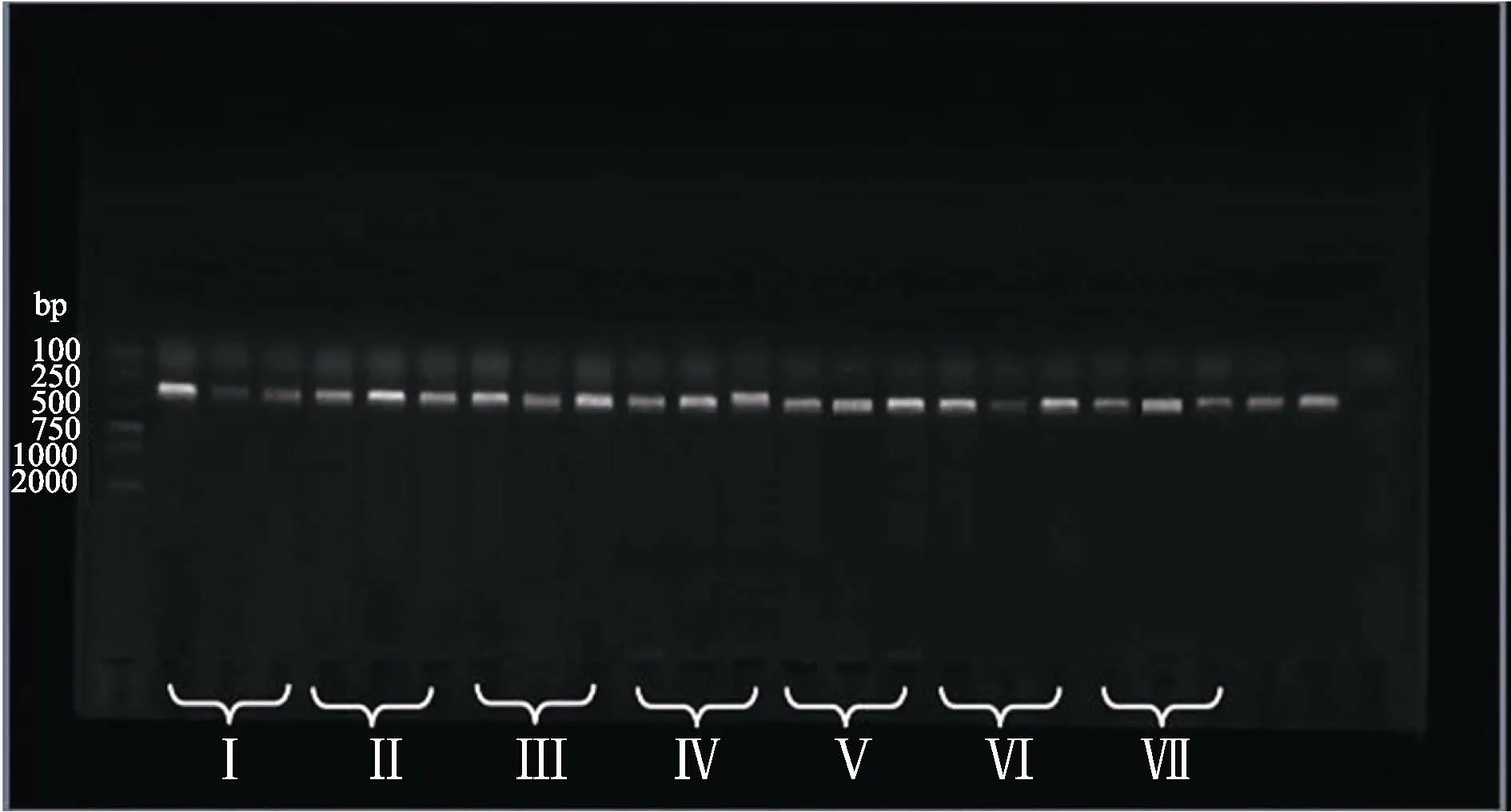
圖1 瓊脂糖凝膠電泳檢測結果Fig.1 Results of agarose gel electrophoresis
2.2 測序數據
2.2.1 高通量測序結果 本研究對7 個實驗組共21 個PCR 產物進行了高通量測序。統計高通量測序獲得的序列長度分布,顯示99.80%以上序列的長度分布于301~350 bp 之間(圖2)。抽平后共計獲得有效序列674,205 條,序列平均長度為312.41 bp。
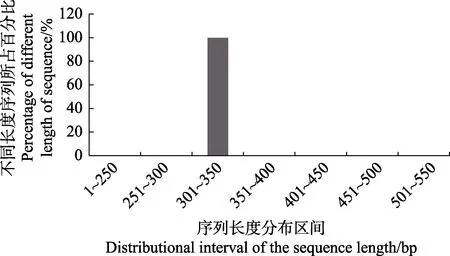
圖2 測序數據長度分布統計Fig.2 Result of distributional interval of the sequence length
2.2.2 分類學信息注釋及reads 數統計結果 將每個樣品reads 總數抽平后,對674,205 條序列進行分類學信息注釋,保留有效序列(序列長度在301~350之間,且分類學信息注釋結果是墨吉對蝦和凡納濱對蝦)的reads。各實驗組高通量測序reads 數及其分類注釋結果詳見表2。實驗組I 的3 組平行樣共獲得94,774 條序列,每個樣品注釋出的墨吉對蝦和凡納濱對蝦平均reads 數分別為11,097 和20,494,二者的比值為13/24;實驗組Ⅱ的3 組平行樣共獲得95,208 條序列,二者reads 數比值為18/23;實驗組Ⅲ的3 組平行樣共獲得 95,572 條序列,二者 reads 數比值為77/73;實驗組Ⅳ的3 組平行樣共獲得95,250 條序列,二者reads 數比值為67/44;實驗組Ⅴ的3 組平行樣共獲得94,408 條序列,二者reads 數比值為43/24;實驗組Ⅵ的3 組平行樣共獲得94,640 條序列,二者reads數比值為 127/35;實驗組Ⅶ的 3 組平行樣共獲得95,526 條序列,二者reads 數比值為187/23。
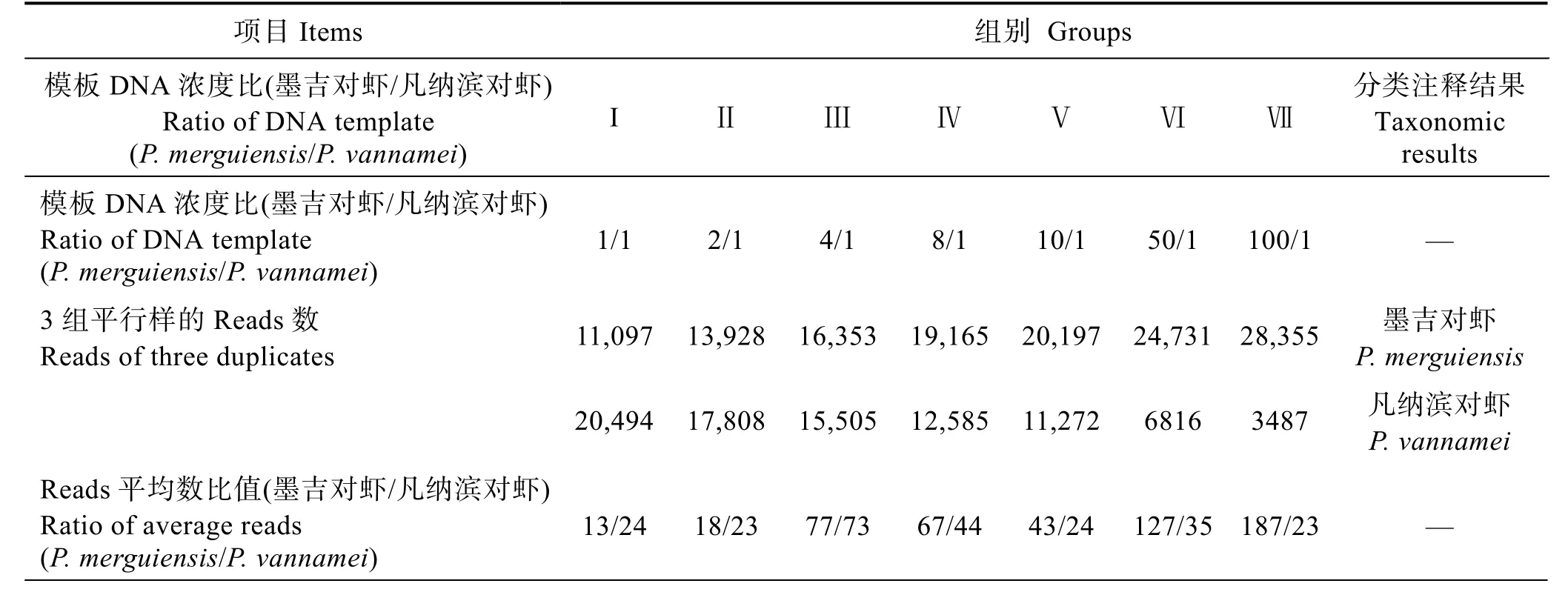
表2 各實驗組獲得的序列數及分類學信息注釋Tab.2 Results of taxonomic information and numbers of reads
2.2.3 混合模板PCR 擴增的引物偏倚率 當模板中2 個物種DNA 濃度比例為1∶1 (墨吉對蝦/凡納濱對蝦)時,2 個物種的模板DNA 含量相同,擴增過程中主要是引物對不同底物的偏好引起的擴增產物差別,此時主要是引物偏倚引起的reads 數差距。根據公式(2)變形得:

代入擴增循環數,由reads 平均數比值,計算得到每個循環中參與反應的2 個物種模板DNA 的平均比率為66/67,換言之,PCR 擴增的每個循環中,平均偏倚率為1/67,約為1.5%,即該引物在同一PCR體系中,每次退火過程對墨吉對蝦DNA 模板的結合率比凡納濱對蝦DNA 模板大約少1.5%。
2.2.4 eDNA 組成與高通量測序reads 數的函數關系
以各實驗組設置的底物中墨吉對蝦DNA 與凡納濱對蝦DNA 的比值為橫坐標,以各實驗組樣品高通量測序結果注釋的墨吉對蝦與凡納濱對蝦reads 平均數的比值為縱坐標,繪制eDNA 組成與高通量測序reads 數的函數關系圖。在公式(2)的前提假設條件下,在高通量結果中,2 種對蝦的reads 數之比和初始PCR模板DNA 中2 種對蝦的DNA 比例應呈現y=kx線性關系,對表2 中的7 個實驗組的reads 平均數比值和PCR 模板DNA 比值的2 組數據進行一元線性回歸分析,最終得出eDNA 組成與高通量測序reads 數的函數關系:y=0.0716x+0.7043 (r2=0.9824)(圖3)。
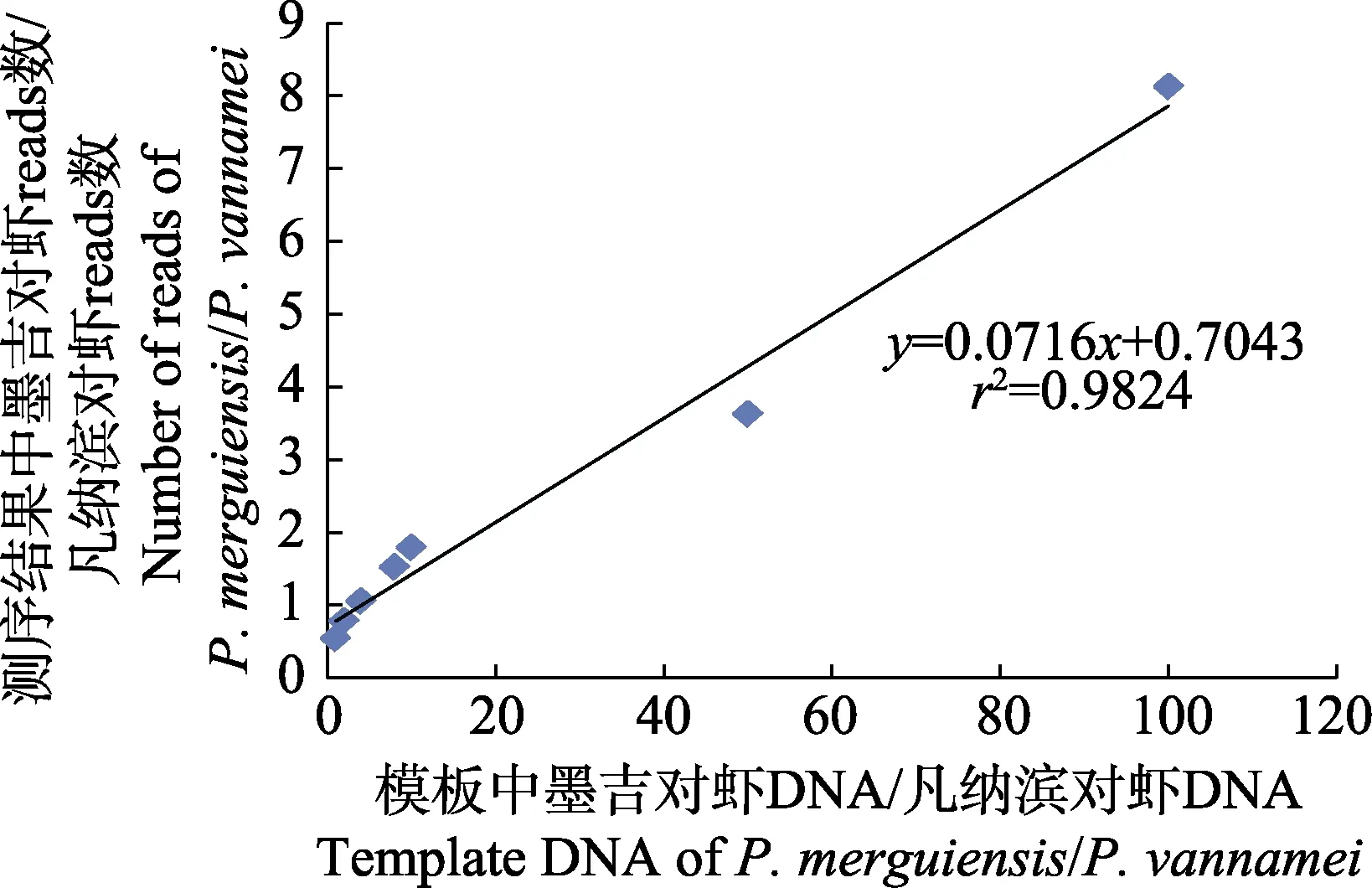
圖3 2 個物種高通量測序結果的序列數比值與模板比例的關系Fig.3 The curve of ratio of two species'reads and ratio of template DNA
3 討論
利用eDNA 宏條形碼技術來評估水體中不同物種的相對數量,是該技術最具潛力的應用前景之一。與傳統調查方法相比,eDNA 宏條形碼技術具有操作簡便、省時高效且對自然資源破壞小等特點。如何使用該技術來監測水體中的生物量,一直是學界關注的焦點,但水體中的eDNA 難以完全富集,且富集到的eDNA 成分復雜;PCR 擴增時,引物對不同種類DNA模板的偏好還會導致擴增效率不同,這些問題亦是eDNA 宏條形碼技術推廣應用的最大障礙。為運用eDNA 宏條形碼技術檢測環境生物相對數量而設計的中觀實驗,大都是在嘗試推導生物量與高通量測序結果reads 數的數學關系,主要包括環境中生物量與eDNA 量、環境中eDNA 量與提取的eDNA 量和提取的eDNA 模板與PCR 產物的高通量測序結果等3 個對應關系,其中任何一個關系不明確,都將影響eDNA 宏條形碼技術的檢測結果,所以,必須逐步理清這3 種對應關系的函數關系,進而得到“高魯棒性”(模式體系固定且成熟,效果準確且穩定)的eDNA宏條形碼方法應用體系。
分解困難是解決問題的有效方法之一,剖析DNA宏條形碼技術全過程,當只關注富集到的eDNA 擴增及測序時,如果能創造一種eDNA 成分簡單、無引物偏好影響的理想條件,進而探索eDNA 高通量測序reads 數和底物eDNA 含量之間的數學關系,便可以收獲“柳暗花明”的結果。
3.1 混合模板PCR 擴增的引物偏倚性
影響多物種eDNA 混合模板PCR 擴增效率的主要原因之一,便是PCR 過程中引物對不同模板DNA堿基組成存在偏好性(Kanagawa, 2003)。理論上,引物的每個脫氧核苷酸和模板DNA 鏈上同源區段的每個脫氧核苷酸可以實現一一對應的關系,但實踐過程中通常難以實現,任何一個錯配位點的出現都會導致相應的擴增效率降低,從而減少產物。為此,通用引物中加入了簡并堿基以降低錯配的影響。然而,在進行復雜模板的PCR 擴增時,即使使用含簡并堿基的通用引物,引物結合能力也會因為不同模板DNA 引物結合區段序列的GC 堿基含量高低而不同(Acinaset al, 2005),GC 含量高的引物結合力較強(Fonseca,2018)。為了提高模板DNA 引物結合區段堿基組成的一致性,從而降低引物偏倚的影響,本研究選擇較為常見的同屬近緣種凡納濱對蝦和墨吉對蝦作為實驗對象。同時,基于基因的進化速率及其數據庫信息的豐富程度等因素,決定以mtDNA 中的COⅠ基因片段作為檢測的目的基因。
通過控制上述引物偏好的條件,最終高通量測序結果中引物的偏倚程度如何。研究分析發現,當eDNA 模板中凡納濱對蝦和墨吉對蝦DNA 組成比例為1∶1 時,高通量測序獲得對應物種reads 數比例為13/24。根據擴增循環數和公式(2),推導此時參與擴增的2 種DNA 比約為66/67,即PCR 擴增的平均偏倚率為1/67,約1.5%。可見選擇凡納濱對蝦和墨吉對蝦制備簡單eDNA 混合模板產生的引物偏倚程度影響較小,符合最初的實驗設計預期。此時,2 個物種的模板濃度相同,高通量測序結果的偏倚主要是由于引物對不同模板的“偏好”程度不同產生的。在后續eDNA 宏條形碼技術的數量性研究探索中,設計實驗時可選用近源種作為實驗對象,從而降低擴增過程中引物對不同模板偏好性不同而導致的“偏倚”現象。
3.2 eDNA 組成與高通量測序reads 數的相關性
當面對實際環境樣本時,如果直接應用eDNA 宏條形碼技術分析高通量測序reads 數與待測樣本生物量的關系,從生物到eDNA 以及從eDNA 再到高通量測序結果的兩個過程中的數學關系則難以理清。鑒于此,本研究采取“化繁為簡”的策略,主要關注eDNA與高通量測序reads 數之間的關系。同時,考慮到復合底物的PCR 過程比較復雜(Harperet al, 2019),最終注釋到每個物種的測序reads 數都受其反應體系的影響,故直接比較樣本間的reads 數會產生一定偏差。充分考慮這些影響因素,本研究采用了樣本內的2 個物種reads 數比值,用于確定其與模板eDNA 濃度的相關性。
統計分析實驗組的reads 平均數比值與PCR 模板eDNA 比值2 組數據,進行一元線性回歸分析,最終得出eDNA 組成與高通量測序reads 數的函數關系:y= 0.0716x+ 0.7043 (r2=0.9824)。等式中的斜率可反應出PCR 擴增過程中的偏倚程度(主要是引物偏倚),當擴增循環數相同,斜率越小,擴增過程中偏倚程度越大。等式中的截距主要是實驗過程中的誤差或者污染導致的。該等式可描述為:當環境水樣中只存在墨吉對蝦和凡納濱對蝦的DNA 時,可通過高通量測序的reads 數推算出2 個物種的相對生物量。
在實際操作中,可能由于模板eDNA 的回收率存在差異,使之不能完全符合預設的比例;另外,PCR擴增時并不是全程以底數為2 的指數式增長,且存在其他不可避免的系統誤差,這些因素都會導致上述線性關系式出現一定偏離。但實驗結果已充分表明,高通量測序reads 數與初始模板eDNA 含量呈線性正相關。

