基于網絡傳播算法預測蛋白質相互作用網絡的方向性
趙巧君,焦 雄
(太原理工大學 生物醫學工程學院,山西 晉中 030600)
隨著蛋白質組學的發展,蛋白質相互作用的研究逐漸成為一個熱門領域。雖然許多學者致力于研究PPIs網絡中的蛋白質和蛋白質功能模塊等,但這些研究結果均缺少信號轉導的方向信息,這些信息通過蛋白質相互作用網絡從一種蛋白質傳遞到另一種蛋白質來參與生命機制的運作,可用于探索病理機制[1]、研究細胞應答過程[2]、遏制疾病的發生[1]、預測蛋白質功能[3]等。在人類PPIs網絡中,PPIs通常參與多個細胞過程的信號轉導,其方向需要進一步的探索和研究。
經典的高通量技術手段往往用來預測蛋白質之間是否發生相互作用,很難發現PPIs間的方向性,隨著生物信息學的發展,研究者們開始利用計算的手段將單個蛋白質的研究轉向PPIs網絡。目前,許多研究小組已經應用生物信息學方法在PPIs網絡中定向信號轉導途徑。例如,STEFFEN et al[4]通過實驗獲得的PPIs與DNA微陣列的表達譜相結合來定向信號轉導網絡,但是該研究結果受到步長的限制;VINAYAGAM et al[5]利用每對PPI的最短路徑數來定向人類信號傳導網絡,這依然受到最短路徑的影響;LIU et al[6]通過使用蛋白功能注釋隱含的上下游關系來區分PPIs的信號流向,但會因蛋白質研究程度的深淺而導致注釋分布不均勻,最終限制該方法的使用。在本文中,為避免這些限制的影響,提出了一種基于網絡傳播具有良好拓展性的定向算法,并將其應用于人類PPIs網絡。
隨著隨機游走[7]和迭代算法的發展,網絡傳播[8]的出現為指導大規模信號轉導網絡的研究提供了基礎。這類方法的原理基于網絡上信息的迭代傳播,使信息遍布整個網絡,它可以度量每個節點的表型在網絡中的相似性而不受最短路徑和步數的限制。網絡傳播可以靈活地應用于各種網絡,因此可以進一步擴展成定向人類復雜PPIs網絡的方法。目前,只有SILVERBUSH et al[9]嘗試將網絡傳播算法應用于人類大規模PPIs網絡。在此方法的基礎上,本文還創新地結合了蛋白質語義相似性度量[10]和重疊聚類算法[11],并將其應用于預測人類大型PPIs網絡的信號流方向。
雖然傳播步數和最短路徑的限制可以被網絡傳播算法彌補,但是復雜的網絡結構自身不對稱性和PPIs權重測量方法的自身缺陷會導致無向PPIs和有向PPIs的區分困難。通過結合重疊聚類算法來檢測無向的PPIs,改善了網絡傳播算法關于識別無向PPIs的問題。在人類PPIs網絡中,蛋白質復合物形成了密集的連接區域,且相應的節點可能屬于一個以上的聚類,即參與多種復合物并發揮生物作用,這樣的節點非常適合采用具有重疊鄰域擴展的聚類算法進行預測。
為了度量蛋白質間的關聯程度,從而為信號游走提供依據,需要計算PPIs的權重。雖然實驗類型的證據可以作為蛋白質相互作用的度量手段,但是因實驗條件與細胞環境的影響容易造成假陰性數據。在信號轉導網絡中,功能相似的蛋白質之間傾向于相互作用。基于GO(gene ontology)[12]語義相似度的simIC[13]和BMA(best-mach average)[14]來計算PPIs的權重,這避免了受實驗類型測量和蛋白質淺注釋的限制。
本文的目的是區分有向PPIs和無向的PPIs,并預測有向PPIs的方向性,從而定向人類的PPIs網絡,為后續信號轉導網絡的研究打下基礎。為實現這一目的,本文通過結合蛋白質語義相似性度量和重疊聚類算法,利用網絡傳播算法來定向人類PPIs網絡的信號流方向。
1 材料和方法
1.1 算法框架
為定向PPIs網絡,算法的過程分以下幾步:1) 利用蛋白質語義相似性計算PPIs的權重;2) 運行網絡傳播算法,獲得每個PPI的得分;3) 利用Cluster ONE區分無向PPIs和有向PPIs;4) 判斷有向PPIs的方向,形成有向的PPIs網絡。其中,步驟2)和3)不分先后順序可同時進行。
算法的輸入為通過預處理的無向的PPIs網絡以及網絡涉及的源蛋白和靶蛋白,這里的源蛋白為整個PPIs網絡的信號起點,靶蛋白為信號終點。算法輸出的是PPIs的分數列表,每對PPI都有對應的分數來代表信號流的方向。算法框架如圖1所示。
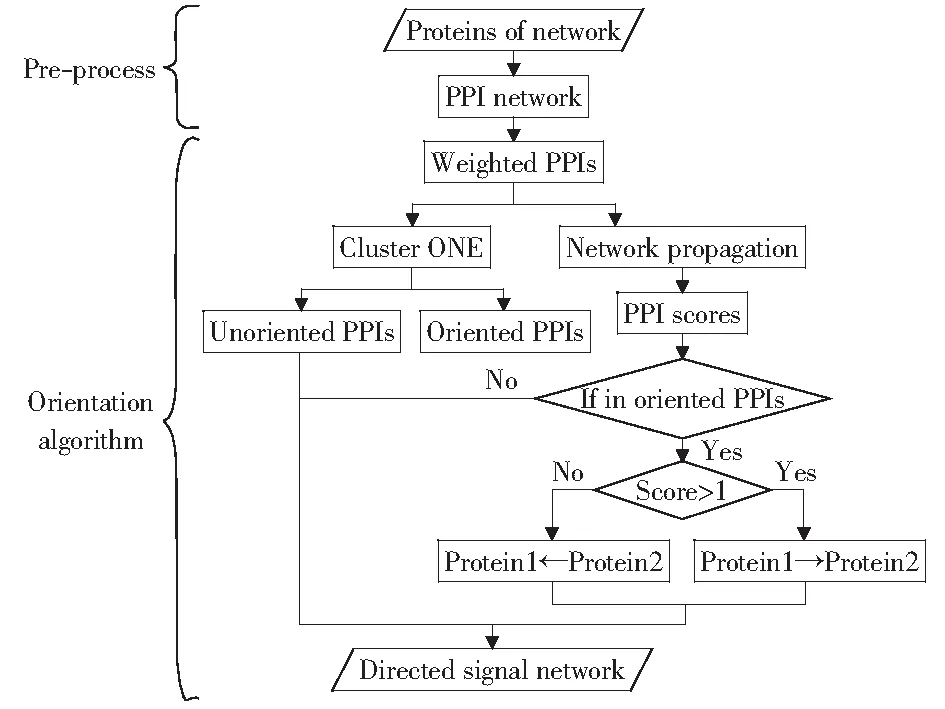
圖1 算法的框架Fig.1 Framework of the algorithm
1.2 數據集
1.2.1網絡和測試集
為了改進算法,本文準備了網絡,并刪除了其方向信息。該網絡來自KEGG(https://www.kegg.jp/kegg/)的VEGF信號轉導途徑,且僅包括蛋白質間的相互作用,PPIs的權重是通過蛋白質功能注釋來計算的。網絡中的源蛋白和靶蛋白均從VEGF信號轉導圖中獲得。
同時,還收集了已知方向的PPIs和經實驗驗證的蛋白質復合物作為測試集,如下:
1) 從CORUM數據庫(http://mips.helmholtz-muenchen.de/corum/)中整理了2 916個蛋白質復合物。
2) 從KEGG數據庫(https://www.kegg.jp/)中下載了567對涉及整合素參與的PPIs,其中包括137對有向的PPIs,430對無向的PPIs.
3) 從VINAYAGAM et al[5]計算的結果中獲得34 814對有向的PPIs.
1.2.2人類PPIs網絡
為構建人類PPIs網絡,收集了473個參與KEGG信號轉導通路的蛋白質(其中有99個蛋白質與整合素參與的網絡有關),并對此數據集做了預處理。首先,在String數據庫(https://string-db.org)中獲得初步的PPIs網絡,并輸入收集到的473個蛋白質,設置蛋白質相互作用來源為文字挖掘、實驗、資料庫、共表達、基因融合等,剔除相互作用的得分低于0.7的PPIs(分數高于0.7的PPIs可靠性高),最終獲得4 668對PPIs;其次,處理PPIs中的假陽性問題,利用蛋白質語義相似性篩選并獲得4 220對權重大于0.4的PPIs(本文設置權重為0.4作為區分蛋白質功能關聯的閾值);最后,構建加權的功能相關的PPIs網絡。
1.3 權重的計算
PPIs的權重將從根本上決定信號傳播方向的準確性問題。本文結合simIC和BMA,借助DaGO-Fun軟件[15]來計算PPIs權重。該方法根據蛋白質的GO注釋來計算蛋白質的語義相似性或者功能相似性,從而表征PPIs權重。計算方法如下:
(1)
(2)
式(1)定義了術語的語義相似性度量。其中,t是注釋蛋白質的術語;MICA表示t1和t2的信息最豐富的共同祖先;IC(t)代表術語t在語料庫中的普及程度。式(2)通過評價注釋兩個蛋白質的所有術語之間的語義相似性來確定PPIs權重。其中,p和q是相互作用的蛋白;x代表生物學過程(BP)本體注釋的一組GO術語,給定的蛋白質n,m分別代表集合中GO項的數量。
1.4 網絡傳播
參照SILVERBUSH et al[9]的方法將網絡傳播應用于PPIs網絡。首先,輸入一個加權的PPIs網絡,源蛋白和靶蛋白(源蛋白為膜蛋白,靶蛋白為肌動蛋白和基因調控蛋白);其次,網絡傳播將信息從源蛋白開始以迭代的方式傳播到附近的節點直到收斂,傳播結束后PPIs網絡中每個蛋白質獲得的分數代表其與源蛋白的接近度,同理,將信息從靶蛋白開始傳播,每個蛋白質獲得的分數代表其與靶蛋白的接近度;最后,算法通過組合兩個蛋白質的得分來比較兩個蛋白質接近源蛋白和靶蛋白的程度,輸出代表PPIs方向的分數。算法過程如下:
Input:G(V,E),ci∈C,ti∈T,w(u,v)
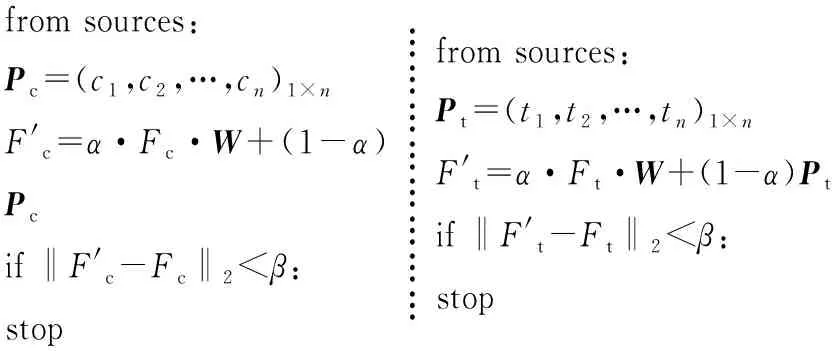
from sources:Pc=(c1,c2,…,cn)1×nF'c=α·Fc·W+(1-α)Pcif ‖F'c-Fc‖2<β:stop????????from sources:Pt=(t1,t2,…,tn)1×nF't=α·Ft·W+(1-α)Ptif ‖F't-Ft‖2<β:stop
if score(u,v)>1:u→v
if score(u,v)<1:u←v
輸入一個無向圖G=(V,E),其中,V表示蛋白質的集合;E表示PPIs的集合;C和T分別是源蛋白和靶蛋白的集合,w(u,v)表示蛋白質u和v相互作用的權重。在算法的過程中:W是歸一化的權重矩陣,N(u)代表蛋白質u的鄰居的集合。Pc和Pt分別是關于源蛋白和靶蛋白的先驗知識的向量(若ci∈C,ci=1,ti同理)。α是網絡和先驗知識的平衡參數,設為0.6.β=10-5是控制傳播停止的參數。算法結束后,獲得PPIs的分數列表。PPIs的分數大于1,意味著u比v更靠近源蛋白,u、v間的信號流方向u→v;當PPIs的分數小于1,u、v間的信號流方向u←v.
1.5 重疊聚類算法
重疊聚類算法用來區分無向PPIs與有向PPIs. ClusterONE算法通過尋找具有內聚性的重疊蛋白質復合物團來預測無向PPIs,算法的實現借助Cytoscape軟件[16]中的ClusterONE插件。在操作過程中,分別選擇了加權的PPIs網絡和未加權的PPIs網絡作為輸入,使用表1中的參數來計算聚類。

表1 ClusterONE參數Table 1 Parameters of ClusterONE
1.6 評估方法
大型的PPIs網絡中,無向PPIs并未經過全面的驗證,使用傳統的準確率、靈敏度等來評估預測的無向PPIs會降低分數。本文使用GO功能富集分析來評估無向PPIs的預測能力,利用具有生物學意義P值范圍內的無向PPIs數量占所有預測的無向PPIs數量的比例,來量化預測的無向PPIs的生物學意義。
為了評估預測的有向PPIs的方向性,計算了ROC曲線。ROC曲線展示了不同閾值下的敏感性和1-特異性,敏感性和特異性用來衡量定向算法在不同閾值下識別有向PPIs中真陽性和假陽性的能力。閾值是分界線,被用來區分PPIs的方向。如果PPI(蛋白質u與蛋白質v相互作用)的分數高于閾值,則方向為u→v,反之亦然。ROC曲線下的面積(AUC)越大,算法的性能越好。
2 結果和討論
2.1 VEGF網絡的結果和分析
為了初步測試PPIs權重對定向的影響,網絡傳播算法對區分無向PPIs和有向PPIs以及預測有向PPIs方向性的性能,選擇了VEGF信號轉導途徑。將加權的(通過蛋白質語義相似性來計算)網絡輸入到網絡傳播中,獲得PPIs分數,從而繪制分數分布的統計圖,如圖2所示。根據網絡傳播算法,分數分布在1左右的PPIs,因兩個蛋白在接近源蛋白與靶蛋白的程度上相近而被歸類為無向的PPIs. 為了檢驗該結論,將PPIs得分進行了劃分(見圖3),并將結果與已知方向的信號路徑進行了比較。結果表明,如果僅使用網絡傳播算法,將限制無向PPIs和有向PPIs的劃分,因此需要結合其他方法來區分無向PPIs和有向PPIs,以實現比VEGF通路更復雜的網絡的定向。

圖2 PPIs分數的分布Fig.2 Score distribution of PPIs

圖3 不同分數段中的PPIs數量Fig.3 Number of PPIs in different fractions
為了消除分布在1左右的分數對區分無向PPIs和有向PPIs的影響,將所有PPIs視為有向PPIs,并以分數1作為分界線來區分信號流的方向,獲得了有向VEGF信號轉導途徑(見圖4).與KEGG中的原始通路相比,本文只有一個PPI被錯誤地預測,而原始方法有4個PPIs被錯誤地預測。因此,當不考慮無向PPIs的區分時,通過使用蛋白質語義相似性來計算小型PPIs網絡的權重提高了信號流方向的預測水平。
圖2中,當不考慮無向PPIs的得分時,期望得分大于1的PPI的方向性為u→v(蛋白質u和v之間的相互作用),得分小于1的PPI的方向性為u←v. 圖3中,有4對PPIs在0.9~1.1的得分范圍內,只有1對屬于真正的無向PPI,其得分為0.910 9,這意味著網絡傳播算法在識別無向PPIs時存在缺陷。當面向更加復雜的網絡時,分數1左右的PPIs的數量將更加龐大,會摻雜更多的假陽性數據。圖4中,節點代表蛋白質,線代表PPIs,箭頭代表預測的信號流方向。該網絡包含28種蛋白質,其中紅色矩形為源蛋白質,藍色菱形為目標蛋白質。在32個PPIs中有一個PPI被錯誤預測,用紅線標記。

圖4 PPIs的方向Fig.4 Direction of PPIS
2.2 人類PPIs網絡的結果及評估
結合蛋白質語義相似性測量和重疊聚類算法,將改良后的算法用于定向大型的人類PPIs網絡。在無向PPIs的預測中,使用加權PPIs網絡和未加權PPIs網絡,分別獲得了兩組預測的無向PPIs,為了評估預測的無向PPIs,分別對其進行GO功能富集分析,并統計了P<0.05以內不同范圍的無向PPIs數量以及占所有預測的無向PPIs數量的比例,結果見表2.雖然兩組預測的結果中,具有顯著的生物學意義的無向PPIs均占較高的比例,分別為86.86%和95.79%,但是未加權PPIs網絡的結果優于加權的PPIs網絡,這也許與該網絡為蛋白質功能相關的網絡而非物理相關的網絡有關。最終選取未加權的PPIs網絡獲得的1 664對PPIs作為預測集的無向PPIs.

表2 GO功能富集分析Table 2 Functional enrichment analysis of GO
預測的2 556對有向的PPIs,其方向性的統計如圖5所示。為了評估算法對有向PPIs的方向性的預測性能,分別將結果與KEGG數據集和VINAYAGAM et al[5]的計算結果相重疊的部分進行統計(如圖6所示)繪制ROC曲線,KEGG測試集中PPIs的方向性得到了大量數據的驗證,當以KEGG作為測試集時,算法的結果獲得了較好的性能(見圖7),這表明本文的結果具有較好的參考價值。

圖5 預測的有向PPIs方向性數量分布Fig.5 Predicted directional quantity distribution of oriented PPIs
圖5中,分數小于1的信號流的方向規定為u←v,分數大于1的信號流的方向規定為u→v.圖6驗證了分數1在大型的復雜網絡中也不能明確區分無向PPIs和有向PPIs,表明改良后的算法在區分無向PPIs和有向PPIs的必要性。由圖7可以看出,算法在經過大量驗證的KEGG數據集上獲得了較好的結果(AUC為0.813).

圖6 預測的無向PPIs和有向PPIs分數分布箱型圖Fig.6 Predicted unerieuted and oriented PPIs box graphs
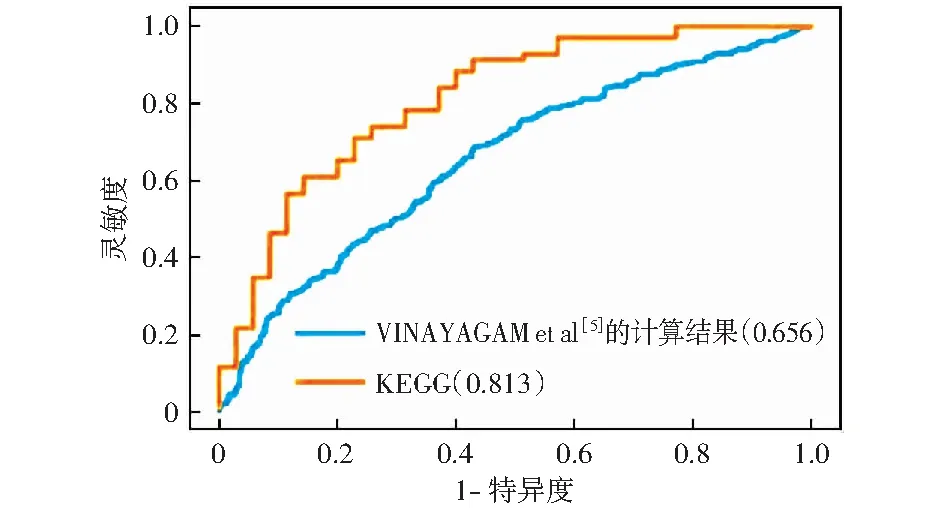
圖7 兩組測試集的ROC曲線Fig.7 ROC curves for two test sets
3 結束語
本文提出了一個新的網絡傳播的方法來預測PPIs網絡信號流的方向,它結合了蛋白質語義相似性度量和重疊聚類算法。將此方法用于人類PPIs網絡并取得了較好的結果,這個結果將有助于進一步研究人類PPIs網絡的相關信息。
雖然方法的性能較好,但是仍存在一些缺陷。1) 此方法只能用于復雜的PPIs網絡,因為重疊聚類算法在簡單通路中不能正確獲得無向的PPIs;2) 源蛋白和靶蛋白來源于已知信息,不能進行源蛋白和靶蛋白的預測;3) 需要進一步考慮范圍在1左右的分數對有向PPIs和無向PPIs區分的影響。

