微型DNA條形碼在水產類物種摻假檢測中的應用
黃曼虹,熊 雄,袁方穎,操 敏,陸利霞,2,熊曉輝,2
(1.南京工業大學 食品與輕工學院,江蘇 南京 211800;2.江蘇省食品安全快速檢測公共技術服務中心,江蘇 南京 211800)
隨著我國經濟的發展和人民生活、文化水平的不斷提高,以低脂肪、高蛋白為主要特征的水產品已經成為我國居民動物蛋白攝入的主要來源之一,水產品消費量不斷增大[1]。在水產品消費量增長的同時,人們對水產品的質量安全也提出了更高的要求,其中一個重要的方面就是對水產食品原料的生物種屬進行準確的標識和鑒定。
水產類物種繁多,各種過敏原和含有天然毒素的有毒物種會給特定消費群體的健康帶來嚴重威脅[2-3]。不同物種的營養價值、口感和價格等方面也存在巨大差異[4-5],由此而產生的商業欺詐問題已經受到人們的廣泛關注[6]。然而,由于不少商販缺乏專業的水產類物種分類知識以及市場缺乏對一些水產類物種命名的統一標準,我國水產品市場上物種錯標現象不斷被曝光,如“真假鱈魚”事件[7]和“真假三文魚”事件[4]。此外,水產品物種摻假的問題并非我國獨有,在世界各國均被廣泛報道(表1)。
為了防止水產品物種摻假,建立一種快速高效的水產品物種鑒定技術迫在眉睫。傳統的形態學檢驗法主要依賴于鑒定者的經驗,對于親緣關系近的物種鑒別難度較大[8],而且被加工成魚片、魚丸和罐頭等產品后,水產類物種特有的形態學特征遭到破壞,無法對物種做出準確鑒定。
隨著分子生物學的發展,基于DNA的PCR技術在水產類物種鑒定中得到運用,已經成為水產類物種摻假檢測的主要技術(表1),其中最常見的一種就是標準DNA條形碼技術[5]。在食品加工中的高溫、高壓、反復凍融和酸堿變化等因素均會使DNA發生降解,與標準DNA條形碼(FDB,650 bp)相比,微型DNA條形碼(MDB,<350 bp)在深加工水產品物種檢測中具有更大的優勢[9]。本文中,筆者綜述MDB的原理、操作及最新研究進展,分析MDB在水產類物種摻假檢測中的應用及規則,并對MDB的關鍵問題進行初步探討。

表1 世界各國水產品摻假現狀
1 FDB檢測水產類物種摻假
FDB是由加拿大科學家Hebert等[34]在2003年首次提出的,其基本原理是利用一段較短的DNA標準序列的多態性來實現快速、準確的物種鑒定。對動物而言,一般為線粒體細胞色素C氧化酶亞單位Ⅰ基因(COⅠ)5’端的一段長約650 bp的片段。目前,FDB技術在水產類物種摻假檢測中得到了廣泛應用(表1)。美國食品藥品監督管理局將DNA條形碼納入魚類監管百科全書(RFE)來進行市場上魚類替代品的檢測[35],巴西聯邦政府也將FDB作為全國范圍內對加工水產品進行常規和系統管理的標準鑒定方法[31]。
然而,隨著FDB研究的深入,研究者們發現其在實際應用中也存在一些問題。如Xiong等[27]利用FDB對市售干制鱈魚樣品進行物種鑒定時發現部分樣品無法成功擴增出650 bp的FDB片段。除了干制品,Guardone等[36]發現魚類罐裝制品也無法擴增出FDB片段。此外,Günther等[37]在魚糜、魚籽醬以及煙熏水產制品中均發現FDB擴增失敗的現象。最新報道表明,烤鱈魚制品也無法成功擴增出FDB片段,其失敗率可達24.2%[7]。
食品加工過程中的高溫、高壓、反復凍融和酸堿變化等因素導致的DNA降解被認為是FDB擴增失敗的主要因素[38]。Gryson等[39]認為pH變化可導致DNA的降解。黃婭琳[40]研究發現高溫對DNA的降解和后續PCR擴增有一定影響,特別是當加熱溫度大于140 ℃、加熱時間超過80 min時,PCR擴增產物條帶明顯減弱。因此,篩選較短的標準DNA片段(即MDB)并探討其對深加工食品進行物種鑒定的可行性引起了人們的廣泛關注。如Günther等[37]設計新的通用引物以擴增更短的線粒體COⅠ基因片段(約326 bp),結果發現,16份FDB擴增失敗的樣品均成功擴增出326 bp的目標基因片段,并且測序結果發現該片段可成功鑒定相應的物種。此外,Xiong等[7]利用一個更短的COⅠ基因片段(約192 bp)實現了對7份FDB技術擴增失敗的市售烤鱈魚樣品的物種準確鑒定。由此可見,MDB概念的提出給深加工魚肉制品中魚肉物種鑒定提供了新思路。
2 MDB檢測水產類物種摻假
2.1 MDB簡介
MDB是由加拿大Guelph大學Hajibabaei等[9]首次提出,該團隊采用序列分析的方法從理論上證實了被分割的短片段序列與原DNA序列具有同等的物種鑒別能力。同時,該團隊從已經發生DNA降解的館藏標本中成功擴增出134 bp的目的基因片段,驗證了該片段在哥斯達黎加地區寄生蜂物種鑒定中的可行性。在此基礎之上,其他學者對MDB的適宜長度進行了深入研究,并提出條形碼長度大于100 bp就可以解決90%以上的物種鑒定問題,即100 bp序列長度就已包含可以進行分類鑒定所需的DNA信息。根據這一結果,該研究團隊基于COⅠ基因設計出能擴增130 bp微型條形碼片段的通用引物,并對該引物的擴增能力進行檢驗,得出該引物擴增MDB的成功率比通常所用的擴增FDB的成功率高。
MDB的應用主要包括以下幾個流程(圖1):①樣品的采集與處理;②DNA的提取和冷凍保藏;③設計引物對目的DNA進行PCR擴增,選擇電泳條帶清晰單一產物;④PCR產物的純化與測序;⑤使用BioEdit軟件進行序列修剪和比對;⑥將測序所得序列提交至NCBI或BOLD數據庫,獲取與目標序列相似度>98%[15]的物種,以此確定樣品的物種。
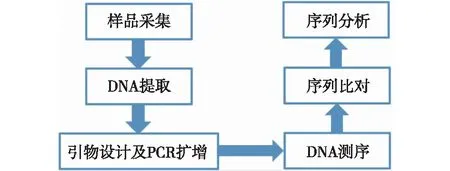
圖1 MDB技術應用流程Fig.1 Process of MDB application
就目前文獻來看,國內外多個研究團隊已經就魚類MDB鑒定技術展開了相關研究(表1、2)。如Armani等[41]利用MDB對太湖銀魚、沙丁魚和鳳尾魚等魚類進行物種鑒定。陳文炳等[42]利用MDB對美洲鰻、歐洲鰻和日本鰻進行物種鑒定。此外,甲殼類動物也可利用MDB技術進行準確鑒定[43]。
盡管如此,關于FDB和MDB物種鑒定能力的比較仍在討論當中。有部分學者認為FDB的物種鑒定能力優于MDB。如Guardone等[36]對意大利市場上277份水產品樣品調研發現,FDB可對55.1%的樣品進行鑒定,顯著高于MDB(14.6%)。此外,Armani等[44]指出,降低片段長度會對條形碼的物種鑒定能力有負面影響。然而,Armani等[10]對壽司中魚類產品的研究發現,FDB和MDB的物種鑒定能力相同。Shokralla等[45]基于COⅠ基因,篩選出了6個MDB片段(127~314 bp),并利用96份魚類樣品對這些MDB片段的可靠性進行驗證,結果發現,93.2%的魚類樣品可以在種或屬水平進行鑒定,MDB可對88.6%的樣品進行鑒定,遠高于FDB(20.5%)。Labrador等[46]對菲律賓的39份沙丁魚樣品進行抽查,結果發現FDB無法擴增出序列片段,而MDB則可對64%的樣品進行鑒定。由此可見:與FDB相比,MDB具有較高的物種鑒定能力。
MDB彌補了FDB由于DNA降解而導致鑒定失敗的缺點,有望成為替代FDB的水產類物種鑒定技術。然而,越來越多的研究證實,MDB的片段長度選擇以及目的基因篩選是影響其物種鑒定能力的重要因素。因此,進行水產品鑒定時,MDB目標片段的適宜長度以及目的基因的選取仍需進一步研究。
2.2 MDB目標片段的長度選擇
目標片段的長度對MDB的物種鑒定能力具有重要的影響。一般而言,隨著目標片段長度的縮短,其物種鑒定能力逐漸減弱。Meusnier等[47]對現有DNA條形碼數據庫中的序列進行模擬計算,結果發現,當COⅠ基因片段長度大于100 bp時即可鑒定90%的物種,當COⅠ基因片段長度大于250 bp時即可鑒定95%的物種。Pollack等[48]對基于COⅠ基因的兩對MDB片段進行比較時發現,長為226 bp的MDB片段(92%~94%),其物種鑒定能力優于長為208 bp的MDB片段(67%~90%)。Sultana等[49]提出,當MDB的目標片段長度≤150 bp時,目標片段無法利用普通一代測序技術進行成功測序;為此,該團隊設計了長約295 bp的魚類通用MDB片段,并證實了該MDB片段可成功鑒定33種魚類。
然而,在水產品品種鑒定中,不同科研團隊并未就MDB片段長度達成一致[32,41-45,47,49-55](表2)。

表2 用于水產類物種摻假檢測中的MDB片段
如Armani等[54]基于16S rRNA基因篩選了長為118 bp的MDB片段,并證明了該MDB片段可以準確鑒定鯡科和鯖科的魚類品種。潘艷儀等[50]基于COⅠ基因篩選了長為136 bp的MDB片段,并利用克隆測序法獲得高質量的測序結果,以此可對魚、蝦等11個肉類物種進行準確鑒定。程鵬等[56]對三段長度分別為100、150和200 bp的魚類COⅠ序列進行物種鑒定能力的比較,結果發現,當序列長度為100 bp時,其物種鑒定能力明顯減弱;基于此,該團隊提出200 bp的MDB片段在魚類樣品鑒定中具有很高的準確性。Shokralla等[45]基于COⅠ基因,篩選出了長為127、129、208、226、227和314 bp的6個MDB片段,研究發現其中長為226 bp的片段可對88.6%的魚類樣品進行物種鑒定,遠優于其他的MDB片段。Günther等[37]利用基于COⅠ基因長為313 bp的MDB片段可對魚類、蝦類進行物種鑒定。最新研究表明,基于16S rRNA長約313 bp的MDB片段也可對太湖銀魚、沙丁魚等魚類[41]以及十足目甲殼類[43]進行物種鑒定。
2.3 MDB目的基因的篩選
一般而言,線粒體基因組和核基因組均可以作為MDB的目的基因。線粒體基因組具有結構簡單、序列和組成比較保守、母系遺傳、無重組以及單拷貝等特點[57]。同時由于線粒體基因組的分子量比較小、進化速度快,所以在物種鑒定的研究中經常被選取為目的基因[58]。
目前,常用于水產類物種鑒定中的目的基因有COⅠ基因、16S rRNA基因和細胞色素b基因(Cytb)(圖2)。Mitchell等[32]基于COⅠ基因開發了一種微型DNA條形碼方法,可用于區分金槍魚罐頭中常見的物種。Spielmann等[43]分別選取16S rRNA和COⅠ基因為目的基因,對甲殼類動物進行物種鑒定,結果發現,基于16S rRNA的條形碼測得序列質量較低,鑒定甲殼類動物最合適的目的基因是COⅠ基因。而Armani等[41]基于16S rRNA基因設計的FOR16spc和REV16spc引物對鱈魚、銀魚等的物種鑒定能力較強。之后,該團隊繼續以16S rRNA為目的基因設計MDB,可對市售97%(207份)的鯡科等魚類物種樣品進行成功鑒定[54]。同時,Sevilla等[55]設計的基于Cytb基因的MDB,可對硬骨魚綱200多種海洋魚類進行物種鑒定。Fernandes等[51]基于Cytb基因設計的MDB可對99.3%的4種常見鱈魚進行物種水平上的準確鑒定。

圖2 微型DNA條形碼在目的基因中的位置Fig.2 Location of the mini DNA barcoding in the target genes
除此之外,基于核基因組的MDB也可對水產類物種進行鑒定。Ma等[59]研究發現,以磷酸烯醇丙酮酸羧激酶(PEPCK)為代表的核基因組,可成功應用于對蝦總科的系統發育研究,為這些物種的分類提供了有力的工具。Tsang等[60]選取PEPCK基因為目的基因對甲殼類動物進行DNA擴增,驗證了PEPCK基因對甲殼類動物DNA樣品的高效性。
3 結論
目前,MDB彌補了FDB由于DNA降解而導致鑒定失敗的缺點,有望成為替代FDB的水產類物種摻假檢測技術。目標片段的長度和目的基因的篩選對MDB技術的物種鑒定能力具有重要的影響。隨著目標片段長度的縮短,MDB的物種鑒定能力逐漸減弱。此外,線粒體基因組和核基因組均可以作為MDB的目的基因。
然而,隨著對MDB研究的深入,研究者們逐漸發現了MDB的缺陷:第一,抑制劑或其他某些物質的存在可能會導致DNA無法進行擴增從而使樣品鑒定失敗,而正常的魚糜加工物質并不是失敗的原因;第二,數據庫中的序列不全,或者序列錯誤導致了鑒定結果的錯誤。由于BOLD和GenBank數據庫均為開放平臺,所有研究者均可提交序列,且數據庫自身并不會核對提交物種序列。MDB對于水產品的鑒定結果依賴于GenBank和BOLD數據庫的準確性、規范性和完整性。因此,在利用FDB和MDB進行物種鑒定時,可通過多次下載序列來對數據庫中的序列進行核查。

